Introducción
La infección persistente con virus de papiloma humano de alto riesgo (VPH- AR) causa cáncer de cuello uterino (CCU) 1,2 y este es el segundo cáncer más frecuente en mujeres a nivel mundial 3. Actualmente se realiza tamización para detectar lesiones precancerosas por medio de la citología, método realizado habitualmente debido a su alta especificidad (90%) 4,5. No obstante, al momento de diagnosticar lesiones escamosas intraepiteliales de grado alto (HSIL) este método alcanza sensibilidad solamente del 50%, principalmente debido a la calidad de la muestra, la pericia del profesional para identificar alteraciones morfológicas y la subjetividad en la interpretación 5,6. Por esta razón se han desarrollado técnicas moleculares con mayor sensibilidad en la detección de tipos específicos de alto riesgo del VPH.
Muchos protocolos de detección se basan en la amplificación del gen L1, debido a que es altamente conservado, sin embargo, el marco de lectura abierto (ORF, open reading frame) de este gen puede perderse en el momento en que el virus se integra en el genoma de la célula hospedadora, fenómeno observado aproximadamente en el 11% de las infecciones por genotipos de VPH-AR 7-11. Una mPCR del gen E7 de VPH-AR podría ofrecer un mejor rendimiento, pues E7 prevalece integrado en todas las células cancerosas, ya que se necesita de la expresión continua de los oncogenes E6 y E7 para que las infecciones sean persistentes y se promueva un fenotipo maligno de las células 12,13.
A continuación se describe un método sencillo, rápido y económico, potencialmente muy sensible y específico para la detección y tipificación de los seis genotipos de VPH-AR de mayor frecuencia en CCU a nivel mundial (VPH -16, -18, -31, -33, -45 y -52), combinando mPCR de primera generación con oligonucleótidos dirigidos al gen E7, específicos para cada genotipo de VPH y detectados por electroforesis en gel de agarosa.
Materiales y métodos
Población de estudio y selección de muestras cervicales
Se obtuvieron muestras de cepillado cervical de mujeres residentes de la zona norte de Bucaramanga, por medio de un dispositivo para auto-toma según el procedimiento y los criterios de inclusión y exclusión descritos previamente 14. Se seleccionaron todas las muestras positivas para VPH (n = 59) que fueron analizadas previamente por amplificación multiplex del gen L1 por PCR, con subsecuente hibridación reversa con la técnica HPV Direct Flow CHIP, hasta el momento de la ejecución del presente estudio.
Se seleccionó una muestra positiva para cada uno de los seis genotipos a detectar y además una muestra negativa para VPH por HPV Direct Flow CHIP para la estandarización de la E7-mPCR. Una vez se estandarizó la técnica, se valoró su desempeño procesando las muestras restantes, de las cuales el 83% se evaluó por duplicado.
Detección de VPH y genotipado por HPV Direct Flow CHIP
Las muestras se conservaron en un líquido de preservación celular (ThinPrep), se centrifugaron 500 μl de muestra a 3.500 rpm por cinco minutos, se lavaron dos veces en 400 μl de solución salina tamponada con fosfato estéril (PBS) y se resuspendieron en 50μL de PBS, según lo indicado por el fabricante (Master diagnóstica, España).
Por medio de una PCR múltiple del segmento de L1 utilizando los cebadores GP5+/GP6+, se amplificaron los genotipos VPH-BR-6, -11, -40, -42, -43, -44, -54, -55, -61, -62, -67, -69, -70, -71, -72, -81, -84 y -89 y VPH-AR -16, -18, -26, -31, -33, -35, -39, -45, -51, -52, -53, -56, -58, -59, -66, -68, -73 y -82. La PCR se realizó con 4 μL de ADN extraído con los siguientes ciclos de amplificación:
1 ciclo de 10 minutos a 25 °C
1 ciclo a 94 °C por 3 minutos
15 ciclos de desnaturalización a 94 °C por 30 segundos, anillamiento a 42°C por 30 segundos y elonga]ción a 72 °C por 30 segundos
35 ciclos de desnaturalización a 94 °C por 30 segundos, anillamiento a 60°C por 30 segundos y elonga]ción a 72 °C por 30 segundos
1 ciclo a 72 °C por 5 min y finalmente por 5 min a 8 °C
Posteriormente, se desnaturalizaron los amplicones biotinilados durante cinco minutos a 95 °C, se enfriaron en hielo durante dos minutos y se hibridaron por transferencia de puntos y detección colorimétrica en membranas CHIP de VPH que contenían cuatro tipos de sondas tridimensionales: para control de hibridación, para el gen de beta-globina, para la secuencia conservada de L1 y las sondas para la detección de VPH específica de genotipo.
La hibridación se realizó usando el equipo semiautomático HybriSpot 12TM (HS12) que une el amplicón biotinilado con las sondas complementarias. La detección colorimétrica se llevó a cabo mediante la adición de sustratos de NBT-BCIP que detectan actividad de fosfatasa alcalina, creando precipitados puros insolubles 15,16.
Extracción de ADN
Se optimizó un protocolo de extracción de ADN por salting out17, por el cual se obtuvieron concentraciones de ADN superiores a 50 ng/uL y un radio de absorbancia a 260/280 por encima de 1,5, las cuales fueron determinadas en el espectrofotómetro NanoDrop 2000 (Thermo Scientific, USA).
Control de extracción de ADN
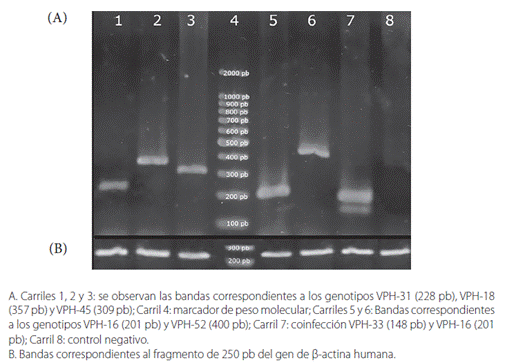
Se confirmó una correcta extracción de ADN y la ausencia de inhibidores de la PCR en las muestras, al amplificar un fragmento de 250 pb del gen de β-actina humana (tabla 1). Seguidamente se corrió una electroforesis en gel de agarosa, donde todas las muestras fueron positivas para este gen.
Diseño de oligonucleótidos específicos para E7
Se utilizaron oligonucleótidos para los tipos de VPH 18, -31, -45 y -52 según Paes, et al. 11, por otro lado, para VPH 16 y -33 se diseñaron nuevos oligonucleótidos utilizando la herramienta Primer-blast (tabla 1). Por medio del programa AutoDimer se verificó que no se formaran estructuras secundarias de cebadores.
Tabla 1 Secuencias de los cebadores dirigidos a la región E7 de VPH y al gen de β-actina, temperaturas de fusión, números de acceso y longitudes de los amplicones
| Nombre | Número de acceso en GenBanka | Cebadores | Tm (C) | Pares de bases |
|---|---|---|---|---|
| β-actina | NM_001101.4 | F: 5' CATGTACGTTGCTATCCAGGC 3' | 59 | 250 |
| R: 5' CTCCTTAATGTCACGCACGAT 3' | 58 | |||
| VPH-16 | NC_001526.4 | F: 5' GCAACCAGAGACAACTGATCTCTAC 3' | 60 | 201 |
| R: 5' GGTCTTCCAAAGTACGAATGTCTACG 3' | 60 | |||
| VPH-18 | GQ180792.1 | F: 5' CGACGCAGAGAAACACAAGTAT 3' | 60 | 357 |
| R: 5' ATTGTTGCTTACTGCTGGGAT 3' | 59 | |||
| VPH-31 | KX638481.1 | F: 5' GGCAACTGACCTCCACTG 3' | 62 | 228 |
| R: 5' ACAGTTGGGGCACACGATT 3' | 63 | |||
| VPH-33 | M12732.1 | F: 5' GGACACAAGCCAACGTTAAAGG 3' | 59 | 148 |
| R: 5' AATCAGCTGT GGCTGGTTGT 3' | 60 | |||
| VPH-45 | KC470260.1 | F: 5' AGGCACGGCAAGAAAGACT 3' | 62 | 309 |
| R: 5' TCTAAGGTCATCTGCCGAGC 3' | 62 | |||
| VPH-52 | LC373207.1 | F: 5' GACCTGTGACCCAAGTGTAAC 3' | 58 | 400 |
| R: 5' GCCTCTACTTCAAACCAGCC 3' | 59 | |||
| a GenBank (https://www.ncbi.nim.nih.gov/genbank | ||||
Fuente: elaboración propia, utilizando el software Primer-blast del Centro Nacional para la Información Biotecnológica (NCBI, por sus siglas en inglés).
Amplificación por E7-mPCR
Se utilizó una PCR Master Mix (Thermo Scientific, DreamTaq Green, 2X) para realizar 20 amplificaciones en las que se modificaron las concentraciones de MgCl2 (1,5 a 4 mM), cebadores (0,033 a 1,00 μM) y ADN (50 a 250 ng). Se crearon diferentes programas de PCR en el termociclador multigene gradient (Labnet, NJ, USA), con variación de la temperatura de anillamiento de 58 a 64 °C y el número de ciclos de 35 a 38.
Electroforesis en gel de agarosa
Se utilizó un sistema de electroforesis horizontal Mini-Sub® Cell GT y la fuente de alimentación básica PowerPac (BioRad, Hercules, USA). Se probaron geles con concentraciones de agarosa que variaron de 2,0% a 3,0%. Se añadieron a cada uno de los pozos del gel 15 uL de los productos de la PCR y se sirvió un patrón de peso molecular de 100 a 2.000 pb (Promega, Madison, WI USA). Se realizaron 20 electroforesis variando la potencia de 60 a 100 voltios y el tiempo de 40 a 80 minutos. Tras la electroforesis se sumergió el gel de agarosa en una solución de bromuro de etidio (0,4 ug/mL) durante 15 minutos. Posteriormente, se observó la separación de los fragmentos de ADN bajo transiluminador con luz ultravioleta a 260 nm, tomándose evidencia fotográfica.
Método estadístico
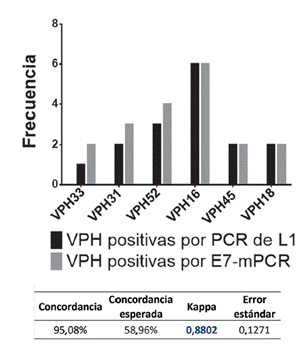
Se calculó la frecuencia de concordancia observada y el coeficiente Kappa de Cohen para evaluar la concordancia entre la prueba VPH direct flow Chip kit y la PCR Multiplex de E7. Se espera un valor de K = 0 cuando la concordancia se dé por azar y un valor de K = 1 cuando la concordancia sea del 100%. Los valores de K > 0,80 representan un acuerdo casi perfecto.
Resultados
Diseño de oligonucleótidos específicos para E7
El diseño de los cebadores para VPH 16 y -33 se fundamentó en la alineación de secuencias en la región del gen E7 de cada uno de los genotipos de VPH-AR, secuencias que fueron obtenidas del GenBank (NCBI, NIH, USA). Los oligonucleótidos tuvieron una longitud que osciló entre 18 y 26 nucleótidos, la temperatura de fusión estuvo entre 58 °C y 63 °C y los tamaños de los productos amplificados variaron entre 148 y 400 pb. De igual modo, se escogieron estas secuencias sentido y antisentido, de tal manera que no se dieran inespecificidades, reacciones cruzadas ni formación de dímeros o bucles entre sí (tabla 1).
Amplificación por E7-mPCR
Se estableció, debido a la existencia de bandas específicas en la electroforesis, que la mezcla más apropiada para la E7-mPCR fue aquella que contuviera cinco unidades de Taq ADN polimerasa, 0,2 mM de desoxirribonucleótidos trifosfatados (dNTP), 4 mM de MgCl2, 0,08 μM de cada cebador y 150 ng de ADN (tabla 2).
Tabla 2 Componentes de la reacción de amplificación
| Componente PCR | Concentración | Volumen | |
|---|---|---|---|
| DreamTaq Green PCR Master Mix (2X) | dNTPs Taq | 0,2 mM 5 u | 7,5 μL |
| polimerasa MgCl2 | 1,5 mM | ||
| MgCl2adicional | 2,5 mM | 1,5 μL | |
| Cebadores | 0,08 μM | 1,25 μL | |
| Agua | _ | 1,75 μL | |
| ADN | 150 ng | 3 μL | |
| Volumen Final | _ | 15 μL | |
Fuente: elaboración propia.
El programa de amplificación con resultados más sensibles y específicos se logró llevando a cabo una fase de desnaturalización inicial a 95 °C durante dos minutos, seguida de 35 ciclos de amplificación (tabla 3), manteniendo la temperatura de extensión a 72 °C durante siete minutos para permitir la polimerización de fragmentos incompletos y finalmente conservar los productos de la PCR a 4 °C hasta el momento de realizar la electroforesis.
Electroforesis en gel de agarosa
Se determinó que una concentración del gel de agarosa al 2,5% con una potencia de 80 voltios y durante 60 minutos, permite una mejor resolución y discriminación de los fragmentos amplificados (figura 1).
Selección de las muestras de estudio
De las 59 muestras seleccionadas de pacientes VPH positivas por amplificación del gen L1 del virus, 11 fueron de genotipo indeterminado, 15 de genotipos correspondientes a los VPH-AR más frecuentes y 33 positivas para otros genotipos (tabla 4).
Tabla 4 Selección de las muestras de estudio y resultados por E7-mPCR
Fuente: elaboración propia, a partir de análisis experimentales comparando los resultados de la prueba de referencia y la E7-mPCR.
Evaluación de la E7-mPCR con muestras de pacientes
Del total de las muestras analizadas, el 25,4% fueron positivas para uno o dos de los seis genotipos de VPH-AR según la PCR del gen L1, en comparación con el 28,8% por E7-mPCR. Las muestras positivas por PCR de L1 para genotipos diferentes a VPH -16, -18, -31, -33, -45 y -52 (sin incluir los genotipos indeterminados) fueron negativas por E7-mPCR en el 93,9% de los casos. Así mismo, el 100% de las muestras de genotipo indeterminado por PCR de L1 fueron negativas por E7-mPCR y no se pudieron clasificar por este método.
Por medio de la E7-mPCR fue posible detectar el ADN viral de tres genotipos de VPH-AR (VPH -31, -33 y -52 ) que no se detectaron mediante amplificación por PCR del gen L1, esto corresponde a un 15,8% de potenciales falsos negativos para la prueba del gen L1. Además, dos de estas muestras fueron positivas por PCR contra L1 para otros genotipos, ya sea de alto o de bajo riesgo (VPH -59 y -72, respectivamente) que no están incluidos en la E7-mPCR. La muestra restante fue positiva por PCR de L1 para VPH-31, sin embargo, por E7-mPCR se detectó adicionalmente una coinfección con VPH-52 (tabla 4).
Los datos evidenciaron una elevada concordancia entre los resultados del ensayo E7-mPCR y PCR de L1 (95,1%) con un valor de K = 0,88, demostrando una excelente concordancia diagnóstica. Además, se obtuvo un 100% de reproducibilidad a partir de las 49 muestras que se procesaron por duplicado, encontrándose mayor número de infecciones por VPH -31, -33 y -52 en el 4,9% con la detección de E7 (figura 2).
Discusión
El número de muestras discordantes entre ambos métodos permitió detectar un 15,8% de falsos negativos por parte de la PCR de L1, lo cual coincide con lo expresado en la literatura. De acuerdo con los esquemas de manejo clínico actuales, a estas pacientes se les recomendó hacerse la citología cervical o una prueba de VPH en los siguientes 6 a 12 meses, con el fin de identificar la posible presencia de infecciones persistentes por VPH-AR.
Dada la evidencia de resolución espontánea en el 90% de las infecciones por VPH 18, la detección inicial del virus no requiere automáticamente una evaluación colposcópica, ya que en las etapas iniciales la probabilidad de encontrar lesiones premalignas es baja, siendo suficiente una prueba de clasificación adicional 19.
Probablemente, el porcentaje de falsos negativos se deba a la pérdida del gen L1 que ocurre durante el proceso de integración viral 2,8. Durante este proceso, además ocurre la interrupción del ORF de los genes E1/E2, provocando un incremento en la expresión de E6 y E7, genes cuya presencia es esencial en el proceso de transformación maligna en el cuello uterino, ya que tienen la capacidad de modular el ciclo celular en las capas epiteliales superiores, así como estimular la proliferación celular en las capas basales y parabasales, razón por la cual la expresión descontrolada de estos oncogenes reguladores del ciclo celular conduce a lesiones premalignas y eventualmente progresan a cáncer en aquellas mujeres que no logran resolver la infección 12,20-22.
En un estudio de lesiones cervicales de alto grado, se calculó la frecuencia de falsos negativos utilizando cebadores de la región L1 en comparación con el empleo de cebadores específicos para las regiones E6/E7. Los resultados mostraron que en las infecciones por VPH-16 el 0,3% de las neoplasias intraepiteliales cervicales (NIC) grado 2 y el 3,94% de NIC 3 fueron negativas por L1 y positivas por E6/E7 23. Asimismo, se ha encontrado que particularmente el ensayo con L1 omite el 8,3% de las infecciones por VPH-16 y el 27,9% de infecciones por VPH-18 24.
Otros estudios de comparación entre PCR con cebadores consenso de L1 (MY09/11) y PCR con cebadores de genotipo específico E6/E7 evidenciaron que el 10,9% de las infecciones por VPH-AR no son detectadas por PCR de L1, mientras que por PCR de E6/E7 sí es posible 7.
Claramente, la variabilidad en la sensibilidad de detección es una de las mayores desventajas de estos ensayos, principalmente como consecuencia de la pérdida de L1 durante la integración viral, aunque también debido a la degradación del ADN o a la falta de coincidencia en la alineación de los cebadores 24. La comparación de estos dos métodos podría proporcionar información acerca de la integración del VPH en el genoma de la célula hospedadora, lo cual es de gran importancia dado que se ha demostrado que la integración viral produce alteraciones en vías que pueden resultar en el crecimiento del tumor, como es el caso de la ruta mediada por proteínas Ras con potencial oncogénico, es decir, puede generar mutaciones conductoras en la carcinogénesis cervical y además promover la expresión de los oncogenes E6 y E7 25-27.
Adicionalmente, se ha encontrado que en pacientes con NIC 1 existe una mayor expresión de la proteína L1 que en pacientes con NIC 2, NIC 3 o carcinoma in situ; esto sugiere que la expresión de L1 tiene una relación inversa con el grado de lesiones cervicales, es decir, la deficiencia de L1 se puede atribuir a la progresión de las lesiones. De esta manera, su ausencia sería un marcador para la predicción de CCU 10,28. Por lo tanto, este ensayo tiene potencial aplicabilidad en la estratificación del riesgo, en la determinación de la persistencia de infección por VPH-AR y en el control y seguimiento post-tratamiento 29.
Por otro lado, el 95,5% (42 de 44) de las muestras negativas para los seis genotipos de VPH-AR, pero positivas para otros VPH-AR o VPH-BR (incluyendo de genotipo indeterminado) por PCR de L1, fueron concordantemente negativas por E7-mPCR, esto llevaría a que esta prueba tenga una excelente especificidad para reconocer los genotipos de VPH, la cual puede explicarse por el uso de oligonucleótidos específicos de genotipo en lugar de oligonucleótidos consenso, ya que la coamplificación de L1 de diferentes genotipos de VPH compite con la amplificación de los genotipos de VPH-AR. Si bien existe una limitación en la cantidad de genotipos de VPH detectados por E7-mPCR, los oligonucleótidos de genotipos específicos de VPH reducen la competencia para la amplificación del ADN de múltiples infecciones por VPH 30.
La limitación en la cantidad de genotipos estudiados podría superarse por medio de un sistema multiparamétrico, como el empleado en el estudio de detección y genotipificación del VPH por PCR Multiplex de L1 y tecnología Luminex en mujeres colombianas con citología anormal, donde la matriz de Luminex permitió la detección de 27 genotipos de VPH y además mostró una sensibilidad significativamente más alta en comparación con la hibridación reversa en fase sólida (RLB, reverse line blotting)31. Otra limitación tiene que ver con la falta de secuenciación de los productos de PCR para corroborar los resultados discordantes entre ambas pruebas. Se tendrá en cuenta este soporte de validación biológica y molecular para futuros estudios en la medida en que se estandaricen nuevas pruebas multiplex y se aumente la cobertura de los genotipos de VPH- AR a analizar.
Conclusiones
A pesar de que la citología disminuye la incidencia de CCU 32, el alto porcentaje de falsos negativos refleja su baja sensibilidad y la necesidad de múltiples citologías para la detección temprana de lesiones precancerosas. Por esta razón, para avanzar en la tamización del CCU es necesaria una prueba lo suficientemente sensible, específica y con un alto valor predictivo negativo que detecte oportunamente el agente causal del CCU, que permita intervalos de detección más espaciados cuando los resultados son negativos y que disminuya los costos, que permita una mayor cobertura y tratamientos más oportunos. Las técnicas moleculares para la genotipificación de VPH ofrecen estas ventajas en comparación con la citología 33,34, además de una mayor protección (de 60 a 70%) contra el carcinoma invasivo de cuello uterino 35 y una alta sensibilidad en la detección de lesiones glandulares en mujeres VPH positivas con resultados normales en la citología 36,37.
De las diferentes técnicas moleculares disponibles para la genotipificación del VPH, la que se usa con mayor frecuencia es la PCR, principalmente empleando cebadores consenso 38,39; sin embargo, se ha demostrado que los conjuntos de cebadores específicos son mejores para la genotipificación de VPH-AR 7,40. Al considerar esto, el presente estudio desarrolló una mPCR con cebadores específicos para el oncogén E7 de los seis genotipos VPH-AR más frecuentes, en la que los amplificados se diferenciaron en función de su tamaño en pares de bases, sin necesidad de procesamientos adicionales, se redujeron tiempos y costos en la identificación en comparación con otros métodos que requieren un procesamiento adicional, ya sea en digestión con enzimas de restricción 41, hibridación 42 o análisis de secuencia 43.
Adicionalmente, se pudo comprobar que un ensayo para la detección del gen L1 puede omitir infecciones que potencialmente evolucionen a fenotipos malignos, por esto las pruebas de detección y genotipificación de VPH que detectan preferentemente genes que se mantienen y contribuyen en la transformación maligna del epitelio infectado, como es el caso del oncogén E7, pueden ofrecer una mayor sensibilidad en la genotipificación de los VPH-AR.
Finalmente, este ensayo puede ser una herramienta diagnóstica más sensible, rápida y económica para detectar los seis genotipos de VPH-AR más frecuentes, cuyo siguiente nivel de aplicación es trasladarse a un sistema de alto desempeño multiparamétrico tipo Luminex®, esta plataforma es más robusta y permite procesar gran cantidad de muestras y ampliar la cobertura de los genotipos de VPH-AR. Asimismo, se deben realizar estudios de validación de la técnica para que posteriormente se pueda utilizar para el diagnóstico temprano de CCU.