Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Ciencia e Ingeniería Neogranadina
Print version ISSN 0124-8170On-line version ISSN 1909-7735
Cienc. Ing. Neogranad. vol.21 no.1 Bogotá Jan./June 2011
Henry Arguello Fuentes2
1M.Sc.(c), Grupo de Investigación en Ingeniería Biomédica, Escuela de Ingeniería Sistemas e Informática, LP 338, Universidad Industrial de Santander, Bucaramanga - Colombia.
fabiancardozo@gmail.com
2Ph.D.(c), Profesor Asistente Universidad Industrial de Santander, Grupo de investigación en Ingeniería Biomédica, Escuela de Ingeniería Sistemas e Informática, Bucaramanga - Colombia.
henarfu@uis.edu.co
Fecha de recepción: 7 de diciembre de 2010 Fecha de aprobación: 19 de mayo de 2011
RESUMEN
Una de las más recientes representaciones de conocimiento bajo incertidumbre son las Redes Bayesianas cuyo mayor atractivo es la propiedad de poder obtener dicha representación a partir de una gran cantidad de datos. El problema radica en que obtener la estructura de una red (procedimiento comúnmente llamado aprendizaje) es un problema NP-Duro, por lo cual se ha realizado una gran cantidad de trabajos para hacer el aprendizaje en los cuales, uno de los enfoques más conocidos es el llamado Basado en puntaje y búsqueda. Este artículo revisa las definiciones básicas de las Redes bayesianas, el enfoque basado en puntaje y las búsquedas y sus derivados, esto es, el enfoque híbrido y la búsqueda de clases de equivalencia; además, describe algunos algoritmos para cada enfoque y presenta un resumen de los resultados de los últimos trabajos realizados.
Palabras clave: redes bayesianas, aprendizaje basado en puntaje y búsqueda, aprendizaje híbrido, aprendizaje de clases de equivalencia.
ABSTRACT
One of the most recent knowledge representations under uncertainty are Bayesian Networks whose main captivation is the property to obtain such a representation from a large amount of data. The issue is that getting a network structure is a NP-hard problem -commonly a learning process-, so there has been a lot of learning work where one of the best known methods is called based scoring and search approach. This paper reviews the basic definition of Bayesian networks, the scoring-and-search-based approach and by-products, that is, the hybrid approach and the search for equivalence classes; in addition, describes some algorithms for each approach and gives a summary of results of recent work.
Key words: bayesian networks, scoring and search learning, hybrid learning, equivalence classes learning.
INTRODUCCIÓN
La representación de redes de interacciones por medio de modelos matemáticos, requiere extraer los aspectos de interés del fenómeno biológico para lograr una apropiada descripción. Según l-Chun Chou et al [1] se debe tener en cuenta los siguientes aspectos en las interacciones de un sistema biológico para llevar a cabo dicha labor: la alta complejidad y la no linealidad de sus relaciones, las respuestas dinámicas y jerarquía de sus elementos, es decir, los elementos en sus interacciones que pueden pertenecer a diferentes niveles - de proteínas, genes o metabolitos - y el comportamiento estocástico de sus elementos, especialmente cuando existen pocas moléculas de una misma especie.
Existen varios modelos matemáticos que se han propuesto para representar redes de interacciones que involucran la regulación de la expresión genética. Diferentes revisiones se han hecho en la bibliografía, desde descripciones generales [2], evaluación de su desempeño [3], estudio de la información en las entradas (datos experimentales), las salidas (inferencias del modelo) [4], y la revisión de los supuestos biológicos y limitaciones de cada uno [5], [6]. Los modelos más utilizados descritos son las Redes Booleanas, las Ecuaciones Diferenciales Ordinarias lineales y no lineales (tales como modelos canónicos o de Michaelis Menten), y los Modelos Gráficos Probabilísticos (los más conocidos, las Redes Bayesianas).
La representación por escoger depende entonces de ciertos criterios [1] que son específicos a los datos disponibles y a las respuestas que se desea responder, que para el tipo de redes que envuelven genes y proteínas, estos datos, en especial de series de tiempo, son muy difíciles de obtener y los efectos medidos son vistos más desde las consecuencias de los procesos (ejemplo: activación o inhibición) que desde los procesos mismos (ejemplo: síntesis, degradación, etc.), haciendo que modelos matemáticos como las redes bayesianas sean muy apropiados.
Las Redes bayesianas han sido recientemente aceptadas y son muy utilizadas para representar redes de regulación de la expresión genética y redes de señalización [7], [8], [9], [10], [11], [12], [13], [14] [15] y [16] entre otros, porque según citan [17], [18], [19] y otros, este tipo de representación presenta las siguientes ventajas: logran presentar las interrelaciones de los elementos como un todo, y no sólo por sus partes, por su representación multivariable; tratan el problema del ruido de los datos experimentales, describen las complejas relaciones de los elementos con naturaleza probabilística y no lineal, representan las relaciones causales de las interacciones y manejan eventos que no han sido observados, y la incertidumbre inherente ellos.
El problema es entonces, obtener una Red Bayesiana como una representación de conocimiento cuantitativo de las interacciones en una red biológica, de tal forma que sea posible hacer inferencias sobre el fenómeno biológico con una red dada. Formalmente, el problema es obtener la estructura y parámetros de una Red Bayesiana a partir de un conjunto de muestras de los elementos de la red. La dificultad radica en que debido al orden exponencial del número de posibles redes bayesianas en el espacio de búsqueda, el problema se define como NP-duro [24]. Por ello, se han propuesto diversos métodos heurísticos para hallar aproximaciones en la predicción de redes [36]. Entre estos métodos, se encuentra el método basado en puntaje y búsqueda que puede ser definido como un problema de optimización para el cual existen diferentes modelos para dar un puntaje a la red de acuerdo con las muestras [25], y diferentes técnicas para reducir el espacio de búsqueda, ya sea utilizando el enfoque híbrido o buscando por clases de equivalencia [26], [36].
Este artículo revisa las definiciones básicas de las Redes bayesianas desde la teoría de grafos y de probabilidad. Además, se hace una descripción formal del problema de aprendizaje sobre el enfoque basado en puntaje, mostrando los diferentes modelos matemáticos y métodos de búsqueda en la literatura. A partir de este enfoque, presenta sus derivaciones, esto es, el enfoque híbrido y la búsqueda de clases de equivalencia. Por último, presenta un resumen de los resultados, al comparar los métodos más utilizados, y a partir de las conclusiones de esas comparaciones, se propone una hipótesis de un método de búsqueda que recoge las ventajas de los métodos anteriores.
1. REDES BAYESIANAS
Para describir qué es el aprendizaje de Redes Bayesianas, primero es necesario entender de donde proviene el concepto de dichas redes, a partir de la teoría de grafos y de probabilidad. Las siguientes definiciones proveen un marco adecuado para describir en detalle, lo que es una Red Bayesiana desde la definición de un dígrafo acíclico, hasta la distribución de probabilidad que la red representa, mostrando su cualidad principal de independencia condicional. Para una descripción mucho más detallada, se puede consultar [20], [21], [22] y [23].
Definición 1: Un Grafo es un par G = (X,A), tal que X es un conjunto finito de elementos llamados nodos, y A es un subconjunto del conjunto {{xi,xj,}; xi,Xj∈ x}, que representa el conjunto de las relaciones entre los nodos (a los elementos de A se les llamará arcos o aristas). Un grafo puede ser representado con una matriz de adyacencia Ma, siendo ésta el arreglo [mi,j]i,j≤ x tal que:
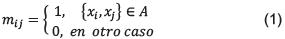
Definición 2: Un grafo dirigido o dígrafo, es un grafo G = (X,A), donde X es un conjunto finito y A es un subconjunto de X2. Nótese que todo elemento (xi,xj) ∈ A, tiene un orden o dirección.
Definición 3: Dado un dígrafo G = (X,A), un camino dirigido es una secuencia {x1,x2,...,xp), tal que para todo i ≤ p se cumple que xi ∈ X y para todo i ≤ p-1 se cumple que (xi,xi+1) ∈ A.
Definición 4: Un grafo acíclico dirigido o dígrafo a... clico es un grafo G tal que para todos los caminos dirigidos de la forma (x1,x2 ,...,xp), en G, se cumple que x1 ≠ xp .
Definición 5: Una red causal es una tripleta (G,fx, Q), tal que G = (X,A) es un dígrafo, Q es un conjunto finito y fx:X → Q es una función donde los elementos de X son llamados variables de la Red (para este caso serán de tipo discreto), y Q es llamado el conjunto de estados de los elementos de X.
Definición 6: Se dice que dos variables xi y xj de una Red Causal están direccionalmente separadas (o d-separadas), si existe una variable intermedia xk (diferente de xi y xj ), y se cumple una de las siguientes condiciones:
- Existe una configuración de la forma xi → xk → xj o de la forma xi ← xk ← xj y se conoce el estado de la variable xk.
- Existe una configuración de la forma xi ← xk → xj y se conoce el estado de la variable xk.
- Existe una configuración de la forma xi → xk ← xj y no se conoce el estado de la variable xk
Si xi está separada de xj, dado el conocimiento del estado de la variable xk, se dice que xi y xj son condicionalmente independientes, dado xk y se denota (xi l Xj ⊥ Xk).
Dado (G,fx,Q), una Red Causal y dado xi, un nodo de G, sea πi el conjunto {xp xp → xi}, llamado el conjunto de padres de xu sea ; una instancia de los estados de las variables que pertenecen aπi (o configuración deπi ), sea qi el tamaño del conjunto {j j es una configuración deπi}, y sea r el tamaño del conjunto de estados Q.
Definición 7: Una Red Bayesiana es una 4-tupla (G,fx,Q,Θ), que representa una Distribución de Probabilidad Conjunta donde:
- (G,fx, Q) es una Red Causal
- G es un dígrafo acíclico.
- El conjunto X de nodos de G es un conjunto {xi i ≤ n}, de variables aleatorias con r estados posibles y Θ es el conjunto {Θi i ≤ n}y Θi = {P(xi = k πi = j) k ∈ Q y j es una configuración de los padres de xi) donde P(xi = k πi = j), denota la probabilidad de que el estado xi sea k, dado que la configuración de padres es j.
De aquí en adelante, se nombrará al dígrafo G de la Red Bayesiana, como la estructura de la red. La Distribución de probabilidad conjunta que representa la Red Bayesiana, de acuerdo con la regla de la cadena, puede ser expresada como la multiplicación de las probabilidades condicionales de cada variable xi, dadas las variables que la preceden, pero si dos variables aleatorias xi,xj ∈ X, son condicionalmente independientes, dado el conocimiento del estado de xk, se cumple que:
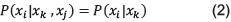
Entonces de acuerdo con esta propiedad, la expresión de la distribución conjunta se reduce a:
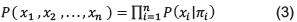
Una característica de una Red Bayesiana, es su capacidad para inferir la probabilidad de cómo puede ser el estado de sus variables, dado que se conoce el estado de una o varias de ellas. A esta probabilidad, se le conoce como la probabilidad a posteriori que es posible obtenerla por medio del Teorema de Bayes.
2. APRENDIZAJE DE LA ESTRUCTURA DE UNA RED BAYESIANA
El problema de aprendizaje de una Red Bayesiana, es el problema que se estudiará en esta propuesta, y se centra en encontrar una estructura que se ajuste a un conjunto de datos, evaluando qué tanto se ajusta una hipotética red a los datos, cuando se escoge la de mejor puntaje. A este enfoque, se le denomina aprendizaje basado en puntaje y búsqueda que se describirá a continuación.
Aprendizaje basado en puntaje y búsqueda: Este enfoque contiene tres elementos (EB,Sf,M), donde:
El Espacio-B, EB , representa el conjunto de todas las estructuras posibles de Redes Bayesianas con un número definido de nodos dado un conjunto de datos. De acuerdo con el número de nodos del grafo, el número de posibles estructuras crece exponencialmente, de manera que para una red de diez nodos, el número de posibles estructuras llega al orden de 10 . El problema de búsqueda de estructuras de acuerdo con este enfoque, es un problema NP-Duro según lo demostrado en [24], por lo cual se ha propuesto el uso de métodos heurísticos de búsqueda para hacer aproximaciones aceptables a posible soluciones.
La Función de puntaje, Sf:EB → 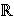 , representa una medición sobre qué tan cercanos están una estructura seleccionada y un conjunto de datos o con qué probabilidad puede una estructura de una Red Bayesiana, generar un conjunto de datos. La función de puntaje tiene las siguientes propiedades [25], [22]:
, representa una medición sobre qué tan cercanos están una estructura seleccionada y un conjunto de datos o con qué probabilidad puede una estructura de una Red Bayesiana, generar un conjunto de datos. La función de puntaje tiene las siguientes propiedades [25], [22]:
Separable: La función de puntaje para le estructura de la red, puede ser definida como la suma de los puntajes para cada nodo, de acuerdo con sus padres en comparación con el conjunto de datos para dicha variable, y sus padres, también llamados puntajes locales o familiares [18].
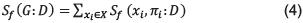
Equivalente: La función de puntaje asigna con el mismo valor a estructuras de Redes Bayesianas equivalentes pero este criterio no se cumple en todos los casos [26].
Para darle un puntaje a cada estructura de una Red Bayesiana, es necesario empezar con la estimación de cada parámetro de su estructura, basado en un simple valor Nijk que es el número de ocurrencias de la variable i en el estado k, dada una configuración j de sus padres en el conjunto de datos. A partir de allí se define el grado de verosimilitud máximo 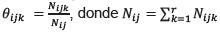 [27].
[27].
Con base en lo anterior se presentarán algunas de las funciones de puntaje que han sido propuestas y que son ampliamente conocidas. Una de las funciones de puntaje más utilizadas [25], es el Criterio de información Bayesiana, basado en la Teoría de la información, que define un balance entre el logaritmo del grado de verosimilitud máximo para todos los parámetros de la estructura y la complejidad de la estructura, sumando lo primero y restando (penalizando), lo segundo así:
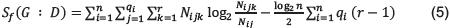
Donde n denota el número de variables, qi el número de configuraciones de los padres de la variable Xi y r el número de estados posibles para cualquier variable.
Una segunda función de puntaje fue propuesta en G.F. Cooperetal [20], llamada Métrica k2 basada en diferentes suposiciones (distribución de variables discretas-multinomial, independencia de los eventos, datos sin valores faltantes, uniformidad en la distribución de probabilidad de los parámetros), y se expresa de la siguiente manera:
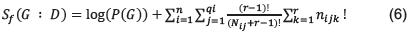
Una generalización de la función anterior es la denominada Función Bayesiana de Dirichlet propuesta por D. Heckerman [21], basada en la probabilidad a posteriori de una estructura de una Red Bayesiana dado un conjunto de datos, expresada así:
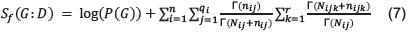
Donde Γ(x)es la función Gamma que es una extensión de la función factorial para los números reales y nijk son los hiperparámetros de la Distribución de Dirichlet a priori, dada la estructura de la red.
Más recientemente, se ha propuesto funciones de puntaje basadas en la métrica de información Mutua [25], o se han hecho formas alternativas para evaluar una estructura, utilizando algoritmos de inferencia [28]. Por último, M es un algoritmo que busca la mejor estructura 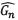 de una Red Bayesiana tal que:
de una Red Bayesiana tal que:
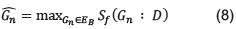
Uno de los primeros algoritmos propuestos fue el llamado K2 por G.F. Cooper [20], que busca el conjunto de padres de cada xx ê X a partir de un conjunto ordenado de variables predefinido (x1 ,x2, ....,xi-1), y un conjunto de estados máximo para las variables, de tal forma que se va añadiendo un elemento al conjunto de padres, de manera que cada adición sea la que más alto puntaje se obtenga según la función de puntaje Sf. Una posible mejora del algoritmo K2, consiste en encontrar un orden para los nodos, y no definirlos en forma arbitraria como lo hace Numata [7] y Xue Wen Chen [11].
Una primera aproximación a este problema utilizando Algoritmos Genéticos (para una introducción a este tipo de algoritmo se encuentra en el artículo de Sivanandam [29]) fue realizada por Larrañaga [30], de tal forma que cada cromosoma en el algoritmo codifica una matriz de adyacencia C que representa a la estructura G de una Red Bayesiana, y cada hijo es corregido por un operador que quita ciclos, si llegan a existir. Este algoritmo tiene la ventaja de que no necesita un orden predefinido de nodos como en k2.
Otro algoritmo evolutivo utilizado es la Programación Evolutiva [31], que utiliza la misma representación de Larrañaga [30] con cinco operadores para la generación de hijos, y recurre a un espacio de búsqueda reducido utilizando el test de Independencia condicional. Este algoritmo surge tras hacer una evaluación de fallas encontradas en los algoritmos genéticos que según el autor, se encuentran en el operador de cruce, y del mejoramiento de un algoritmo basado en programación evolutiva que realiza el mismo autor.
Existe también el algoritmo de Optimización por Colonia de Hormigas (para una completa explicación de esta clase de algoritmo consultar a M. Dorigo [32]) propuesto por L. M. de Campo [33] donde cada camino de una hormiga k es representado como un 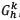 , de tal forma que cada arista añadida en el camino, representa la unión de dos nodos cualquiera (xi, xi ), en una estructura G de una Red Bayesiana y la probabilidad con la cual una hormiga k añade una arista en su camino, depende de una información heurística nij = ƒ(xi ,πi U xj)- ƒ(xi,πi). Este algoritmo utiliza el algoritmo K2 para generar un conjunto inicial de hormigas y el Hill Climbing CH [18], para hacer búsquedas intermedias cada tstep pasos, y, según el autor, es mejor en rendimiento que el algoritmo CH.
, de tal forma que cada arista añadida en el camino, representa la unión de dos nodos cualquiera (xi, xi ), en una estructura G de una Red Bayesiana y la probabilidad con la cual una hormiga k añade una arista en su camino, depende de una información heurística nij = ƒ(xi ,πi U xj)- ƒ(xi,πi). Este algoritmo utiliza el algoritmo K2 para generar un conjunto inicial de hormigas y el Hill Climbing CH [18], para hacer búsquedas intermedias cada tstep pasos, y, según el autor, es mejor en rendimiento que el algoritmo CH.
Una mejora a este algoritmo, es realizada por Jun-Zhong [34], que añade un parámetro w a la información heurística basado en la métrica de Información Mutua, ya que define la dependencia entre los nodos candidatos a unirse. Además, antes de hacer la búsqueda, utilizando la métrica de información Mutua de orden cero, se reduce el espacio de búsqueda.
Otros métodos utilizados para la búsqueda de estructuras, son los propuestos por Zhihua Du [9] y P. A. D. Castro [35] los cuales utilizan el método de Optimización por Enjambre de partículas y Sistemas Inmunes Artificiales respectivamente.
Aprendizaje Híbrido: Este enfoque, en vez de seguir el enfoque anterior, mide la independencia condicional de las variables, siguiendo una medida llamada Información Mutua con base en el test ch i-cu adrad o [22], y se compara con un umbral a. La información mutua de dos variables xi y xj, dado un conjunto z de variables intermedias entre ellas, se define como:
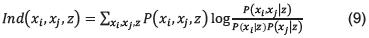
El orden de la medida de Información Mutua es el número de elementos del conjunto z. A partir de esta medida se construye una red que contiene las separaciones direccionales correspondientes a las independencias condicionales medidas.
Después del procedimiento anterior, el resultado es una reducción del espacio de búsqueda EB, descartando relaciones entre nodos que no cumplen con la métrica de Información Mutua dado el umbral a y luego, basado en el espacio factible generado, se realiza la búsqueda con base en puntaje y búsqueda. Por lo tanto, este enfoque se llama híbrido por que une el enfoque de aprendizaje basado en puntaje y búsqueda, con la predicción dada por la medida de independencia condicional.
Entre los trabajos realizados con este enfoque, se encuentra el de Man Leung Wong [31], que utiliza una búsqueda basada en Programación Evolutiva, utilizando los cuatro operadores mencionados en la sección anterior; el realizado por Jun-Zhong Jl [34], que añade al trabajo de Luis M. de Campos [33], el test de Independencia condicional para reducir el espacio de búsqueda; el desarrollado por Xue wen Chen [11], que construye un grafo inicial no dirigido basado en la métrica de información mutua y luego, obtiene un conjunto ordenado de nodos para hacer la búsqueda por el algoritmo K2; el desarrollado por Laura E. Brown loannis Tsamardinos [36], que llama a su algoritmo Max-Min Hill-Climbing (MMHC), haciendo una reducción del espacio de búsqueda con un algoritmo llamado MMPC (basado en el análisis de las dependencias entre variables); el de Gui Xia Liu [8], que realiza una búsqueda posterior basada en un algoritmo genético que utilizan operadores basados en computación inmune propuesto en Licheng Jiao [37], (mas aplicaciones de estos operadores y su eficiencia en algoritmos como Colonia de Hormigas en [38], [39] y [40]), y más reciente el trabajo de PC. Pinto [41], que es una mejora al algoritmo MMHC, pero utiliza una representación con Colonias de Hormigas llamado MMACO que según el autor, obtiene mejores resultados que los trabajos anteriores.
3. APRENDIZAJE DE LAS CLASES DE EQUIVALENCIA
El concepto de clases de equivalencia de Redes Bayesianas, provee un componente clave para el problema de aprendizaje, ya que dos estructuras diferentes pueden describir la misma distribución de probabilidad; por esto, se dará una revisión rápida a las definiciones básicas sobre la equivalencia entre estructuras y clases de equivalencia; para más detalle, consultar en [26], [42], [43] y [44].
Definición 8: Dos estructuras G y G' son Markov Equivalentes si y sólo si para toda Red Bayesiana B = (G,f,Q,Θ), existe una Red Bayesiana B' = (G',f,Q,Θ') tal que B y B' tienen la misma distribución de probabilidad y describen el mismo conjunto de independencias.
Definición 9: Dado un dígrafo acíclico G, el esqueleto de G es el grafo no dirigido que se obtiene al omitir la dirección de las aristas de G.
Definición 10: Una estructura-v es una tripleta (xi,xj,xk) EX tal, que se cumple que: xi → xj ,xj ← xk y la arista (xi,xk), no pertenece a A. En otras palabras, es una estructura de la forma xi→ xi ← xk.
Teorema 1: Dos estructuras G y G' son equivalentes si y solo si tienen el mismo esqueleto y las mismas estructuras-v.
Definición 11: Un dígrafo acíclico parcial es un grafo acíclico que contiene aristas dirigidas y no dirigidas.
Dado el espacio no vacío EB de estructuras de Redes Bayesianas, la relación de equivalencias entre estructuras en dicho espacio, denotada como ∼, cumple las siguientes propiedades :
Reflexividad: ∀G ∈ Eb G ∼ G (10)Simetría: ∀G, G'∈ EB SIG∼G'⇒ G' ∼ G (11)
Transitividad: ∀G,G',G''∈EBSIG∼G'∧G'∼G⇒G''' (12)
Luego dicha relación es de equivalencia.
Definición 12: Dado un dígrafo acíclico parcial P, se define su clase de equivalencia así:
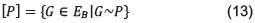
Si la arista xi → xj está presente en todo G £ [P], se dice que xi → xj es forzada. Si una arista xi → xj no es forzada, se dice que es reversible.
Definición 13: Un dígrafo acíclico parcial completo es un dígrafo acíclico parcial tal, que toda arista forzada es una arista dirigida, y toda arista reversible es una arista no dirigida.
Teorema 2: Dado un espacio de estructuras de Redes Bayesianas EBexiste un único dígrafo acíclico parcial completo por cada clase de equivalencia en dicho espacio.
El aprendizaje de Clases de Equivalencia difiere del aprendizaje estructural en que el Espacio de Búsqueda de las clases de equivalencia, denominado EE, tiene las siguientes propiedades:
- Contiene un conjunto de estados que son las clases de equivalencia que conforman dicho espacio.
- Cada estado tiene una representación que es un grafo acíclico parcial completo.
- Existe un conjunto de operadores para hacer posible el movimiento entre los estados en un solo paso.
Luego la principal ventaja de este enfoque, es que dado que los operadores permiten el paso de una clase a otra, se evita el movimiento dentro de una misma clase que desfavorecía la búsqueda en el espacio EB, puesto que la función de puntaje es equivalente (para más detalle consultar en [26], [42], [43] y [44]). A pesar de la ventaja que tiene la búsqueda de clases de equivalencias (más precisamente de grafos acíclicos parciales completos), la búsqueda no es trivial, puesto que el número de clases de equivalencia también crece exponencialmente, de acuerdo con la forma como crece el número de nodos [45], [46] y [47].
Este enfoque ha sido revisado por algunos autores, como [27], que hace una revisión de búsqueda de clases de equivalencia utilizando algoritmos evolutivos [48], que utilizan los operadores para moverse en distintas clases de equivalencia al momento de la construcción de un camino, utilizando el método de optimización de Colonia de Hormigas propuesto por [33], [49], [50], que desarrolló un mejoramiento en la búsqueda en EE, utilizando un algoritmo híbrido entre un algoritmo genético, utilizando características de Sistemas Inmunes Artificiales que aplican un operador de vacuna, utilizando el test de Independencia Condicional y por último, el algoritmo realizado por [51], denominado ACO-E que mejora los algoritmos anteriores, utilizando Colonia de Hormigas (extendiendo el trabajo expuesto en Roñan Daly [48]), y utilizando una técnica para manejar más rápidamente las restricciones de los operadores [53].
4. EVALUACIÓN DEL APRENDIZAJE
La metodología de Evaluación del aprendizaje que se usa en gran parte de la literatura (entre las más recientes [36], [41] y [51]), con el siguiente protocolo:
- Se escoge un conjunto de Redes Bayesianas conocidas (que son estándar sus estructuras y parámetros1).
- Para cada una de las redes se generan diferentes conjuntos de datos que varían desde 50 muestras por variables dentro de la red, hasta 10000.
- Para cada uno de los conjuntos de datos, se ejecuta cada algoritmo por evaluar.
- Para cada algoritmo, tanto en ejecución como con la red obtenida, se hace la evaluación, utilizando las siguientes métricas [36] y [52]:
Función de Puntaje: Es la métrica utilizada en el algoritmo para evaluar la cercanía de la red construida y los datos, y como tal, es la que primero evalúa los resultados del algoritmo.
Distancia Estructural de Hamming (SHD, por sus siglas en inglés): Es el promedio entre los arcos añadidos, omitidos e invertidos (en dirección), entre la estructura generada por el algoritmo y la estructura de la cual se generaron los datos iniciales. La obtención de esta métrica se describe en el Algoritmo 1, donde H y G son los dígrafos acíclicos parciales obtenidos por el algoritmo como el inicial de donde se generaron los datos respectivamente.
Número de evaluaciones o llamadas estadísticas (NSC, por sus siglas en inglés): Devuelve el número de llamadas que hace el algoritmo a la función objetivo o a otras funciones matemáticas que se utilicen en el algoritmo, como por ejemplo: la función de análisis de dependencia. Cabe anotar que es una métrica que mide la complejidad del algoritmo.
Algoritmo 1: Algoritmo SHD tomado de [36]
Entrada: H construida, G Verdadera
Salida: shd
1. shd = 0;
2. para cada arco E en H diferente en G hacer
3. si E no está en H entonces
4. shd = shd + 1;
5. fin si
6. si E esta en H y no en G entonces
7. shd = shd + 1;
8. fin si
9. si E esta en H en diferente dirección que G entonces
10. shd = shd + 1;
11. fin si
12. fin para
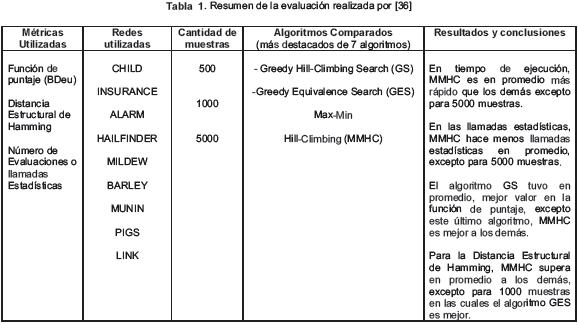
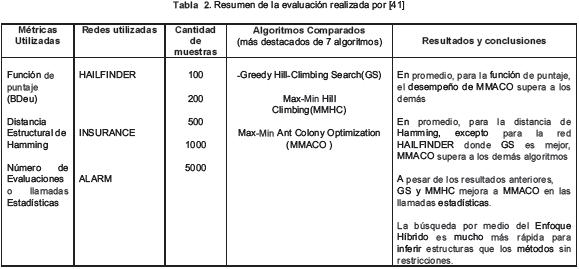
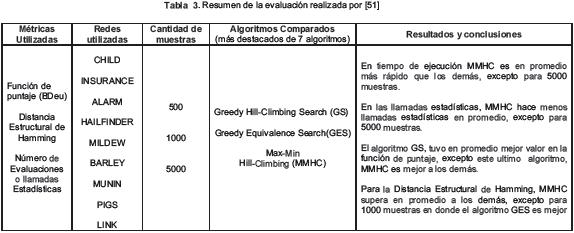
Se escogieron los resultados obtenidos en los trabajos de [36], [41] y [51], por que presentan una comparación de los últimos y más conocidos algoritmos, utilizando el protocolo ya mencionado. Para cada uno, como se muestra en los cuadros I, II y III del Anexo A, se especifica las métricas utilizadas para evaluar las Redes Bayesianas utilizadas, la cantidad de muestras, los algoritmos evaluados, los resultados obtenidos de acuerdo con cada métrica y una conclusión.
5. CONCLUSIONES Y DISCUSIÓN
En este trabajo, se encontró que la obtención de una estructura de una Red Bayesiana que mejor se ajuste a un conjunto de datos, se ha tratado de diferentes maneras, pero se puede concluir lo siguiente:
Hay mayor rendimiento, si se evita la búsqueda redundante (e ineficiente), en una misma clase de equivalencia, utilizando operadores que permitan el movimiento entre diferentes clases de equivalencia en la búsqueda, como se ha demostrado en David Maxwell [26], utilizando el algoritmo Greedy-E.
Hay mayor rendimiento del algoritmo, si se utiliza un enfoque híbrido, es decir, si antes de hacer una búsqueda heurística, se reduce el espacio de búsqueda haciendo análisis de dependencia, como lo es el trabajo de Laura E. Brown loannis Tsamardinos [36], que utiliza el algoritmo MMPC para reducir el espacio de búsqueda.
El trabajo de Laura E. Brown loannis Tsamardinos [36], describe que cuando se utiliza el algoritmo MMPC para reducir el espacio de búsqueda, se obtiene mayor rendimiento que el algoritmo Greedy-E [26], a pesar de que realiza la búsqueda entre clases de equivalencia.
Basados en los trabajos de David Maxwell [26] y Laura E. Brown loannis Tsamardinos [36], se han realizado varios trabajos, pero entre los más recientes, se destacan dos de ellos que utilizan Optimización basado en Colonia de Hormigas, se hace referencia al algoritmo llamado ACO-E de [51], que utiliza los operadores introducidos en David Maxwell [26] y al de P.C. Pinto [41], llamado MMACO que utiliza el algoritmo MMPC diseñado por Laura E. Brown loannis Tsamardinos [36]. Ambos trabajos siguen la metodología de evaluación descrita en el apartado anterior, y los resultados muestran un mayor desempeño que los algoritmos que los anteceden.
Se han realizado pocos trabajos [49], [50] que combinen el enfoque híbrido que utiliza el análisis de dependencia, y el enfoque del aprendizaje de las clases de equivalencia.
De acuerdo con lo anterior, se plantea realizar, en trabajos futuros, el aprendizaje de la Red Bayesiana, utilizando como base el algoritmo de Colonia de Hormigas y combinando el enfoque híbrido para reducir el espacio de búsqueda según el algoritmo MMACO y el uso de operadores para moverse entre clases de equivalencia de acuerdo con el algoritmo ACO-E.
AGRADECIMIENTOS
Los autores agradecen a la Universidad Industrial de Santander (UIS) y al Departamento Administrativo de Ciencia, Tecnología e Innovación de la República de Colombia (COLCIENCIAS), por al apoyo a este trabajo parcial bajo el programa de Jóvenes Investigadores e Innovadores con número P-2009-0189. Este trabajo fue financiado por la Vicerrectoría de Investigación y Extensión de la Universidad Industrial de Santander, con el proyecto de investigación de código interno 5537 y titulado: Sistema en tiempo real de verificación de identidad a través de la imagen facial.
Pie de página
1http://www.compbio.cs.huji.ac.il/Repository/
REFERENCIAS BIBLIOGRÁFICAS
[1] l-Chun Chou and Eberhard O. Voit, (2009). Recent developments in parameter estimation and structure identification of biochemical and genomic systems. Mathematical Biosciences, 219 (2): 57-83. [ Links ]
[2] Hidde de Jong, (2002). Modeling and simulation of genetic regulatory systems: A literature review. I n: Journal of Computational Biology, 9(1): 67-103. [ Links ]
[3] Alberto Ambesi-lmpiombato & Diego di Bernardo Mukesh Bansal, Vin- cenzo Belcastro, (2007). How to infer gene networks from expression profiles. In: Molecular systems biology, 3. [ Links ]
[4] K.-H. Cho, S.-M. Choo, S.H. Jung, J.-R. Kim, H.-S. Choi, and J. Kim, (2007). Reverse engineering of gene regulatory networks. In: Systems Biology, IET, 1(3): 149-163. [ Links ]
[5] Feng He, Rudi Balling, and An-Ping Zeng, (2009). Reverse engineering and verification of gene networks: Principles, assumptions, and limitations of present methods and future perspectives. In: Journal of Biotechnology, Press, Corrected Proof. [ Links ]
[6] Valdimir Filkov, (2005). Identifying gene regulatory networks from gene expression data. Handbook of Computational Molecular Biology, Handbook of Computational Molecular Biology. [ Links ]
[7] K. Numata, S. Imoto, and S. Miyano, (2007). A structure learning algorithm for inference of gene networks from microarray gene expression data using bayesian networks. In: Bioinformatics and Bioengineering. BIBE 2007. Proceedings of the 7th IEEE International Conference, pp. 1280-1284. [ Links ]
[8] Gui xia Liu, Wei Feng, Han Wang, Lei Liu, and Chun guang Zhou, (2009). Reconstruction of gene regulatory networks based on two-stage bayesian network structure learning algorithm. In: Journal of Bionic Engineering, 6 (1):86 -92. [ Links ]
[9] Zhihua Du, Yiwei Wang, and Zhen Ji, (2009). A new structure learning method for constructing gene networks. In: Bioinformatics and Biomedical en gineering, 2009. ICBBE 2009. 3rd International Conference, pp 1-4. [ Links ]
[10] Cedric Auliac, Vincent Frouin, Xavier Gidrol, and Florence d'Alche Buc, (2008). Evolutionary approaches for the reverse-engineering of gene regulatory networks: A study on a biologically realistic dataset. In: BMC Bioinformatics, 9(1):91. [ Links ]
[11] Xue wen Chen, Gopalakrishna Anantha, and Xinkun Wang, (2006). An effective structure learning method for constructing gene networks. In: Bioinformatics, 22(11): 1367-1374. [ Links ]
[12] Dana Pe'er, (2003). From gene expression to molecular path ways. Ph.D. thesis, the Hebrew University. [ Links ]
[13] Alexander J. Hartemink, David K. Gifford, Tommi S. Jaakkola, and Richard A. Young, (2002). Bayesian methods for elucidating genetic regulatory networks. In: IEEE Intelligent Systems, 17(2):37-43. [ Links ]
[14] Alexander John Hartemink, (2001). Principled Computational Methods for the Validation and Discovery of Genetic Regulatory Networks. PhD thesis, Massachusetts institute of technology. [ Links ]
[15] Amira Djebbari and John Quackenbush, (2008). Seeded bayesian networks: Constructing genetic networks from microarray data. In: BMC Systems Biology, 2(1):57. [ Links ]
[16] Chris Needham, lain Manfield, Andrew Bulpitt, Philip Gilmartin, and David Westhead, (2009). From gene expression to gene regulatory networks in arabidopsis thaliana. In: BMC Systems Biology, 3(1):85. [ Links ]
[17] Chris J. Needham, James R. Bradford, Andrew J. Bulpitt, and David R. Westhead, (2007). A primer on learning in bayesian networks for computational biology. In: PLoS Comput Biol, 3(8):129. [ Links ]
[18] Dana Pe'er, (2005). Bayesian network analysis of signaling networks: A primer. Sci. STKE, 2005(281 ):pl4. [ Links ]
[19] Karen Sachs, Omar Perez, Dana Pe'er, Douglas A. Lauffenburger, and Garry P. Nolan, (2005). Causal protein-signaling networks derived from multiparameter single-cell data. In: Science, 308(5721 ):523-529. [ Links ]
[20] Gregory F. Cooper and Edward Herskovits, (1992). A bayesian method for the induction of probabilistic networks from data. In: Machine Learning 9, Number 4:309-347. [ Links ]
[21] David Heckerman, (1995). A tutorial on learning with bayesian networks. In: Technical report, Microsoft Research. [ Links ]
[22] Finn V. Jensen and Thomas D. Nielsen, (2007). Bayesian Networks and Decision Graphs. Springer Science + Business Media, LLC [ Links ]
[23] Richard E. Neapolitan, (2003). Learning Bayesian Networks. Prentice Hall; illustrated edition. [ Links ]
[24] Dan Geiger David M. Chickering and David Heckerman, (1994). Learning bayesian networks: is NP-Hard. In: Technical report, Microsoft Research. [ Links ]
[25] Luis M. de Campos, (2006). A scoring function for learning bayesian networks based on mutual information and conditional independence tests. In: The Journal of Machine Learning Research, 7:2149-2187. [ Links ]
[26] David Maxwell Chickering, (2002). Learning equivalence classes of bayesian network structures. In: The Journal of Machine Learning Research, 2:445-498. [ Links ]
[27] Jorge Muruzabal and Carlos Cotta, (2004). A primer on the evolution of equivalence classes of bayesian network structures, volume 3242, pages 612-621. [ Links ]
[28] Adamo L. de Santana, Carlos R. Frances, Claudio A. Rocha, Solon V. Carvalho, Nandamudi L. Vijaykumar, Liviane P. Rego, and Joao C. Costa, (2007). Strategies for improving the modeling and interpretability of bayesian networks. In: Data & Knowledge Engineering, 63(1):91-107. Data Warehouse and Knowledge Discovery (DAWAK05), 7th International Congress on Data Warehouse and Knowledge Discovery (DAWAK'05). [ Links ]
[29] S.N. Sivanandam and S.N. Deepa, (2007). Introduction to Genetic Algorithms. Springer-Verlag, 1st edition. [ Links ]
[30] Pedro Larrañaga, Mikel Poza, Yosu Yurramendi, Roberto H. Murga, and Cindy M.H. Kuijpers, (1996). Structure learning of Bayesian networks by genetic algorithms: A performance analysis of control parameters. In: IEEE Transactions on Pattern Analysis and Machine Intelligence, 18(9):912-926. [ Links ]
[31] Man Leung Wong and Kwong Sak Leung, (2004). An efficient data mining method for learning bayesian networks using an evolutionary algorithm- based hybrid approach. Evolutionary Computation, In: IEEE Transactions on, 8(4):378-404. [ Links ]
[32] Marco Dorigo and Thomas Stutzle, (2004). Ant colony Optimization. Massachusetts Institute of Technology. [ Links ]
[33] Luis M. de Campos, Juan M. Fernández-Luna, José A. Gamez, and José M. Puerta, (2002). Ant colony optimization for learning Bayesian networks. In: International Journal of Approximate Reasoning, 31(3):291 -311. [ Links ]
[34] Jun-Zhong J.I., Hong-Xun ZHANG, Ren-Bing HU, and Chun-Nian LIU, (2009). A bayesian network learning algorithm based on independence test and ant colony optimization. In: Acta Automática Sínica, 35(3):281 -288. [ Links ]
[35] Pablo A.D. Castro and Fernando J. Von Zuben, (2005). An immune inspired approach to Bayesian networks. In: HIS'05: Proceedings of the Fifth International Conference on Hybrid Intelligent Systems, pages 23-28. Washington: IEEE Computer Society. [ Links ]
[36] Laura E. Brown loannis Tsamardinos and Constantin F. Aliferis, (2006). The max-min hill-climbing bayesian network structure learning algorithm. Machine Learning, 65(1):31-78. [ Links ]
[37] Licheng Jiao and Lei Wang, (2000). A novel genetic algorithm based on immunity. Systems, Man and Cybernetics, Part A: Systems and Humans. In: IEEE Transactions, 30(5):552-561. [ Links ]
[38] Zne-Jung Lee, Chou-Yuan Lee, and Shun-Feng Su, (2002). An immunity-based ant colony optimization algorithm for solving weapon target assignment problem. Applied Soft Computing, 2(1 ):39 - 47. [ Links ]
[39] Chui-Yu Chiu, l-Ting Kuo, and Chia-Hao Lin, (2009). Applying artificial immune system and ant algorithm in airconditioner market segmentation. In: Expert Systems with Applications 36(3, Part 1):4437 - 4442. [ Links ]
[40] Xianjin Fang and Longshu Li, (2008). An artificial immune model with vaccine operator for network intrusion detection. In Computational Intelligence and Industrial Application. PACIIA'08. Pacific-Asia Workshop, volume 1, pp. 488-491. [ Links ]
[41] P.C. Pinto, A. Nagele, M. Dejori, T.A. Runkler, and J.M.C. Sousa, (2009). Using a local discovery ant algorithm for Bayesian network structure learning. Evolutionary Computation. In: IEEE Transactions, 13(4):767-779. [ Links ]
[42] Paul Munteanu and Denis Cau, (2000). Efficient score based learning of equivalence classes of bayesian networks. In PKDD '00: Proceedings of the 4th European Conference on Principles of Data Mining and Knowledge Discovery, pp. 96-105. London: Springer-Verlag. [ Links ]
[43] P. Munteanu and M. Bendou, (2001). The eq framework for learning equivalence classes of bayesian networks. In: Data mining. ICDM 2001, Proceedings IEEE International Conference, pages 417-424. [ Links ]
[44] Silva Acid and Luis M. de Campos, (2003). Searching for bayesian network structures in the space of restricted acyclic partially directed graphs. In: Journal of Artificial Intelligence Research, 18:445-490. [ Links ]
[45] Steven Gillispie Department and Steven B. Gillispie, (2001). Enumerating markov equivalence classes of acyclic digraph models. In: Proc. of the Conf. on Uncertainty in Artificial Intelligence, pages 171-177. Morgan Kaufmann. [ Links ]
[46] Steven B. Gillispie and Michael D. Perlman, (2002). The size distribution for markov equivalence classes of acyclic digraph models. Artificial Intelligence, 141(1 -2): 137-155. [ Links ]
[47] Steven B. Gillispie, (2006). Formulas for counting acyclic digraph markov equivalence classes. In: Journal of Statistical Planning and Inference, 136(4):1410-1432. [ Links ]
[48] Ronan Daly, Qiang Shen, and Stuart Aitken, (2006). Using ant colony optimisation in learning bayesian network equivalence classes. Proceedings of the 2006 UK Workshop on Computational Intelligence, pages 111-118. [ Links ]
[49] Haiyang Jia, Dayou Liu, Juan Chen, and Xin Liu, (2007). A hybrid approach for learning markov equivalence classes of bayesian network. In: KSEM, pp. 611-616. [ Links ]
[50] Haiyang Jia, Dayou Liu, Juan Chen, and Jinghua Guan, (2008). Learning markov equivalence classes of bayesian network with immune genetic algorithm. In: Industrial Electronics and Applications. ICIEA 2008. 3rd IEEE Conference, pp.197-202. [ Links ]
[51] Ronan Daly and Qiang Shen, (2009). Learning bayesian network equivalence classes with ant colony optimization. Artificial Intelligence Research, 35:391-447. [ Links ]
[52] Shen Q. & Aitken-S Daly, R. (2006). Speeding up the learning of equivalence classes of bayesian network structures. In Proceedings of the Tenth IAST- ED International Conference on Artificial Intelligence and Soft Computing, pp.34-39. [ Links ]
ANEXO A. RESUMEN DE RESULTADOS
A continuación, se presenta un resumen de los resultados obtenidos en los trabajos de [36], [41] y [51] en las tablas 1, 2 y 3 respectivamente. Se especifican las métricas utilizadas para evaluar las Redes Bayesianas, la cantidad de muestras, los algoritmos evaluados, los resultados obtenidos de acuerdo con cada métrica y una conclusión. En ellos, se resume la comparación de diferentes algoritmos, utilizando el protocolo mencionado en la sección V.