Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Facultad Nacional de Agronomía Medellín
Print version ISSN 0304-2847
Rev. Fac. Nac. Agron. Medellín vol.59 no.2 Medellín July/Dec. 2006
Aplicación de una técnica de Cromatografía de Exclusión molecular para la purificación de ADN en plantas de Coffea sp.
APPLICATION OF A TECHNIQUE OF MOLECULAR EXCLUSION CHROMATOGRAPHY FOR THE PURIFICATION OF DNA FROM Coffea sp. PLANTS
Ana María García Cepero1; Yamel López Forero2 y Nestor Miguel Riaño Herrera3
1 Microbióloga. Clínica Cardiovascular Santa María. Calle 8B No. 75-21, Medellín, Colombia. <instinve@une.net.co>
2 Profesor Asociado. Universidad Nacional de Colombia, Sede Palmira. Facultad de Ciencias Agropecuarias. A.A. 0237, Palmira, Colombia. <yamel.lopez@cafedecolombia.com>
3 Investigador Científico II. Fisiología Vegetal. Centro Nacional de Investigaciones de Café. CENICAFÉ, Chinchiná, Caldas, Colombia. <nestorm.riano@cafedecolombia.com>
Recibido: Junio 1 de 2005; aceptado: Febrero 6 de 2006.
RESUMEN
Uno de los mayores inconvenientes en la extracción y purificación de biomoléculas a partir de plantas del género Coffea, es un alto contenido de polifenoles y compuestos tánicos. En el presente artículo se describe una metodología que permite obtener ADN de alta pureza. La extracción del ADN del homogeneizado de tejido foliar en siete genotipos de Coffea sp., se realizó mediante la técnica citada por Chaparro (1993) y su purificación se logró mediante cromatografía de exclusión molecular sobre una fase estacionaria de Sephacryl S-1000. Los resultados muestran que la alta eficiencia de separación de ARN degradado, proteínas, pigmentos y compuestos que absorben entre 220 y 300 nm, permiten obtener un ADN de alta pureza a juzgar por los datos espectrofotométricos y electroforéticos.
Palabras clave: Coffea arabica L., café, ADN, cromatografía de exclusión molecular.
ABSTRACT
One of the greatest difficulties in extracting and purifying biomolecules from plants in the genus Coffea is the high polyphenol and tannin contents. In this study a methodology is described that allows obtaining high purity DNA from leaf tissues of seven genotypes of Coffea sp. by means of the technique desribed by Chaparro (1993) and its further purification was achieved by molecular exclusion chromatography on Sephacryl S-1000 (Pharmacia). The results showed that the high separation efficiency of degraded RNA, proteins, pigments, and other compounds that absorb between 220 and 300 nm allowed obtaining high purity DNA as judged by the spectophometric and electroforetic data.
Key Words: Coffea arabica L., coffee, DNA, molecular exclusion chromatography.
 MATERIALES Y MÉTODOS
MATERIALES Y MÉTODOS
 RESULTADOS Y DISCUSIÓN
RESULTADOS Y DISCUSIÓN
 AGRADECIMIENTOS
AGRADECIMIENTOS
 BIBLIOGRAFÍA
BIBLIOGRAFÍA
Por las características bioquímicas del café y otras plantas relacionadas, en el momento de la homogenización de tejido foliar, se liberan metabolitos secundarios presentes en vacuolas, tales como complejos fenólicos que sirven de sustrato polifenóloxidasas (PPO), además de liberar clorofilas y taninos que se encuentran en los organelos de las células del mesófilo. Estos componentes son indeseables en los procesos de extracción de ADN y producen oxidaciones rápidas y de gran magnitud, en comparación con las de otras especies como Pisum sativum, cuyas características bioquímicas y metabólicas hacen que posea un contenido más bajo de tales metabolitos y menores tasas oxidativas (Guillemaut y Drovard 1992, Kanazawa y Tsutsumi 1992).
La utilización de PVP (Polivinipirrolidona), PVPP (Polivinilpolipirrolidona) y DIECA (Diotiocarbamato de sodio), inhiben la actividad de PPO facilitando la extracción de las biomoléculas de los tejidos (Couch y Fritz 1990).
En café a pesar de la utilización en altas concentraciones de los compuestos antes mencionados, no hay inhibición total de la actividad oxidativa, esto conlleva a utilizar métodos adicionales que permitan obtener muestras de ADN altamente purificadas para lograr realizar exitosamente los diferentes procesos enzimáticos indispensables en el desarrollo de la mayoría de las técnicas de biología molecular.
En café no se logra una inhibición total de PPO y otras oxidasas a pesar del uso de PVP, PVPP y DIECA en altas concentraciones, lo cual exige la modificación de las metodología para obtener muestras de ADN altamente purificadas, útiles en trabajos de biología molecular (Fuchs y Blakesley 1983).
MATERIALES Y MÉTODOS
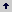
Material vegetal. Se utilizaron hojas jóvenes del segundo par de ramas de plantas de 1 año de edad, de la especie Coffea arabica cv. Caturra, Coffea arabica cv. Colombia, Coffea canephora, Coffea congensis, Coffea eugenioides y el Híbrido de Timor.
Aislamiento de ADN total. Se tomó tejido foliar (1-3 g) y se pulverizó en presencia de nitrógeno líquido. Al macerar se adicionaron 60 mg de PVPP y 90 mg de Dieca por cada gramo de tejido. Luego se agregaron 2 volúmenes de buffer de homogeneización (SDS 1 %, sacarosa 0,25 M, NaCl 50 mM, EDTA 20 mM, Tris-HCl 50 mM, pH 8,0), se agitó en vortex por 3 min y se llevó a incubación por 30 min a temperatura ambiente. La muestra fue extraída dos veces con fenol saturado en Tris-HCl 0,1 mM, pH 8,0 y cloroformoalcohol isoamilico (24:1) y una vez con cloroformoalcohol isoamílico (24:1). Se precipitó con etanol absoluto, se lavó con etanol al 70 %, y se resuspendió en agua destilada estéril (Chaparro 1993).
Purificación del ADN por columna de exclusión molecular. Se empacó una columna Econocolumn (Biorad) de 1 cm de diámetro y 30 cm de longitud con 15,7 ml de Sephacryl S-1000 superfine (PharmaciaBiotech). La columna se equilibró con 100 ml buffer (Tris-HCl 10 mM, pH 8,0, NaCl 200 mM, EDTA 0.5 mM) con flujo de 0,5 ml/min., para eliminar contaminantes e impurezas del soporte. En la columna se inyectaron entre 400-600 ml de la muestra y la cromatografía se corrió con un flujo de 1 ml min-1, utilizando el mismo buffer de cromatografía. Se recolectaron 23 fracciones de 2 ml cada una (50 ml en total), a partir del ml 4-5 (Bookjans, Stumman y Henningsen 1984).
Evaluación de las fracciones. Cada una de las fracciones fue evaluada por espectrofotometría haciendo un barrido entre 220-320 nm y se determinó la relación A260/280 de cada fracción. Las fracciones que presentaron picos de absorbancia alrededor de A260 y relaciones A260/280 mayor de 1,8 se precipitaron con etanol absoluto y acetato de sodio 3M, resuspendidas en 50 ml de agua desionizada estéril y corridas en una electroforesis en gel de agarosa al 0,8 % para determinar integridad y presencia de ARN. Las fracciones con mayor contenido de ADN, que no revelaron presencia de ARN y con relación A260/280 mayor de 1,8 se reunieron y fueron utilizadas para continuar el proceso (Sambroock, Fritsch y Maniatis 1989).
Análisis por electroforesis en gel de agarosa. El ADN se analizó por electroforesis horizontal en gel de agarosa al 0,8 % con bromuro de etidio (0,2 mg ml-1) y se utilizó como buffer TBE 1X (Tris-HCl 45 mM, pH 8,0, EDTA 10 mM, ácido bórico 45 mM) y 1 ml de buffer de carga 6X (Glicerol 30 %, azul de bromofenol 0,25 %, xilenocianol 0,25 %). La electroforesis se corrió a 60 voltios durante 2h y las bandas se visualizaron en un transiluminador (Sambroock, Fritsch y Maniatis 1989).
Análisis por espectrofotometría. Se determinaron las absorbancias a A260 y A280 nm., la relación A260/280 y se realizó un barrido entre 220 y 320 nm (Sambroock, Fritsch y Maniatis 1989).
Análisis de restricción. Se realizó análisis de restricción del ADN con la enzima EcoRI, siguiendo los protocolos descritos por Sambrook (1989). La muestra fue evaluada a través de una electroforesis horizontal en gel de agarosa al 0,8 % teñido con bromuro de etidio (Frischauf 1987, Palmer y Shields 1984, Sambroock, Fritsch y Maniatis 1989).
Evaluación de la concentración de ADN. Se evalúo por electroforesis horizontal en gel de agarosa al 0,8 % con bromuro de etidio y se utilizó el marcador de tamaño molecular y concentración Low DNA Mass Ladder de BRLGibco.
Extracción inicial de ADN total. Se obtuvo una concentración de 320 mg de ADN por gramo de tejido, evaluada por espectrofotometría, con una buena integridad evidenciada por las bandas de alto peso molecular en la electroforesis. La relación A260/280 (1,28) y la inhibición en el corte con EcoRI mostraron una baja pureza de la muestra, por la presencia de productos fenólicos, pigmentos y/o proteínas no eliminados en el proceso de extracción (Figura1).
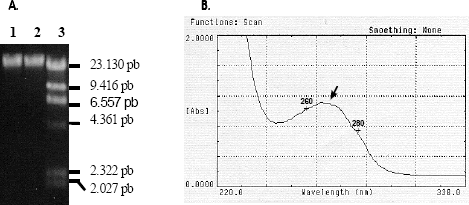
Figura 1. A. Electroforesis en gel de agarosa del ADN C. arabica cv. Caturra extraído con la técnica descrita por Chaparro (1993), en gel de 0,8 % con bromuro de etidio. Carril 1: ADN total. Carril 2: ADN total cortado con EcoRI. Carril 3, Marcador de peso molecular λ HindIII. B. Curva espectrofotométrica del ADN. La flecha muestra el pico máximo de obsorción hacia 270 nm.
Análisis de la cromatografía de exclusión molecular. Se obtuvieron tres grupos de fracciones a, b y c, cada una de ellas con moléculas diferentes evidenciadas por espectrofotometría y electroforesis (Tabla 1, Figura 2).
Tabla 1. Evaluación por espectrofotometría de las fracciones obtenidas de la cromatografía de exclusión molecular de C. arabica cv. Caturra.
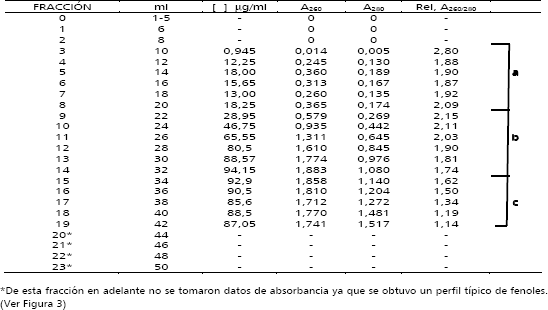
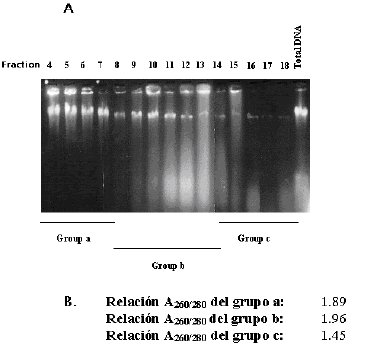
Figura 2. Comportamiento electroforético y espectrofotométrico de las muestras de ADN de C. arabica cv. Colombia, extraído por la técnica de Chaparro (1993) y purificado a través de columna de exclusión molecular Sephacryl S1000. A. Gel de agarosa al 0,8 % con bromuro de etidio de las fracciones eluidas de la columna de exclusión molecular. b. Relaciones 260/280 nm promedio de los grupos de fracciones a, b y c.
El componente de mayor tamaño molecular de la muestra a purificar fue el ADN total, el cual comenzó a eluir en la fracción 3, continuando su elución hasta la fracción 7, a este se le denominó grupo a. Su calidad determinada por la relación A260/280 fue muy buena (1,88-1,92) con un promedio de 1,89. La curva del espectro de absorción entre 220-320 nm, tuvo el comportamiento típico para ácidos nucleicos, con un pico máximo cerca a 260 nm, el cual aumenta en magnitud a medida que se incrementa el volumen de elución (Tabla 1). Este grupo fue elegido como muestra óptima para continuar el trabajo. La electroforesis del grupo b de fracciones correspondientes a las fracciones 8 a 13, muestra bandas de alto peso molecular y barridos a lo largo de los carriles, posiblemente debido a la presencia de ARN degradado. Las relaciones A260/280 mostraron valores aparentemente normales (entre 1,81 y 2,15), con un promedio de 1,96 y la curva del espectro de absorción entre 220-320 nm fue normal. Aunque la pureza de la muestra fue buena y aun había ADN total íntegro, estas fracciones fueron rechazadas por la presencia de ARN.
En el grupo c correspondiente a las fracciones 14 a 18, la relación A260/280 comienza a bajar a medida que aumenta el volumen de elución, obteniéndose valores entre 1,14 y 1,7, con un promedio de 1,45. Hay deformación de la curva del espectro de absorbancia entre 220-320 nm, con aparición de picos de absorbancia hacia 270 nm que corresponden probablemente a fenoles y en algunos casos a 280 nm correspondientes a proteínas (Figura 3). Además en la electroforesis desaparece casi totalmente la presencia de bandas y barridos lo que muestra ausencia de ácidos nucleicos pero presencia de otras moléculas que absorben en este rango de longitud de onda.
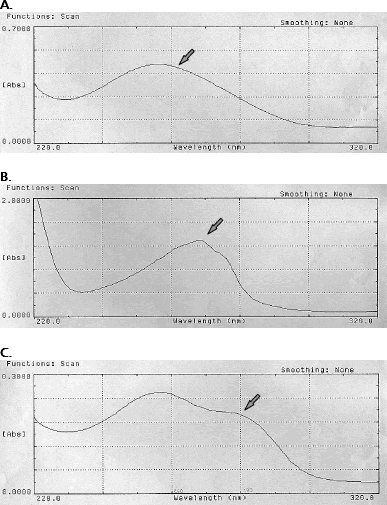
Figura 3. Espectros de absorción. A. ADN total puro B. ADN total contaminado con fenoles. C. ADN total contaminado con proteínas. Las flechas muestran la ubicación de los picos de absorción predominantes.
Mas allá de la fracción 19, la curva toma el aspecto de los productos fenólicos y los valores de absorbancia disminuyen hasta llegar casi a cero.
Análisis del ADN purificado. La electroforesis del ADN total de todos los genotipos extraídos y purificados, muestra bandas de alto peso molecular sin degradación que al ser tratadas con la enzima de restricción EcoRI producen barridos de mediano peso molecular, típicos de la acción de esta enzima (Figura 4 A).
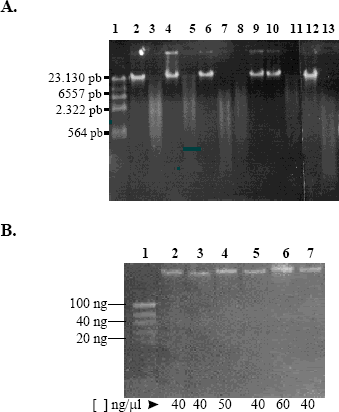
Figura 4. Análisis del ADN extraído y purificado de todos los genotipos. A. Análisis del ADN sin cortar y cortado con EcoRI en gel de agarosa al 0.8% con bromuro de etidio. Carril 1: λ HindIII. Carril 2: C. arabica cv. Caturra. Carril 3: C. arabica cv. Caturra EcoRI. Carril 4: C. arabica cv. Colombia. Carril 5: C. arabica cv. Colombia EcoRI. Carril 6: Hibrido de Timor. Carril7: Hibrido de Timor EcoRI. Carril 8: C. canephora EcoRI. Carril 9: C. canephora. Carril 10: C. congensis. Carril 11: C. congensis EcoRI Carril 12: C. eugenioides Carril 13: C. eugenioides EcoRI B. Evaluación de la concentración de las muestras por electroforesis en gel de agarosa al 0.8% con bromuro de etidio.Carril 1: DNA Mass Ladder. Carril2: C. arabica cv. Caturra. Carril 3: C. arabica cv. Colombia. Carril 4: Hibrido de Timor. Carril 5: C. canephora. Carril 6: C. congensis. Carril 7: C. eugenioides.
Con el marcador de tamaño molecular se encontró que la concentración de ADN obtenido de cada genotipo estaba entre 20-80 ng ml-1, concentración aparentemente baja (Figura 4 B). Sin embargo, el volumen de dilución del ADN nunca fue menor de 200 ml, lo cual proporciona material suficiente para realizar más de 200 reacciones de PCR por muestra. Al evaluar la calidad de los materiales aislados por espectrofotometría, se encontró que las relaciones A260/280 fueron óptimas mostrando la alta pureza del material obtenido de la columna (Tabla 2).
Tabla 2. Análisis espectrofotométrico del ADN en los diferentes genotipos de café aislados.

De estos resultados se pude concluir que la cromatografía de exclusión molecular utilizando Sephacryl S-1000, es una buena alternativa para la purificación de ADN originado de genotipos con alto contenido de metabolitos secundarios como es el caso del género Coffea, ya que las moléculas presentes en la muestra se separan en fracciones discriminantes, obteniendo un ADN de excelente calidad.
Este trabajo fue cofinanciado por Colciencias y la Federación Nacional de Cafeteros de Colombia, dentro del marco del proyecto Estudio de la Actividad Fotosintética de Hojas y Frutos de diferentes Especies de Café Coffea sp, código:2251-07-002-93.
Bookjans, G., Stumman, B. M. and Henningsen, K. W. 1984. Preparation of chloroplast DNA from pea plastides isolated in a medium of high ionic strenght. En: Analytical Biochemistry. Vol. 141; no.1; p. 244-247. [ Links ]
Couch, J. A. and Fritz, P. 1990. Isolation of DNA from plants high in polyphenolics. En: Plant Molecular Biology Reporter. Vol. 8, no.1; p. 8-12. [ Links ]
Chaparro, K. 1993. Contribución al estudio del genoma del cafeto, construcción de una biblioteca genómica de Coffea arabica L. variedad caturra. Bogotá. 82 h. Tesis Maestría en Ciencias Agrarias. Universidad Nacional de Colombia. Facultad de Agronomía. [ Links ]
Frischauf, A. M. 1987. Digestion of ADN: size fractionation. p. 183-189 En: Berger, Shelby L. and Kimmel, Alan R. Guide to molecular cloning thechniques. En: Methods in Enzymology. Vol. 152. 818 p. [ Links ]
Fuchs, E. and Blakesley, R. 1983. Guide of the use of type II restriction endonucleases. Recombinant DNA. Part B. En: Methods in Enzymology. Vol. 100; p. 3-38. [ Links ]
Guillemaut, P. and Drovard, L. M. 1992. Isolation plant DNA: a fast inexpensive and reliable method. En: Plant Molecular Biology Reporter. Vol. 10, no.1; p. 60-65. [ Links ]
Kanazawa, A. and Tsutsumi, N. 1992. Extraction of restrictable DNA from of the genus Nelumbo. En: Plant Molecular Biology Reporter. Vol. 10, no. 4; p. 316-318. [ Links ]
Palmer, J. D. and Shields, C. R. 1984. Tripartite structure of Brassica campestris mitochondrial genome. En: Nature. Vol. 307; p. 437-440. [ Links ]
Sambroock, J., Fritsch, E. F. and Maniatis, T. 1989. Molecular cloning: a laboratory manual. 3 ed. New York: Cold Spring Harbor Laboratory. 2300 p. [ Links ]














