Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Facultad Nacional de Agronomía Medellín
Print version ISSN 0304-2847
Rev. Fac. Nac. Agron. Medellín vol.61 no.2 Medellín July/Dec. 2008
IDENTIFICACIÓN DE GENES CANDIDATOS DE PATOGENICIDAD EN LA INTERACCIÓN DE LA CEPA CENICAFE 9501 CON EL NEMÁTODO DEL NUDO RADICAL Meloidogyne spp.
IDENTIFICATION OF PATHOGENIC CANDIDATES GENES IN THE INTERACTION OF THE CENICAFE 9501 STRAIN WITH THE ROOT KNOT NEMATODE Meloidogyne spp.
Nadya Lorena Cardona Bustos1, Jhon Fredy Betancur Pérez2, Luis Fernando Rivera Serna3 y Alvaro Gaitán Bustamante4
1 Docente-Investigadora. Universidad de Antioquia. Grupo Ecoepidemiología y Control Biológico. Instituto de Biología. A.A. 1226, Medellín, Colombia. <nadyaloren@gmail.com>
2 Investigador Asociado. Disciplina de Fitopatología. Centro Internacional de Investigaciones del Café, CENICAFE. Sede Planalto, km. 4 vía Chinchiná, Caldas, Colombia. <fredy.betancur@gmail.com>
3 Investigador Asociado. Disciplina de Fitopatología. Centro Internacional de Investigaciones del Café, CENICAFE. Sede Planalto, km. 4 vía Chinchiná, Caldas, Colombia. <fernando.rivera@cafedecolombia.com>
4 Investigador Científico. Disciplina de Fitopatología. Centro Internacional de Investigaciones del Café, CENICAFE. Sede Planalto, km. 4 vía Chinchiná, Caldas, Colombia. <alvaro.gaitan@cafedecolombia.com>
Recibido: Agosto 28 de 2007; aceptado: Septiembre 26 de 2008.
Resumen. En 1997 se registró por primera vez el aislamiento de un hongo Hyphomycete de suelos de Chinchiná (Caldas, Colombia) atacando huevos y otros estadios biológicos de las especies de nematodos fitoparásitos Meloidogyne incognita y M. javanica provenientes de lotes comerciales con café. Debido a la imposibilidad de clasificarlo taxonómicamente en los géneros actuales, este aislamiento se ha denominado temporalmente como CENICAFE 9501. Dado su potencial como biocontrolador, se propuso identificar genes candidatos involucrados en el proceso de patogenicidad de huevos de Meloidogyne. Con este fin se construyeron librerías diferenciales mediante el método de hibridación sustractiva. La secuenciación de 188 clones obtenidos permitió identificar 80 unigenes, de los cuales el mayor porcentaje correspondió a secuencias sin homología (32%), seguidas por genes candidatos a funciones de patogénesis (22%), transporte celular (17%), síntesis de proteínas (11%) y en menor proporción aquellos involucrados con transcripción y metabolismo primario (18%). Dentro de aquellos genes que contienen marcos de lectura con homología a proteínas que intervienen en la patogenicidad se encuentran una peptidasa, un receptor para sitios de ubiquitinación, una deubiquinasa, una ubiquinona oxidoreductasa, proteína relacionada con la degradación de pared celular, glicosil hidrolasa e hidroxilasa de ácidos grasos, asi como una serin proteasa. Se hace necesaria la validación de las funciones putativas de estos genes candidatos con el fin de incrementar el conocimiento básico de la fisiología de este hongo con potencial biorregulador.
Palabras claves: Hongo nematófago, genes de hongos biocontroladores, genes de patogenicidad, serín proteasas, interacción hongo: Meloidogyne.
Abstract. In 1997 it was reported for the first time from soil isolation of a Hyphomycete fungus from Chinchiná ( Caldas, Colombia ), attacking eggs and other biological stages of the plant parasite nematodes Meloidogyne incognita and M. javanica, from commercial coffee plots. Due to the impossibility to classify it taxonomically under current genera, this isolate has been labeled in the mean time as CENICAFE 9501. Given its potential as biological control agent, the identification of canditate genes involved in pathogenicity processes on Meloidogyne eggs was proposed. With this purpose, differential libraries were constructed using the subtractive hybridization method. Sequencing of 188 clones allowed the identification of 80 unigenes, with the highest percentage corresponding to sequences without homology (32%), followed by candidate genes for pathogenesis (22%), cellular transport (17%), protein synthesis (11%) and in lesser degree those involved with transcription and primary metabolism (18%). Among those genes with reading frames showing homology to proteins involved in pathogenicity can be found a peptidase, a ubiquitination receptor, a deubiquitinase, a ubiquinone oxydoreductase, a protein related to the degradation of the cell wall, a glycosyl hydrolase and fatty acid hydrolase, as well as a serine protease. A validation of the putative function of these genes is neccesary in order to increase the basic knowledge of the physiology of this fungus with bioregulation potential.
Key words: Nematophagous fungi, fungi biocontrol genes, pathogenicity genes, serín proteases, fungi: Meloidogyne Interactions.
Los nemátodos del género Meloidogyne, atacan a más de 2000 especies de plantas, incluyendo a la mayoría de las especies cultivadas. Afectan su crecimiento al debilitar las puntas de la raíz y al inhibir su desarrollo o estimular la formación excesiva de raíces, así como inducir la formación de hiperplasia con la generación de células gigantes. Cuando las plantas susceptibles son infectadas en estados de plántula, las pérdidas son considerables, y pueden dar lugar a la destrucción total de los cultivos. En plantas adultas, pueden disminuir severamente la producción (Stirling, 1991; Sirohi, et al., 2005). Las pérdidas económicas ocasionadas por los nemátodos fitoparásitos, han sido estudiadas ampliamente en otros países; en Estados Unidos por ejemplo, se presentan pérdidas anuales del orden de 78 billones de dólares (Sirohi, et al., 2005), de las que aproximadamente el 70% son ocasionadas por los nemátodos del nudo radical (Kerry y Brown,1987; Stirling, 1991). En Colombia, algunos de los cultivos afectados por este parásito son flores, pithaya, tomate de árbol, maracuyá, granadilla, y café entre otros (Bobadilla et al., 1999; Giraldo y Leguizamón, 1999; Montoya, 1999; Munera, 1999; Price et al., 1997).
En Colombia los nemátodos del género Meloidogyne incognita-javanica se constituyen en uno de los principales problemas sanitarios durante las primeras etapas del cultivo del café. Leguizamón (1994), demostró como en la medida en que se aumentó el porcentaje de infección de éste género sobre plántulas de café, disminuyó el número de hojas formadas, los pesos secos de las raíces y de la parte aérea de las plántulas inoculadas. Algunas de las medidas de control de nemátodos consisten en prácticas culturales que incluyen la solarización, el retiro de material vegetal contaminado del campo, así como la vaporización del suelo, o medidas de control genético, como sería la utilización de material resistente, que no está disponible para todos los tipos de cultivos. Otra de las alternativas más comunes para el control es el uso de nematicidas, pero debido a la alta tasa de desarrollo y a la fecundidad de los nemátodos, las poblaciones son difícilmente controladas por estos productos. Así mismo se han presentado dificultades en el control químico, debido a la composición de los huevos, a su capacidad de sobrevivir durante periodos largos en residuos de cosecha, en donde los nematicidas difícilmente penetran. Uno de los principales inconvenientes del control químico es la residualidad de nematicidas en plantas y aguas subterráneas, lo cual los hace inadecuados para incluirlos dentro de programas de agricultura sostenible (Davies et al., 1994; Jatala, 1980; Lumsden et al., 1995).
Debido a las dificultades de su control, en Colombia se ha trabajado desde hace algunos años en el empleo de biocontroladores para el manejo de nemátodos. En la zona cafetera existe una gran biodiversidad y es así como se han realizado investigaciones para el manejo de nemátodos en café, con microorganismos aislados de estas regiones. Se han encontrado resultados promisorios con micorrizas, los cuales deben ser aplicadas al momento del transplante de la plántula (Leguizamón, 1995); otros microorganismos incluyen los hongos Paecilomyces lilacinus, Beauveria bassiana, Metarhizium anisopliae y Verticillium chlamydosporium, que han sido utilizados principalmente en el control de huevos de Meloidogyne spp en almácigo (Giraldo et al., 1996.; Hincapié et al., 1998, 1999.; Leguizamón y Padilla, 2001). Dentro de los estudios tendientes a la búsqueda de biocontroladores promisorios, en la década de los 90s un hongo Hyphomycete se aisló de suelos de Chinchiná (Caldas) atacando huevos y otros estadios biológicos de las especies de nemátodos fitoparásitos M. incognita y M. javanica provenientes de lotes comerciales con café (Cardona y Leguizamón 1997). Inicialmente, el IMI (Internacional Mycologycal Institute) insinuó que se trataba de un hongo que hasta ese momento no había sido descrito. Morfológicamente el hongo presenta tres tipos de estructuras de reproducción asexual denominadas Tipo I, Tipo II y conidias en sentido estricto, y se le denominó cepa CENICAFE 9501 (Cardona y Leguizamón 1997.)
Teniendo en cuenta que este microorganismo es un recurso de la biodiversidad de la zona cafetera, con potencial de ser incorporado en programas de manejo integrado de nemátodos, es importante adelantar trabajos de caracterización morfológica y molecular con el propósito de explorar su taxonomía y biología. El objetivo de este estudio consistió en identificar genes expresados durante la interacción In vitro de la cepa CENICAFE 9501 con huevos de M. incognita y M. javanica, con el propósito de determinar similitudes con otros genes registrados como biocontroladores en las bases de datos, así como genes novedosos asociados a la función de patogenicidad en nemátodos.
MATERIALES Y MÉTODOS
Cultivo y reactivación del hongo. El estudio fue realizado con el aislamiento CENICAFE 9501 registrado en la Colección de Microorganismos de CENICAFÉ, el cual se obtuvo en el año 1995 a partir de estadios de nemátodos de las especies M. incognita y M. javanica afectando raíces de café en el departamento de Risaralda (Cardona y Leguizamón, 1997). Debido a que el hongo se mantuvo por un periodo prolongado en medio de cultivo sintético y almacenado en nevera, fue necesario realizar su reactivación previo montaje de pruebas que permitieran estudiar su expresión. La reactivación de la cepa a partir de su almacenamiento bajo condiciones de baja temperatura se realizó mediante crecimiento en medio de cultivo e infección en huevos de Meloidogyne spp. extraídos de raíces de plántulas de café afectadas por el nemátodo. El hongo creció a partir de porciones de micelio de la cepa original, las cuales se sembraron en Medio Completo (MC: Na2HPO4 1,4 g; KCl 1,0 g; MgSO4.7H2O 0,6 g; NH4NO3 0,7 g; Extracto de levadura 5,0 g; Glucosa; 10,0 g; Agar 18 g, Agua Destilada 1 L) por dos semanas.
A partir de las colonias desarrolladas se prepararon suspensiones acuosas de esporas (1x107conidias mL-1) para realizar las pruebas de infectividad sobre huevos de Meloidogyne spp. La prueba de infectividad consistió en la adición de 5 mL de la suspensión de esporas a cajas petri de 5 cm de diámetro, a las cuales se les había depositado previamente 1 mL de una solución de que contenía 200 huevos y estados juveniles (J2) de M. incognita-javánica, obtenidos de plántulas de café, suministrados por CENICAFE. La mezcla se agitó suavemente hasta homogenizar y luego se incubó a temperatura ambiente ( 23 a 28 ºC) por una semana. Se realizaron observaciones al microscopio cada dos días. La infectividad del hongo sobre los huevos y juveniles del nemátodo se determinó mediante observación microscópica como el porcentaje de estructuras del nemátodo (huevos o larvas) con daño en estos o en el embrión por presencia de micelio.
Multiplicación de Meloidogyne en material vegetal. Se incrementaron los nemátodos (M. incógnita–javanica) en material vegetal bajo condiciones de invernadero. Para esta multiplicación, y posterior obtención de huevos, se utilizaron plántulas de tomate Chonto variedad Santa Cruz, crecidas bajo condiciones de invernadero a una temperatura promedio de 23 ºC, humedad relativa aproximada del 60%, con luz natural. La población inicial para la multiplicación fue suministrada por el laboratorio de Fitopatología de CENICAFE, el cual consistió en estadios de Meloidogyne desarrollados en plántulas de café variedad Colombia de 6 meses de edad. La extracción de los nemátodos y la aplicación para su multiplicación en plantas de tomate, se realizó siguiendo los procedimientos descritos por Leguizamón (1994). Plantas de Impatiens sp. con presencia de abundantes nodulaciones en las raíces fueron colectadas en la zona cafetera, vereda Plan Alto, en la parcela La Guamera en predios de CENICAFE (Chinchiná, Caldas) con el fin de extraer masas de huevos y aumentar la concentración de estos en las pruebas de inducción de genes en la interacción hongo-nemátodo. Para identificar la especie de Meloidogyne presente en las plantas de Impatiens sp., se realizaron cortes perineales de las hembras extraídas de la raíz.
Obtención de huevos de Meloidogyne spp. Los huevos se obtuvieron a partir de las raíces mediante un lavado inicial con NaOCl al 10%, seguido de agitación manual durante 5–10 minutos, y finalmente se hizo atravesar un juego de tamices de 16, 32, 60, 150, 250 y 500 mesh. El contenido de este último tamiz fue colectado por medio de lavados con agua corriente. Para la separación y limpieza de los huevos del resto de residuos de suelo y raíces, se hizo una centrifugación con glucosa al 50% a una velocidad de 1.500 rpm por 20 minutos. El sobrenadante se filtró a través de un tamiz de 500 mesh con el fin de recuperar los huevos presentes. Los huevos fueron entonces sometidos a varios lavados sucesivos con agua destilada estéril, con el fin de retirar los residuos de azúcar y se almacenaron en recipientes limpios a 4 °C hasta el momento de su utilización.
Preparación del inóculo de la cepa CENICAFE 9501 para la prueba de hibridación sustractiva. Todo el material utilizado para la realización de esta fase del estudio, específicamente para la obtención de ARN total y ARNm, se trató con agua DEPC al 0,1%, con el fin de eliminar la presencia de ARNasas que pudieran interferir con el proceso. Para la multiplicación de la cepa CENICAFE 9501, se utilizó el medio SDB (Saboureau Dextrosa Broth; Glucosa 40 gL-1, Peptona 10 gL-1), a un pH de 5,5. El medio fue distribuido en erlemeyers de 250 ml, a cada erlemeyer con medio se le adicionó un disco de 5 mm de agar con micelio del hongo crecido por 10 días en el medio Agar Quitina (Villa, 2006). Posteriormente, los cultivos fueron incubados bajo condiciones de luz y agitación constante (150 rpm) por cuatro días. Cumplido el tiempo de incubación, se tomó una alícuota del cultivo del hongo para ser observada al microscopio y determinar la presencia o no de estructuras de Tipo II. Los cultivos que presentaron estas estructuras fueron transferidos a tubos Falcon de 50 ml y luego se centrifugaron a 2500 rpm por 20 minutos. El sobrenadante se descartó y el pellet se sometió a tres lavados sucesivos con agua destilada estéril, con el fin de eliminar los residuos de medio de cultivo. A continuación, se realizó una observación al microscopio del contenido del pellet con el fin de garantizar la pureza de las estructuras presentes y la proporción de éstas con respecto a las conidias propiamente dichas del hongo.
Inducción de genes en la cepa CENICAFE 9501 en su interacción sobre huevos de M. incognita-javanica. Para estudiar la inducción de genes se hicieron pruebas de patogenicidad de la Cepa CENICAFE 9501 sobre huevos de M. incognita-javánica con suspensiones acuosas de estructuras de Tipo II ajustadas a una concentración final de 2x107 estructuras/ml de medio SDB. La concentración se escogió teniendo como base los resultados de los estudios preliminares realizados en el Laboratorio de Sanidad Vegetal de la Universidad Nacional de Colombia, Sede Medellín para determinar la DMI50 (Villa, 2006). Adicionalmente, se preparó bajo condiciones asépticas una solución acuosa de huevos de Meloidogyne spp ajustada a una concentración final de 20.000 huevos mL-1, la cual se calculó teniendo en cuenta las pruebas de patogenicidad realizadas por Cardona y Leguizamón (1997). La suspensión resultante se adicionó luego en un volumen de 10 ml a un erlemeyer que contenía los 25 ml de medio con la concentración de las estructuras Tipo II del hongo.
Los huevos del nemátodo utilizados para esta prueba se esterilizaron con vapor caliente en un autoclave a 15 lb de presión por 15 minutos, corroborando su integridad mediante observación al microscopio. La esterilización de huevos permitió garantizar que los genes expresados provenían de uno solo de los organismos presentes en la interacción (huevos vs. hongo), considerando que al momento de iniciar este estudio, no existía un registro de base de datos con genes del género Meloidogyne. Después de los tiempos de incubación se tomaron de manera aséptica alícuotas de la suspensión hongo-huevos de nemátodo con el fin de ser observados bajo microscopio (40X) y establecer la presencia del hongo parasitando los huevos, de acuerdo con metodología establecida previamente por Cardona y leguizamon (1997).
Se realizaron 4 tratamientos así: huevos más el hongo interactuando por 24 horas (Inducido 24) y por 48 horas (Inducido 48), y hongo sin adición de huevos (No inducido 24 y No inducido 48). Cada tratamiento se hizo por duplicado. Los erlemeyer que contenían los tratamientos se colocaron en agitación constante de 150 rpm a 27 °C durante 24 y 48 horas. La extracción de ARN total se realizó a partir de 100 mg de las muestras incubadas por 24 y 48 horas, para los dos tratamientos. Antes de realizar la extracción, de cada muestra se tomó una alícuota y se observó al microscopio para verificar la ausencia de contaminación bacteriana y la presencia de huevos infectados por el hongo.
Para la extracción se siguió el procedimiento descrito para el kit RNAgents Total RNA Isolation System (Promega) y Polyatract mRNA isolation Systems (Promega). Para comprobar la presencia de ARN total y ARNm en la muestra, se tomaron 5 µl del producto de la extracción y se visualizaron por electroforesis en un gel de agarosa al 2% desnaturalizado a 65 °C. Para comparar el peso de las bandas, se utilizó el marcador ΦX174 DNA/Hae III Markers (Promega). El ARNm obtenido a partir del ARN total de cada muestra, se marcó de acuerdo con la composición de cada uno de los tratamientos.
Síntesis de primera y segunda cadena de cDNA. Para la síntesis del cDNA (ss) se utilizó el kit Advantage RT- para PCR (BD Biosciences). Para la síntesis de la segunda cadena, se utilizó el suguiente protocolo de síntesis manual: a 20,5 µl de cada ssDNA se agregaron 9,5 µl de una mezcla conformada por 2,5 µl de buffer 10X, 1 µl de hexámeros, 2,5 µl de dNTPs, 3,0 µl de MgCl2 y 0,5 µl de taq polimerasa (Promega), para un volumen total de 30 µl por reacción. La reacción de PCR fue llevada a cabo por 35 ciclos, siguiendo el esquema: un ciclo inicial de desnaturalización a 90 °C por 5 minutos, seguido de 34 ciclos de desnaturalización a 92 °C por 20 segundos, alineamiento a 35 °C por 15 segundos, extensión a 72 °C por 2 minutos y 30 segundos y una extensión final a 72 °C por 5 minutos. Los productos fueron observados en un gel de agarosa al 2% en buffer TAE. Como patrón de comparación de bandas, se utilizó el marcador ΦX174 DNA/Hae III Markers (Promega)(Cardona, 2007).
Hibridación sustractiva. Para la hibridación sustractiva se siguió la metodología descrita por Clontech (Cat.No.K1804-1). Con los productos obtenidos a partir de la hibridación sustractiva, se procedió a realizar la amplificación por PCR de las muestras sustraídas y no sustraídas. Los tratamientos que se amplificaron fueron (tester vs. driver): SA= Sustraído A (Inducido 24 vs. No Inducido 24), SB= Sustraído B (Inducido 48 vs. No Inducido 48), UA= No sustraído A (No inducido 24), UB= No sustraído B (No inducido 48).
Se realizó una primera PCR utilizando 2 µl de cada una de las muestras y 23 µl de los componentes de la reacción en las siguientes proporciones: 16 µl de H20 ultra pura, 2,5 µl de Buffer PCR 10X, 0,5 µl de mezcla de dNTPs ( 10 mM), 1 µl de cebador 1(10 µM), 0,5 µl Taq (Promega), 2,5 µl de MgCl2 (2,5 mM), para un volumen final de 25 µl. La reacción de PCR se llevó a cabo de acuerdo al siguiente esquema: un ciclo inicial de precalentamiento a 75 ºC por 5 minutos, seguido de 27 ciclos de desnaturalización a 94 ºC por 25 segundos, alineamiento a 66 ºC por 30 segundos y extensión a 71 ºC por 90 segundos. Los productos de la amplificación fueron diluidos utilizando 3 µl de reacción primaria y 24 µl de buffer de PCR (10X). La dilución resultante se utilizó como muestra para la segunda reacción de PCR.
El producto de la amplificación de la PCR1 a la dilución indicada se utilizó como base para la PCR2. A 1 µl de cada muestra diluida se le adicionaron 24 µl de una mezcla compuesta por 16 µl de H2O, 2,5 µl de buffer PCR (10X) (Promega), 1 µl de cebador anidado 1 (10 mM), 1 µl de cebador anidado 2 (10 mM), 0,5 µl de mezcla de dNTPs (10 mM), 0,5 µl de Taq polimerasa (Promega), 2,5 µl de MgCL2 (2,5 mM), para un total de 25 µl por reacción. Con los productos obtenidos en la PCR2, se procedió a realizar la clonación (Cardona, 2007).
Clonación de fragmentos y secuenciación. El proceso de ligación fue realizado utilizando el kit PGEM-TEasy System I (Promega), y con células competentes JM109 (Promega). La selección de colonias recombinantes se realizó en medio LB (Bactopeptona 10g /L, extracto de levadura 5 gL-1, NaCL 5 gL-1) más ampicilina (100 µg mL-1), IPTG (0,5 mM) y X-Gal (80 µg mL-1). Los cultivos fueron luego incubados a 37 ºC durante 16 horas. Se escogieron al azar un número similar de colonias de cada una de las librerías para secuenciar sus insertos. Para comprobar la presencia del inserto, las muestras se analizaron por colonia PCR (Lee y Cooper, 1995), separando los fragmentos amplificados por electroforesis en geles de agarosa al 2% en buffer TBE 1X. Con las colonias seleccionadas se realizaron minipreparaciones de plásmidos de acuerdo con la metodología de Quiagen (Qiaprep-Miniprep), los cuales fueron enviados para secuenciación a Macrogen (Corea) (Cardona, 2007).
Análisis de la información. Con las secuencias obtenidas se realizó un análisis de su calidad, así como la limpieza de vectores y adaptadores, utilizando el Programa Codon Code Aligner (CodonCode Aligner V1.5.1 CodonCode Corporation, Tokyo Japan). No se incluyeron dentro de los análisis secuencias que presentaron alguno de los siguientes casos: el inserto correspondía a secuencias de adaptadores, carecían de inserto, o al final de la edición los insertos presentaban menos de 100 nucleótidos. Las lecturas de secuenciación tenían que presentar valores de Phred por encima de 30 (Primrose, 2001). Una vez procesados los cromatogramas, se construyeron secuencias consenso correspondientes a Contigs (grupos de secuencias alineadas que representan un solo transcrito) o a Singletons (transcritos representados por una sola secuencia). Con este juego de unigenes se realizó la búsqueda con Blastn y Blastx utilizando la base de datos no redundante (nr). Para determinar la función y la similitud con dominios proteínicos, se hicieron búsquedas con el Programa InterproScan, y la base de datos CDD, cuyo enlace se originó a partir de la base de datos del Genbank. Se consideraron como similares aquellas secuencias que presentaron un valor esperado e menor o igual a 10-2 (Guilleroux y Osbourn, 2004). Con los datos obtenidos se determinó el porcentaje de secuencias que cumplían con las condiciones establecidas de calidad y longitud, al igual que los porcentajes de secuencias contaminantes tales como ADNr y Colifagos, y secuencias que no presentaban similitud con las bases de datos (Cardona, 2007).
RESULTADOS Y DISCUSIÓN
Con la metodología establecida fue posible lograr la expresión de genes, para evaluar la interacción de la Cepa CENICAFE 9501 con huevos de M. incógnita-javanica. Con el medio de cultivo utilizado se obtuvo una proporción del 80% de estructuras de Tipo II en 4 días, con las cuales se realizaron las pruebas de patogenicidad para la hibridación sustractiva. Al esterilizar los huevos de Meloidogyne por medio de vapor de agua sólo se obtuvieron al final estadios maduros de J2, próximos a eclosionar. Los estadios inmaduros no resistieron las condiciones estudiadas, de tal forma que los resultados sólo son aplicables a huevos con estadios maduros. A pesar de que los huevos se esterilizaron, se encontró la expresión de genes candidatos para continuar con posteriores estudios de expresión de genes en otros estadios de madurez de los huevos de Meloidogyne spp. utilizando hongos biocontroladores. La observación de que el organismo blanco podría ser esterilizado para estudios de interacción con organismos blancos fue reportada por Góngora et al. (2003, 2005), quienes utilizaron materiales estériles de la broca del café, para estudios de la expresión de genes de B. bassiana, con resultados importantes.
Para las cuatro librerías construídas, una por cada tratamiento, se secuenciaron en total 188 clones, de los cuales se analizaron 155 (Se descartaron 33 secuencias). Como resultado del ensamblaje de los clones, se obtuvieron un total de 62 secuencias únicas (Singletons) y 18 representados por dos o mas clones (Contigs), para un total de 80 unigenes para la cepa CENICAFE 9501. Los singletons oscilaron entre 111 a 695 pb, mientras que los Contigs variaron en extensión entre 153 y 694 pb. Se obtuvo un mayor número de singletons en las librerías correspondientes a las 24 horas (34 secuencias en UA y 20 secuencias en SA) que a las 48 horas (4 de UB y 5 de SB).
Las librerías no sustraídas presentaron un número mayor de singletons con funciones más diversas si se comparan con las sustraídas. En la librería sustraída de 24 horas se presentó un porcentaje más alto de secuencias sin identidad (62%), cuando se compara con la no sustraída (31%). En esta última fue posible encontrar secuencias relacionadas con genes constitutivos como lo son aquellos que intervienen en el transporte celular, metabolismo de carbohidratos, y síntesis y metabolismo de proteínas, entre otros.
La anotación de los unigenes indicó que el mayor porcentaje corresponde a secuencias sin homología (32%), seguidas con un 22% por secuencias que pueden tener una función en la patogénesis, 17% relacionadas con transporte celular, 11% con síntesis de proteínas y en menor proporción (18%) aquellas relacionadas con la transcripción, metabolismo primario y ADN foráneo (Figura 1). En relación con los productos que intervienen en el metabolismo primario, se encontraron hidrolasas de carbohidratos, que corresponden a una Gliceraldehido-3-fosfato dehidrogenasa y una enolasa, que intervienen en el metabolismo de carbohidratos, las cuales se registraron igualmente por Ahren et al.,(2005) en el hongo Monacrosporium sp. sobre Caenorhabditis elegans.
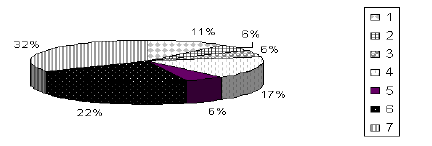
Figura 1. Proporción de contigs en huevos de Meloidogyne spp. de acuerdo con la función en las cuatro librerías. 1= Síntesis de proteínas; 2= Transcripción, 3= Metabolismo primario, 4= Transporte celular, 5= ADN foráneo, 6= Posible relación con la patogénesis, 7= Sin identidad.
En el grupo de los transportadores se obtuvieron secuencias relacionadas con receptores extracelulares de proteínas y aminoácidos, al igual que para el transporte de pequeños solutos. Por otra parte, se detectó una familia de receptores y moduladores de señales de calcio, elemento importante en diferentes funciones metabólicas celulares (Resultados no mostrados).
Dentro de las funciones de los contigs y singletons, se encontraron algunas secuencias que podrían estar relacionados con genes que intervienen en la patogenicidad. Dentro de ellos se tienen una peptidasa S8 (Clon UA33) (Tabla 1), un receptor para sitios de ubiquitinación (Clon SB40)(Tabla 1), una hidrolasa de Ubiquitina (Deubiquinasa) (Clon C11) (Tabla 2), Ubiquinona oxidoreductasa, proteína relacionada con la degradación de pared celular (Clon C16) (Tabla 2), Glicosil hidrolasa (Clon UA6)(Tabla 1) e Hidroxilasa de ácidos grasos (Clon UA39) (Tabla 1). Quizá una de las secuencias que reviste más interés en el estudio corresponde a un fragmento de una Serin proteasa (Clon UA 33)(Tabla 1), que ha mostrado en la búsqueda con Blast, identidades que han oscilado entre el 50 y el 65% con géneros como Tolypocladium, Metharizium, (Joshi et al., 1997), Lecaniciliium, Beauveria y Paecilomyces, entre otros, los cuales han sido estudiados debido a sus características como organismos biocontroladores, (Joshi et al., 1995). Este grupo de enzimas, ya ha sido estudiado en hongos nematófagos, como Monacrosporium haptoglosum (Ahren et al., 2005; Siroi, et al.,2005), Monacrosporium melagosporum, cuyo gen fue clonado y caracterizado (Kanda, et al., 2008), al igual que ha sido mencionado en Arthrobotrys oligospora, (Ahman, 1996), Dactylella shizishanna, denominada como Ds1, capaz de degradar la cutícula de Penagrellus redivivus y un amplio número de proteínas (Wang et al., 2006).
Tabla 1. Anotación de algunos singletons producto de la hibridación sustractiva en la interacción la cepa CENICAFE 9501– huevos de Meloidogyne spp. (Cardona, 2007).
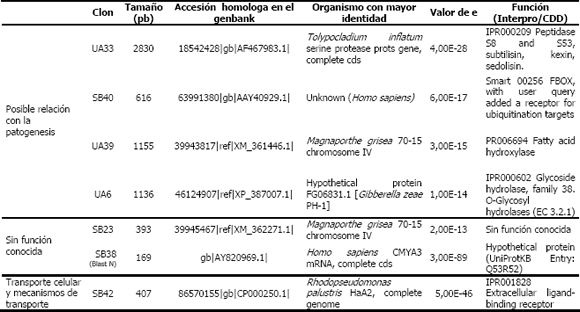
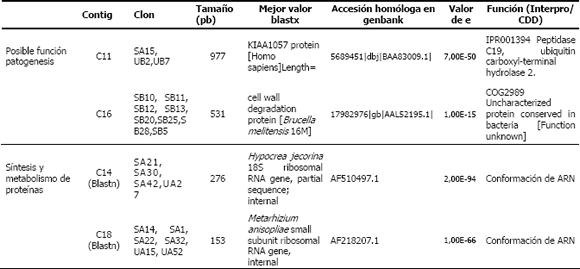
Tabla 2. Anotación de algunos contigs producto de la hibridación sustractiva en la interacción la cepa CENICAFE 9501– huevos de Meloidogyne spp.(Cardona, 2007)
Dado el bajo número de clones secuenciados en las librerías no sustraídas, los singletons pueden estar representando genes comunes en el metabolismo celular, y los contigs genes sobreexpresados en esas condiciones. La proporción de singletons en la librería no sustraída a las 24 horas (UA) refleja este hecho al presentar un bajo porcentaje de secuencias sin homología (31%), seguido por clones con homología pero sin función conocida (18%) y clones con homólogos conocidos (51%). Los singletons que podrían estar relacionados con la patogenicidad conformaron un 15% (Figura 2). En las librerías sustraídas, los singletons y los contigs representan genes diferencialmente expresados, específicos para una condición dada, en este caso interacción con huevos de nemátodos. En este sentido, en la librería sustraída a las 24 horas (SA), se observó como un alto porcentaje de singletonsque correspondió a secuencias sin homología significativa (62%) y sólo un 5% presentó homología con genes previamente relacionados con la patogenicidad (Figura 2).
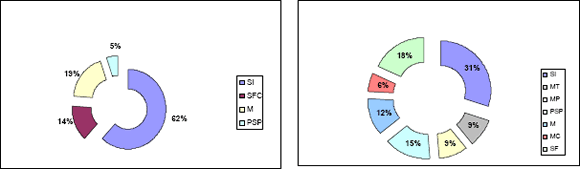
Figura 2. Proporción de singlets de librería sustraída (SA) y no sustraída (UA) a las 24 horas en huevos de Meloidogyne spp. SI= sin identidad, MT= mecanismos de transporte, M= misceláneas, MP= metabolismo de proteínas, PSP= posibles mecanismos de patogenicidad, MC= metabolismo de carbohidratos, SF- SFC= sin función conocida.
En los singletons obtenidos a partir de las librerías de 48 horas (SB y UB), solo se recuperaron 2 secuencias sin función conocida pero con homólogos presentes en Magnaporthe grisea y Homo sapiens (SB23, SB38), una secuencia posiblemente relacionada con la patogénesis (SB40), una relacionada con transporte celular-mecanismos de transporte (SB42) y una sin homología conocida (SB43, resultado no mostrado) (Tabla 1).
Por su parte, se ensamblaron 18 contigs con un rango entre 2 y 13 clones. Para 6 clones no fue posible identificar homologías significativas (C3-C17SH) mientras que 4 contigs, estuvieron asociados con patogénesis (Peptidasa, Ubiquitin Hidrolasa y Oxidasa, Proteína de degradación celular). En este sentido, es importante observar como los genes de patogenicidad que estuvieron diferencialmente expresados en las librerías sustraídas a las 24 horas, no se expresaron en forma diferencial a las 48 horas (Figura 3).
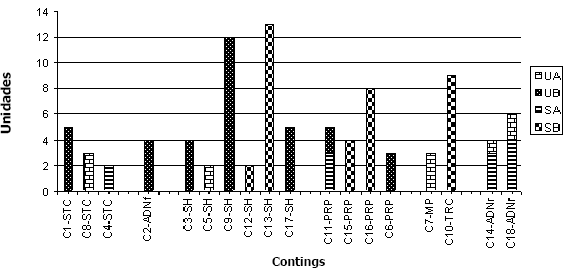
Figura 3. Agrupación de contigs en huevos de Meloidogyne spp., de acuerdo con su función en las librerías.
UA= No sustraída 24 horas; UB= No sustraída 48 horas, SA=Sustraída 24 horas; SB= Sustraída 48 horas. C1,8,4 STC= Sistema de transporte celular; C2 ADNf= ADN foráneo; C3,5,9,12,13,17 SH= Sin homología; C11, 15,16,6 PRP= Posible relación con la patogénesis; C7 MP=Metabolismo primario; C10 TRC= Transcripción; C14,18 ADNr= ADN ribosomal
En aquellos contigs con combinación de clones de las librerías, se podría evidenciar una alta transcripción de genes al mismo tiempo bajo las dos condiciones estudiadas (24-48 horas), como serían C14 y C18 (UA y SA), anotados en conformación de RNA; o en general, como el C11 (SA y UB), asociado a una ubiquitin carboxilasa. Para este tipo de genes es igualmente importante revisar la expresión relativa de los mismos, que con la muestra analizada no hace tan evidente su expresión diferencial. En la Figura 3, es posible observar la agrupación de contigs de acuerdo con su función en las librerías, de tal forma que los contigs agrupados como SH (Sin homología), incluyen el mayor número de clones de las 4 librerías procesadas. Con respecto a los contigs agrupados dentro del grupo identificado como Posible relación con la patogénesis (PRP), se puede observar como el contig 16 agrupa 8 secuencias (Tabla 2), que corresponden a una proteína de degradación de pared celular reportada en Brucella melitensis, cuya función es desconocida, la cual podría estar involucrada en procesos de degradación de los contenidos internos de Meloidogyne a las 48 horas. El hecho de haber encontrado en las librerías trabajadas un gran número de secuencias sin homología o sin identificar, denota la necesidad de continuar realizando investigaciones en este sentido, que permitan generar aún mas información para la comprensión de los procesos de interacción hongo–nemátodo.
De acuerdo con los resultados se observa que, a pesar de haber encontrado a las 48 horas algunos clones relacionados con la patogénesis, 24 horas es un tiempo adecuado para llevar a cabo estudios futuros, relacionados con la expresión diferencial en huevos maduros de Meloidogyne spp. Lo anterior, en virtud de que el huevo en este momento conserva las diferentes capas que podrían estimular la expresión de distintos tipos de genes, relacionados con la degradación del nemátodo. Villa (2006) encontró como a las 24 horas, en la concentración 2x107 conidias mL-1, se presentaba el 50% de la patogenicidad y el hongo podía ser observado tanto en la superficie como en el interior del huevo de Meloidogyne spp. Esto hace suponer que las actividades de transcripción y traducción son altas en esos momentos para producir enzimas relacionadas con la degradación de la pared del huevo, conformado por proteínas, carbohidratos y lípidos, pero al mismo tiempo para otras proteínas novedosas que se expresen diferencialmente y podrían estar relacionadas con patogenicidad, que corresponden al grupo del 62% de singletons sustraídos a las 24 horas sin homología conocida. En este mismo grupo pueden estar fragmentos poco conservados de genes conocidos, que por procesos evolutivos no presentan similitudes significativas. Para conocer secuencias mas representativas de estos genes es necesario utilizar técnicas como RACE (Chenchik et al, 1998) o Genome Walker (Siebert et al., 1995) que permitan dilucidar con certeza la novedad de las proteínas codificadas.
Villa (2006) indica que a las 48 horas se presentó un porcentaje alto de colonización de los huevos, cuyos contenidos son reemplazados por las hifas del hongo. Es de esperarse entonces que para este tiempo la transcripción de genes de patogenicidad haya disminuido debido al agotamiento del sustrato y a que fisiológicamente el hongo inicia procesos relacionados con su estado reproductivo de su ciclo de vida, que hace que comiencen a expresarse nuevos genes, distintos a la degradación del nemátodo. En este momento se presenta entonces una producción relativa mas abundante de un juego particular de proteínas, que se vería reflejada en una mayor proporción y riqueza (en número de clones) de contigs para este tiempo.
En el caso de Paecilomyces lilacinus, Bonants et al., (1995) purificaron mediante cromatografía una proteasa extracelular con capacidad para degradar la capa vitelínica de los huevos inmaduros de Meloidogyne hapla, pero sin afectar los estadios juveniles. Los autores concluyeron que la serin proteasa podría jugar un papel importante en la penetración del hongo a través de las envolturas del huevo de los estados juveniles. Estudios más recientes han demostrado como las serin protesasa de P. lilacinus pueden presentarse en tres formas estructurales, cuando su expresión es inducida con yema de huevo (Khan et al., 2003). Uno de los sitios potenciales de acción de las serin proteasas son las capas de colágeno de los estadios juveniles preparasíticos de los nemátodos. En este sentido, Koltai et al. (1997) caracterizaron un gen del colágeno (Mjcol-3) de M. incognita, presente tanto en huevos preparasíticos y en menor cantidad en estadíos J3-J4 y hembras jóvenes, cuya estructura básica de proteína predicha ( MJCOL-3) presentó un sitio de clivaje para la serin proteasa. Lo anterior corrobora la importancia que podría tener esta enzima en el proceso de patogénesis de la cepa CENICAFE 9501, en la interacción con huevos de distintos estadios, incluyendo huevos preparasíticos que contienen en su interior J2 próximos a emerger.
En relación con la influencia que puede tener el medio en la producción de serin proteasas, Ahman et al., (1996) observaron como su producción era estimulada cuando se sembraba el hongo con fragmentos de la cutícula de los nemátodos. Al igual que otros autores, observaron como su producción era inhibida por la presencia de glucosa en el medio. Joshi et al. (1995) realizaron una observación similar en B. bassiana, en donde la producción de proteasas se veía disminuída en SDB (Saboureaud Dextrosa Agar). Lo anterior podría explicar la baja cantidad de clones obtenidos con la secuencia de serín proteasa, a pesar de la alta concentración de huevos puestos en contacto con la cepa CENICAFE 9501. El medio utilizado contenía dextrosa, lo cual pudo inhibir la expresión de los genes. Para futuros ensayos sería conveniente probar nuevos medios que permitieran el desarrollo del hongo, cuya dextrosa sea reemplazada por otro tipo de azúcar metabolizable por el hongo y que no inhiba la expresión de este tipo de genes.
Teniendo en cuenta la presencia de serin proteasas en la cepa CENICAFE 9501, para futuros estudios sería necesario realizar amplificaciones con primers específicos probados para otros hongos nematófagos tales como el P307 y el P308, que fueron utilizados para la amplificación de genes de serin proteasa en Monacrosporium haptotylum (Ahren et al., 2005). Por otra parte, conociendo que la serin proteasa fue detectada a las 24 horas y que coincide con la TL50 a una concentración de 2x107 estructuras de Tipo II/ml (Villa, 2006), la cuantificación de los niveles de transcripción del gen por medio de Q-PCR a las 24 y 48 horas es importante para validar las hipótesis relacionadas con su función.
De otro lado, se hallaron genes registrados por Ahren et al., (2005) cuyo destino es la Ubiquitina. Ésta es una proteína pequeña que solo se encuentra en organismos eucarióticos, altamente conservada y que funciona como marcador para la acción de enzimas líticas en los mecanismos de degradación de proteínas. Aquellas proteínas que deben ser degradadas, tienen que conjugarse primero con la Ubiquitina. Una vez cumplida su función, la Ubiquitina es liberada de la unión con la proteína objetivo por medio de proteasas conocidas como deubiquinasas. Estas proteasas se han clasificado en clanes y subdivididas en familias, con base en la arquitectura de su sitio catalítico. Dentro de ellas están las pertenecientes al Clan A, que contiene, entre otras, a la familia de las peptidasas específicas para ubiquitina, como las homólogas a C12 y C19. Esta última es una peptidasa intracelular que remueve la Ubiquitina de péptidos por medio de clivaje, con el fin de reciclar la enzima, para que pueda interactuar en otros procesos líticos (Barret et al., 2001). El hecho de haber encontrado un receptor para sitios de ubiquitinación (SB40), una hidrolasa de Ubiquitina (Deubiquinasa Peptidasa C19) y una Ubiquinona oxidoreductasa (Clon 11), provee evidencia para sugerir que en el hongo ocurren procesos proteolíticos, posiblemente relacionados con el reciclaje de aminoácidos en las etapas primarias de invasión del sustrato que implica la degradación de los estadios larvarios, así como en el aprovechamiento de las proteínas cuticulares y de glicoproteínas del tipo colágeno (Abrantes, 2002) allí presentes.
Respecto a la degradación de proteínas cuticulares, el hallazgo de una Glycosyl hidrolasa (UA6) es interesante, dado que esta enzima ha sido encontrada en la degradación de proteínas asociadas a carbohidratos como la Manosa (Bairoch, 1996) que podrían estar formando parte del colágeno de la cutícula de los estadíos inmaduros en los huevos. Aún cuando éste carbohidrato no ha sido directamente estudiado en el género Meloidogyne, existen registros de su presencia en géneros de anélidos (Spiros y Bhoyroo, 1980), quienes utilizaron colagenasas para permitir la liberación de los carbohidratos de la cutícula del género Nereis, entre ellos la Manosa. Debido a esto, el gen de la Glicosil hidrolasa puede ser un buen candidato para continuar estudios posteriores relacionados con la expresión de genes de patogenicidad del hongo, debido a que podría estar directamente implicado en la degradación observada de huevos de Meloidogyne spp.
Otro gen de interés relacionado con la degradación de proteínas pertenece al clon SB40, el cual mostró una alta similitud (93%) con un dominio encontrado en humanos, asignando como F-Box, el cual está presente en varias proteínas que participan en la regulación de distintos procesos incluyendo la división celular, transcripción, transducción y desarrollo. El dominio F-Box forma un complejo conocido como SCF (Skp1-Cullin-F-box), el cual juega un papel similar a la E3 ligasa en la vía de la ubiquitación proteínica. Los complejos SCF intervienen en las vías de señalización de proteín-quinasas que controlan la las proteínas. Aún cuando se han dilucidado las estructuras, los mecanismos específicos de acción no estan claros (Patton et al., 1998; Passmore y Bardford, 2004; Skowyra et al., 1997).
El hallazgo de una hidroxilasa de ácidos grasos (UA39), podría indicar uno de los mecanismos de acción del hongo sobre la degradación de los huevos, los cuales tienen una capa lipídica (Wharton,1980; Morgan-Jones et al., 1983). Es interesante observar como hasta la fecha, en las bases de datos revisadas, no fue posible encontrar registros de hongos nematófagos en los cuales se hubiera estudiado este tipo de enzimas relacionadas con la infección. Igualmente es notorio el hecho de no haber encontrado dentro de los clones trabajados alguna secuencia relacionada con la degradación de la quitina, componente importante dentro de la composición de los huevos (Fanelli et al., 2005). De acuerdo con reportes realizados por Villa (2006), la cepa de CENICAFE 9501 presenta la capacidad de degradar la quitina en medio de cultivo. Sin embargo, Cohen y Chet (1998) han registrado como la expresión de las quitinasas se ve inhibida por la presencia de glucosa, carbohidrato que estuvo presente en el medio utilizado en la interacción hongo-huevos de nemátodos en este estudio. Lo anterior podría explicar la ausencia de clones que presentaran identidad con quitinasas de hongos, lo cual deberá ser corroborado en futuros estudios.
Con base en los resultados obtenidos, se cuenta con una técnica establecida para lograr la expresión de genes diferencialmente expresados durante la interacción de la cepa CENICAFE 9501 con huevos de Meloidogyne spp. Sin embargo, se hace necesario continuar las investigaciones con genes candidatos con el fin de incrementar el conocimiento básico de la fisiología de este hongo con potencial biorregulador y de las proteínas relacionadas con la patogénesis, para optimizar en un futuro la producción y formulación de agentes más eficientes para ser usados en programas de manejo integrado de enfermedades.
AGRADECIMIENTOS
Los autores agradecen al Ministerio del Medio Ambiente, al Centro Nacional de Investigaciones del Café (CENICAFE), y al Departamento de Investigaciones de la Universidad Nacional de Colombia, Medellín (DIME) por el soporte financiero y logístico para la realización del trabajo, al igual que a la Corporación para Investigaciones Biológicas de Medellín (CIB), por la asesoría brindada. Así mismo al Dr. Pablo Gutiérrez y la Dra. Lucía Afanador de la Universidad Nacional de Colombia, Sede Medellín, por sus aportes y correcciones a los resultados del trabajo y al grupo de Bioinformática de CENICAFE, por la colaboración en el procesamiento de las secuencias obtenidas.
BIBLIOGRAFÍA
Abrantes, I.M. 2002. Immunolocalization of a putative cuticular collagen protein in several developmental stages of Meloidogyne arenaria, Globodera pallida and G. rostochiensis.J Helminthol. 76(1):1- 6. [ Links ]
Ahren, D., M. Tholander, C. Fekete, B. Rajashekar, E. Friman, T. Johansson and A. Tunlid. 2005. Comparison of gene expression in trap cells and vegetative hyphae of the nematophagous fungus Monacroporium haptotylum. Microbiology 151: 789-803. [ Links ]
Ahman, J., B. Ek, L. Rask and A. Tunlid. 1996. Sequence analysis and regulation of a gene encoding a cuticle degrading serine protease from the nematophagous fungus Arthrobotrys oligospora. Microbiology 142: 1605-1616. [ Links ]
Bairoch, A. 1996. Updating the sequence -based classification of glycosyl hydrolases. Biochem. J. 316: 695-6. [ Links ]
Barrett, A.J. and N.D. Rawlings. 2001. Evolutionary lines of cysteine peptidases. Biol. Chem. 382: 727-733. [ Links ]
Bobadilla, C., J. Mejía y R. Jurado. 1999. Identificación y distribución de las enfermedades de tomate de árbol (Solanum betacea (cav) Sendt), en la región de Sumapaz. pp. 3. En: Memorias. XX Congreso Nacional de Fitopatología y Ciencias Afines. ASCOLFI. Manizales. [ Links ]
Bonants, P.J.M., P.F.L. Petters, H. Thijs, de E. Belder, C. Waalwijk and J.W.D.M. Henfling. 1995. A basic serine protease from Paecilomyces lilacinus with biological activity against Meloidogyne hapla eggs. Microbiology, 141: 775-784. [ Links ]
Cardona, N. 2007. Caracterización molecular de la cepa CENICAFE 9501, y anotación de genes involucrados en la interacción con huevos de Meloidogyne spp. provenientes de zona cafetera. Tesis Ph.D. Ciencias Agropecuarias, área Agraria. Facultad de Agronomía. Universidad Nacional de Colombia. Bogotá. 175 p. [ Links ]
Cardona, N. L. y J.E. Leguizamón. 1997. Aislamiento y patogenicidad de hongos y bacterias al nemátodo del nudo radical del café Meloidogyne spp. Goeldi. Fitopatología Colombiana 21(1): 39-52. [ Links ]
Chenchik, A., Y. Zhu, L. Diatchenko, R. Li, J. Hill, and P.D. Siebert. 1998. Generation and use of high-quality cDNA from small amounts of total RNA by SMART PCR. In Gene Cloning and Analysis by RT-PCR (BioTechniques Books, MA): 305–319. [ Links ]
Cohen, R. and I. Chet. 1998. The molecular biology of chitin digestion. Current Opinion in Biotechnology 9: 270-277. [ Links ]
Davies, K.G., B.R. Kerry and C.A. Flynn. 1994. Microbial agents for the biological control of plant parasitic nematodes in tropical agriculture. Tropical Pest Management 37 (4): 303-320. [ Links ]
Fanelli, E., M. di Vito, J.T. Jones and C. De Giorgi. 2005. Analysis of chitin synthase function in a plant parasitic nematode, Meloidogyne artiellia, using RNAi. Gene. 11 (349): 87-95. [ Links ]
Giraldo, M.A., J.E. Leguizamón y C.B. Chaves. 1996. Evaluación de Paecilomyces lilacinus (Thom) Samson en el control de Meloidogyne spp. Goeldi en almácigo de café Coffea arabica L. var. Caturra. p. 58. En: Congreso de la Asociación Colombiana de Fitopatología y Ciencias Afines, Encuentro Internacional de la Biotecnología Agronómica. Paipa, Colombia. [ Links ]
Giraldo, M.A. y J.E. Leguizamón. 1999. Hongos biocontroladores de Meloidogyne spp. Asociados al café en seis departamentos del país. p. 99. Memorias XX Congreso Nacional de Fitopatología. Ascolfi. Manizales, Colombia. [ Links ]
Góngora, C. y M. Rodríguez. 2003. Transformación de Beauveria bassiana con genes involucrados en la patogenicidad de Metarhizium anisopliae y evaluación del efecto de la expresión de estos en la patogenicidad de Beauveria bassiana contra la broca. p. 87. En: XXX Congreso de la Sociedad Colombiana de Entomología. Cali, Colombia. [ Links ]
Góngora, C. y M. Rodríguez. 2005. Transformación de Beauveria bassiana cepa Bb9205 con los genes pr1A, pr1J y ste1 de Metarhizium anisopliae y evaluación de su patogenicidad sobre la broca del café. Revista Colombiana de Entomologia 31(1): 51-58. [ Links ]
Guilleroux, M. and A. Osbourn. 2004. Gene expresión during infection of wheat roots by the Take all fungus Gaeumannomyces graminis. Molecular Plant Pathology 5(3): 203–216. [ Links ]
Hincapié, R.D. y C.J.E. Leguizamón. 1992. Efecto de Meloidogyne spp. En almácigos de café variedad caturra. Centro Nacional de Investigaciones del café CENICAFE. Disciplina de Fitopatología. Chinchiná, Colombia. [ Links ]
Hincapié, D., J. Leguizamón y J. Arroyave. 1998. Parasitismo de aislamientos nativos de Verticillium chlamydosporium Goddar en estados de Meloidogyne spp. p. 77. En: Memorias. IXX Congreso de la Asociación Colombiana de Fitopatología y Ciencias Afines. Pasto, Colombia. [ Links ]
Hincapié, R.D., C.J. Leguizamón. 1999. Efecto de Verticillium chlamydosporium Goddar en el control de Meloidogyne spp. Goeldi en almácigos de café var. Caturra. CENICAFE 50 (4): 286-298. [ Links ]
Jatala, J. A. 1980. Biological control of plant parasitic nematodes. Annual Review of Phytopathology 12: 253- 259. [ Links ]
Joshi, L., R.J. Leger, D.W. Roberts. 1997. Isolation of a cDNA encoding a novel subtilisin-like protease (Pr1B) from the entomopathogenic fungus, Metarhizium anisopliae using differential display-RT-PCR. Gene. 197: 1-8. [ Links ]
Joshi, L., R.J. Leger and M.J. Bidochka. 1995. Cloning of a cuticle-degrading protease from the entomopathogenic fungus, Beauveria bassiana. FEMS Microbiol Lett. 125: 211-7. [ Links ]
Kanda, S., T. Aimi, S. Kano, S. Ishihara, Y. Kitamoto and T. Morinaga. 2008. Ambient pH signaling regulates expression of the serine protease gene (spr1) in pine wilt nematode-trapping fungus, Monacrosporium megalosporum. Microbiological Research 163: 63-72. [ Links ]
Kerry, B.R. and N.H. Brown. 1987. Principles and practices of nematode control in crops. Academic Press. Sidney. 447 p. [ Links ]
Khan, A., K. Williams and H. Nevalainen. 2003. Testing the nematophagous biological control strain Paecilomyces lilacinus 251 for paecilotoxin production. FEMS Microbiol. Lett. 10. 227 (1): 107-111. [ Links ]
Koltai, H., N. Chejanovsky, B. Raccah and Y. Spiegel. 1997. The first isolated collagen gene of the root-knot nematode Meloidogyne javanica is developmentally regulated. Gene. 196(1-2):191-199. [ Links ]
Lee, A. and T.A. Cooper. (1995). Improved direct PCR screen for bacterial colonies: wooden toothpicks inhibit PCR amplification. Biotechniques 18: 225-226. [ Links ]
Leguizamón, J.E. 1994. Efecto de Meloidogyne spp. en plántulas de Coffea arabica L. variedad Caturra en etapa de vivero. p. 5. En: XV Congreso de la Asociación Colombiana de Fitopatología y Ciencias Afines. Santafé de Bogotá, Colombia. [ Links ]
Leguizamón, J.E. 1995. Interacción de una mezcla de micorrizas y el complejo Meloidogyne spp. en almácigos de café. p. 50-56. En: XVI Congreso de la Asociación Colombiana de Fitopatología y Ciencias Afines. Medellín, Colombia. [ Links ]
Leguizamón, J.E. y B.E. Padilla. 2001. Efecto de Beauveria bassiana y Metarhizium anisopliae en el control del nemátodo del nudo radical del café. CENICAFE 52 (1): 29-41. [ Links ]
Liu, G.Y. and S.S. Tzean. 1997. Phylogeny of the genus Arthrobotrys and allied nematode trapping fungi based on rDNA sequences. Mycologia 89 (6): 876-884. [ Links ]
Lumsden, R.D., J.A. Lewis and D.R. Fravel. 1995. Formulation and delivery of biocontrol agents for use against soilborne plant pathogens. In: Biorational pest control agents. Formulation and delivery, American Chemical Society, Washington, D.C. ACS Symposium Series 595: 166-182. [ Links ]
Montoya, C. A. 1999. Enfermedades del maracuyá causadas por hongos, bacterias, virus y nemátodos. pp. 354-374. En: Memorias Curso Nacional de Frutas Tropicales. Universidad Nacional de Colombia. Palmira. [ Links ]
Morgan, G., J.F. White, R. Rodriguez-Kabana. 1983. Phytonematode pathology: ultraestructural studies. Parasitism of Meloidogyne arenaria in eggs by Verticillium chlamydosporium. Nematropica 13(2): 245-260. [ Links ]
Múnera, U.U.E. 1999. Interacción Meloidogyne spp.- Fusarium spp. en granadilla (Passiflora ligularis). pp. 5. En: Memorias. XX Congreso Nacional de Fitopatología Ascolfi, Manizales. [ Links ]
Passmore, L.A. and D. Barford. 2004. Getting into position: the catalytic mechanisms of protein ubiquitylation. Biochem J. 379 (3): 513-525. [ Links ]
Patton, E.E., A.R. Willems and M. Tyers. 1998. Combinatorial control in ubiquitin-dependent proteolysis: don't Skp the F-box hypothesis. Trends Genet. 14 (6): 236-43. [ Links ]
Price, N.S., J. Machon and M. Realpe. 1997. Nematodos asociados a cultivos de flores en Colombia. Boletín de información científica y tecnológica. CIB-Asocolflores. 1 (6):1-6. [ Links ]
Primrose, S.B., R.M. Twyman and R.W. Old. 2001. Principles of gene manipulation. 6 ed. Blackwell Science, Oxford. 263 p. [ Links ]
Siebert, P., A. Chenchik, D. Kellogg, K. Lukyanov and S. Lukyanov. 1995. An improved PCR method for walking in uncloned genomic DNA. Nucleic Acids Research. 23: 1087-1088. [ Links ]
Sirohi, A., K.C. Mohanty, S.J. Pankaj, A. Eapen, K. Ganguly and U. Rao. 2005. Molecular basis of plant-nematode interaction and genetic engineering for their management. National Symposium on Recent Advances and Research Priorities in Indian Nematology 9 - 10. p. 3. [ Links ]
Spiros, R.G. and V.D. Bhoyroo. 1980. Studies on the carbohydrate of collagens: characterization of a glucuronic acid-mannose disaccharide unit from nereis cuticle collagen. The Journal of Biological Chemistry. 255 (11): 5347-5354. [ Links ]
Stirling, G.R. 1991. Biological control of plant parasitic nematodes. pp. 137-142. En: Progress, problems, and prospects. Wallinford. C.A.B. International. England . [ Links ]
Skowyra, D., K.L. Craig, M. Tyers, S.J. Elledge and J.W.F. Harper. 1997. F-box proteins are receptors that recruit phosphorylated substrates to the SCF ubiquitin-ligase complex. Cell 91 (2): 149-51. [ Links ]
Villa C. 2006. Estudios in vitro sobre la patogenicidad de cultivos monospóricos de Hyphomycete sp. (CENICAFE 9501) sobre huevos de Meloidogyne spp. provenientes de café, Trabajo de grado. Facultad de Ciencias. Universidad de Antioquia. 51 p. [ Links ]
Wang, R.B., J.K. Yang, C. Lin, Y. Zhang and K.Q. Zhang. 2006. Purification and characterization of an extracellular serine protease from the nematode-trapping fungus Dactylella shizishanna. Lett Appl. Microbiol. 42 (6): 589-94. [ Links ]
Wharton, D. 1980. Nematode egg-shells. Parasitology 81: 447-463. [ Links ]














