Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Facultad Nacional de Agronomía Medellín
Print version ISSN 0304-2847
Rev. Fac. Nac. Agron. Medellín vol.64 no.1 Medellín Jan./June 2011
DETECCIÓN DE LOS VIRUS AMV, CMV Y PLRV EN CULTIVOS DE TOMATE DE ÁRBOL (SOLANUM BETACEUM CAV.) EN ANTIOQUIA, COLOMBIA
DETECTION OF VIRUSES AMV, CMV AND PLRV IN TAMARILLO (SOLANUM BETACEUM CAV.) ORCHARDS IN ANTIOQUIA, COLOMBIA
Margarita Jaramillo Zapata1; Pablo Andrés Gutiérrez Sánchez2; José Miguel Cotes Torres3; Elena Paola González Jaimes4 y Mauricio Marín Montoya5
1 Docente Tiempo Completo. Corporación Universitaria Lasallista. Facultad de Ciencias Administrativas y Agropecuarias. Carrera 51 118 sur - 57 Caldas, Antioquia, Colombia. majaramillo@lasallistadocentes.edu.co.
2 Profesor Asociado. Universidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias. Laboratorio de Microbiología Industrial. A.A. 3840, Medellín, Colombia. <paguties@unal.edu.co>
3 Profesor Asociado. Universidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias Agropecuarias. Departamento de Ciencias Agronómicas. A.A. 3840, Medellín, Colombia. <jmcotes@unal.edu.co>
4 Profesora Asistente. Politécnico Colombiano Jaime Isaza Cadavid. Facultad de Ciencias Agrarias. Laboratorio de Sanidad Vegetal. Carrera 48 No. 7-151, Medellín, Colombia. <epgonzalez@elpoli.edu.co>
5 Profesor Asociado. Universidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias. Laboratorio de Biología Celular y Molecular. A.A.3840, Medellín, Colombia. <mamarinm@unal.edu.co>
Recibido: Mayo 12 de 2010; aceptado: Marzo 06 de 2011
Resumen. El tomate de árbol es uno de los principales frutales cultivados en la región andina de Colombia. Durante los últimos años las áreas dedicadas a su plantación se han reducido drásticamente, debido entre otras razones, al efecto detrimental de la enfermedad conocida como Virosis. A pesar de la importancia económica de esta enfermedad, el nivel de conocimiento que se tiene en el país, sobre la identidad taxonómica de los virus asociados a su ocurrencia es precario y por tanto, se carece de buena parte de la información necesaria para el diseño de estrategias de manejo efectivas contra esta enfermedad. En este trabajo se evaluó la presencia e incidencia de los virus isométricos AMV, CMV y PLRV en cultivos de tomate de árbol establecidos en el departamento de Antioquia, mediante el empleo de pruebas de ELISA, RT-PCR y secuenciación de la cápside viral. Las pruebas serológicas permitieron detectar la presencia de los tres virus, aunque las secuencias generadas sólo fueron confirmatorias para el virus PLRV, lo que posiblemente sugiere que existen diferencias en las secuencias dirigidas a los sitios de alineamiento de los cebadores para AMV y CMV. La incidencia de los virus CMV y PLRV en las muestras analizadas fue de un 57 y 41% respectivamente, mientras que para AMV ésta solo llegó a un nivel del 2%. Los hallazgos obtenidos permiten confirmar la presencia de un complejo viral asociado a la enfermedad de la Virosis del tomate de árbol en el territorio mencionado.
Palabras clave: ELISA, RT-PCR, secuenciación, virus de ARN.
Abstract. Tamarillo is one of the main fruit crops in the Colombian andean region. However, during the past years, there has been a drastic reduction in the total crop area due to the detrimental effect of a disease known as Virosis. In spite of the economic impact of this disease, there is little knowledge about the taxonomical identity of its associated viruses; therefore, no information is available for the design of appropriate control strategies. In this work, was evaluated the presence and incidence levels of AMV, CMV and PLRV viruses in tamarillo crops in the department of Antioquia by ELISA, RT-PCR and DNA sequencing of the viral capsid. Even though serological tests detected the presence of all three viruses, DNA sequencing could only confirm the presence of PRLV. The failure to detect AMV and CMV at the DNA level suggests differences in the target sequences used for primer design. CMV and PLRV were detected in 57 and 41% of all the analysed samples in contrast to AMV whose incidence level was only 2%. These findings confirm the presence of viral complex associated to the Virosis disease of tamarillo orchards in the mentioned territory.
Key words: ELISA, RT-PCR, sequencing, RNA viruses.
El tomate de árbol (Solanum betaceum Cav.), es un frutal de origen andino que se cultiva principalmente en Colombia y Ecuador, para el consumo fresco y para la industria de alimentos. En Colombia la producción de tomate de árbol se realiza en 18 departamentos con un área estimada de 7.646 ha, aunque cerca de dos terceras partes de la producción nacional proceden de los departamentos de Antioquia (50%) y Cundinamarca (14%). En el caso de Antioquia los rendimientos ascienden a 32 t/ha, valor muy superior al promedio nacional que es del orden de 18 t/ha. En sentido opuesto, el departamento de Nariño participa con un 5% del área total cultivada en el país, pero su producción tan sólo alcanza el 1% del total nacional, por cuanto los rendimientos son de 7,2 a 10,9 t/ha (Corporación Colombia Internacional, 2007).
Los rendimientos del cultivo en Colombia se ven seriamente afectados por problemas fitopatológicos como la antracnosis (Colletotrichum acutatum), los nemátodos de los nudos radicales (Meloidogyne spp.) y la enfermedad denominada como Virosis (Tamayo, 1990; Tamayo, 1996; Betancourth et al., 2003, Cruz, 2005; Ayala, 2009). Todos estos factores han conducido a que los agricultores deban realizar renovaciones de la plantación luego de 16–18 meses de producción, cuando anteriormente la vida útil se extendía hasta por 48 meses.
En Colombia la primera enfermedad asociada a virus en el cultivo del tomate de árbol se detectó en el departamento de Boyacá y fue denominada necrosis anular del tomate de árbol. El agente causal de esta enfermedad se caracteriza por presentar partículas alargadas y flexuosas de aproximadamente 750 nm de largo y transmisión mecánica a partir de inóculo de frutos enfermos. Se cree que este virus hace parte del género Potyvirus aunque no se realizaron estudios serológicos o moleculares para su confirmación (Sánchez, 1982a; Sánchez 1982b; Maldonado y Sánchez, 1984; Tamayo, 1996). El segundo problema viral fue reportado por Sañudo y Orellana (1989), quienes registraron la presencia de síntomas virales en plantas de tomate de árbol en el Valle de Sibundoy (Putumayo). Esta enfermedad se caracterizaba por inducir el amarillamiento general de la planta y causar clareamiento de venas, vejigas y ampollas, enrollamiento foliar y reducción del área fotosintética; sin embargo, la identidad de su agente causal no fue determinada. Posteriormente, se reportaron en cultivos de los municipios de Rionegro y Marinilla (Antioquia) árboles de tomate de árbol con mosaicos. Tamayo (1990) observó la presencia de partículas isométricas de aproximadamente 30 nm de diámetro asociados a este síntoma. Para su detección se desarrolló un anticuerpo policlonal denominado Col-7 (Tamayo et al., 1999). Aparentemente este virus no se transmite por semilla sexual pero sí de forma mecánica a plantas de tomate de árbol y para el momento de su detección, se registraba que su incidencia era muy baja en cultivos del norte de Antioquia (Tamayo, 1990).
El cuarto problema viral denominado virosis del tomate de árbol, se detectó en 1991 en el municipio de Santa Rosa de Osos y en 1992 en el oriente antioqueño (Saldarriaga y Bernal, 1994). Los síntomas de esta enfermedad se manifiestan en hojas y frutos y se caracterizan por la presencia de mosaicos, engrosamiento de nervaduras, ampollas y formación de rosetas. En los frutos verdes se presentan manchas moradas que cambian a diferentes tonalidades rojizas con la maduración; además, es frecuente encontrar frutos deformes y daños en la calidad de la pulpa. Aparentemente el virus no se transmite por semilla, pero sí mecánicamente y presenta partículas alargadas y flexuosas de aproximadamente 750 nm de largo (Tamayo, 1996; Saldarriaga et al., 1997; Saldarriaga et al., 2000). La alta incidencia de esta enfermedad en el norte antioqueño quedó demostrada a partir de un estudio realizado por Tamayo et al. (1999), donde se encontró que de 83 muestras foliares con síntomas aparentes de Virosis, 22 reaccionaron con el antisuero CoL-11 desarrollado en el laboratorio del Centro Internacional de la Papa para la detección de este virus. La especificidad de estos anticuerpos no se ha determinado, pudiendo presentar reacciones cruzadas con otros patógenos virales que afectan este cultivo en Colombia (Ayala, 2009).
En el Valle del Cauca se reportó una quinta enfermedad de etiología viral asociada a cultivos de tomate de árbol, cuyos síntomas se manifiestan inicialmente con la presencia de un mosaico leve en hojas jóvenes, además de presencia de vejigas, deformación y enrollamiento de la lámina foliar. A medida que la enfermedad avanza las hojas se presentan más pequeñas, arrugadas y tienden a caerse prematuramente. En la etapa final hay defoliación y las ramas comienzan a secarse de manera descendente. Su agente causal fue detectado mediante microscopía electrónica, encontrándose partículas isométricas y flexuosas que se transmiten mecánicamente y por áfidos (Chávez y Varón, 2001). Betancourth et al. (2003), registraron la ocurrencia de una sexta enfermedad viral en el departamento de Nariño caracterizada por inducir manchas aceitosas, clorosis, mosaicos, distorsiones en color, tamaño y forma de las hojas y frutos. Su agente causal presentaba morfología de varilla flexuosa, de 800 nm y su transmisión fue confirmada mediante áfidos de la especie Myzus persicae e inoculación mecánica. Adicionalmente, existen informes preliminares basados en pruebas de detección por RT-PCR de la presencia de un posible cucumovirus y de un polerovirus en cultivos de tomate de árbol en Cundinamarca con síntomas de malformación en hojas, clorosis, baja talla, volcamiento y llagas (Cruz, 2005). Más recientemente, Ayala (2009) determinó mediante análisis serológicos y moleculares que al menos dos especies de potyvirus (Potato virus Y, PVY y Tamarillo leaf malformation virus, TaMLV) estaban asociadas a la Virosis en cultivos del departamento de Antioquia.
En el mundo sólo existen registros de virus en cultivos de tomate de árbol en Nueva Zelanda, India y Ecuador. En el primer país se han identificado los virus: Tamarillo mosaic virus (TaMV, Potyvirus), Cucumber mosaic virus, (CMV, Cucumovirus), Potato aucuba mosaic virus (PAMV, Potexvirus), Alfalfa mosaic virus (AMV, Alfamovirus), Tomato spotted wilt virus (TSWV, Tospovirus), Tomato mosaic virus (ToMV, Tobamovirus) (Fletcher, 1987; Eagles et al., 1990; Eagles, 1994; Eagles et al., 1994). Adicionalmente, Chamberlain (1954) y Bhargava y Joshi (1959), confirmaron la presencia del PVY (Potyvirus) en cultivos de Nueva Zelanda e India, respectivamente. En Ecuador, se han encontrado sintomatologías severas de enfermedades virales del tomate de árbol en los valles de la provincia de Pichincha. Estos síntomas corresponden a amarillamiento de venas, manchas aceitosas, moteados y manchas cloróticas. Mediante pruebas de ELISA fue posible asociar el amarillamiento de venas y mosaicos con la infección por Potato leafroll virus (PLRV, Polerovirus), Tomato ringspot virus (ToRSV, Nepovirus) y PVY; mientras que las manchas cloróticas se relacionaron con PLRV, ToRSV y AMV (Vizuete et al., 1990; Ochoa e Insuasti, 2005).
Los efectos de las enfermedades virales sobre los planes de expansión del cultivo de tomate de árbol en Colombia, son evidentes en regiones como el departamento de Antioquia, donde durante el año 2007 se redujo en 1.533 ha el área cultivada con este frutal (Gobernación de Antioquia, 2008). Esta problemática sumada a referencias nacionales e internacionales sobre la presencia de múltiples especies de virus asociadas a las enfermedades de este cultivo, condujeron a plantear el presente estudio, tendiente a identificar los virus isométricos asociados a la Virosis del tomate de árbol en Antioquia y determinar sus niveles de incidencia en las principales regiones productoras del departamento, de manera que sea posible en el mediano plazo el planteamiento de estrategias de manejo acordes con cada uno de los componentes del complejo viral asociado a esta enfermedad. Debido a la gran diversidad de síntomas virales encontrados en los cultivos de tomate de árbol y a la imposibilidad de asociar alguno de los virus reportados con un grupo de síntomas en particular, en esta investigación se seguirá el planteamiento de Ayala (2009) que utiliza la denominación de “Virosis” del tomate de árbol para referirse al conjunto de enfermedades de origen viral que afectan este cultivo.
MATERIALES Y MÉTODOS
Localización. Esta investigación se desarrolló en el Laboratorio de Biología Celular y Molecular de la Universidad Nacional de Colombia, Sede Medellín. Las muestras de plantas afectadas por Virosis del tomate de árbol fueron obtenidas en las cinco principales regiones cultivadoras de la especie en Antioquia: oriente cercano (Carmen de Viboral, Marinilla, Rionegro), oriente (La Unión, Sonsón), norte cercano (Entrerríos), norte (Santa Rosa, Yarumal) y suroeste (Jardín) (Tabla 1).
Tabla 1. Procedencia de las muestras de tomate de árbol utilizadas para determinar los niveles de incidencia de virus isométricos en Antioquia, Colombia.
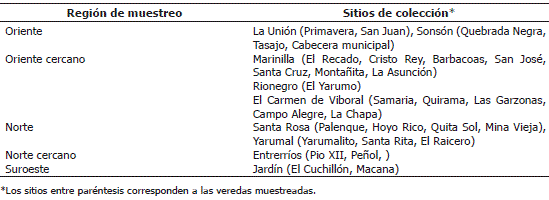
Colección de muestras y descripción de síntomas. Con el fin de determinar la presencia y los niveles de incidencia de los virus isométricos AMV, CMV y PLRV, seleccionados para su evaluación por considerarse que afectan los cultivos de los andes ecuatorianos y Nueva Zelanda (Vizuete et al., 1990; Eagles, 1994), se realizó un muestreo aleatorio en 10 lotes de las regiones norte cercano, suroeste y oriente y en 20 lotes del oriente cercano y norte; en cada lote, se tomaron 30 muestras de cuatro hojas, las diferencias en los números de lotes evaluados, se deben a que el oriente cercano y norte del departamento presentan una mayor área plantada con este cultivo.
Detección serológica de los virus. De cada muestra compuesta se pesaron 100 mg de tejido foliar afectado y se maceró con el buffer de extracción 1X. Con el macerado se realizó la prueba de ELISA mediante el uso de anticuerpos específicos para los virus AMV, CMV y PLRV, adquiridos en la compañía Agdia (Indiana, USA). Los resultados colorimétricos fueron cuantificados en un equipo Multiscan (Labsystem, Finlandia). Cada prueba incluyó un control positivo suministrado por el fabricante en forma liofilizada y un control negativo (buffer de extracción). Los pozos considerados con reacción positiva fueron aquellos en los cuales la lectura de absorbancia a 405 nm presentaba un valor mínimo del doble de la lectura obtenida en el control negativo (Matthews, 1993).
Los resultados fueron analizados a través de un modelo lineal generalizado aleatorio considerando una distribución binomial para la variable incidencia y utilizando una función de ligamiento logit. Los efectos aleatorios fueron: región, lotes de cultivo dentro de región, plantas dentro de lotes de cultivo. Para el análisis estadístico se utilizó el procedimiento GLIMMIX de SAS v. 9.1.3.
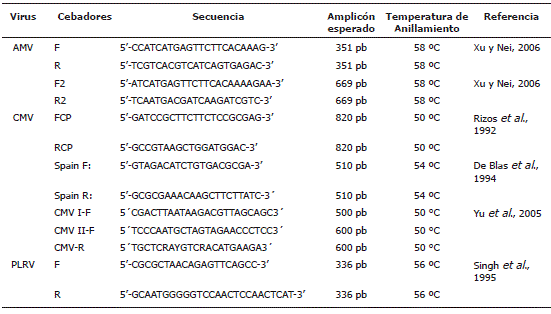
Identificación molecular de virus. El ARN molde necesario para la técnica de RT-PCR se obtuvo a partir del ARN total extraído de un mínimo de diez muestras que resultaron positivas en las pruebas de ELISA para los virus AMV, CMV y PLRV, para lo cual se utilizó el kit RNeasy plant mini (Qiagen, California, USA). Para la extracción de ARN se maceraron 100 mg de tejido foliar en 450 mL de buffer RLT y 10 mL de β-mercaptoetanol, siguiendo las instrucciones del fabricante. Al finalizar el procedimiento, el ARN obtenido fue diluido en 40 mL de agua destilada estéril libre de ARNasas. Las reacciones de RT-PCR se realizaron con el kit Qiagen OneStep RT-PCR, con 5 mL de ARN, 0,6 mM de cada cebador específico, 1X de Buffer Qiagen OneStep RT-PCR Buffer, 400 mM de dNTPs y 2 mL de mezcla de enzimas Qiagen (transcriptasa reversa Omniscript, transcriptasa reversa Sensiscript y Polimerasa de ADN HotStarTaq). Inicialmente se realizó la síntesis de la primera cadena a 50 °C por 30 min, posteriormente se activó la Taq polimerasa HotStar a 95 °C por 15 min y finalmente se procedió al programa de amplificación con 35 ciclos a 94 °C por 1 min, 50 a 58 °C por 1 min (dependiendo de los cebadores), 72 °C por 1,30 min y una extensión final de 10 min a 72 °C. Los cebadores fueron seleccionados con base en secuencias disponibles en el GenBank y en reportes de su uso en otros hospedantes (Tabla 2).
Tabla 2. Cebadores específicos para la región de la cápside de tres virus isométricos evaluados en muestras de tejido de tomate de árbol afectado por la virosis en Antioquia, Colombia.
Los productos amplificados fueron separados por electroforesis en gel de agarosa al 1,5% suplementado con 4 mL de bromuro de etidio (10 mg•mL-1), visualizados en un transiluminador UV (Biometra, Göttingen, Alemania) y su tamaño verificado por comparación con el marcador de peso molecular Generuler 100 pb DNA ladder (Fermentas).
De los amplicones del tamaño esperado para cada uno de los virus bajo análisis, se purificaron para su secuenciación al menos cinco de ellos mediante el kit QIAquick PCR Purification (Qiagen®). Cuando las reacciones no eran específicas se purificaron los fragmentos del tamaño esperado con el kit QIAquick Gel Extraction (Qiagen). La secuenciación se realizó en ambos sentidos utilizando los mismos cebadores del RT-PCR en un secuenciador ABI Prism 3730xl (PE Applied Biosystems) de la compañía Macrogen (Corea del Sur).
Las secuencias obtenidas con cada cebador fueron editadas mediante el software BioEdit 6.0.6 y Chromas 1.45, construyéndose secuencias consenso y confirmándose su identidad con genes virales por comparación con las bases de datos moleculares, mediante el programa Blastn (http://www.ncbi.nlm.nih.gov/BLAST/Blast.cgi). Paralelamente se obtuvieron secuencias del GenBank de diferentes variantes del virus que se logró amplificar en este trabajo, con éstas se realizó un alineamiento mediante el software Clustal W y se determinaron sus niveles de identidad con la región secuenciada de las cepas que afectan el cultivo de tomate de árbol en Antioquia.
RESULTADOS
Colección de muestras y descripción de síntomas. En total se obtuvieron muestras de tomate de árbol con síntomas de Virosis en 29 veredas de los ocho municipios antioqueños evaluados (Tabla 1). En términos generales los cultivos evaluados presentaban síntomas de afecciones virales de diferente índole, aunque fue evidente la ocurrencia de un patrón común de sintomatologías en árboles provenientes de plantaciones de las diferentes regiones de Antioquia, caracterizado por un mosaico acompañado de ampollamientos, engrosamiento y bandeamiento de venas; deformación de brotes, grabados foliares y en frutos; cambios en la coloración de las flores y frutos, endurecimiento de la pulpa, deformación de frutos, aborto floral y finalmente decaimiento general de las plantas (Figura 1).

Figura 1. Síntomas característicos de Virosis del tomate de árbol en el departamento de Antioquia, Colombia. (A) Bandeamiento de venas. (B) Ampollamiento y engrosamiento de venas. (C-D) Mosaicos y moteados foliares. (E-F) Grabados foliares y en frutos. (G) Amarillamiento intervenal. (H) Blanqueamiento intervenal.
Detección serológica de los virus. La detección de virus isométricos en cultivos de tomate de árbol en Antioquia mediante pruebas de ELISA, arrojó resultados positivos para la presencia de AMV, CMV y PLRV. El análisis de incidencia determinó que el virus CMV se presenta en un nivel más alto, con un promedio general de 57%, siendo más frecuente en plantaciones de la región del suroeste (93%) y menos incidente en el municipio de Entrerríos (norte cercano) (Figura 2). Por otra parte, el virus PLRV presentó una incidencia promedia del 41%, con bajos niveles de detección en el suroeste (25%, municipio de Jardín) y mayor presencia (58%) en los cultivos de la región del oriente cercano. Finalmente, el virus AMV se detectó en forma marginal en las cinco regiones muestreadas, su incidencia promedio fue del 2% y en ninguna región estos valores superaron el 7%. El análisis estadístico de los niveles de incidencia de cada virus entre regiones, no arrojó diferencias significativas (α=0,05), con excepción de la presencia de CMV en los cultivos del suroeste, que fue significativamente superior con respecto a las otras regiones estudiadas (Figura 2).
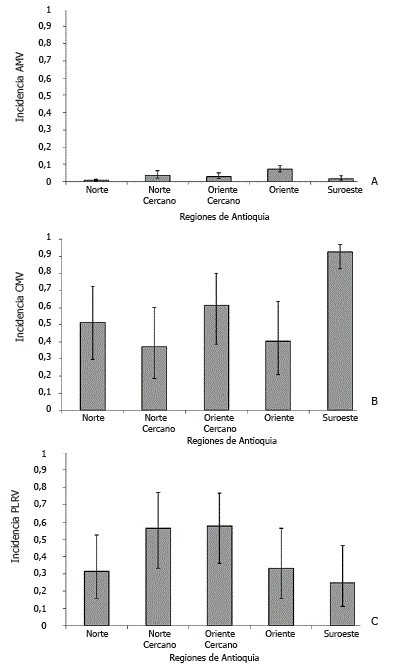
Figura 2. Niveles de incidencia determinados a partir de pruebas de ELISA en tomate de árbol para los virus AMV (A), CMV (B) y PLRV (C) en cinco regiones del departamento de Antioquia, Colombia. Las líneas indican los intervalos de confianza al 95%.
Identificación molecular de virus. A partir de diez muestras de tejido foliar previamente evaluadas como positivas en las pruebas de ELISA para los diferentes virus evaluados en el presente estudio, se realizaron extracciones de ARN total con el fin de utilizarlas para amplificar regiones diagnósticas de dichos virus mediante RT-PCR con cebadores específicos. De esta forma, para el virus AMV se obtuvieron amplicones cercanos a 310 pb con los cebadores AMV F y R; mientras que para CMV los cebadores CMV F y R condujeron a la generación de dos bandas de aproximadamente 1000 y 800 pb; los cebadores CMV-spain F, R y CMV I-F, II-F, R, resultaban en múltiples bandas inespecíficas. Para el caso de PLRV, los cebadores PLRV F y R generaron un solo amplicon de ~330 pb. Una vez enviados los amplicones a secuenciar, el análisis de Blast de las secuencias obtenidas indicó que solamente las bandas obtenidas con los cebadores PLRV F y R correspondían a las regiones que codifican para la cápside viral en dicho virus, mientras que las secuencias de los demás amplicones estaban relacionadas con diferentes genes de origen vegetal, especialmente de la familia Solanaceae. Por esto, no fue posible obtener información de las secuencias de la cápside viral de AMV y CMV.
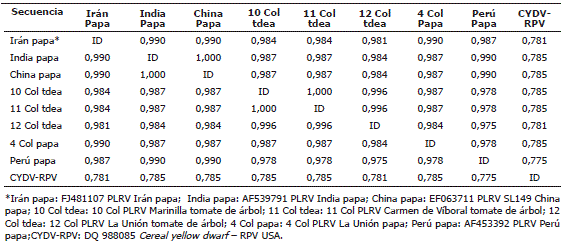
El análisis de identidad utilizando dichas secuencias y otras de referencia obtenidas del GenBank de cepas de PLRV procedentes de diferentes cultivos de papa, indicó un alto nivel de identidad para la región estudiada, siendo en todos los casos superior al 97% (Tabla 3). Los niveles de identidad entre las cepas de PLRV obtenidas en cultivos de tomate de árbol de diferentes regiones de Antioquia fueron superiores al 98% e incluso compartieron este nivel de identidad con una cepa de PLRV obtenida en cultivos de papa del municipio de La Unión (Antioquia) (Gil et al., 2009a).
Tabla 3. Matriz de identidad para una región de 330 pb de la cápside viral de aislamientos del PLRV procedentes de cultivos de tomate de árbol y papa de diferentes lugares del mundo.
DISCUSIÓN
La Virosis del tomate de árbol es en la actualidad el problema fitosanitario más limitante en la producción de este cultivo en Colombia (Mejía et al., 2009). Estudios realizados por Tamayo et al. (1999), Saldarriaga et al. (1997), Saldarriaga et al. (2000), Sañudo y Orellana (1989), Betancourth et al. (2003), Chávez y Varón (2001), Gil et al. (2009b), determinaron con base en pruebas biológicas y de microscopía electrónica la posible asociación de un potyvirus con dicha enfermedad; situación que fue recientemente confirmada por Ayala (2009), quien utilizando pruebas serológicas y moleculares identificó al menos dos especies de este género afectando cultivos sintomáticos de diferentes regiones del departamento de Antioquia; dichas especies pertenecen al género Potyvirus: Potato virus Y (PVY) y a un posible nuevo virus denominado Tamarillo leaf malformation virus (TaLMV). Adicionalmente a la ocurrencia de potyvirus en este cultivo en Colombia, trabajos preliminares realizados por Tamayo (1996) y Tamayo et al. (1999), sugerían la presencia de al menos un virus isométrico, que Cruz (2005) ha asociado a PLRV y/o CMV, aunque esta situación no fue confirmada mediante análisis de secuencias. En este trabajo mediante pruebas serológicas basadas en ELISA con anticuerpos específicos, se logró determinar la presencia en cultivos de tomate de árbol de Antioquia, de éstos virus isométricos (CMV y PLRV), además del virus AMV.
Aunque en el mundo, los trabajos de virología en cultivos de tomate de árbol son muy reducidos, los pocos realizados han planteado la ocurrencia de complejos virales en este frutal, hipótesis inicialmente soportada por la diversidad de síntomas presentes en cultivos de Nueva Zelanda (Chamberlain, 1954; Eagles, 1994; Eagles et al., 1994) y posteriormente confirmada mediante pruebas de ELISA en Ecuador (Vizuete et al., 1990). Los virus más frecuentemente reportados en dichos complejos incluyen por lo menos un potyvirus, que para el caso del Ecuador corresponde al PVY, mientras que en Nueva Zelanda se reportan incidencias hasta del 100% del TaMV. Adicionalmente, se ha detectado en forma frecuente la presencia de los virus PLRV, ToRSV y AMV en los valles de la región de Pichincha de Ecuador, mientras que CMV, PaMV, AMV, TSWV y ArMV han sido diagnosticados en cultivos de Nueva Zelanda (Fletcher, 1987; Eagles, 1994; Eagles et al., 1994).
El efecto detrimental de la Virosis del tomate de árbol en los cultivos del país, fue evidenciado en las salidas de colección, donde se observó una drástica disminución en las áreas explotadas con este frutal en municipios tradicionalmente productores como Entrerríos y Santa Rosa (Antioquia), disminución confirmada por las alcaldías y UMATAS (Unidades Municipales de Asistencia Técnica Agropecuaria). Debido a los problemas inherentes a la falta de estadísticas en el sector agropecuario colombiano, estas reducciones en el área dedicada al cultivo no han sido completamente registradas, ya que oficialmente a nivel nacional sólo se obtienen cifras de este frutal hasta el año 2004 (Ministerio de Agricultura y Desarrollo Rural, 2006). Sin embargo, en fuentes locales como el Anuario Estadístico de Antioquia de 2008, se indica que en este departamento para el año 2007, se perdieron o fueron erradicadas por los agricultores cerca de 1.533 ha del cultivo (Gobernación de Antioquia, 2008). Paralelamente a la reducción del área establecida con este frutal, aquellos agricultores que poseen la capacidad financiera para mantenerse en esta actividad, desplazan sus cultivos a nuevas regiones con la idea de encontrar zonas libres de los patógenos virales o de sus vectores, pero siguiendo las mismas prácticas agronómicas deficientes (falta de semilla certificada, exceso de fertilización, ausencia de podas sanitarias o podas de formación inadecuadas, ausencia en el control de focos de la enfermedad, transporte de material de propagación desde las zonas de producción actual ya afectadas por la virosis, etc.) que finalmente desencadenan en la misma problemática viral. Esta situación fue evidente en regiones como el municipio de Jardín (suroeste antioqueño), donde a partir de 2006, se generó una plantación masiva de tomate de árbol por parte de productores del norte de Antioquia y luego de dos años de cultivo, se procedió a erradicar.
Con respecto a la sintomatología de la enfermedad presente en las plantas de diferentes regiones de Antioquia, se encontró un patrón común consistente en un mosaico con ampollamientos acompañado por diversas deformaciones foliares y cambios en la apariencia de flores y frutos. Estos síntomas han sido mencionados de forma repetida por diversos autores como Tamayo (1996) y Saldarriaga et al. (1997, 2000), Gil et al. (2009b) en Antioquia; Sánchez (1982a,b) en Boyacá, Cuspoca (2007) en Cundinamarca, Betancourth et al. (2003) en Nariño y Sañudo y Orellana (1989) en Putumayo. De esta forma la denominación propuesta por Tamayo (1996) para diferenciar la “Virosis” del “Mosaico” del tomate de árbol actualmente no es aplicable, pues existe un traslape de síntomas entre ambas enfermedades y tal como lo expresó recientemente Ayala (2009), es frecuente encontrar plantas con síntomas exclusivos de mosaico en las que se detectan potyvirus, desvirtuándose la idea que este síntoma es causado únicamente por un virus isométrico. De esta forma se ha propuesto emplear en términos genéricos la denominación de Virosis del tomate de árbol para el conjunto de enfermedades de origen viral que ocurre en este cultivo (Ayala, 2009).
El estudio de los niveles de incidencia en las zonas productoras de tomate de árbol de Antioquia de tres de los virus isométricos reportados en otros países (AMV, CMV y PLRV) y seleccionados con el fin de complementar el trabajo realizado por Ayala (2009) en las mismas regiones pero evaluando la incidencia de potyvirus, confirmó la frecuente asociación de los virus CMV y PLRV con la Virosis del tomate de árbol, encontrándose niveles de incidencia promedios de 57 y 41% en los cultivos bajo estudio, respectivamente; mientras que el virus AMV fue detectado en forma minoritaria, no superando el 7% de incidencia en ninguna de las zonas muestreadas. Estos resultados basados en un universo de 2.100 muestras procesadas, obtenidas en al menos 10 lotes en cada una de las cinco regiones en estudio, conducen a plantear que los programas de manejo integrado de la enfermedad, especialmente los dirigidos en mejoramiento genético, deben orientarse no sólo a los potyvirus, sino también a CMV y PLRV, siendo importante realizar análisis de incidencia detallados para estos virus en las otras regiones productoras del país.
Cuando se analizaron los niveles de incidencia de los tres virus entre las regiones de estudio, no se encontraron diferencias significativas, con excepción de la presencia de CMV en el municipio de Jardín, que alcanzó un valor del 92,5%, muy superior a lo encontrado en las otras zonas (rango de 37 a 61%). La explicación a este hallazgo requiere de estudios más profundos que permitan evaluar las diferentes variables epidemiológicas involucradas en el desarrollo de las enfermedades virales, como lo son los métodos de transmisión, sanidad de la semilla primaria, estado fisiológico de los cultivos, condiciones medioambientales de las regiones productoras, el manejo agronómico, la presencia de otros cultivos y arvenses que puedan actuar como hospedantes alternos de los virus o sus vectores, entre otras. No obstante, el hecho de que la región del suroeste antioqueño, es una zona nueva en la producción de este frutal, puede aportar un factor a considerar cuando se realicen los estudios planteados.
La infección de plantas con múltiples virus es un fenómeno común, que puede resultar o no en la interacción entre virus, en el neutralismo ó alternativamente generar reacciones de antagonismo o sinergismo. En el antagonismo, la infección o acumulación de uno de los virus es completa o parcialmente reducida por efecto de la presencia del otro y frecuentemente puede conducir al fenómeno de la protección cruzada; mientras que en el sinergismo, la interacción resulta en el aumento de la severidad de los síntomas y en la acumulación de partículas de los grupos virales participantes (Matthews, 1993; Untiveros et al., 2007). Los sinergismos mejor estudiados son los ocurridos en tabaco con los virus PVX y PVY, que en dobles infecciones resultan en el incremento de la acumulación de PVX en comparación con plantas infectadas sólo con este virus (González et al., 2004). Así mismo, son bien conocidos los trabajos que han confirmado el sinergismo de CMV con un amplio rango de otros virus, especialmente de potyvirus (Wang et al., 2004) y los múltiples sinergismos que ocurren entre algunos de los más de 20 virus que afectan la batata (Ipomoea batatas), siendo notable aquel que resulta en la enfermedad denominada (SPVD) (Sweet potato virus disease), causada por la interacción entre Sweet potato chlorotic stunt virus (SPCSV, Crinivirus) y Sweet potato feathery mottle virus (SPFMV, Potyvirus) y entre SPCSV con los potyvirus: Sweet potato latent virus, Sweet potato mild speckling virus; el ipomovirus: Sweet potato mild mottle virus y el cucumovirus: CMV, entre otros (Untiveros et al., 2007). Ya que la naturaleza de la interacción entre los diferentes virus detectados en esta investigación en plantas de tomate de árbol en Colombia se desconoce, es necesario profundizar en estudios que permitan determinar la posible existencia de sinergismos, antagonismos o neutralismos entre dichos virus ó variantes intraespecíficas.
Las reacciones de RT-PCR dirigidas a la detección de los diferentes virus previamente identificados mediante anticuerpos específicos con pruebas de ELISA, permitieron obtener amplicones de tamaños esperados para los virus AMV, CMV y PLRV, aunque la confirmación de la naturaleza viral de dichos fragmentos mediante secuenciación, solamente se logró para el último virus, siendo las secuencias para los demás, representativas de diferentes regiones del genoma del hospedante. Estos resultados pueden ser analizados desde dos perspectivas básicas: 1) Que los cebadores utilizados al ser diseñados con base en secuencias de cepas virales obtenidas en otros cultivos y regiones del mundo, podrían no ser lo suficientemente conservados para cubrir todo el rango de variación de cada especie viral, más cuando se considera que no existe ninguna secuencia depositada en las bases de datos moleculares para virus de estas especies procedentes de S. betaceum y 2) Que los cebadores presentan regiones complementarias en el genoma de S. betaceum, que conducen a amplificaciones inespecíficas, favorecidas por las temperaturas relativamente bajas de alineamiento (menores a 58 °C) sugeridas por la literatura para el uso de dichos cebadores (Rizos et al., 1992; De Blas et al., 1994; Singh et al., 1995; Yu et al., 2005; Xu y Nie, 2006). De otra parte, estos resultados refuerzan la necesidad de incluir durante los procesos de estandarización de las pruebas de detección molecular de virus de plantas, la fase de confirmación mediante secuenciación de los amplicones aparentemente de tamaños esperados, pues de no ser así se podrían presentar diferentes falsos positivos, que conducen a diagnósticos erráticos. Adicionalmente, la ausencia de amplificación en algunos de los virus evaluados, hacen necesario formular propuestas de investigación que permitan secuenciar completamente los genomas de las cepas virales presentes en cultivos de tomate de árbol, partiendo de metodologías clásicas de virología vegetal como la purificación de virus, separación de mezclas de virus a partir de inoculaciones en diferentes plantas indicadoras y la generación de ADN copia (ADNc) con cebadores genéricos como hexámeros y oligo-dt (Hull, 2004).
El análisis de la secuencia parcial del gen de la cápside viral en cepas de PLRV obtenidas a partir de cultivos de tomate de árbol, indicó un muy alto nivel de identidad (superior al 98%) entre las cepas de Antioquia, y entre éstas y la cepa de referencia 4 Col obtenida en un cultivo de papa del municipio de La Unión (Antioquia). Esta situación conlleva a plantear que ambos cultivos comparten las mismas variantes de PLRV, aunque es necesario realizar estudios que permitan evaluar esta situación en pruebas de patogenicidad cruzada. Los altos niveles de identidad encontrados en este estudio entre cepas de PLRV, han sido registradas en trabajos previos realizados con variantes del virus de diferentes países (Guyader y Ducray, 2002; Taliansky et al., 2003; Mukherjee et al., 2003; Plchova et al., 2009), en donde reiteradamente se encuentra que los niveles de variación en la secuencia completa del genoma de PLRV no supera el 6%, siendo el ORF 3 que codifica para la cápside viral, aún más conservado entre cepas de diferentes orígenes geográficos, con un rango de identidad entre el 96,5% y 99,8%. Estos estudios basados en la secuenciación de los genomas completos de PLRV, han encontrado que los ORF 0 y el extremo 5´ del ORF1, son las regiones del genoma que ofrecen un mayor nivel de variación en esta especie viral, aunque los niveles de identidad siguen siendo muy altos, ya que superan el 94,5% (Guyader y Ducray, 2002; Plchova et al., 2009). Sin embargo, con base en secuencias de estos ORF se han identificado tres linajes filogenéticos en el virus PLRV: el linaje II corresponde a cepas exclusivamente del continente australiano, mientras que los linajes I y III están conformados por cepas de varias regiones geográficas (Guyader y Ducray, 2002) que no presentan diferencias en su eficiencia de transmisión por áfidos o grado de virulencia en papa. En este sentido, resulta de gran interés la secuenciación de estas regiones del genoma viral de las cepas colombianas de PLRV en papa y tomate de árbol, de tal manera que se evalúe su afinidad filogenética con los linajes mencionados. Adicional a esta evaluación, la realización de pruebas de patogenicidad con aislamientos de tomate de árbol en Physalis floridana, Datura stramonium, Capsicum spp., S. lycopersicum, permitiría identificar sí dichas variantes corresponden a una de las razas tradicionalmente designadas para aislamientos de este virus de hospedantes diferentes a papa: Tobacco yellow top virus, Tomato yellow top strain (Thomas, 1984) y Capsicum yellows virus (Gunn y Pares, 1990; ICTV, 2006).
CONCLUSIONES
Este estudio representa un avance en el nivel de conocimiento de los virus asociados a la Virosis que afecta el cultivo de tomate de árbol en Antioquia. Los resultados permiten complementar los trabajos recientes de Ayala (2009) y Gil et al. (2009a) relacionados con la detección de al menos dos especies de potyvirus en cultivos de tomate de árbol de Antioquia, confirmándose así la presencia de un complejo de virus flexuosos e isométricos afectando a este cultivo en Colombia, con tres grupos virales predominantes: potyvirus, CMV (Cucumovirus) y PLRV (Polerovirus). La manifestación de este complejo viral hace necesario profundizar en el estudio de las diferentes variables epidemiológicas involucradas en el establecimiento y expansión de esta enfermedad en las regiones andinas del país, estos estudios incluyen la evaluación de los mecanismos de transmisión de cada uno de los virus, la susceptibilidad de los diferentes ecotipos de tomate de árbol disponibles en el mercado, la presencia de hospedantes alternos de estos virus entre plantas arvenses y cultivos de importancia económica que comparten las zonas agroecológicas del tomate de árbol, la ocurrencia de fenómenos de sinergismo y/o antagonismo entre virus, la asociación de sintomatologías específicas a cada grupo viral, el desarrollo de herramientas de detección eficientes basadas en la genotipificación de cada virus y de su rango de variación intraespecífica y finalmente el perfeccionamiento de métodos de manejo que conduzcan de nuevo a la expansión de este frutal para su utilización en la industria de bebidas y alimentos y para su exportación al mercado internacional como fruta exótica.
AGRADECIMIENTOS
Esta investigación fue financiada por el proyecto COLCIENCIAS Código 1118-405-20317 Contrato 408-2007 “Desarrollo de métodos de detección serológica y molecular del complejo de virus asociado al mosaico del tomate de árbol en Colombia”. Se agradece a los productores de tomate de árbol de Antioquia que permitieron la colección de las muestras y a los integrantes del Laboratorio de Biología Celular y Molecular de la Universidad Nacional de Colombia, Sede Medellín.
BIBLIOGRAFÍA
Ayala, M. 2009. Caracterización del Potyvirus asociado a la virosis del tomate de árbol en Antioquia. Tesis Magíster en Ciencias Agrarias. Facultad de Ciencias Agropecuarias. Universidad Nacional de Colombia, Medellín. 96 p. [ Links ]
Betancourth, C., R. Goyes y D. Bravo. 2003. Caracterización biológica de un virus del tomate de árbol (Solanum betaceum Sendt) en el departamento de Nariño. Fitopatología Colombiana 27(1): 7-10. [ Links ]
Bhargava, K. and R. Joshi. 1959. A virus disease of tree tomato, Cyphomandra betacea Sendt. due to Potato virus Y. American Journal of Potato Research 36(8): 288-291. [ Links ]
Chamberlain, E. 1954. Plant viruses in New Zealand. Department of Scientific and Industrial Research, Australia. 129 p. (Bulletin No. 108). [ Links ]
Chávez, B. y F. Varón. 2001. Enfermedad de etiología viral en cultivos de tomate de árbol. Epidemiología Agrícola. pp. 39-43. [ Links ]
Corporación Colombia Internacional CCI. 2007. Sistema de información de precios de insumos y factores asociados a la producción, costos de producción por hectárea, http://www.agronet.gov.co/; consulta: agosto 2008. [ Links ]
Cruz, L. 2005. Identificación de virus en Solanum betaceum. Trabajo de grado de Ingeniería Agronómica. Facultad de Agronomía, Universidad Nacional de Colombia, Bogotá. 48 p. [ Links ]
Cuspoca, J. 2007. Evaluación de virus de tomate de árbol (Solanum betaceum) en plantas indicadoras y su detección por PCR. Trabajo de grado de Ingeniería Agronómica. Facultad de Agronomía. Universidad Nacional de Colombia, Bogotá. 31 p. [ Links ]
De Blas, C., M.J. Borja, M. Saiz and J. Romero. 1994. Broad spectrum detection of Cucumber mosaic virus (CMV) using the polymerase chain reaction. Journal of Phytopathology 141(3): 323-329. [ Links ]
Eagles, R., R. Gardner and R. Forster. 1994. Incidence and distribution of six virus infecting tamarillo (Cyphomandra betacea) in New Zealand. New Zealand Journal of Crop Horticultural Science 22: 453-458. [ Links ]
Eagles, R., R. Gardner and R. Forster. 1990. Nucleotide sequence of the Tamarillo mosaic virus coat protein gene. Journal of Nucleic Acids Research 18(23): 7166. [ Links ]
Eagles, R. 1994. Tamarillo mosaic potyvirus: characterization and resistance. Tesis Doctor of Philosophy. School of Biological Sciences, University of Auckland, Australia. [ Links ]
Fletcher, J. 1987. New plant diseases records in New Zealand: Additional hosts of Alfalfa mosaic virus and Cucumber mosaic virus. New Zealand Journal of Agricultural Research 30(4): 505-506. [ Links ]
Gil, J., M. Quintero, P. González, J. Cotes y M. Marín. 2009a. Detección serológica y molecular de virus en cultivos de papa de tres regiones de Antioquia. En: Memorias XXIX Congreso Asociación Colombiana de Fitopatología ASCOLFI, Medellín, Colombia. 121 p. [ Links ]
Gil, J., M. Ayala, M. Marín y P. González. 2009b. Identificación de potyvirus en cultivos de tomate de árbol (Solanum betaceum cav.) en Antioquia mediante detección serológica. Revista Politécnica 5: 112-120. [ Links ]
Gobernación de Antioquia. 2008. Anuario Estadístico Agropecuario Año 2007: Estadísticas Agropecuarias por consenso. En: Gobernación de Antioquia, http://www.antioquia.gov.co; consulta: junio 2009. [ Links ]
González, P., F. Tenllado, B. Martínez, F. Atencio, D. Barajas, M. Vargas, J. Díaz y J.R. Díaz-Ruíz. 2004. Host-dependent differences during synergistic infection by Potyviruses with Potato virus X. Molecular Plant Pathology 5(1): 29-35. [ Links ]
Gunn, L. and R. Pares. 1990. Capsicum yellows a disease induced by a Luteovirus in glasshouse peppers (Capsicum annuum) in Australia. Journal of Phytopathology 129(3): 210-216. [ Links ]
Guyader, S. and D. Ducray. 2002. Sequence analysis of Potato leafroll virus isolates reveals genetic stability, major evolutionary events and differential selection pressure between overlapping reading frame products. Journal of General Virology 83: 1799-1807. [ Links ]
Hull, R. 2004. Matthew’s plant virology. Cuarta edición. Elsevier Academic Press, USA. 1001 p.
International Committee on Taxonomy of Viruses. 2006. Potato leafroll virus. En: ICTVdB - The Universal Virus Database, http://www.ncbi.nlm.nih.gov/ICTVdb/ICTVdB/; consulta: noviembre 2008. [ Links ]
Maldonado, C. y C. Sánchez. 1984. Caracterización, purificación y serología del virus de la necrosis anular del tomate de árbol (Cyphomandra betacea Sendt). En: Memorias VI Congreso Asociación Colombiana de Fitopatología ASCOLFI, Santa Marta, Colombia. [ Links ]
Matthews, R.E.F. 1993. Diagnosis of plant virus diseases. CRC Press, Boca Raton (USA). 374 p. [ Links ]
Mejía, D., E. Rodas, L. Patiño y E. González. 2009. Efecto del acibenzolar-s-metil sobre el desarrollo de la virosis causada por potyvirus en tomate de árbol. Agronomía Colombiana 27(1): 87-93. [ Links ]
Ministerio de Agricultura y Desarrollo Rural. 2006. Observatorio Agrocadenas Colombia. En: MADR, http://www.agrocadenas.gov.co; consulta: enero 2008. [ Links ]
Mukherjee, K., Y. Verma, S. Chakrabarti, M. Singh and S. Khurana. 2003. Cloning and sequencing of coat protein gene of an Indian Potato leafroll virus (PLRV) isolate and its similarity with other members of Luteoviridae. Virus Genes 26(3): 247-253. [ Links ]
Ochoa, L. y A. Insuasti. 2005. Etiología de las enfermedades virales del tomate de árbol en Ecuador. Instituto Nacional de Investigaciones Agropecuarias, Quito (Ecuador). Informe Técnico Anual - INIAP. Est. Exp. Santa Catalina. Departamento Nacional de Protección Vegetal. Estudios agronómicos, fitopatológicos y entomológicos de frutales nativos andinos. 4 p. [ Links ]
Plchova, H., N. Cerovska, T. Moravec and P. Dedic. 2009. Molecular analysis of Potato leafroll virus isolates from the Czech Republic. Virus Genes 39(1): 153-155. [ Links ]
Rizos, H., L. Gunn, R. Pares and M. Gillings. 1992. Differentiation of Cucumber mosaic virus isolates using the polymerase chain reaction. Journal of General Virology 73: 2099-2103. [ Links ]
Saldarriaga, A. y J.A. Bernal. 1994. Virus en tomate de árbol (Cyphomandra betacea Sendt). En: Memorias XV Congreso Asociación Colombiana de Fitopatología ASCOLFI, Bogotá, Colombia. 5 p. [ Links ]
Saldarriaga, A., J. Bernal y P. Tamayo. 1997. Enfermedades del cultivo de tomate de árbol en Antioquia: Guía de Reconocimiento y Control. Editorial Piloto, Medellín. 43 p. [ Links ]
Saldarriaga, A., J. Bernal y P. Tamayo. 2000. Reconocimiento y manejo de las enfermedades del cultivo del tomate de árbol en Antioquia. Corporación Colombiana de Investigación Agropecuaria (CORPOICA). Instituto Colombiano Agropecuario (ICA), Medellín, Colombia. 44 p. (Boletín de Sanidad Vegetal No. 31). [ Links ]
Sánchez, C. 1982a. Estudios de hospedantes de un nuevo virus en el tomate de árbol (Cythomandra betacea Sendt). pp. 41-42. En: Memorias V Congreso Asociación Colombiana de Fitopatología ASCOLFI; XXII Reunión Anual de la Sociedad Americana de Fitopatología. División Caribe, ASP- CD. Cali, Colombia. [ Links ]
Sánchez, C. 1982b. Presencia de virus en tomate de árbol (Cyphomnadra betacea (Cav) Sendt) en Colombia. p. 3. En: Memorias V Congreso Asociación Colombiana de Fitopatología ASCOLFI; XXII Reunión Anual de la Sociedad Americana de Fitopatología. División Caribe, ASP- CD. Cali, Colombia. [ Links ]
Sañudo, B. y G. Orellana. 1989. Un virus afectando tomate de árbol (Cyphomandra betacea) en valle del Sibundoy, Putumayo. ASCOLFI Informa 15, 24. [ Links ]
Singh, R., J. Kurz, G. Boiteau and G. Bernard. 1995. Detection of Potato leafroll virus in single aphids by the reverse transcription polymerase chain reaction and its potential epidemiological application. Journal of Virological Methods 55(1): 133-143. [ Links ]
Taliansky, M., M. Mayo and H. Barker. 2003. Pathogen profile Potato leafroll virus: a classic pathogen shows some new tricks. Molecular Plant Pathology 4(2): 81-89. [ Links ]
Tamayo, P. 1990. Mosaico del tomate de árbol. ASCOLFI Informa 16: 54-55. [ Links ]
Tamayo, P. 1996. Enfermedades virales del tomate de árbol (Cyphomandra betacea (Cav).Sendt.) en Colombia. ASCOLFI Informa 22: 26-29. [ Links ]
Tamayo, P., J. Zapata y L. Salazar. 1999. El mosaico y la virosis del tomate de árbol en el altiplano norte de Antioquia. Revista Facultad Nacional de Agronomía, Medellín 52(2): 781-785. [ Links ]
Thomas, J. 1984. Characterisation of an Australian isolate of Tomato yellow top virus. Annals of Applied Biology 104(1): 79-86. [ Links ]
Untiveros, M., S. Fuentes y L.F. Salazar. 2007. Synergistic Interaction of Sweet potato chlorotic stunt virus (Crinivirus) with Carla-, Cucumo-, Ipomo-, and Potyviruses Infecting Sweet potato. Plant Disease 91: 669-676. [ Links ]
Vizuete, B., M. Insuasti, J. Ochoa and M. Ellis. 1990. Biological and serological characterization of tree tomato virus diseases in Ecuador. In: Ohio State University, http://www.oired.vt.edu/ipmcrsp/communications/annrepts//Ecuador/ecuador_topic11.pdf. 3 p.; consulta: noviembre 2008. [ Links ]
Wang, Y., K. Lee, V. Gaba, S. Wong, P. Palukaitis and A. Gal-On. 2004. Breakage of resistance to Cucumber mosaic virus by co-infection with Zucchini yellow mosaic virus: enhancement of CMV accumulation independent of symptom expression. Archives in Virology 149(2): 379-396. [ Links ]
Xu, H. and J. Nie. 2006. Identification, characterization, and molecular detection of Alfalfa mosaic virus in potato. Phytopathology 96(11): 1237-1242. [ Links ]
Yu, C., W. Jianxiang y Z. Xueping. 2005. Detection and subgrouping of Cucumber mosaic virus isolates by TAS-ELISA and immunocapture RT-PCR. Journal of Virological Methods 123(2): 155-161. [ Links ]














