Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Facultad Nacional de Agronomía Medellín
versão impressa ISSN 0304-2847
Rev. Fac. Nac. Agron. Medellín v.64 n.2 Medellín jul./dez. 2011
Caracterización Morfológica y Molecular de 18 Introducciones de Uchuva Physalis peruviana L. de la Colección de la Universidad de Nariño
Morphological and Molecular Characterization of 18 Introductions of Cape Gooseberry Physalis peruviana L. collection of the University of Nariño
Anjuly Tatiana Morillo Paz1; Diana Elizabeth Villota Cerón2; Tulio César Lagos Burbano3 y Héctor Ramiro Ordóñez Jurado4
1 Ingeniera Agroforestal. Universidad de Nariño - Facultad de Ciencias Agrícolas. Ciudad Universitaria Torobajo, Calle 18 Carrera 50, San Juan de Pasto, Colombia. <anjulymorillo@gmail.com>
2 Ingeniera Agroforestal. Universidad de Nariño - Facultad de Ciencias Agrícolas. Ciudad Universitaria Torobajo, Calle 18 Carrera 50, San Juan de Pasto, Colombia. <dianitavillota@hotmail.com>
3 Profesor Asociado. Universidad de Nariño - Facultad de Ciencias Agrícolas. Ciudad Universitaria Torobajo, Calle 18 Carrera 50, San Juan de Pasto, Colombia. <tclagosb@udenar.edu.co>
4 Profesor Asociado. Universidad de Nariño - Facultad Ciencias Agrícolas. Ciudad Universitaria Torobajo, Calle 18 Carrera 50, San Juan de Pasto, Colombia. <hectoramiro@hotmail.com>
Recibido: Febrero 11 de 2010; aceptado: Agosto 06 de 2011.
Resumen. Fueron caracterizadas morfológica y molecularmente con marcadores RAM, 18 introducciones de uchuva de la colección de la Universidad de Nariño. Para la caracterización morfológica se sembraron 10 plantas de cada introducción. A través del análisis de componentes principales (ACP) se determinó que el 81,75% de la variabilidad total de la población estudiada, respecto a las variables cuantitativas, está representada por tres componentes principales, definidos por características relacionadas con el fruto y la semilla. En cuanto a las cualitativas, el análisis de correspondencias múltiples (ACM) indica que el 61,51% de la variabilidad total, está explicada por cinco factores, definidos principalmente por variables relacionadas con la flor y el cáliz. El análisis de clasificación (AC) con base en la caracterización morfológica, agrupó las introducciones por sólidos solubles totales y longitud del cáliz. En la caracterización molecular se encontraron 66 locí polimórficos con bandas de 500 a 3.000 pb. El valor de heterocigosidad para las 18 introducciones fue de 0,44. Con base en el criterio de distancia genética de Nei, se encontró la menor distancia genética (0,095) entre UN-34 y Silvania, la mayor distancia genética (1,42) se dio entre el ecotipo Colombia y Perú. Los agrupamientos de la caracterización morfológica y molecular no fueron coincidentes, probablemente debido a que los caracteres fenotípicos y genotípicos estudiados son gobernados por diferentes factores.
Palabras clave: Variabilidad, análisis de componentes principales, análisis de correspondencias múltiples, análisis de clasificación.
Abstract. Eighteen cape gooseberry introductions from the collection of Nariño's University were characterized through morphological and molecular RAM markers. For the morphological characterization were used 10 plants of each entry. Through the principal component analysis (PCA) was found that 81.75 % of the total variability of the study population, regarding quantitative variables, is represented by three main components, defined by characteristics related to fruit and seed. The qualitative multiple correspondence analysis (MCA) indicated that 61,51% of the total variability was explained by five factors, defined mainly by traits related to the flower and calyx. The classification analysis (CA) based on morphological characterization, grouped introductions by total soluble solids and length of calyx. In the molecular characterization 66 polymorphic loci were found with bands of 500 to 3,000 bp. The value of heterozygosity for the 18 introductions was 0.44. Based on the Nei genetic distance criterion, it was found the less genetic distance (0.095) between A-34 and Sylvania, the largest genetic distance (1.42) occurred between Ecotype Colombia and Peru. Clusters of morphological and molecular characterization are mismatched, probably because phenotypical and genotypical characteristics, studied are governed by different factors.
Key words: Variability, principal component analysis, multiple correspondence analysis, classification analysis.
En años recientes, el consumo de la uchuva Physalis peruviana L., se ha incrementado paulatinamente, al igual que la posibilidad de ampliar sus exportaciones como fruta exótica, lo cual ha incentivado su desarrollo como cultivo comercial (Ligarreto et al., 2005), reconociéndose entre los frutales andinos de mayor potencialidad y grado de desarrollo junto con el lulo (Solanum quitoense), el tomate de árbol (Cyphomandra betacea) y la mora (Rubus glaucus) (Lobo, 2006). El departamento de Nariño, cuenta con zonas potenciales para el cultivo de uchuva, junto con los departamentos de Antioquia, Boyacá, Cundinamarca, Cauca, Huila, Magdalena y Tolima; además, Nariño cuenta con una ubicación estratégica con relación a los mercados (Flórez et al., 2000; Miranda, 2005).
El crecimiento de P. peruviana como cultivo enfocado principalmente a la exportación, hace necesario utilizar la variabilidad genética existente en Colombia, principalmente en actividades de selección de mejores materiales o de premejoramiento. Lobo (2006) señala que la utilización plena del potencial genético de cualquier cultivo, incluidos los frutales, depende de la disponibilidad de una base genética amplia para poder aplicar procesos de selección. Así mismo, se relaciona con la existencia de colecciones representativas de la diversidad, así como de un buen conocimiento de los atributos presentes en estas, de lo cual se deriva la importancia de conocer, conservar y manejar los recursos genéticos de especies de gran potencial como P. peruviana, sobre todo en regiones como Nariño.
La Universidad de Nariño cuenta con una colección de 60 introducciones, la mayoría de las cuales fueron colectadas en Nariño y caracterizadas morfológicamente por Lagos et al. (2003). Otras introducciones fueron donadas por la Universidad Nacional de Colombia, Sede Palmira, que posee la mayor colección de Colombia con más de 220 accesiones (Bonilla et al., 2008a). Dicha colección puede ser de gran utilidad en programas de mejoramiento genético y para el desarrollo de estrategias de conservación de la variabilidad genética existente en P. peruviana. Por lo tanto, es necesario conocer la estructura y nivel de variabilidad genética existente entre las distintas introducciones, con el objeto de trazar estrategias de conservación y mejoramiento.
En Colombia, las primeras referencias de trabajos de evaluación y caracterización morfológica de P. peruviana y especies relacionadas corresponden, a los realizados por Pulgarín (1989), donde se realizó una caracterización fenotípica preliminar de 13 materiales de uchuva (P. peruviana L.) procedentes de Ecuador y Colombia. Este autor encontró la mayor variabilidad en la forma y tamaño del cáliz, forma y peso del fruto. Sin embargo, para los 36 descriptores utilizados, 16 no presentaron ninguna variación para el grupo de materiales evaluados, bajo las condiciones en que esta se realizó.
Arbeláez y Mora (1990) llevaron a cabo una caracterización y evaluación fenotípica de 68 materiales del género Physalis, de los 36 descriptores empleados, seis no presentaron variabilidad alguna en las colecciones de las dos especies. Estos descriptores corresponden a características cualitativas poco influenciadas por el ambiente, como antocianina en las venas foliares, pubescencia en el estilo, color de la corola, color de las manchas de la corola, color del fruto inmaduro y forma del cáliz.
En la Universidad Nacional de Colombia, Sede Palmira, Bonilla et al. (2008b) caracterizaron morfológicamente 24 genotipos, los cuales no presentaron un agrupamiento acorde a su lugar de origen. En la caracterizaron molecular se evaluaron 43 genotipos encontrando un total de 44 locus polimórficos y una heterocigosidad de 0,22.
Trillos et al. (2008) caracterizaron morfológicamente 46 accesiones de uchuva provenientes del banco de germoplasma de la nación colombiana, a cargo de Corpoica, en el Centro de Investigación La Selva. Emplearon 69 descriptores, 40 de ellos cualitativos y 29 cuantitativos, 56 de los cuales (81,16%) fueron útiles en la diferenciación de las accesiones. Para las variables cualitativas estimaron los coeficientes de disimilaridad de Gower, que fluctuaron desde 0 a 0,20; y para las variables cuantitativas se estimaron los valores de distancia Euclediana, que fluctuaron entre 0,25 y 1,22.
En el departamento de Nariño se cuenta con los trabajos realizados por Lagos et al. (2003), donde se caracterizaron morfológicamente 50 genotipos y se encontró que el mayor aporte a la variabilidad de la especie se da por las variables relacionadas principalmente con el fruto, de igual manera Lagos (2001) caracterizó molecularmente 19 introducciones de uchuva, y encontró una heterosis de 0,30, y un total de 66 locus polimórficos.
Es necesario continuar con los estudios básicos para establecer la estructura genética de estas poblaciones y conocer el grado de divergencia entre ellas, que permita el desarrollo de nuevas investigaciones sobre esta especie.
Acorde con lo anterior, los objetivos de este trabajo fueron caracterizar morfológica y molecularmente 18 introducciones de uchuva de la colección de la Universidad de Nariño, evaluar la diversidad genética de la población estudiada a través de la técnica RAM (Random Amplified Microsatellites) y determinar la distancia genética entre las diferentes introducciones.
MATERIALES Y MÉTODOS
La investigación se desarrolló en dos fases. La primera correspondió a la caracterización morfológica que se realizó en la Granja Experimental Botana localizada en el Altiplano de Pasto (Nariño) a 2.820 msnm, en las coordenadas 01°09'12'' Latitud Norte y 77°18'31'' Longitud Oeste, con temperatura promedia de 13 °C y 970 horas sol/año, precipitación pluvial promedio de 803 mm/año y humedad relativa del 82% (Lagos et al., 2001). La segunda se llevó a cabo en el Laboratorio de Biología Molecular de la Universidad Nacional de Colombia, Sede Palmira.
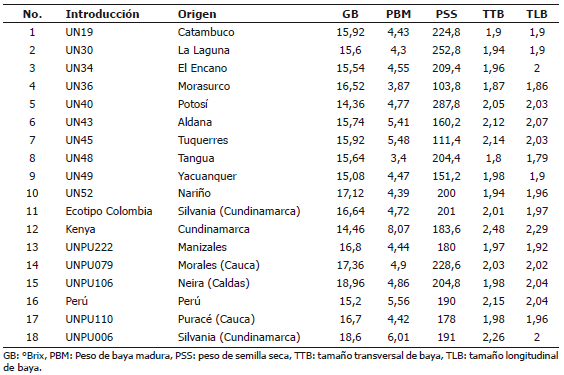
En la Tabla 1 se describen las características más importantes de las 18 introducciones de uchuva, de la colección de la Universidad de Nariño. El 55% de las introducciones provienen del departamento de Nariño, el 16% de Cundinamarca, el 11% de Caldas, el 11% de Cauca y el 5% de Perú. Las introducciones codificadas con UNPU y Perú son donaciones de la Universidad Nacional de Colombia, Sede Palmira, grupo de investigación de Diversidad Biológica, las introducciones restantes fueron caracterizadas por Lagos et al. (2003).
Tabla 1. Origen y características principales de 18 introducciones de Physalis peruviana L. pertenecientes a la colección de la Universidad de Nariño, Colombia.
Caracterización morfológica. Para la caracterización morfológica se sembraron en la Granja Experimental Botana 10 plantas de cada introducción, con distancia entre plantas de 2 m y 2,50 m entre surcos. De las variables evaluadas y propuestas por Lagos et al. (2003) y Bonilla et al. (2008c), se tomaron para este estudio 11 cuantitativas y 8 cualitativas.
Las variables cuantitativas correspondieron a sólidos solubles totales, SST (°Bx), número de semillas por baya (NSB), ancho de hoja (AH), longitud de hoja (LH), diámetro de flor (DIAF), longitud de cáliz acrescente (LCA), ancho de cáliz acrescente (ACA), peso de baya madura (PBM), eje polar de la baya (EPB), diámetro ecuatorial de la baya (DEB) y peso de semilla seca (PSS). Las variables cualitativas correspondieron a color de semilla seca (CSS), prolongación de la mácula (PM), pegajosidad (PG), pubescencia (PB), forma de cáliz (FC), forma de fruto (FF), color de fruto maduro (CFM) y hábito de crecimiento (HC).
Caracterización molecular. Esta fase se desarrolló en tres etapas, que comprendieron la extracción de ADN, el establecimiento de los perfiles térmicos para los marcadores RAM y el tránsito de los amplificados a través de la electroforesis vertical.
Para la extracción de ADN se trabajó con base en la metodología de Dellaporta et al. (1983). La amplificación de los microsatélites RAM vía PCR se realizó con seis primers (Zietkiewicz et al., 1994); Tabla 2, escogidos por manifestar polimorfismo y repetibilidad en especies como Rubus spp. (Morillo et al., 2005), Psidium guajava (Sanabria et al., 2006), Heliconia spp. (Arcos et al., 2004) y P. peruviana (Lagos, 2001 y Bonilla et al., 2008b). Se determinaron las condiciones óptimas de cloruro de magnesio, cebador y buffer Taq polimerasa. Se utilizaron 200 µM de DNTP y 1U de Taq polimerasa (Promega®), en un volumen total de reacción de 12,5 µL.
Tabla 2. Cebadores utilizados en la técnica microsatélites RAM para el estudio de la diversidad genética de 18 introducciones de Physalis peruviana L.
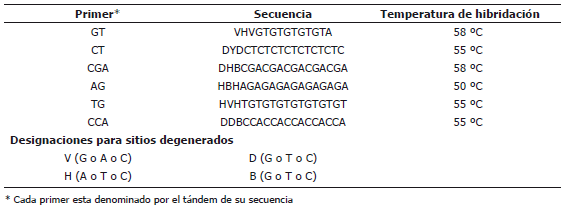
La amplificación se realizó en un termociclador PTC-100 Programmable Termal Controller de MJ Research®. La desnaturalización inicial fue de 95 ºC durante 5 min, 37 ciclos de desnaturalización a 95 ºC por 30 seg. La temperatura de hibridación para los diferentes cebadores fue entre 50 y 58 ºC por 45 seg (Tabla 2), una extensión a 72 ºC por dos min y finalmente 72 ºC durante siete min.
Los amplificados se separaron por electroforesis (1 hora en Tampón TBE 0,5 X a 160 V) en geles de poliacrilamida al 8%, usando sales de plata para la tinción. La visualización de los geles se hizo directamente sobre ellos, utilizando un fondo de luz blanca. El peso de los amplificados, se estimó por comparación con el marcador Lambda de 100 pb (Promega®).
Análisis estadístico. La información de las variables cuantitativas se sometió a un análisis de componentes principales (ACP) y las cualitativas a un análisis de correspondencias múltiples (ACM). Con base en los resultados, se realizó el Análisis de Clasificación Jerárquica teniendo en cuenta los criterios de Ward (Morineau, 1984).
La información proveniente de la caracterización molecular, se organizó en una matriz binaria, donde la presencia de la banda fue uno y la ausencia cero. Con el uso del software para el análisis de datos moleculares TFPGA (Tools for Population Genetic Analysis) (Miller, 1997), se obtuvieron las estadísticas descriptivas de frecuencias alélicas y de heterocigotos, las heterocigosidades observada y esperada, el porcentaje de locus polimórficos. Las distancias genéticas se calcularon mediante las distancias mínimas insesgadas de Nei (1978) y se construyó un dendrograma con base en el método de agrupamiento UPGMA (Unweighted Pair Group Method with Arithmetic Mean), el cual utiliza el promedio aritmético no ponderado.
RESULTADOS Y DISCUSIÓN
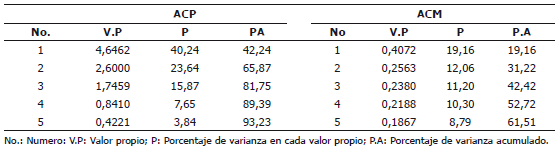
El ACP indica que los tres primeros componentes principales (CP) expresan el 81,75% de la variabilidad total, donde el primer CP explica el 42,24%, el segundo el 23,64% y el tercero el 15,87% de la variación encontrada en la población estudiada (Tabla 3).
Tabla 3. Análisis de componentes principales (ACP) y de correspondencias múltiples (ACM) para variables cuantitativas y cualitativas para 18 introducciones de Physalis peruviana L., pertenecientes a la Universidad de Nariño, Colombia.
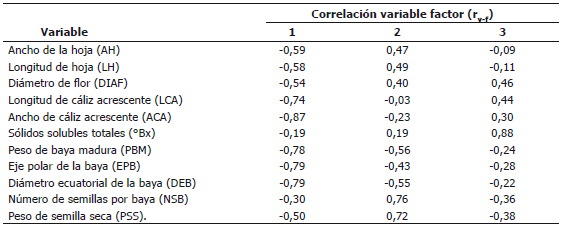
El primer CP está determinado por las variables relacionadas con el fruto como el ancho de cáliz acrescente (ACA), peso de baya madura (PBM), diámetro ecuatorial de la baya (DEB) y eje polar de la baya (EPB), con correlaciones variable factor que van entre 0,79 y 0,87. El segundo CP está determinado por el número de semillas por baya (NSB) y peso de semilla seca (PSS) con valores rv-f de 0,76 y 0,72, respectivamente, y el tercer CP está definido por los °Bx, con un aporte al factor del 0,88 (Tabla 4). Estas variables son de gran importancia en estudios de caracterización de uchuva, debido a la variabilidad que se ha encontrado en este trabajo como en investigaciones similares realizadas por Bonilla et al. (2008c); Lagos et al. (2003); Pulgarín (1989).
Tabla 4. Aporte a la construcción de los tres primeros CP en el ACP de las variables cuantitativas evaluadas en 18 introducciones de Physalis peruviana L., pertenecientes a la Universidad de Nariño, Colombia.
El dendrograma del análisis de clasificación generado a partir del ACP (Figura 1A) de las 18 introducciones de uchuva, muestra cinco grupos a una distancia de siete. En el primer clúster se encuentran agrupadas las introducciones UN-40 y UN-30, que representan el 11,11% del total de las introducciones. Este grupo presenta características de fruto como TLB (1,97 cm), TTB (2 cm), ACA (2,74 cm), PBM (4,54 g), LCA (3,68 cm) y SST (14,98 ºBx) menor respecto al promedio general (1,99 cm, 2,04 cm, 2,82 cm, 4,90 g, 3,93 cm y 16,23 ºBx en su orden), a excepción de NSB (270) por encima del promedio general (192). Las introducciones que forman este grupo, fueron colectadas en el departamento de Nariño en el municipio de Potosí (2.669 msnm) y en el municipio de Pasto, vereda La Laguna (2.795 msnm).
Figura 1. Dendrogramas del análisis de clasificación jerárquica con base en el ACP (A) y en el ACM (B) de 18 introducciones de Physalis peruviana L., pertenecientes a la Universidad de Nariño, Colombia.
El segundo grupo está conformado por nueve introducciones (UN-49, UN-19, UN-34, Manizales, Perú, UN-52, Puracé, UN-43 y UN-45) que representan el 50% de la población estudiada. En este clúster, los valores de los descriptores evaluados como SST (17,89 ºBx), DIAF (2,08 cm), LCA (4,35 cm), ACA (3,10 cm), PSS (0,22 g), PBM (5,13 gr), TTB (2,08 cm) y TLB (2,01 cm) presentaron valores superiores al promedio general (16,23 ºBx, 1,95 cm, 3,96 cm, 2,82 cm, 0,20, 4,90 g, 2,04 cm y 1,99 cm, en su orden). La mayoría de las introducciones dentro del grupo fueron colectadas en el departamento de Nariño entre los 1.900 y 3.100 msnm.
A la distancia de siete, Kenya no se agrupó con ninguna introducción; sin embargo, se encuentra cercana al cuarto y quinto clúster, agrupándose con estos a una distancia de 26,46. Esta introducción presenta un PBM de 8,07 g superior al promedio general (4,90 g) y con un menor valor para SST (14,46 °Bx) respecto al promedio general (16,23 °Bx). Esta población se introdujo a Colombia desde África en 1988 por Fischer et al. (2005) y proviene del departamento de Cundinamarca.
El cuarto clúster está formado por cuatro introducciones (Morales, Neira, Colombia y Silvania) que representan el 22% de las poblaciones caracterizadas, las cuales presentan características por encima del promedio general. Las variables que agruparon estas introducciones fueron los SST, los DIAF y la LCA, siendo los SST (17,89 ºBx) superiores al promedio general (16,23 ºBx), dentro de este grupo se encuentran introducciones cultivadas en los departamentos de Caldas, Cauca y Cundinamarca.
El quinto clúster está formado por las introducciones UN-48 y UN-36, las cuales se agruparon principalmente por su LH (7,21cm) menor al del promedio general (8,54 cm), SST (16,08 ºBx) menor al promedio general (16,23 ºBx), NSB (154) menor al promedio general (192), PBM (3,64 g) menor al promedio general (4,90 g), LCA (3,46 cm) menor que el promedio general (3,96 cm) y ACA (2,45 cm) menor que el promedio general (2,82 cm). Estas introducciones fueron colectadas en el departamento de Nariño, en los municipios de Tangua (2.400 msnm) y Morasurco (2.800 msnm).
En el ACM los cinco primeros ejes explican el 61,51%, de la variabilidad total. A esta, los tres primeros valores propios le aportan el 19,16, el 12,02 y el 11,2%, en su orden (Tabla 3). El primer eje está definido por la PM y la FC con un aporte (Av-f) a la construcción del eje del 18,0 y 16,6%, respectivamente. La modalidad de mayor importancia para PM fue la presencia (Av-f = 12%) y para FC, el ligulado (Av-f = 8,2%).
El segundo eje está definido por la PG (Av-f = 23,6%) y el CSS (Av-f = 29,9%). La modalidad de mayor importancia para la PG fue intermedia (Av-f = 15,7%) y para CSS fue crema oscuro (Av-f = 9,7%). El tercer eje está conformado por el CSS (Av-f = 36,2%) y CFM (Av-f = 21,4%). Para estas variables las modalidades de mayor aporte fueron CSS ocre claro y CFM amarillo - naranja, con aportes del 17,6 y 10,1%, respectivamente.
En el análisis de clasificación con base en el ACM de las 18 introducciones de uchuva se encontraron cuatro clúster formados a una distancia de 9, siendo los más importantes el primer y cuarto que agrupan al 44 y 28% de la población, en su orden. El primer clúster lo conforman las introducciones UN-48, UN-40, UN-19, UN-43, Perú, UN-45, UN-49 y Kenya. Las variables que agruparon estas introducciones fueron FC urceolado y PB abundante (Figura 2).
Figura 2. Representación gráfica en un plano factorial del aporte de las variables cualitativas a los factores 1 y 2 en el ACM de 18 introducciones de Physalis peruviana L., pertenecientes a la Universidad de Nariño, Colombia.
El segundo clúster está formado por la introducción Manizales, a la distancia de 9 no se agrupó con ninguna introducción; sin embargo se encuentra cercano al primer y tercer clúster, agrupándose a una distancia de 10,07. Esta introducción se caracteriza por presentar color de semilla crema oscuro.
El tercer clúster lo componen las introducciones UN-34, Silvania, Morales y UN-52, que corresponde al 22,22%, agrupadas por la PG abundante, la FC ligulado. Finalmente el cuarto clúster lo forman las introducciones Neira, UN-36, UN-30, Colombia y Purace, agrupadas principalmente por la FF ovoide.
Respecto a la caracterización molecular, los cebadores RAM utilizados en la caracterización molecular generaron un total de 1188 registros de presencia y ausencia de bandas, de los cuales el 71,29% corresponden a presencia y el 28,70% a ausencia. Se identificaron 66 locus con bandas que van desde 500 a 3.000 pb. Al respecto, Bonilla et al. (2008b) en un estudio similar, encontraron 42 locus polimórficos en 43 genotipos evaluados y Lagos (2001) encontró un total de 66 locus polimórficos en 19 accesiones evaluadas.
Los marcadores RAM utilizados en este trabajo, fueron polimórficos en un 98,48% (P<0,01). AG, CCA, GT, TG y CT (Figura 3) presentaron el 100% de locus polimórficos. De igual manera, se mostraron altos valores de heterocigosidad insesgada (He), con un valor mínimo de 0,36 para el primer CGA y un valor máximo de 0,47 y 0,46 para los cebadores GT y AG, respectivamente. Estos dos últimos cebadores pueden ser útiles para obtener una mayor discriminación entre genotipos de uchuva, por presentar mayores valores de He.
Figura 3. Marcadores RAM obtenidos con el cebador CCA en las introducciones de Physalis peruviana L., pertenecientes a la Universidad de Nariño, Colombia, evaluadas en gel de poliacrilamida.
La He para las 18 introducciones fue de 0,44, siendo mayor a la mencionada por Lagos (2001) y Bonilla et al. (2008b) que fueron de 0,3 y 0,2, en su orden. Esto se debe a que en las introducciones evaluadas existe mayor variabilidad en las procedencias y las pertenecientes al departamento de Nariño son poblaciones con una menor presión de selección por encontrarse en estado semidomesticado.
Berg y Hamrick (1997) indican que las frecuencias alélicas son el parámetro genético básico de una población, puesto que introducciones con diferentes frecuencias genotípicas pueden tener frecuencias alélicas idénticas; además, la evolución se ha definido como un cambio en las frecuencias alélicas a través del tiempo, lo cual conlleva a una mayor o menor distancia genética entre dos poblaciones.
La menor distancia genética de Nei (1978) se encontró entre las introducciones UN-34 Encano (Nariño) y Silvania (Cundinamarca), con un valor de 0,095. La similitud genética entre dos introducciones se debe al movimiento de germoplasma dado entre las diferentes regiones por acciones antrópicas.
La mayor distancia genética (1,42) se dio entre el ecotipo Colombia y Perú. Esta separación genética se explica por el amplio distanciamiento geográfico entre los sitios de origen de las dos introducciones, lo cual limita considerablemente el flujo de genes. Este distanciamiento genético puede ser útil en trabajos de mejoramiento, debido a que pueden generar una amplia variabilidad genética. El agrupamiento de las 18 introducciones de P. peruviana de manera general se muestra en el dendograma generado por el método UPGMA (Figura 4).
Figura 4. Dendrograma de la estructura genética de 18 introducciones de Physalis peruviana L., pertenecientes a la Universidad de Nariño, Colombia, basado en el coeficiente de similaridad de Nei y calculado de los datos combinados de los 6 cebadores microsatélites RAM con el método de clasificación UPGMA.
En el dendrograma (Figura 4) se identifican cuatro grupos (A, B, C y D) a un nivel de similaridad superior al 50%; las introducciones evaluadas no se agruparon de acuerdo al origen geográfico, a excepción de los materiales colectados en los departamentos de Caldas y Cauca.
En las cuatro agrupaciones del dendrograma están presentes introducciones provenientes de Nariño, demostrando que la mayor parte de la variabilidad se encuentra presente en este departamento; además, el nivel de alogamia de la especie y el uso que el agricultor le da como especie semidomesticada, son fenómenos que aportan a la variación.
El grupo B se encuentra conformado por introducciones provenientes de Nariño (UN-30, UN-19 y UN-48) y la introducción Kenya, aunque se presentaron diferencias a nivel morfológico, estos materiales se agruparon de acuerdo a características genotípicas, debido a que los marcadores moleculares, en general, son neutros con relación a los efectos fenotípicos y contienen mayor información genética por locus que los marcadores morfológicos (Ferreira y Grattapaglia, 1998).
El grupo D está formado por la introducción proveniente del departamento de Nariño UN-36 y la introducción Perú, las cuales muestran un grado de similitud mayor del 75%. Este valor fue el menor distanciamiento encontrado entre los cuatro grupos. Esto se debe al flujo de genes que existe, desde los Andes peruanos extendiéndose por toda la cadena montañosa suramericana que atraviesa Colombia y va hasta Venezuela.
Los agrupamientos de la caracterización morfológica y molecular no son coincidentes, dado que los caracteres fenotípicos están influenciados por el ambiente y algunos de ellos no son heredables y los caracteres genotípicos pueden no encontrarse expresados en el fenotipo (Lobo, 2006).
Las introducciones UN-34 y UN-52 provenientes de Nariño, se encuentran altamente relacionadas dentro de la colección debido a que se agruparon en el ACP, ACM y el dendrograma de estructura genética. Por otra parte, la introducción Kenya se encuentra agrupada en el ACM y en clasificación generada por los marcadores RAM con las poblaciones UN-19 y UN-48, provenientes de Catambuco y Tangua; sin embargo, se encuentran en grupos distantes en el ACP.
Lo anterior, refleja la estrecha relación existente en las características morfológicas cualitativas y las moleculares para las introducciones en mención, ya que las variables cualitativas son las de mayor heredabilidad (Lobo, 2006). El agrupamiento dado por el ACM y el dendrograma de estructura genética, también es coincidente para las introducciones Silvania, Morales y UN-34, provenientes de Cundinamarca, Cauca y Nariño, contrario a lo informado por Trillos et al. (2008), quienes no obtuvieron concordancia entre los dendogramas obtenidos de descriptores cualitativos y cuantitativos.
Ferreira y Grattapaglia (1998) afirman que como resultado complementario a la caracterización morfológica, existen estudios de valor agregado que permiten perfeccionar el conocimiento de los recursos genéticos y promueven su utilización, como los marcadores moleculares.
CONCLUSIONES
Los caracteres de mayor aporte a la variabilidad de P. peruviana son los relacionados con el fruto (forma del cáliz, peso de baya madura, ancho de cáliz acrescente), siendo útiles en estudios de variabilidad de la especie.
Los valores de polimorfismo y heterocigosidad insesgada, en las 18 introducciones de P. peruviana de la colección de la Universidad de Nariño fueron altos, especialmente los mostrados por los marcadores moleculares GT y AG.
La mayor distancia genética se presentó entre el ecotipo Colombia y Perú, siendo estas introducciones útiles para programas de fitomejoramiento, debido a su amplia variabilidad genética.
Los análisis de clasificación de la caracterización molecular y morfológica no permitieron establecer grupos por origen geográfico, evidenciándose un flujo genético entre las introducciones evaluadas.
BIBLIOGRAFÍA
Arbeláez, C. y M. Mora. 1990. Caracterización fenotípica de uchuva (Physalis sp.). Trabajo de grado Ingeniería Agronómica. Facultad de Ciencias Agropecuarias. Universidad Nacional de Colombia. Medellín. 69 p. [ Links ]
Arcos, A., A. Mondragón, J. Muñoz y S. Botero. 2004. Colecta y caracterización molecular con marcadores tipo RAM (Microsatélites aleatorios) de heliconias y especies relacionadas. pp. 346-;347. En: Memorias. II Congreso Colombiano de Botánica: Botánica Diversidad y Cultura. Universidad del Cauca, Popayán. [ Links ]
Berg, E. and J. Hamrick. 1997. Quantification of genetic diversity at allozyme loci. Canadian Journal of Forest Research 27:415-424. [ Links ]
Bonilla, M., K. Espinosa, A. Posso, H. Vásquez y J. Muñoz. 2008a. Establecimiento de una colección de trabajo de uchuva del suroccidente colombiano. Acta Agronómica (Palmira) 57(2): 95-99. [ Links ]
Bonilla, M., K. Espinosa, A. Posso, H. Vásquez y J. Muñoz. 2008b. Caracterización molecular de 43 accesiones de uchuva de seis departamentos de Colombia. Acta Agronómica (Palmira) 57(2): 109-115. [ Links ]
Bonilla, M., K. Espinosa, A. Posso, H. Vásquez y J. Muñoz. 2008c. Caracterización morfológica de 24 accesiones de uchuva del banco de germoplasma de la Universidad Nacional de Colombia, Sede Palmira. Acta Agronómica (Palmira) 57(2): 101-108 [ Links ]
Dellaporta S., J. Wood and J. Hicks. 1983. A plant DNA minipreparation: Versión II. Plant Molecular Biology Reporter 1(4): 19-;21. [ Links ]
Ferreira, M. y D. Grattapaglia. 1998. Introducción al uso de marcadores moleculares en el análisis genético. EMBRAPA- Cenargem, Brasilia D.F. 221 p. [ Links ]
Flórez, V.J., G. Fischer y A.D. Sora. 2000. Producción, poscosecha y exportación de la uchuva Physalis peruviana L. Universidad Nacional de Colombia, Bogotá. 175 p. [ Links ]
Fischer, G., D. Miranda, W. Piedrahita y J. Romero. 2005. Avances en cultivo, poscosecha y exportación de la uchuva Physalis peruviana L. en Colombia. Universidad Nacional de Colombia, Bogotá. 221 p. [ Links ]
Lagos, T., H. Criollo y C. Mosquera. 2001. Evaluación preliminar de cultivares de uvilla o uchuva (Physalis peruviana L.) para escoger materiales con base en la calidad del fruto. Revista de Ciencias Agrícolas 18(2): 82-94. [ Links ]
Lagos, T. 2001. Diversidad genética de 19 accesiones de Physalis peruviana L., de la colección de la Universidad de Nariño. Trabajo de ascenso a Profesor Asociado. Facultad de Ciencias Agrícolas. Universidad de Nariño, Pasto. 45 p. [ Links ]
Lagos, T., H. Criollo, A. Ibarra y H. Hejeile 2003. Caracterización morfológica de la colección Nariño de uvilla o uchuva Physalis peruviana L. Fitotecnia Colombiana 3(2): 1-9. [ Links ]
Ligarreto, G., M. Lobo y A. Correa. 2005. Recursos genéticos de la uchuva en Colombia. pp. 9-26. En: Fischer, G., D. Miranda, W. Piedrahita y J. Romero (eds). Avances en el cultivo, poscosecha y exportación de uchuva P. peruviana L. en Colombia. Universidad Nacional de Colombia, Unibiblos, Bogotá. 221p. [ Links ]
Lobo, M. 2006. Recursos genéticos y mejoramiento de frutales andinos: una visión conceptual. Revista Corpoica -; Ciencia y Tecnología Agropecuaria 7(2): 40-54 [ Links ]
Miller, M. 1997. Tools for population genetic analysis (TFPGA) 1.3: A Windows program for the analysis of allozyme and molecular population genetic data, http://herb.bio.nau.edu/-miller; consulta: enero 2010. [ Links ]
Miranda, D. 2005. Criterios para el establecimiento, los sistemas de cultivo, el tutorado y la poda de la uchuva pp. 29-;54. En: Fischer, G., D. Miranda, W. Piedrahita y J. Romero (eds.). Avances en cultivo, poscosecha y exportación de la uchuva (Physalis peruviana L.) en Colombia. Universidad Nacional de Colombia, Unibiblos, Bogotá. 221 p. [ Links ]
Morillo, A., Y. Morillo, A. Zamorano, H. Vásquez y J. Muñoz. 2005. Caracterización molecular con microsatélites aleatorios RAM de la colección de mora Rubus spp. de la Universidad Nacional de Colombia, Sede Palmira. Acta Agronómica (Palmira) 54(2): 15-24. [ Links ]
Morineau, A. 1984. Note sur la caractérisation statistique d'une classe et les valeurs-test. Bulletin Technique du Centre de Statistique et d'Informatique Appliquées 2(1-2): 20-27. [ Links ]
Nei, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 89: 583-590. [ Links ]
Pulgarín, O. 1989. Caracterización fenotípica preliminar de 13 colecciones de uchuva (Physalis peruviana L.). Trabajo de grado de Biología. Departamento de Biología. Universidad de Antioquia, Medellín. 136 p. [ Links ]
Sanabria, O., M. García, H. Díaz, J. Muñoz. 2006. Caracterización molecular con marcadores RAM de árboles nativos de Psidium guajava (guayaba) en el Valle del Cauca. Acta Agronómica (Palmira) 55(1): 23-30. [ Links ]
Trillos, O., J. Cotes, C. Medina, M. Lobo y A. Navas. 2008. Caracterización morfológica de cuarenta y seis accesiones de uchuva (Physalis peruviana L.), en Antioquia (Colombia). Revista Brasileira de Fruticultura 30(3): 708-715. [ Links ]
Zietkiewicz, E., Rafalski, A. y Labuda, D. 1994. Genome fingerprinting by Simple Sequence Repeat (SSR) - Anchored Polymerase Chain Reaction Amplification. Genomics 20: 176-183. [ Links ]














