Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Actualidades Biológicas
Print version ISSN 0304-3584
Actu Biol vol.36 no.101 Medellín July/Dec. 2014
ARTÍCULOS DE INVESTIGACIÓN
Estimación de heredabilidad y correlaciones genéticas en caracteres morfológicos y fisiológicos para una población de Zamia obliqua A.Br. (Zamiaceae: Cycadales)
Estimation of heritability and genetic correlations in morphological and physiological traits for a population of Zamia obliqua A.Br. (Zamiaceae: Cycadales)
Natalia Gómez-Lopera1, Cristina López-Gallego2
1 Instituto de Biología, Universidad de Antioquia. A. A. 1226. Medellín (Antioquia), Colombia. autor para correspondencia: natalia.gomezl@udea.edu.co
2 Docente, Instituto de Biología, Universidad de Antioquia. A. A. 1226. Medellín (Antioquia), Colombia. mariac.lopez@udea.edu.co.
Recibido: enero 2014; aceptado: junio 2014.
Resumen
La respuesta a la selección natural en caracteres cuantitativos en una población natural depende de la magnitud de variabilidad genética y las correlaciones genéticas en los rasgos, y estos parámetros genéticos pueden diferir entre categorías de rasgos. En este estudio se caracterizaron los patrones de varianza y covarianza fenotípica y se realizaron estimaciones de heredabilidad (h2) y correlaciones genéticas de rasgos morfológicos y fisiológicos en una población de Zamia obliqua (Zamiaceae: Cycadales). Se probaron las hipótesis de que la varianza y la heredabilidad de rasgos morfológicos son mayores que las de rasgos fisiológicos, y que las correlaciones fenotípicas y genéticas son mayores dentro que entre las categorías de rasgos. Los valores de varianza fenotípica fueron mayores en los rasgos fisiológicos comparados con los caracteres morfológicos. Los estimativos de heredabilidad sugieren que los caracteres morfológicos presentan mayor varianza genética que los rasgos fisiológicos. Por otro lado, no se obtuvieron correlaciones genéticas significativas entre los rasgos. Sin embargo, las correlaciones fenotípicas muestran mayor correlación dentro de los caracteres morfológicos que dentro de los fisiológicos o entre rasgos morfológicos y fisiológicos. Estas estimaciones de parámetros genéticos permiten construir hipótesis sobre la evolución de caracteres fenotípicos en poblaciones naturales, y son aportes importantes al estudio de la ecología evolutiva de especies no modelo y sus poblaciones en hábitats naturales.
Palabras claves: correlaciones fenotípicas, parentesco, similitud fenotípica, variabilidad fenotípica, Zamiaceae.
Abstract
The response to natural selection of quantitative traits in a natural population depends on the magnitude of genetic variability and genetic correlations between traits, and these genetic parameters can differ between trait categories. In this study we characterized the patterns of phenotypic variance and covariance and estimated heritability and genetic correlation of morphological and physiological traits in a population of Zamia oblique (Zamiaceae: Cycadales). We tested the hypothesis that variance and heritability values are higher for morphological than for physiological traits, and that phenotypic and genetic correlations are larger within than between trait categories. Phenotypic variance values were higher for physiological traits than for morphological characters. Heritability estimates suggested that morphological traits had a higher genetic variance than physiological traits. On the other hand, significant estimates of genetic correlations among traits were not obtained. Nevertheless, phenotypic correlations show a higher correlation within morphological traits than within physiological traits or among physiological and morphological traits. These kinds of estimates for genetic parameters can help generate hypotheses about the evolution of phenotypic traits in natural populations, and represent important contributions to the study of evolutionary ecology in non-model species and their populations in natural habitats.
Key words: phenotypic correlations, relatedness, phenotypic similarity, phenotypic variability, Zamiaceae.
INTRODUCCIÓN
Un debate importante sobre la evolucion de caracteres cuantitativos es la cantidad de variacion mantenida en poblaciones naturales para diferentes tipos de rasgos. Se ha propuesto que existe un patron de jerarquizacion en los valores de heredabilidad (h2) entre categorias de rasgos de la siguiente manera: morfologia > fisiologia > comportamiento > historias de vida (Roff 1997). Este patron es consistente con la hipotesis de que para rasgos expuestos a una seleccion natural direccional fuerte y cercanamente relacionados a la adecuacion biologica, la varianza genetica aditiva es baja en las poblaciones debido a que se han fijado alelos en el tiempo, en contraste con los rasgos expuestos a una seleccion debil que presentan altos niveles de variacion genetica aditiva (Teplitsky et al. 2009). Una segunda hipotesis para explicar este patron es la plasticidad fenotipica alta de algunos caracteres, como la de los fisiologicos. Esto se debe a que la heredabilidad es inversamente relacionada a la magnitud de los efectos ambientales sobre el rasgo, entonces, rasgos altamente plasticos como aquellos a nivel de la fisiologia de la hoja presentan menor heredabilidad (Geber y Griffen 2003).
Otro aspecto importante a considerar en la evolucion de caracteres cuantitativos es que pueden existir complejos de rasgos ligados por correlaciones geneticas. En los escenarios en que la integracion entre caracteres es beneficiosa la seleccion deberia aumentar la correlacion genetica entre rasgos funcionalmente relacionados, lo que lleva a la evolucion de complejos estructurales de caracteres integrados (McGlothlin et al. 2005). Si esto se cumple, la correlacion genetica seria mas fuerte entre rasgos funcionalmente relacionados que entre rasgos independientes. Por ejemplo, la correlacion genetica entre caracteres vegetativos y florales suele ser menor que dentro de la categoria de rasgos florales o dentro de los propios vegetativos (Gomez et al. 2009).
Las estimaciones de heredabilidad y correlaciones geneticas son entonces fundamentales para entender los cambios fenotipicos en las poblaciones en respuesta a la seleccion natural y otras fuerzas evolutivas. Estos dos parametros clasicamente se estiman a partir del grado de semejanza fenotipica entre parientes en una poblacion. Este estudio es relativamente sencillo en poblaciones artificiales de individuos en cautiverio con una genealogia conocida y bajo condiciones del ambiente controladas. Pero en poblaciones naturales, y mas aun en especies de larga vida, la aplicacion de metodos clasicos de la genetica cuantitativa se vuelve impractica, pues es dificil establecer las relaciones de parentesco entre individuos de manera precisa (Ritland 2000). Ademas, muchos organismos no se reproducen regularmente y el fenotipo de interes puede tardar mucho tiempo en desarrollarse (Herrera y Bazaga 2009). Sin embargo, la mayor disponibilidad de marcadores geneticos para especies no modelo ha permitido que en los ultimos anos se desarrollen metodos alternativos que puedan ser aplicados al analisis de la variacion genetica de rasgos cuantitativos de poblaciones silvestres (Garant y Kruuk 2005).
Uno de los principales metodos propuesto para estimar parametros de genetica cuantitativa en poblaciones naturales ha sido desarrollado por Ritland (1996, 2000). Este metodo propone el uso de marcadores moleculares para estimar el parentesco entre individuos y correlacionarlo con la similitud fenotipica y asi determinar la heredabilidad y correlaciones geneticas de los rasgos de interes. Esta aproximacion depende de la eficiencia de los estimadores de parentesco y de una varianza real de la relacion genetica entre los individuos de una poblacion. El metodo de Ritland se ha usado en varios estudios para estimar heredabilidad de rasgos cuantitativos en poblaciones naturales obteniendo resultados positivos. Por ejemplo, Andrew et al. (2005) encontraron una heredabilidad significativa para la sintesis de los compuestos sideroxynal, cineol y el contenido de nitrogeno, responsables de la defensa foliar de la planta Eucalyptus melliodora A.Cunn. ex R.T.Baker & H.G.Sm. Mas recientemente, Castellanos et al. (2011) encuentran heredabilidades significativas para categorias de rasgos florares y vegetativos, y concluyen que el metodo de Ritland es una buena opcion para estimar este parametro cuantitativo en poblaciones de plantas de larga vida.
En nuestro estudio, se comparo la variabilidad genetica de categorias de rasgos morfologicos y fisiologicos en una poblacion de la especie de larga vida Zamia obliqua A.Br. Para estimar la heredabilidad de cada rasgo y potenciales correlaciones geneticas entre los caracteres se uso el metodo de Ritland (1996, 2000). La hipotesis con respecto a la heredabilidad fue que los rasgos morfologicos tienen mayor heredabilidad que los fisiologicos. Por otro lado, la hipotesis con respecto a las correlaciones geneticas fue que dentro del grupo de rasgos morfologicos y dentro del grupo de rasgos fisiologicos las correlaciones geneticas son mayores que las correlaciones entre rasgos morfologicos y fisiologicos. Para probar estas hipotesis se hizo una caracterizacion de la variacion fenotipica de cuatro caracteres morfologicos y cuatro fisiologicos, se estimo la magnitud de la heredabilidad para los rasgos y se establecio potenciales correlaciones a nivel fenotipico y genotipico dentro y entre categorias de caracteres.
MATERIALES Y METODOS
Sistema de estudio. Zamia obliqua A.Br. (Zamiaceae: Cycadales) es una especie de gimnosperma tropical que se encuentra en bosques humedos tropicales primarios y secundarios del sur de Panama y las regiones del Darien y el Choco biogeografico de Colombia, desde 0 hasta 500 m de altitud (figura 1). Esta es una gimnosperma arborescente con un tallo entre 0,5-5 m de alto y de 5-12 cm de diametro que tiene de 5 a 20 hojas compuestas con 10-40 foliolos. Son plantas dioicas en las cuales los conos masculinos producen abundante polen mientras que los femeninos producen hasta 200 semillas (Stevenson 2004). Los eventos reproductivos son anuales y sincronicos, pero cada ano solo un pequeno porcentaje de individuos de la poblacion produce organos reproductivos. La polinizacion es llevada a cabo por coleopteros curculionidos, pero se desconoce la capacidad de dispersion del polen por estos insectos. El desarrollo de las semillas tiene una duracion de varios meses y la mayoria de estas tienen una dispersion restringida al ser movidas por gravedad cuando las partes del cono empiezan a descomponerse (Norstog y Nicholls 1997).
En este estudio se examinaron rasgos fenotipicos y genotipicos de 181 individuos correspondientes a casi la totalidad de plantas de una poblacion de Z. obliqua. La poblacion (parche de individuos aislada por al menos un kilometro de otros parches) esta compuesta por adultos y juveniles que se encuentran distribuidos en un area de una hectarea en el sotobosque de un bosque humedo tropical, lo cual es tipico de las poblaciones de esta especie en el Choco biogeografico. La poblacion esta localizada en la Estacion Biologica ''El Amargal'', en el corregimiento de Arusi del municipio de Nuqui en la region de Cabo corrientes del departamento del Choco (Colombia).
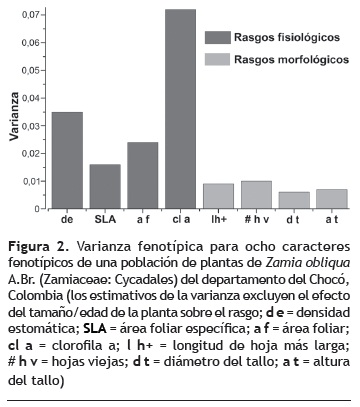
Caracterizacion fenotipica de rasgos morfologicos y fisiologicos. Se midieron fenotipos cuantitativos de categorias de rasgos morfologicos y fisiologicos. Los caracteres morfologicos fueron: diametro del tallo en la base (d t), altura total del tallo (a t), numero de hojas viejas (# h v) y longitud de la hoja mas larga (l h+). Los caracteres fisiologicos fueron: densidad estomatica (d e), area foliar (a f), area foliar especifica (SLA) y cantidad de clorofila a (cl a). Los rasgos morfologicos y fisiologicos de interes pueden cambiar de magnitud con el tamano/edad de la planta, por lo cual para cada individuo se registro el numero de foliolos de la hoja mas larga para usar esta medida como una covariable en los analisis de variabilidad de los caracteres. El numero de foliolos aumenta progresivamente con el tamano/edad de las plantas de Zamia y esta variable ha sido usada ampliamente en estudios de ecologia de cycadales para dar cuenta de las diferencias entre individuos debidas al tamano (Norstog y Nicholls 1997).
La caracterizacion de los rasgos morfologicos se realizo en el campo con la ayuda de una cinta metrica. Para la evaluacion de los rasgos fisiologicos se tomaron cuatro foliolos (partes de la hoja compuesta) representativos de cada uno de los individuos para ser analizados posteriormente en el laboratorio. Del enves de cada foliolo se determino la densidad estomatica por la tecnica de improntas con esmalte transparente. Por otro lado, se usaron secciones de foliolos de 0,1 g para cuantificar la cantidad de clorofila a con una medicion espectrofotometrica a longitudes de onda de 652 nm. Para establecer el area foliar y el area foliar especifica se midio el area de cada foliolo usando fotografias digitales que fueron analizadas en el software Image J (Schneider et al. 2012). Posteriormente, se secaron los foliolos en un horno a 80 °C por 72 horas y se obtuvo el peso seco en una balanza analitica. El area foliar especifica de cada foliolo fue calculada como la relacion entre el area y peso seco del foliolo.
Para comparar la variabilidad a nivel fenotipico en la poblacion entre los rasgos morfologicos y fisiologicos se uso la varianza para cada rasgo, despues de estandarizar los valores de los diferentes caracteres (con medias iguales a cero) para evitar sesgos por las escalas de medicion. Para determinar si existian correlaciones fenotipicas entre rasgos se realizaron correlaciones lineares de Spearman con los registros originales de cada individuo. Sin embargo, varios de los rasgos morfologicos y fisiologicos presentan una relacion significativa con el tamano de la planta estimado segun el numero de foliolos (tabla 1). Por esto, fue necesario obtener los residuales de la regresion entre cada caracter y el tamano de la planta para realizar los analisis de la variacion fenotipica y las correlaciones fenotipicas excluyendo el efecto del tamano de la planta y de manera estandarizada. Los analisis estadisticos se llevaron a cabo en el programa SPSS version 17 (SPSS Inc. 2008).
Caracterizacion genotipica de los individuos. Para realizar las estimaciones de heredabilidad y correlaciones geneticas de los caracteres cuantitativos es necesario determinar el grado de parentesco entre individuos dentro de la poblacion, para lo cual usamos marcadores moleculares microsatelites o STRs. Primero, se extrajo el ADN de foliolos secos utilizando el metodo de CTAB (Doyle y Doyle 1990). Posteriormente se analizaron seis marcadores polimorficos STRs para la especie Zamia fairchildiana L.D. Gomez, dado que no existen marcadores moleculares desarrollados para Z. obliqua (Lopez-Gallego y O'Neil 2009). De los seis STRs probados usamos los cuatro marcadores que amplificaron exitosamente en Z. obliqua y que fueron polimorficos en la poblacion de estudio.
Con los marcadores seleccionados, se amplificaron 4 loci para los 181 individuos muestreados en la poblacion. Las reacciones de amplificacion se prepararon en un volumen final de 15 µl teniendo en cuenta unas concentraciones finales: buffer (NH4) 1X, dNTPs 0,2 mM, primers F y R 0,16 µM (primer R marcados con fluoroforos), Taq polimerasa 0,5 U, DNA 30 ng, MgCl2 entre 1,5 mM y 2,5 mM. Las diferentes soluciones se incubaron a 5 min a 94 °C, seguido por 32 ciclos de 30 s de desnaturalizacion a 94 °C, 30 s de alineamiento (la temperatura varia de acuerdo al STR genotipificado), 30 s de extension a 72 oC y 10 minutos adicionales de extension a 72 oC al final de los 32 ciclos. Una vez se comprobo que las muestras habian amplificado a traves de electroforesis en agarosa; se realizo la tipificacion de los marcadores en un analizador genetico automatico ABI PRISM 3130 mediante la aplicacion del software GENEMAPPER (Applied Biosystems).
Con los genotipos de la poblacion se determino el grado de parentesco entre individuos para usarse en las estimaciones de parametros de genetica cuantitativa. Antes de estimar el grado de parentesco entre los individuos, se hicieron varios analisis con los genotipos de todos los loci para determinar si eran adecuados para este tipo de estimacion. Primero se analizaron los loci con el programa Micro-Checker version 2.2.3 (Oosterhout et al. 2004) para calcular la probabilidad de alelos nulos. Se obtuvieron tambien las frecuencias alelicas y genotipicas observadas y se probo la hipotesis de equilibrio de Hardy-Weinberg, el desequilibrio de ligamento entre los loci y se estimo la diversidad genetica en la poblacion con el programa Arlequin v.3.01 (Excoffier y Lischer 2010).
Estimativos de parentesco entre individuos. Varios estimadores de parentesco han sido desarrollados, entre los cuales se encuentran los metodos de Queller y Goodnight (1989), Ritland (1996), Lynch y Ritland (1999) y Wang (2002); y su efectividad depende de la poblacion de estudio. Para obtener estimativos razonables del coeficiente de parentesco (r), cuando los loci exhiben codominancia son necesarios muchos loci bialelicos o relativamente pocos loci variables (Hedrick 2005). Se uso una simulacion Monte-Carlo implementada en el programa Mark version 3.1 (Ritland 1996) para escoger el mejor estimador de parentesco para nuestra poblacion de estudio. El analisis indico que el mejor coeficiente de parentesco para esta poblacion es el de Queller y Goodnight (1989), ya que presenta mayor varianza de r y varianza real [var (r), necesaria para la estimacion de heredabilidad y correlaciones geneticas usando el metodo de Ritland] comparado con los otros estimadores (tabla 2). Se estimo la relacion de parentesco entre todos los posibles pares de individuos en la poblacion y su varianza real en el programa Mark para su posterior uso en los estimados de heredabilidad y correlaciones geneticas segun el metodo de Ritland (1996).
Estimacion de heredabilidad y correlaciones geneticas. Se usaron los valores de relacion genetica o parentesco entre individuos inferidos de los marcadores microsatelites y su correlacion con la similitud fenotipica para estimar heredabilidad y correlaciones genotipicas de las caracteristicas morfologicas y fisiologicas en la poblacion. Para estimar el grado de similitud fenotipica entre individuos en cada uno de los rasgos es necesario eliminar el efecto del tamano/edad de la planta sobre la similitud, por lo cual para las estimaciones de heredabilidad y correlaciones geneticas se usaron los residuos estandarizados de la regresion entre cada rasgo y el numero de foliolos del individuo.
La heredabilidad y correlaciones geneticas para los caracteres, se estimaron segun el metodo de regresion Ritland (1996, 2000) usando el programa Mark (Ritland 1996). El metodo de Ritland (1996) propone que la heredabilidad en sentido estrecho podria ser estimada por la regresion de una medida de similitud fenotipica sobre las relaciones de parentesco genetico entre los individuos obtenidas con los marcadores moleculares. La heredabilidad para cada rasgo puede ser estimada como:
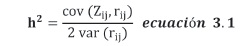
donde cov (Zij, rij) es la covarianza entre la relacion genetica estimada y la similitud fenotipica y var (rij) es la varianza real de los relacionados geneticamente. La estimacion de la varianza real es descrita con detalle en Ritland (2000).
La correlacion genetica aditiva se obtiene a partir de la expresion:
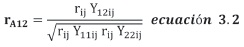
donde rij es el coeficiente de correlacion genetica, Y12ij es la covarianza fenotipica entre los individuos i e j. El signo de la correlacion genetica fue estimado a partir de la covarianza entre la similitud fenotipica y la relacion genetica estimada (cov (Zij, rij)). A diferencia de los estimativos de heredabilidad, este parametro no requiere la medida de la varianza real del parentesco entre los individuos.
La estimacion de heredabilidad segun el metodo de Ritland exige la eleccion de una distancia minima entre los individuos que seran comparados en una poblacion, para evitar comparaciones entre individuos con grados de parentesco con valores poco precisos (Ritland 1996). Para explorar la relacion entre el grado de parentesco y la distancia fisica entre individuos se caracterizo la estructura genetica espacial fina de la poblacion en el programa Spagedi (Hardy y Vekemans 2002), y se uso una distancia minima de 10 m entre individuos que resulto ser la distancia en la que los estimados de heredabilidad eran mas precisos. Para determinar si las estimaciones de heredabilidades y correlaciones geneticas para los caracteres son significativamente diferentes de cero se uso un analisis de bootstraping (1.000 bootstraps) implementado en el programa Mark. En este analisis un estimado es significativamente diferente de cero si el 95% de los valores del bootstraping estan por debajo del valor base estimado.
RESULTADOS
Caracterizacion fenotipica de rasgos morfologicos y fisiologicos. En terminos generales se observo un patron en el cual los rasgos fisiologicos tienen mayor varianza fenotipica cuando se comparan con los rasgos morfologicos. La cantidad de clorofila a presenta la mayor varianza fenotipica en la poblacion, seguido por la densidad estomatica, el area foliar, y por ultimo, el area foliar especifica. Por otro lado, la varianza fenotipica en los rasgos morfologicos es similar entre estos, y es mucho menor que para cualquiera de los rasgos fisiologicos (figura 2).
Cuando se consideran correlaciones fenotipicas con los valores originales de los rasgos, la mayoria de los rasgos morfologicos y fisiologicos estan correlacionados entre si, excepto con la cantidad de clorofila a (figura 3). En general, se observa un patron en el que los rasgos morfologicos estan mas altamente correlacionados entre ellos mismos que los rasgos fisiologicos. Todas las posibles correlaciones entre los rasgos morfologicos son fuertes (coeficiente de correlacion > 0,8) y positivas, mientras que en los rasgos fisiologicos la mitad de las posibles correlaciones son moderadas (coeficiente de correlacion entre 0,3 y 0,5). Entre los rasgos fisiologicos, el area foliar y el SLA estan moderadamente correlacionados, mientras que la densidad estomatica tiene una debil correlacion con estos dos rasgos y adicionalmente con los rasgos morfologicos. Por otro lado, la cantidad de clorofila a no se relaciona con ningun rasgo.
Cuando el efecto del tamano/edad de la planta es removido, se encuentra un numero menor de correlaciones fenotipicas entre rasgos, sin embargo se observa el mismo patron general que con los datos originales. En la poblacion, los rasgos morfologicos estan mas altamente correlacionados entre ellos mismos que los rasgos fisiologicos, y ocasionalmente se encuentran correlacionados rasgos morfologicos con fisiologicos (figura 4). La unica relacion fuerte es entre la altura y el diametro de tallo (coeficiente de correlacion = 0,8). Para los otros rasgos morfologicos se observan correlaciones moderadas (coeficientes de correlacion entre 0,2 y 0,4). Por otro lado, los rasgos fisiologicos presentan correlaciones debiles entre ellos mismos (coeficientes de correlacion entre 0,2 y 0,3). La densidad estomatica esta debilmente correlacionada con el area foliar y el SLA, y estas dos ultimas estan mutuamente relacionadas, mientras que la cantidad de clorofila a no esta correlacionado con ningun otro rasgo. Las correlaciones entre los caracteres morfologicos y fisiologicos son menos comunes y debiles, exceptuando por el area foliar y el SLA que estan relacionados a los rasgos morfologicos (coeficientes de correlacion entre 0,2 y 0,3).
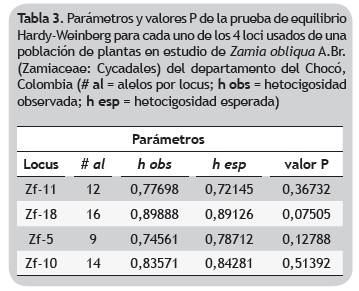
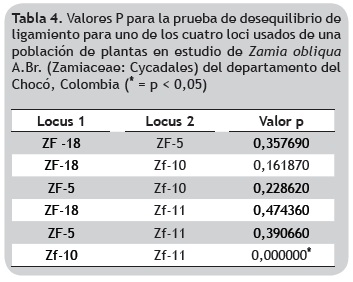
Caracterizacion genotipica. Los cuatro loci microsatelites evaluados para estimar el parentesco entre los individuos fueron polimorficos con 13 alelos promedio por locus, con marcadores altamente informativos como el Zf-18 y Zf-10 con 16 y 14 alelos, respectivamente. En esta poblacion de Z. obliqua, la heterocigocis observada no difirio significativamente de la esperada bajo el supuesto de equilibrio Hardy-Weinberg para los cuatro marcadores evaluados (tabla 3) y ningun loci demostro la presencia de alelos nulos. En cuanto a la prueba de desequilibrio de ligamiento, se obtuvieron valores significativos para la relacion entre los loci Zf-10 y Zf-11, indicando que se encuentran en desequilibrio de ligamiento (tabla 4).
Estimaciones de heredabilidad y correlaciones geneticas. Cinco de los ocho rasgos analizados presentaron valores de heredabilidad significativamente distintos de cero, mientras que la longitud de la hoja, el SLA y la densidad estomatica no parecen tener variabilidad aditiva genetica en la poblacion (figura 5). En general se observa que los rasgos morfologicos presentan heredabilidades significativas mas altas que los fisiologicos. La altura y el diametro del tallo tiene valores cercanos 0,4, y el numero de hojas tienen alta heredabilidad con valor aproximado de 0,7. El area foliar tiene valor de heredabilidad bajo cercano a 0,2, y la cantidad de clorofila a tiene una heredabilidad de menos de 0,1. Los estimativos de heredabilidad fuera del rango teorico (0 < h2 < 1) son consecuencia del metodo del Ritland que podria representar un problema si se esta interesado en valores precisos de heredabilidad, pero en este caso se desea realizar comparaciones entre categorias de rasgos.
No se obtuvieron correlaciones geneticas significativamente diferentes a cero entre los rasgos, probablemente porque el error del metodo usado para estimar este parametro es muy grande (Castellanos et al. 2011). Por lo tanto, se usaron las correlaciones fenotipicas como un indicativo de la potencial correlacion evolutiva entre rasgos en esta poblacion.
DISCUSION
Las dificultades inherentes para estimar heredabilidad en campo han limitado su uso extensivo en estudios evolutivos en plantas (Castellanos et al. 2011). Sin embargo, en este trabajo fue posible estimar heredabilidades significativas para dos categorias de rasgos morfologicos y fisiologicos en una poblacion natural chocoana de Z. obliqua sin previa informacion de la estructura familiar de la poblacion a traves del metodo de Ritland (1996). En sintesis, en este estudio se encontro soporte para las dos hipotesis planteadas: 1) que la heredabilidad tiende a ser mayor en caracteres morfologicos que en caracteres fisiologicos (aunque algunos rasgos parecen no tener variacion genetica); 2) las correlaciones fenotipicas sugieren que los rasgos morfologicos estan mas estrechamente relacionados entre si que los fisiologicos entre ellos o los morfologicos con los fisiologicos.
En este trabajo fue posible obtener heredabilidades significativas en casi todos los rasgos evaluados a traves del metodo del Ritland gracias al polimorfismo alto de los marcadores utilizados y a la estructuracion genetica espacial de la poblacion (Andrew et al. 2005). Segun Ritland (1996) para una adecuada estimacion de parentesco son necesarios entre 2 y 10 loci, cada uno con 10 alelos promedios. En este estudio se usaron 4 marcadores con un promedio de 13 alelos por locus, cumpliendo con el requisito de alto polimorfismo. En cuanto a la estructuracion genetica espacial, se observa que plantas mas cercanas geograficamente estan mas emparentadas, posiblemente como consecuencia de una dispersion de semillas restringida que generalmente se da por la gravedad. Gracias a esta estructuracion espacial fina, se espera tener individuos relacionados en varios grados (e. g., hermanos completos, hermanos medios, individuos no relacionados) a distintas distancias geograficas y una varianza de parentesco entre pares de individuos significativa.
La ausencia de varianza significativa en el grado de parentesco entre individuos representa uno de los mayores obstaculos para la aplicacion del metodo de Ritland en poblaciones naturales (Ritland 1996, Shikano 2008). Por este motivo, la exactitud de los estimativos de heredabilidad hallados con este metodo han sido cuestionados por varios autores (Bouvet et al. 2008, Garant y Kruuk 2005). Para varias poblaciones con conocimiento de la genealogia se estimo heredabilidad a traves del metodo de Ritland y el metodo clasico, encontrando que en el primero los valores de heredabilidad son menos exactos comparados con el segundo (Coltman 2005, Frentiu et al. 2008, Thomas et al. 2002). Sin embargo, Bessega et al. (2009) encuentran una consistencia entre los estimativos de heredabilidad obtenidos por los metodos clasicos y a traves del metodo de Ritland en la planta Prosopis alba, gracias a que en la poblacion existe una varianza de parentesco significativa. En el caso hipotetico que los estimativos de heredabilidad hallados no sean exactos por los errores estadisticos inherentes al metodo, estos pueden seguir siendo usados para la comparacion de los rasgos dentro de la poblacion como se hizo en este estudio. Asumiendo que los estimativos de heredabilidad en este estudio son comparables entre caracteres, las heredabilidades en esta poblacion mostraron una marcada diferencia entre categorias de rasgos. Los caracteres morfologicos presentaron una heredabilidad mas alta comparados con los rasgos fisiologicos (aunque uno de los caracteres morfologicos y dos de los fisiologicos no exhibieron heredabilidades distintas de cero). Este mismo patron de diferencias en la heredabilidad se ha observado para otras especies tanto modelo como no modelo. Por ejemplo, en un estudio realizado en 20 variedades de arroz se encontro heredabilidades mas altas en rasgos relacionados con el tamano y arquitectura de la planta que para rasgos fisiologicos (Jahn et al. 2011). De manera similar, Garcia-Verdugo et al. (2010) detectaron heredabilidades significativas para todos los rasgos morfologicos mientras que ninguna variacion genetica para los caracteres fisiologicos en poblaciones naturales de Olea europeae L.
En este estudio, los rasgos fisiologicos presentaron una menor heredabilidad y mayor varianza fenotipica que los caracteres morfologicos. La baja heredabilidad de los rasgos fisiologicos puede deberse a que estos estan expuestos a una seleccion natural direccional mas fuerte que los morfologicos. Roff (1997) propone un patron de jerarquizacion en la heredabilidad de las categorias de rasgos, en el cual los caracteres mas estrechamente conectados con la adecuacion biologica presentan menor heredabilidad. Dado lo anterior, puede suponerse que los rasgos fisiologicos han fijado alelos beneficos a traves del tiempo mas rapidamente que los morfologicos, lo cual tiene como resultado una disminucion de la varianza genetica, y por ende menor heredabilidad (Teplitsky et al. 2009). Esta hipotesis es apoyada por varios estudios en poblaciones naturales que han registrado que los rasgos mas cercanamente asociados a la adecuacion biologica tienen menor heredabilidad que los menos asociados a esta (Coltman et al. 2005, McCleery et al. 2004, Teplitsky et al. 2009).
La plasticidad fenotipica alta de los rasgos fisiologicos puede ser otro posible factor responsable de su menor heredabilidad. Esto se debe a que la heredabilidad es inversamente relacionada a la magnitud de los efectos ambientales sobre el rasgo, entonces rasgos altamente plasticos presentan menor heredabilidad (Geber y Griffen 2003). Culley et al. (2006) sugieren que los estimativos de heredabilidad negativa o muy bajos para rasgos fisiologicos son consecuencia de la alta plasticidad que ellos presentan. En este estudio, el patron observado de mayor varianza fenotipica en rasgos fisiologicos podria estar asociado a mayor plasticidad fenotipica en estos caracteres. Varios estudios han informado de heredabilidades bajas en caracteres con alta plasticidad fenotipica, como el realizado por Johnson et al. (2009) que estudia la varianza genetica de 24 rasgos de la planta Oenothera biennis y encuentra que algunos caracteres eco-fisiologicos con alta plasticidad fenotipica, como la conductancia estomatica, la eficiencia en el uso del agua (WUE) y la tasa fotosintetica en funcion del area foliar presentan una baja heredabilidad.
No se pudieron obtener estimativos significativos de correlaciones geneticas entre los rasgos. Sin embargo, analizado las correlaciones fenotipicas se encontro que los rasgos morfologicos estan mas altamente correlacionados entre ellos mismos que los rasgos fisiologicos y que entre las dos categorias de los rasgos, tanto con los datos originales como con los corregidos por edad/tamano de la planta (donde las correlaciones fueron mas debiles). Este patron esta de acuerdo con la hipotesis en la cual se sugiere que existe una correlacion mas fuerte entre rasgos funcionalmente relacionados, como los que estan involucrados con el crecimiento de planta, que entre rasgos independientes (Gomez et al. 2009). Este patron en las correlaciones podria resultar del efecto de la seleccion sobre combinaciones ventajosas de rasgos que pertenecen a la estructura del mismo complejo funcional (McGlothlin et al. 2005). La baja correlacion entre rasgos fisiologicos podria deberse a la flexibilidad que presentan estos caracteres a las diferentes demandas ecologicas, como lo sugieren Buehler et al. (2012).
Este estudio ha mostrado la utilidad e importancia que tiene el metodo de Ritland para inferir parametros cuantitativos como heredabilidad directamente en poblaciones naturales. A pesar de las potenciales limitaciones este enfoque ofrece una herramienta util para estudios de genetica cuantitativa en poblaciones naturales de plantas de largos ciclos de vida (Andrew et al. 2005).
En conclusion, usando el metodo de Ritland se encontro que en esta poblacion de la especie Z. obliqua la heredabilidad de los rasgos morfologicos tiende a ser mayor que la de los rasgos fisiologicos, y que la varianza fenotipica de los rasgos fisiologicos es mayor que la de los morfologicos. Ademas, se encontro que usando las correlaciones fenotipicas se observa mayor correlacion entre los rasgos morfologicos que dentro de los fisiologicos o entre rasgos morfologicos y fisiologicos. Este estudio genera informacion importante sobre parametros basicos de genetica cuantitativa que describen la varianza y covarianza fenotipica y genetica para caracteres de importancia ecologica dentro de una poblacion de una especie de larga vida, y es una importante contribucion al estudio de la ecologia evolutiva de especies no-modelo en sus ambientes naturales.
AGRADECIMIENTOS
Las autoras de este articulo agradecemos a Montgomery Botanical Center y The Cycad Society (E. U. A.) por la financiacion del proyecto. El proyecto fue financiado mientras las investigadoras estaban asociadas al Grupo de Biodiversidad de la Corporacion para Investigaciones Biologicas (CIB, Medellin). Agradecemos tambien al personal del grupo de investigacion Ecologia Evolutiva y Conservacion (EECO), Universidad de Antioquia por su colaboracion en el trabajo de campo y al personal del grupo Genetica Molecular (Genmol), Universidad de Antioquia donde realizamos los analisis geneticos y donde la investigadora principal desarrollo su trabajo de grado para el titulo de Biologa.
REFERENCIAS
Andrew RL, Peakall R, Wallis IR, Wood JT, Knight EJ, Foley WJ. 2005. Marker-based quantitative genetics in the wild?: the heritability and genetic correlation of chemical defenses in eucalyptus. Genetics, 171 (4): 1989-1998. [ Links ]
Bessega C, Saidman BO, Darquier MR, Ewens M, Sanchez L, Rozenberg P, Vilardi JC. 2009. Consistency between marker- and genealogybased heritability estimates in an experimental stand of Prosopis alba (Leguminosae). American Journal of Botany, 96 (2): 458-465. [ Links ]
Bouvet J-M, Kelly B, Sanou H, Allal F. 2008. Comparison of markerand pedigree-based methods for estimating heritability in an agroforestry population of Vitellaria paradoxa C. F. Gaertn. (shea tree). Genetic Resources and Crop Evolution, 55: 1291-1301. [ Links ]
Buehler M, Vezina F, Goymann W, Schwabl I, Versteegh M, Btieleman I. 2012. Independence among physiological traits suggests flexibility.in the face of ecological demands on phenotypes. Journal of Evolutionary Biology, 25: 1600-1613. [ Links ]
Castellanos M, Alcantara J, Rey P, Bastida J. 2011. Intra-population comparison of vegetative and floral trait heritabilities estimated from molecular markers in wild Aquilegia populations. Molecular Ecology, 20: 3513-3524. [ Links ]
Coltman D. 2005. Testing marker-based estimates of heritability in the wild. Molecular Ecology, 14: 2593-2599. [ Links ]
Culley T, Dunbar-Wallis A, Sakai A, Weller S, Mishio S, Campbell D, Herzena M. 2006. Genetic variation of ecophysiological traits in two gynodioecious species of Schiedea (Caryophyllaceae). New Phytologist, 169: 589-601. [ Links ]
Doyle J, Doyle J. 1990. Isolation of plant DNA from fresh tissue. Focus, 12: 13-15. [ Links ]
Excoffier L, Lischer H. 2010.Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 10: 564-567. [ Links ]
Frentiu F, Clegg S, Chittock J, Burke T, Blows M, Owens I. 2008. Pedigreefree animal models: the relatedness matrix reloaded. Proceeding of Royal Society B: Biological Sciences, 275: 639-647. [ Links ]
Garant D, Kruuk L. 2005. How to use molecular marker data to measure evolutionary parameters in wild populations. Molecular Ecology, 14: 1843-1859. [ Links ]
Garcia-Verdugo C, Mendez M, Velazquez-Rosas N, Balaguer L. 2010. Differential morphological and physiological responses across insular environments: phenotypic expression and heritability of light-related traits in Olea europaea. Oecologia, 164: 647-655. [ Links ]
Geber M, Griffen L. 2003. Inheritance and natural selection on functional traits. International Journal of Plant Sciences, 164 (3 Suppl.): S21-S42. [ Links ]
Gomez J, Abdelaziz M, Munoz J, Perfectti F. 2009. Heritability and genetic correlation of corolla shape and size in Erysimummedio hispanicum. Evolution, 63 (7): 1820-1831. [ Links ]
Hardy J, Vekemans X. 2002. SPAGeDi: a versatile computer program to analyse spatial genetic structure at the individual or population levels. Molecular Ecology Notes, 2: 618-620. [ Links ]
Hedrick PW. 2005. Genetics of populations. 3rd ed. Boston (U. S. A.): Jones and Bartlett. p. 274. [ Links ]
Herrera C, Bazaga P. 2009. Quantifying the genetic component of phenotypic variation in unpedigreed wild plants: tailoring genomic scan for within-population use. Molecular Ecology, 18: 2602-2614. [ Links ]
Jahn C, Mckay J, Mauleon R, Stephens J, Mcnally K, Bush D, Leung H, Leach J. 2011. Genetic variation in biomass traits among 20 diverse rice varieties. Plant Physiology, 155: 157-168. [ Links ]
Johnson M, Agrawal A, Maron I, Salminen J. 2009. Heritability, covariation and natural selection on 24 traits of common evening primrose (Oenothera biennis) from a field experiment. Journal of Evolutionary Biology, 22: 1295-1307. [ Links ]
Lopez-Gallego C, O'neil P. 2010. Life-history variation following habitat degradation associated with differing fine-scale spatial genetic structure in a rainforest cycad. Population Ecology, 52: 191-201. [ Links ]
Lynch M, Ritland K. 1999. Estimation of pairwise relatedness with molecular markers. Genetics, 152: 1753-1766. [ Links ]
McCleery R, Pettifor R, Armbruster P, Meyer K, Sheldon B, Perrins M. 2004. Components of variance underlying fitness in a natural population of the great tit Parus major. The American Naturalist, 164 (3): E62-E7. [ Links ]
Mcglothlin J, Parker P, Nolan V, Ketterson E. 2005. Correlational selection leads to genetic integration of body size and an attractive plumage trait in dark-eyed juncos. Evolution, 59: 658-671. [ Links ]
Norstog K, Nicholls T. 1997. The biology of the cycads. New York (U. S. A.): Cornell University Press. p. 363. [ Links ]
Oosterhout C, Hutchinson W, Wills P, Shipley P. 2004. MICROCHECKER: software for indentifying and correcting genotyping errors in microsatellite data. Molecular Ecology Notes, 4: 535-538. [ Links ]
Queller D, Goodnight K. 1989. Estimating relatedness using genetic markers. Evolution, 43: 258-275. [ Links ]
Ritland K. 1996. A marker-based method for inferences about quantitative inheritance in natural populations. Evolution, 50: 1062-1073. [ Links ]
Ritland K. 2000. Marker-inferred relatedness as a tool for detecting heritability in nature. Molecular Ecology, 9: 1195-1204 [ Links ]
Roff D. 1997. Evolutionary quantitative genetics. New York (U. S. A.): Chapman & Hall. p. 493. [ Links ]
Schneider C, Rasband S, Eliceiri K. 2012. NIH Image to Image J: 25 years of image analysis. Nature Methods, 9: 671-675. [ Links ]
Shikano T. 2008. Estimation of quantitative genetic parameters using marker-inferred relatedness in Japanese flounder: A case study of upward bias. Journal of Heredity, 99 (2): 94-104. [ Links ]
SPSS Inc. 2008. SPSS Statistics for Windows, Software Version 17.0. Chicago: SPSS Inc. Released. [ Links ]
Stevenson D. 2004. Cycads of Colombia. The Botanical Review, 70 (2): 194-234. [ Links ]
Teplitsky C, Mills J, Yarrall J, Merila J. 2009. Heritability of fitness components in a wild bird population. Evolution, 63-3: 716-726. [ Links ]
Thomas S, Hil W. 2000. Estimating quantitative genetic parameters using sibships reconstructed from marker data. Genetics, 155: 1961-1972. [ Links ]
Wang J. 2002. An estimator for pairwise relatedness using molecular markers. Genetics, 160: 1203-1215. [ Links ]