INTRODUCCIÓN
Los xenartros (Gardner, 2008) constituyen un superorden de mamíferos que, de acuerdo con su registro fósil, son originarios de Suramérica y se distribuyen por todo el continente desde el sur de los Estados Unidos hasta el sur de Argentina (Abba et al., 2012; Wilson y. Mittermeier, 2018). Este superorden comprende tres grupos vivientes, los perezosos (Pilosa), los hormigueros (Vermilingua) y los armadillos (Cingulata), los cuales se dividen en 14 géneros y 31 especies identificadas (Gaudin, 2003). Estos animales son de gran importancia histórica y ecológica para el continente por su proceso evolutivo y su estado de conservación, pero no han sido lo suficientemente estudiados debido a su naturaleza críptica.
El uso de técnicas en genética molecular para responder preguntas en biología de la conservación y ecología del comportamiento está en aumento. Tal es el caso de los estudios de taxonomía filogenética, en los que el uso de marcadores moleculares ha permitido identificar individuos, especies y poblaciones (Avise, 1989). La realización de estos procedimientos moleculares requiere de ADN de alta calidad (concentración adecuada, alta pureza e integridad), lo cual depende del tipo de muestra y del método de extracción usado (Blanco-Jarvio et al., 2014). En los últimos años, para el monitoreo genético, los métodos de muestreo mínimamente invasivos han evolucionado, dando la ventaja de generar datos sin tener que capturar el animal, ni manipularlo y en algunos casos ni siquiera observarlo (Carroll et al., 2018).
Los diferentes métodos usados para extraer ADN no son usualmente utilizados para muestras en organismos específicos y deben analizarse y ajustarse a las características de cada caso. Existen pocos estudios acerca de xenartros donde usen muestras de animales vivos (Barros et al., 2003); otros, como el llevado a cabo por Coimbra et al. (2017), usaron un amplio rango de muestras como hígado, músculo, sangre, pelos o muestras de colecciones de museos; sin embargo, para obtener alguna de las muestras usadas en estos estudios, no solo se requiere la captura e inmovilización del animal, sino también gastos en tiempo y dinero, lo cual podría ser optimizado conociendo la eficiencia del método de extracción de ADN que se seleccione para este fin y las muestras que, con menores intervenciones en los animales, generen los mejores resultados.
Por otro lado, los diferentes métodos anteriormente mencionados muestran resultados variables en cuanto a la eficiencia en la obtención de ADN para diferentes tipos de muestras. El método más comúnmente usado es fenol-cloroformo y en algunos casos la extracción con kit comercial como QUIAGEN®; sin embargo, ha sido ampliamente documentado que el método de fenol-cloroformo aunque es simple y eficiente, tiene componentes muy tóxicos para los investigadores y el ambiente (Silva et al., 2013).
Existen otros métodos de extracción que toman aprovechan ciertas capacidades de algunos materiales para absorber el ADN y tienen importantes ventajas que evitan la degradación química y física del ADN durante la purificación, haciendo al ADN menos susceptible a la degradación enzimática y maximizando la degradación de proteínas (contaminantes) (Tian et al., 2000). Por ejemplo, los kits de extracción basados en tecnologías de sílica, son un método con una buena relación entre costos y resultados, con un proceso de extracción orgánica fácil y rápida (Liu et al., 2016). Otra técnica ampliamente usada es la separación por resinas magnéticas, este es un método rápido que puede ser automatizado y puede ser un poco más costoso que el anterior (Dhaliwal, 2020).
Dada la relevancia de los xenartros en el neotrópico y el creciente interés en estudios de genética molecular, en esta investigación se compararon dos métodos de extracción de ADN, separación magnética con el kit PrepFiler™ (Thermo Fisher Scientific, Waltham, Massachusetts, U.S.) y un método basado en sílica con el kit GeneJET™ (Thermo Fisher Scientific, Waltham, Massachusetts, U.S.), para 5 tipos de muestras: sangre, tejido, saliva, pelos y heces o frotis anales. El objetivo del estudio fue seleccionar el mejor método de extracción, en términos de pureza, calidad e integridad de ADN, en combinación con el tipo de muestras menos invasiva posible.
MATERIALES Y METODOS
Muestras
Las muestras provienen de animales de diferentes regiones de Colombia, que se encontraban en la fundación AIUNAU, institución localizada en Caldas, Antioquia, Colombia, la cual se encarga de su proceso de rehabilitación y reintroducción a la vida silvestre; allí se desarrolló el protocolo de muestreo. Todo el material biológico recolectado está amparado por el permiso otorgado por la autoridad nacional de licencias ambientales (ANLA) a la universidad CES, resolución 790 de 2014 y una vez concluida la investigación las muestras fueron depositadas en la colección biológica CBUCES-L (constancia de depósito 114) con registro nacional de colecciones 209; además, el proyecto fue aprobado por el comité de bioética de la universidad CES en acta No 7 del 19 de enero de 2018.
Se obtuvieron muestras de sangre, tejido, saliva, pelos y heces o frotis anales de 29 individuos distribuidos en cuatro especies del superorden Xenarthra: Myrmecophaga tridactila (n=2), Tamandua mexicana (n=5), Bradypus variegatus (n=3) y Choloepus hoffmanni (n=19). Para la toma de la muestra se siguió el siguiente protocolo:
Sangre: esta solo se obtuvo de 14 animales, de dos especies T. mexicana y C. hoffmani, que murieron durante el proceso de rehabilitación o que llegaron muertos a la fundación AIUNAU; a cada animal se le tomó una muestra de 5 mL de sangre en tubo con anticoagulante EDTA y se almacenó a -20 ºC hasta su procesamiento (Muñoz-García et al., 2016).
Tejidos: hace referencia a muestras diferentes a sangre y pelos, por ejemplo: músculo, intestino, pulmón, corazón, entre otros; como en el caso anterior, solo se tomó de animales muertos e inmediatamente después del fallecimiento; se tomaron muestras a 11 animales de las especies T. mexicana, B. variegatus y C. hoffmani, de aproximadamente 1 cm2 de hígado, pulmón o intestino. Las muestras fueron almacenadas en un tubos eppendorfs a -20 ºC hasta su procesamiento (Muñoz-García et al., 2016).
Saliva: se colocó un hisopado estéril en la boca del animal permitiendo que lo masticara, igualmente se frotó contra las paredes internas de la cavidad oral (Gallina y López González, 2011). Las muestras siempre se tomaron en la mañana antes de la alimentación, después los hisopos se dejaron secar a temperatura ambiente, se empacaron en bolsas de papel y plásticas y se almacenaron a temperatura ambiente hasta su procesamiento. En este caso, las muestras fueron recolectadas en 18 animales de las especies M. tridactyla, T. mexicana, B. variegatus y C. hoffmani.
Pelos: estas muestras fueron tomadas a 14 animales vivos y muertos de las especies M. tridactyla, B. variegatus y C. hoffmani; los animales fueron inmovilizados mecánicamente y se arrancó, aproximadamente, entre 20-30 pelos que tuvieran el folículo capilar y se almacenaron en bolsas de papel y plásticas a temperatura ambiente (Gallina y López González, 2011).
Heces/frotis anales: estas muestras se recolectaron de cuatro animales vivos de las especies M. tridactyla y C. hoffmani; las muestras se tomaron en su encierro de rehabilitación justo después de su deposición; entre 3-4 excretas se tomaron por animal con guantes estériles y se depositaron en bolsas de papel y se almacenaron a -20 ºC hasta su procesamiento (Gallina y López González, 2011). En algunos casos se procedió a realizar frotis anal directamente en el animal con el uso de un hisopo húmedo.
Extracción de ADN
Para probar los kits de extracción de ADN (PrepFiler™ and GeneJET™), las muestras biológicas usadas fueron: 60 µL de sangre, medio hisopo con saliva, 20 mg de tejido macerado, ocho cabellos o un hisopo impregnado de heces.
El kit de extracción PrepFiler™ (cat. 4463351) (Thermo Fisher Scientific, Waltham, Massachusetts, U.S.) fue usado siguiendo el protocolo del fabricante con algunas modificaciones como se describe a continuación: el proceso de lisis celular para sangre, saliva y heces fue realizado usando 300 µL de búfer de lisis para muestras biológicas e incubado a 70 ºC por 20 min para sangre y 40 min para saliva y heces con agitación y vórtex; para las muestras de tejidos y pelos, la digestión se hizo acorde al protocolo descrito previamente por Loreille et al. (2007) usando 180 µL de búfer de digestión y 5 uL de proteinasa K con incubación a 56 ºC de 10-12 h. Una vez se obtuvo el ADN, este se almacenó a 4 ºC y una vez se terminaron los análisis, se almacenó a -20 ºC en las colecciones biológicas de la universidad CES (CBUCES).
La extracción con GeneJET™ (cat. K0722) (Thermo Fisher Scientific, Waltham, Massachusetts, U.S.) fue realizada siguiendo las indicaciones del fabricante, con algunas modificaciones que se describen a continuación: la lisis celular se realizó adicionando 200 µL de la solución de lisis para muestras biológicas e incubadas a 56 ºC por 10 min con agitación y vórtex. Las muestras de saliva y heces fueron sumergidas en solución de lisis por 20 min e incubadas por 15 min a 56 ºC con agitación y vórtex; los tejidos y los pelos fueron digeridos siguiendo el protocolo de Loreille et al. (2007) descrito previamente.
Concentración integridad y pureza del ADN
Una vez el material genético fue obtenido, se determinó la concentración en ng/µL y se cuantificó la pureza por espectrofotometría usando un nanodrop 2000 (Thermo Fisher Scientific Inc., U.S). Considerando que la reacción en cadena de la polimerasa (PCR) requiere de 100-200 ng de ADN, las concentraciones iguales o superiores a 5 ng/µL se consideraron óptimas. La relación de absorbancia 260/280 fue considerada para evaluar la pureza, donde una razón entre 1,8-2,0 se considera como un indicador de pureza, valores inferiores o superiores son indicios de contaminación con residuos orgánicos o inorgánicos como proteínas o solventes (Alejos-Velázquez et al., 2014). La integridad del ADN se evaluó a través de una electroforesis en agarosa al 1% teñida con SYBR™ Safe (Invitrogen). La electroforesis se llevó a cabo a 80V por 30 min y la visualización se realizó con luz ultravioleta (UV) en un foto-documentador BIO-RAD ChemiDoc™ MP; se usó un marcador de peso molecular 50 pb (Bioline, U.S). Se consideró una integridad óptima cuando la banda de ADN tenia alto peso molecular (mayor a 2000 pb).
Análisis estadístico
Para comparar la cantidad y pureza del ADN, entre los métodos de extracción y los diferentes tipos de muestras, se utilizó un análisis de varianza de efectos mixtos con dos factores fijos (el método de extracción y el tipo de muestra) y con el individuo como factor aleatorio. Se utilizó una prueba de comparaciones múltiples de Tukey para identificar las diferencias específicas entre medias (concentración de ADN y pureza). Todas las pruebas fueron *realizadas con un nivel de significancia del 5%. La cantidad de ADN fue transformada con el logaritmo natural para asegurar una distribución aproximadamente normal y los resultados para esta variable se reportaron en dicha escala. Todos los análisis fueron realizados en el programa estadístico R (Core Team, 2008).
RESULTADOS
Concentración del ADN
La concentración de ADN obtenida, usando los kits PrepFiler™ y GeneJET™, presentó diferencias entre ellos y los resultados se muestran en la Figura 1 (ANOVA, F4,110 = 4,35; p = 0,0026). La concentración media más alta de ADN (5,25 ng/µl) fue obtenida de muestras de tejido con el kit PrepFiler™ y la más baja concentración con muestras de pelo con el kit GeneJET™ kit (1,37 ng/µl) (Tabla 1). Se encontraron diferencias (prueba Tukey, p < 0,0001) entre muestras de tejido y sangre y muestras de tejido y pelo con el kit PrepFiler™; mientras que con el kit GeneJET se observaron diferencias (prueba Tukey, p < 0,0001) entre muestras de tejidos y heces y muestras de tejido y cabello (Figura 1).
Tabla 1 Media de la concentración de ADN (transformada con un logaritmo) y su desviación estándar (DS) obtenido a través de los métodos de extracción de ADN PrepFiler™ and GeneJET™
| Concentración de ADN (ng/µl) (Media ± EE) | ||
|---|---|---|
| Muestra | Kit PrepFiler™ | Kit GeneJET™ |
| Tejido (n=12) | 5,25 ± 0,39 | 2,89 ± 0,14 |
| Saliva (n=18) | 3,56 ± 0,24 | 2,54 ± 0,24 |
| Sangre (n=14) | 2,44 ± 0,40 | 2,53 ± 0,23 |
| Heces (n=4) | 2,81 ± 0,34 | 1,94 ± 0,11 |
| Pelos (n=14) | 2,20 ± 0,20 | 1,37 ± 0,08 |
n= número de muestras
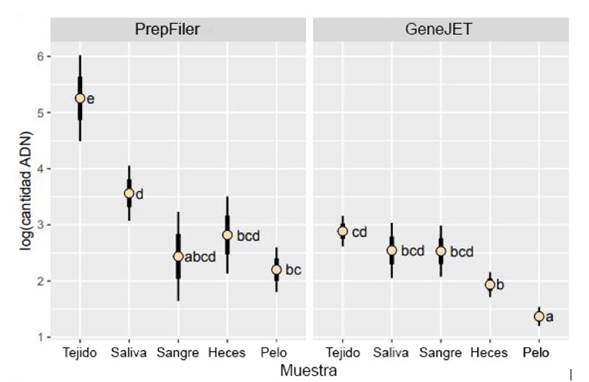
Figura 1 Concentración de ADN (log media transformado) usando los métodos PrepFiler™ (izquierda) y GeneJET™ (derecha) a través de las cinco muestras biológicas ensayadas. Los grupos de muestras identificadas con la misma letra no muestran diferencias con la prueba de Tukey's test (α=0,05). Las barras de error gruesas indican ± error estándar, y las barras delgadas (más amplias) indican un intervalo de confianza del 95%.
Pureza del ADN
La variación obtenida en cuanto a la pureza del ADN se muestra en la Figura 2 (ANOVA, F4,110 = 4,12; p = 0,0038). La pureza más alta fue obtenida para muestras de tejido con el kit PrepFiler™ y la más baja con el kit GeneJET™ en muestra de pelo (Tabla 2). Se encontraron diferencias (Prueba de Tukey, p<0,0001) con el kit PrepFiler™ entre muestras de tejido y pelos y entre saliva y pelos. Por otro lado, las diferencias observadas con el kit GeneJET™ fueron entre las muestras de tejido y cabello, tejido y heces, saliva y heces, sangre y cabello, y heces y cabello (Figura 2).
Tabla 2 Medias de la Pureza A260/280 nm del ADN con su error estándar (DS) obtenido mediante los métodos de extracción con el kit PrepFiler™ y el kit GeneJET™
| Pureza de ADN (Media ± EE) | ||
|---|---|---|
| Muestra | Kit PrepFiler™ | Kit GeneJET™ |
| Tejido (n=12) | 1,87 ± 0,04 | 1,77 ± 0,04 |
| Saliva (n=18) | 1,85 ± 0,04 | 1,61 ± 0,06 |
| Sangre (n=14) | 1,55 ± 0,11 | 1,72 ± 0,05 |
| Heces (n=4) | 1,59 ± 0,15 | 1,46 ± 0,04 |
| Pelos (n=14) | 1,48 ± 0,04 | 1,19 ± 0,03 |
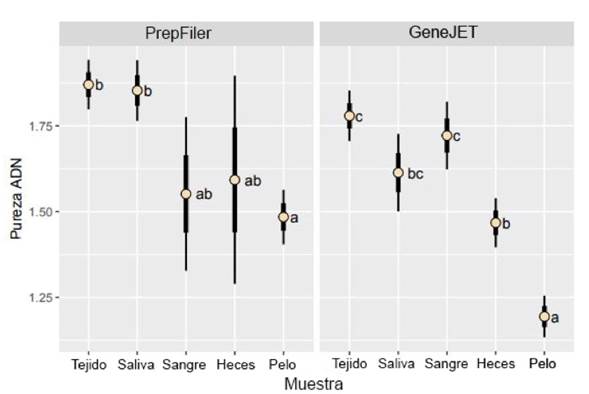
Figura 2 Medias de la pureza del ADN (izquierda: kit PrepFiler™; derecha: kit GeneJET™). Grupos con la misma letra no resultaron diferentes en la prueba de Tukey (α=0,05). Las barras de error gruesas indican ± error estándar y las barras delgadas (más amplias) indican un intervalo de confianza del 95%.
Integridad del ADN
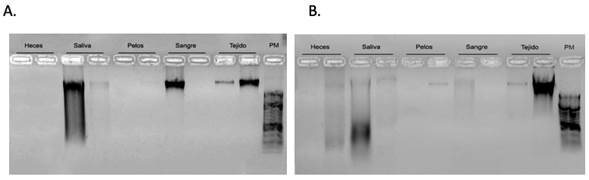
La integridad del ADN fue analizada por electroforesis al 1%; en la Figura 3 se muestra el patrón electroforético obtenido del ADN de los cinco tipos de muestras, con los dos métodos de extracción empleados. Se obtuvo ADN de diferentes pesos moleculares (50 - >2000pb), con variación entre muestras y métodos de extracción. Para el método de extracción GeneJET™ (Figura 3A), se obtuvieron bandas bien definidas para las muestras de saliva, sangre y tejidos, pero no se observaron resultados para heces y pelo; por otro lado, con el método de extracción PrepFiler™ (Figura 3B), se observaron bandas en todos los tipos de muestras, pero las mismas evidencian un proceso de fragmentación, lo que sugiere degradación en el ADN obtenido.
DISCUSIÓN
La aplicación de técnicas moleculares comienza con la extracción de ADN a partir de muestras adecuadas. El éxito en la obtención de resultados confiables y reproducibles depende en gran medida de la obtención de un ADN de alta calidad. Esta calidad es determinada por el tipo de muestra, su manipulación, almacenamiento y el método de extracción de ADN usado. Aunque la cantidad de ADN de un organismo es igual y constante a través de sus células, la cantidad que se puede extraer difiere ampliamente entre los tipos de muestras (González y Martínez, 2001).
Un método de extracción de ADN ideal es corto, con un número mínimo de pasos y con reactivos que no sean nocivos para los investigadores y el ambiente (Osorio et al., 2009); en este sentido, los métodos que usan esferas magnéticas (PrepFiler™) y membranas de sílice (GeneJET™) ofrecen simplicidad y rapidez en su implementación.
Los resultados de este estudio muestran que la extracción con PrepFiler™ tiene mayores concentraciones de ADN y pureza para todas las muestras empleadas; este resultado es comparable con la validación realizada por Brevnov et al. (2009) donde la calidad de ADN obtenida de saliva y sangre fue adecuada para la aplicación posterior de técnicas como PCR. Sin embargo, el protocolo de este kit incluye mucha manipulación y lavados de las muestras, lo cual explica la baja integridad observada con respecto a los resultados obtenidos con GeneJetTM (Figura 3). Por otro lado, el método de extracción GeneJetTM consigue purificar un ADN menos fragmentado, pero la concentración y la pureza es menor que la obtenida con PrepFiler™; sin embargo, GeneJetTM ha sido probado con muestras tan complejas como restos óseos y tejido de insectos (Pérez et al., 2016) mostrando que tiene alta capacidad de recuperar ADN funcional.
Con respecto a la eficiencia de las muestras usadas, el método de extracción PrepFiler™ recuperó la más alta concentración de ADN, la más alta pureza y mostró la menor degradación a partir de tejido; además, hubo diferencia en la concentración de ADN con otras muestras comúnmente usadas como sangre y pelos. Como esta muestra solo se tomó de animales muertos, se infiere que no está disponible en el trabajo rutinario con animales vivos, dado que su obtención causa gran dolor y estrés.
Un aspecto importante a considerar es el bienestar del animal; la obtención de muestras para estudios genéticos requiere captura, inmovilización e invasión del animal, causando estrés y estados de sufrimiento, que pueden desencadenar reacciones sistémicas que lleven a la muerte del animal (Valdespino et al., 2007), por lo que es recomendable buscar alternativas mínimamente invasivas.
Por lo tanto, a fin de garantizar el bienestar animal, el trabajo científico requiere la búsqueda de otros métodos, independiente del costo o la conveniencia (Jar, 2014). Los métodos mínimamente invasivos pueden ser los de elección en muchos estudios de monitoreo genético en vertebrados. La principal razón es que se pueden obtener muestras de ADN de animales en libertad y ser usadas para identificar individuos a través del espacio y el tiempo y generar datos genéticos sin necesidad de atraparlos, manipularlos y en algunos casos observarlos (Beja et al., 2009; Carroll et al., 2018; Schwartz et al., 2007). Por esta razón, se probaron muestras como pelos, saliva y heces, las cuales son fáciles y rápidas de obtener y requieren un contacto mínimo con el animal; estas muestras han sido útiles en proveer información genética de un gran rango de especies con altos niveles de individualización (Taberlet et al., 1999).
De las fuentes mínimamente invasivas probadas en este estudio, la obtención de ADN de saliva fue una de las que mostró mejores resultados, en particular cuando se usa el método de extracción PrepFiler™, el cual no mostró diferencias significativas en cuanto a pureza, concentración e integridad cuando se compara con las muestras de sangre. Este mismo resultado fue obtenido por Abraham et al. (2012), donde concluyen que, en muchos casos, la saliva puede reemplazar la muestra de sangre en muchos procedimientos para obtención de ADN. Por otro lado, la muestra de saliva también presentó mejores valores de calidad con respecto a pelo, una muestra que es comúnmente usada en estudios de este tipo en el superorden Xenarthra (Coimbra et al., 2017).
Por otro lado, cabe mencionar que los resultados de ADN obtenidos de heces cuando se usa el método de extracción de ADN PrepFiler ™, mostraron niveles de concentración y pureza similares a los obtenidos de sangre y pelos (sin diferencia significativa), por lo cual se podría considerar el uso de esta muestra, que no es comúnmente empleada como fuente de ADN en este clado; apoyando esta idea, Chiou y Bergey (2017) concluyeron que las heces son una fuente importante y confiable de ADN para estudios moleculares en animales salvajes. Sin embargo, aquí no analizamos suficientes muestras de heces para sacar una conclusión confiable sobre este tipo de muestra, por lo que recomendamos reconsiderarlo y volver a estudiarlo en futuras investigaciones.
Estos resultados permiten orientar los estudios genéticos y mejorar las metodologías de toma de muestra y obtención de ADN en los Xenarthra, evitando protocolos como los descritos por Barros et al., (2003) y Miranda et al., (2017), dado que se demuestra que hay otras fuentes de ADN que no requieren la muerte o una intervención mayor de los animales.
CONCLUSIONES
Los resultados obtenidos en estos estudios permiten concluir que la saliva es una muestra mínimamente invasiva que permite obtener ADN de muy buena calidad y cuyo uso con métodos como PrepFiler™ facilita su extracción y purificación. En conclusión, usar muestras de saliva y métodos de extracción de ADN como los descritos, es recomendable para la obtención de ADN en los xenartros bajo estudio. La obtención de ADN a partir de tejidos es una excelente fuente de ADN, pero se sugiere que solo sea usada en casos de contar con animales muertos.