Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Caldasia
Print version ISSN 0366-5232On-line version ISSN 2357-3759
Caldasia vol.30 no.2 Bogotá July/Dec. 2008
HERNANDO DEL CASTILLO
JEAN PAUL VERNOT
Laboratorio de Fisiología Celular y Molecular, Facultad de Medicina, Universidad Nacional de Colombia, Bogotá D.C., Colombia. hdelcs@yahoo.com; jpvernoth@unal.edu.co
RESUMEN
La cadena z es una proteína transmembranal que se encuentra en los linfocitos T, uno de los tipos de células más importantes en el sistema inmune adaptativo, haciendo parte de un complejo multimérico que se ubica en la superficie de la célula y que se encarga, por un lado, del reconocimiento antigénico y por otro, de la transmisión de señales durante la activación de la misma. Esta proteína es una de las que primero se fosforila luego de la estimulación a través del receptor y se sabe que es esencial en los primeros eventos de señalización. Dobles mutantes de esta molécula muestran una respuesta adaptativa inadecuada; así mismo, se ha visto que su expresión está alterada en cáncer y en algunas enfermedades auto-inmunes. La fosforilación en residuos de tirosina que se encuentran en los motivos de activación de residuos de tirosina de los receptores inmunes (ITAMs), de los cuales hay tres en la región citoplasmática, ha demostrado ser uno de los eventos principales durante la activación. En este reporte se presenta la caracterización de la estructura primaria de la cadena z en los monos del nuevo mundo Aotus nancymaae; la secuencia de aminoácidos se dedujo a partir de la secuencia nucleotídica de su cADN. Las moléculas de Aotus presentaron una identidad del 95.5 y del 95.7% con la molécula de humano en las secuencias de nucleótidos y de aminoácidos, respectivamente. Una comparación de la proteína por regiones, mostró una identidad del 100% en la región extracelular y la conservación de residuos y regiones importantes en el resto de la molécula; esto incluye aminoácidos polares en la región transmembranal y los ITAMs en la región citoplasmática. Los resultados sugieren que la proteína de Aotus es plenamente funcional y que los procesos de activación de los linfocitos T se suceden de manera similar a lo descrito en humanos. El resultado mostrado aquí soporta las evidencias previas en el sentido de que las estructuras moleculares involucradas en la respuesta inmune adaptativa se hallan conservadas.
Palabras clave. Aotus nancymaae, inmunidad adaptativa, cadena z, complejo asociado, ITAMs
ABSTRACT
The z chain is a transmembrane protein belonging to a multimeric complex in association with the T-cell receptor (TCR). The TCR is responsible for antigen recognition while the associated complex is responsible for signalling during activation. It has been shown that z chain expression becomes reduced in cancer and some autoimmune diseases. Tyrosine phosphorylation in immunoreceptor tyrosine-based activation motifs (ITAMs), present in the complex's different proteins is a critical event during activation. This report characterises the z chain in the new world monkey Aotus nancymaae; the amino acid sequence was deduced from its cDNA sequence. Aotus molecules revealed 95.5% and 95.7% identity with the human counterpart in nucleotide and amino acid sequences, respectively. Comparing different regions revealed 100% identity in the extra-cellular domain and conservation of important residues and domains in other regions of the protein: these included polar amino acids in the transmembrane region and the ITAMs. Our results suggest that the Aotus protein is fully functional and T-lymphocyte activation proceeds as in humans, supporting previous evidence that molecular structures involved in adaptive immune response are extremely conserved.
Key words. Aotus nancymaae, adaptive immunity, z chain, associated complex, ITAMs.
INTRODUCCIÓNLa respuesta inmune representa uno de los aspectos biológicos más fascinantes de los seres vivos; la capacidad de diferenciar lo propio de lo ajeno, permitiendo la preservación de lo propio y a la eliminación de lo extraño (Hoebe et al. 2004, Schulenburg et al. 2004). Se han descrito básicamente dos tipos de inmunidad: la innata y la adaptativa (Marchalonis et al. 2002, Bartl et al. 2003). La primera es una respuesta en la cual moléculas receptoras presentes en la superficie celular, reconocen estructuras o patrones moleculares en diferentes organismos, haciendo que se desencadene un ataque directo sobre ellos. Este tipo de respuesta se encuentra en invertebrados, plantas, hongos, pero también en los vertebrados (Schulenburg et al. 2004). La segunda es una respuesta con un grado muy fino de especificidad, en la cual se distinguen dos características sobresalientes: 1-requiere de la presentación antigénica, que es llevada a cabo por las células presentadoras de antígeno (CPA) en el contexto de moléculas del complejo mayor de histocompatibilidad (CMH) y 2-los sistemas de reconocimiento específico se basan en receptores moleculares los cuales generan su repertorio de posibilidades usando un sistema de recombinación a través de las enzimas recombinasas RAG1 y RAG2 (Cooper et al. 2006). La inmunidad adaptativa es la responsable de la memoria inmunológica (Anderson et al. 2004).
La inmunidad adaptativa se encuentra únicamente a partir de los vertebrados mandibulados y sugiere una explosión súbita que llevo a la emergencia de esta propiedad (Flajnik et al. 2004). En primer lugar una duplicación parcial o total del genoma en un ancestro común de los vertebrados mandibulados creó un escenario con nuevas posibilidades para el desarrollo de las estructuras moleculares responsables de este tipo de inmunidad (Kasahara et al. 2004); por otra parte, la adquisición, posiblemente por transferencia lateral, de los genes que codifican las enzimas recombinasas, los cuales quedaron inmersos dentro del ADN que determina a los receptores (Flajnik et al. 2004, Kasahara et al. 2004), dotaron a este ancestro con las herramientas que permitieron el desarrollo del sistema inmune adaptativo. Si bien en los vertebrados superiores la inmunidad adaptativa parece haberse desarrollado independientemente de la inmunidad innata, no se produce en estos organismos una respuesta inmunitaria satisfactoria sin el concurso de los dos sistemas, la inmunidad adaptativa permite reconocer estructuras antigénicas muy particulares, pero no parece ser capaz de detectar la presencia del patógeno (Janeway et al. 2002).
Uno de los tipos celulares más importantes y característico de la respuesta adaptativa son los linfocitos T (Krogsgaard et al. 2005). Estas células presentan en su superficie un complejo compuesto por diferentes proteínas transmembranales; tienen una capacidad de reconocimiento que se fundamenta en la presencia del denominado receptor de células T (TCR) y tienen además una capacidad de señalización determinada por el complejo asociado al receptor, el cual está formado por los heterodímeros (ed y ey) y por los homodímeros zz (Cantrell 1996).
Todas las cadenas de este complejo asociado tienen en su dominio intracelular los llamados motivos de activación basados en tirosina de los receptores inmunes (ITAMs). Estos ITAMs no son exclusivos de las cadenas del complejo asociado, se han encontrado en otro tipo de moléculas y tienen la estructura característica YxxLxxxxxxxxYxxL, en donde las Y son tirosinas, las L son leucinas y las x cualquier aminoácido (Humphrey et al. 2005); la fosforilación por proteínas quinasas de los residuos de tirosina (Y) dentro del ITAM promueve la asociación de otras proteínas adaptadoras y enzimas que portan los denominados dominios SH2 (Reth et al. 2004), en esta dinámica la señal progresa. Una vez que una célula T reconoce un péptido antigénico en el contexto de una CPA, se da inicio a un complicado programa bioquímico compuesto de muchos pasos interconectados, el cual puede llevar en algunos casos a una respuesta proliferativa y efectora, en otros no dar respuesta alguna o también progresar hacia la muerte celular programada, apoptosis (Germain et al. 1999).
La cadena z al igual que las otras asociadas al receptor (TCR) es una molécula proteica transmembranal, que presenta un dominio extracelular relativamente pequeño y un muy importante dominio intracelular en donde se destaca la presencia de 3 ITAMs (Yamamoto et al. 2005). Esta molécula es de las primeras que aparecen fosforiladas luego de la estimulación de la célula T; se han distinguido dos formas de la proteína fosforilada, la p21 y la p23; con base en evidencia experimental se han propuesto dos maneras para explicar las fosforilación de los ITAMs, que lleva a estas dos formas; en una de ellas, trabajando con anticuerpos que reconocen cada uno de los ITAMs fosforilados, se mostró que se produce una fosforilación ordenada, en la que la fosforilación del ITAM más proximal a la membrana precede a las demás (Kersh et al. 1998). En la otra, trabajando con mutantes en los cuales se han reemplazado las tirosinas por fenilalaninas, se mostró que la fosforilación es sucesiva y que empieza por el ITAM más distal de la membrana, siguiendo con el intermedio, para rendir la forma p21 y terminando con el más proximal, para producir la forma p23, en la que todos los ITAMs están fosforilados (Pitcher et al. 2003a). Se ha sugerido que la fosforilación diferencial de z entre las formas p21 y p23 se correlaciona con la activación de la célula (Stefanová et al. 2003); sin embargo, la evidencia experimental es controversial en este sentido; se ha propuesto también que las cadenas z pueden ser relevantes en la discriminación de señales, lo cual puede determinar respuestas diferentes por parte del linfocito T (Pitcher et al. 2003b).
En este reporte se presenta la caracterización de la estructura primaria de la cadena z (secuencia deducida de aminoácidos) en los monos del nuevo mundo Aotus nancymaae. La investigación de esta molécula se hace muy importante en inmunología, teniendo en cuenta la importancia que tiene en la respuesta adaptativa a través del papel que juega durante la activación del linfocito T. Por otra parte, la determinación de la estructura primaria de esta proteína en monos del nuevo mundo como el Aotus, ofrece opciones de comparación en análisis filogenéticos; por último hay que resaltar la utilidad de estos animales como modelo experimental en el estudio de enfermedades parasitarias de humanos, teniendo en cuenta su susceptibilidad a las mismas.
MATERIALES Y MÉTODOS
Muestras Biológicas
Se obtuvieron células mononucleares de sangre periférica (CMSP) de monos Aotus nancymaae, capturados en la Amazonía colombiana; todas las muestras de sangre se analizaron para descartar la presencia de filarias; solamente aquellas muestras libres de parásitos se procesaron. Las CMSP se aislaron usando centrifugación en un gradiente de Ficoll-Hypaque (Sigma, St Louis MO); las células se contaron y la viabilidad se determinó por exclusión del colorante azul de tripán; las muestras con más de un millón de células se usaron para extraer el RNA total. Todos los animales se manejaron de acuerdo a la normatividad contenida en la resolución 008430 de 1993 del Ministerio de Salud y a los protocolos aprobados por el comité de ética de la FIDIC.
Extracción de RNA total y síntesis de cDNA
El RNA total se aisló de las CMSP de A. nancymaae utilizando el método del reactivo de Trizol (Invitrogen, Life Technologies, Carlsbad CA) y se almacenó a -70ºC. RNA total de humano se aisló en las mismas condiciones y se utilizó como control. La síntesis de cDNA se realizó usando el sistema One-step RT-PCR (Invitrogen, Life Technologies, Carlsbad CA). En la reacción se utilizaron los primers específicos: TGCCTCAGCCTCTGCCTC (sentido) y CCCCTGGCTGTTAGCGAG (antisentido). Se utilizó el siguiente perfil de síntesis: 30 min a 55ºC; 2 min a 94ºC; 40 X (15 seg a 94ºC; 30 seg a 60ºC; 60 seg a 68ºC); 5 min a 68ºC.
Ligación, secuenciación y análisis de secuencias
Los productos de amplificación se observaron en geles de agarosa al 1% y después de comprobar el tamaño adecuado del producto se realizó la ligación en el vector plasmídico pGEM-T easy (Promega, Madison). Todas las reacciones de secuencia se realizaron por Macrogen Inc. (www.macrogen.com); se utilizaron los primers universales PUC-M13 dirigidos contra el vector, la metodología de Sanger, usando terminadores bigdye en condiciones cíclicas y el sistema ABI3730XL (PE Applied Biosystems).
Las secuencias obtenidas se editaron con el software Chromas (versión 1.45) y la secuencia de aminoácidos se dedujo con Generunner (versión 3.05); los alineamientos se realizaron usando ClustalX (versión 1.83) y se editaron con el Genedoc (versión 2.6.001); se usó la matrix PAM 250.
RESULTADOS
Generación del cDNA de z de A. nancymaae
En general se obtuvieron pocas células de cada una de las muestras de sangre periférica de A. nancymaae (1'100,000-1'900,000), y por consiguiente, poco ARN total pudo ser aislado, la figura 1 A muestra el resultado de una de las electroforesis después de la extracción. Teniendo en cuenta este resultado, se decidió combinar el ARN total de 8 muestras diferentes antes de realizar la amplificación.
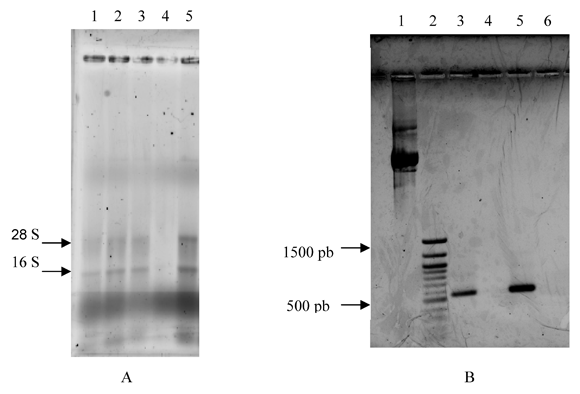
Los primers para la amplificación de la cadena z de A. nancymaae, se sintetizaron con base en la secuencia de la molécula de humanos, cuidando que toda la estructura codante quedara incluida. El producto de amplificación se observó en gel de agarosa (Figura 1 B); se aprecia una banda por debajo de 600 pb, carriles 3 y 5. El cDNA producido a partir del ARN total de Aotus se ligó en el vector pGEM-T easy (Promega, Madison), la presencia del inserto se confirmó por PCR usando primers contra el vector o los específicos de la cadena z en reacciones independientes.
Secuencia de la cadena z de Aotus nancymaae
La secuencia nucleotídica de la cadena z de A. nancymaae se envió el Genbank y recibió el número de acceso EF656481.
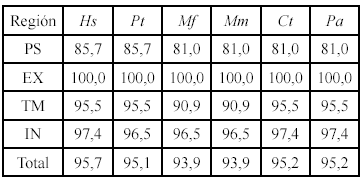
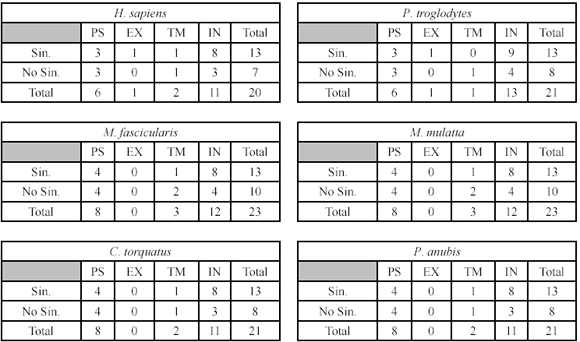
La secuencia obtenida fue de 547 nucleótidos; los análisis de marco abierto de lectura realizados con el GeneRuner (3.05), permitieron determinar en el marco de lectura 1 una proteína de aprox. 20.2 kDa que se corresponde con el tamaño reportado para la cadena z; utilizando este mismo software, se predijo la secuencia de aminoácidos de la cadena z de A. nancymaae en este marco de lectura. Los respectivos alineamientos de la molécula de Aotus con la de otros primates se hicieron usando ClustalX (1.83). Los resultados obtenidos muestran una identidad por encima de 95% en secuencia de nucleótidos entre Aotus y otros primates del viejo mundo incluido el humano y una identidad por encima del 93% en secuencia de aminoácidos en la misma comparación (Tabla 1). Un total de 13 cambios sinónimos se encontraron en la comparación Aotus cf. primates del viejo mundo y entre 6-10 cambios no sinónimos (Tabla 2). Con respecto a los humanos y los chimpancés el Aotus mostró la inserción de 2 codones en la región intracelular; con los macacos un codón también en la región intracelular; ni con los babuinos ni con los papiones se encontró esta diferencia.
La comparación de la estructura primaria de la proteína z de Aotus como ya se mencionó, mostró un alto grado de identidad con su contra parte humana, la región de la molécula con mayor variación resulta ser la del péptido señal (Tabla 1), la cual no queda incluida en la proteína madura. Residuos que se han descrito como esenciales para la funcionalidad de la molécula como la cisteína (C) (posición 11) y el ácido aspártico (D) (posición 15) en la región transmembranal, así como las tirosinas (Y) (posiciones 51, 62, 90, 102, 121 y 132) en los ITAMs, están absolutamente conservados; todas las posiciones de los residuos se dan con base en la proteína madura. En la figura 2 se muestra un alineamiento entre las cadenas z de humano y de Aotus; se señalan las regiones de la proteína y se destacan algunos residuos y sub-regiones importantes en cada una de ellas.

DISCUSIÓN
La estructura primaria de la cadena z de los monos nocturnos A. nancymaae, presentada en este reporte, mostró una identidad de 95.5% en secuencia nucleotídica y de 95.7% en secuencia de aminoácidos con su contra parte humana; con otros primates del viejo mundo entre 95-95.5% en secuencia de nucleótidos y 93.5-94% en secuencia de aminoácidos, respectivamente.
Nueve exones codifican esta proteína; en mamíferos se han descrito 3 isoformas: z1, z2 y n; las formas z y n se producen por splicing alternativo de los dos últimos exones, en ellas la proteína se diferencia en la región carboxi-terminal; las formas z1 y z2 se diferencian en que en z1 hay un codón adicional en la región citoplasmática, entre el primero y el segundo ITAM, la variante se produce por un segmento localizado en el extremo 3' del cuarto intrón (Atkinson et al. 2003). La secuencia de Aotus reportada aquí es más semejante a la isoforma z1; se ha propuesto que la expresión de las dos isoformas z está conservada en diferentes organismos. Sin embargo, establecer si en Aotus también se expresan las dos, requiere analizar un mayor número de muestras.
Un análisis por regiones mostró que en la región del péptido señal es donde se encuentra el mayor porcentaje de variación en todas las comparaciones realizadas entre Aotus y los primates del viejo mundo (Tabla 1); hay de 6-8 cambios entre sinónimos y no sinónimos en esta región que es de 21 codones, en comparación con de 13-15 cambios en el resto de la molécula que es de 145 codones (Tabla 2), hay que señalar que el péptido señal no hace parte de la proteína madura.Caracterizaciones previas de otras moléculas transmembranales de Aotus (Montoya et al. 2002, 2004), así como de moléculas del complejo CD3 en otros primates (Uda et al. 2001, Liu et al. 2007), han mostrado una variación importante en la región extracelular. Este no es el caso de la cadena z, en donde se encontró una identidad del 100% entre Aotus y otros primates. Esta región es bastante pequeña, solamente tiene 9 aminoácidos y se ha sugerido que es importante tanto en la asociación del complejo como en la señalización (Bolliger et al. 1997); se ha mostrado también que la cadena z es esencial en la expresión del complejo en la superficie celular y en la internalización del mismo que se produce después de la interacción con las células presentadoras, la región extracelular podría jugar un papel importante en esta movilización (La Gruta et al. 2004). Con base en lo anterior y con un tamaño tan reducido de la región extracelular, tal vez se pueda explicar la identidad absoluta que se encuentra en la comparación con los otros primates.
La cadena z forma homodímeros z-z y ocasionalmente heterodímeros z-n, los cuales se producen por un puente disulfuro; una cisteína en la posición 2 de la porción transmembranal es la responsable de este enlace (Orloff et al. 1989). La región transmembranal de las proteínas del complejo asociado y las cadenas a y ß del receptor tienen como característica la presencia de aminoácidos polares; los de las cadenas a y ß son de carácter básico y los del resto de las cadenas son ácidos. Se ha establecido que estos aminoácidos polares son esenciales para el ensamblaje del complejo (Call et al. 2005). Se ha mostrado adicionalmente que el ácido aspártico (D) en la posición 6, la leucina (L) en la posición 9, la tirosina (Y) en la posición 12, la treonina (T) en la posición 17 y la fenilalanina (F) en la posición 20, resultan esenciales para la formación de los dímeros (Call et al. 2006). En la cadena z de Aotus que se está reportando, todos estos aminoácidos, se encuentran conservados.
En cuanto a la región intracelular de la cadena z, se encontró nuevamente un alto grado de identidad entre Aotus y los primates del viejo mundo, los tres ITAMs que son las subregiones más características de esta porción de la proteína están plenamente conservados. Se ha propuesto que la fosforilación de estos ITAMs es un evento clave en la señalización tanto en la maduración de los timocitos, como en la activación del linfocito T en la periferia (Kersh et al. 1999). Sin embargo, algunos hallazgos experimentales son controversiales acerca del papel que juega la fosforilación en estos eventos. Se han planteado dos modelos para explicar la activación que no son del todo excluyentes: en uno, teniendo en cuenta la multiplicidad de ITAMs en todo el complejo, se habla de una posible redundancia y que diferentes combinaciones de ITAMs fosforilados se encargan de la transmisión de la señal. En el otro se habla de independencia de los módulos (z y CD3), siendo el complejo CD3 el predominante para la progresión de la señal y z ejerciendo el papel de sensores (Pitcher et al. 2003b). Los diferentes estados de fosforilación de los ITAMs en la cadena z pueden ser esenciales para determinar cuales moléculas se asocian con ellos y de esta forma establecer tipos diferentes de respuesta (Chae et al. 2004).
El alto grado de conservación que se encuentra en la cadena z dentro de los primates, incluido el Aotus, es indicio de su relevancia fisiológica y sugiere que la funcionalidad de la molécula de Aotus es semejante a humanos en aspectos como: la fosforilación (Pitcher et al. 2003c, 2005); la dinámica de la recirculación del TCR que involucra a z (D'Oro et al. 2002, Lauritsen et al. 2004, Myers et al. 2005); y en algunos cambios estructurales que podría exhibir esta cadena y que estarían relacionados con los eventos de fosforilación de los ITAMs (Aivazian et al. 2000, Sigalov et al. 2004). En el caso de otras cadenas del complejo como la CD3z se ha mostrado en un estudio en humanos que cambios en algunos aminoácidos, tienen efectos dramáticos en la asociación con CD3e y en la expresión en superficie de todo el complejo (Thomassen et al. 2006). Quizás haya controversia en cuanto a la función específica de la fosforilación de los ITAMs de la cadena z durante la activación del linfocito T, pero es claro que esta molécula es esencial para una adecuada respuesta inmune adaptativa, tal es así, que en cáncer y en enfermedades auto-inmunes como el lupus eritematoso se han reportado alteraciones en la expresión de esta proteína (Whiteside 2004).
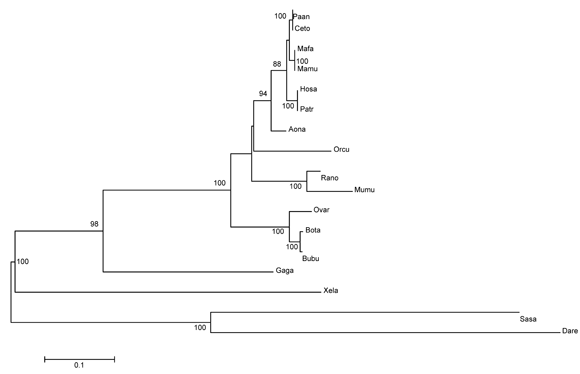
Un análisis filogenético con base en la secuencia de aminoácidos de z (Figura 3), agrupó al A. nancymaae con los otros primates, como era de esperarse. La secuencia de Aotus mostró una inserción de 2 aminoácidos en la región intracelular con respecto a humanos y chimpancé. Sin embargo, ni con los macacos, ni con el papión ni con el mangabey se encontró esta diferencia. Esto sugiere que el carácter visto en Aotus es ancestral y que más bien humanos y chimpancés perdieron los dos codones después de la divergencia entre los primates del viejo y del nuevo mundo, la cual se produjo hace aproximadamente 500 millones de años.
La estructura primaria de la cadena z de A. nancymaae mostrada aquí, confirma que esta proteína tiene un alto grado de identidad entre diferentes organismos, a pesar de los tiempos de divergencia entre los mismos, esto muestra que está sometida a una gran restricción funcional. Ya se ha planteado que las moléculas del complejo TCR y las rutas de señalización se hallan conservadas (Gouaillard et al. 2001) y si bien estos hechos apoyarían la utilización de Aotus como modelo experimental para probar vacunas y medicamentos con uso potencial en humanos, habría que decir también que las comparaciones muestran que otros organismos tienen características semejantes.
AGRADECIMIENTOS
Agradecemos a los Drs. Alberto Pérez y Carlos Suárez, por la lectura crítica del manuscrito y por los análisis filogenéticos respectivamente; a la Fundación Instituto de Inmunología de Colombia (FIDIC), por el suministro de las muestras de sangre de Aotus y a la Fundación para la Promoción de la Investigación y la Tecnología del Banco de la República, por la financiación que permitió el desarrollo del presente trabajo.
LITERATURA CITADA
1. AIVAZIAN, D. & L.J. STERN. 2000. Phosphorylation of T cell receptor z is regulated by a lipid dependent folding transition. Nature Structural Biology 7: 1023-1026. [ Links ]
2. ANDERSON, M. R. PANT, A.L. MIRACLE, X. SUN, C.A. LUER, C.J. WALSH, J.C. TELFER, G.W. LITMAN & E.V. ROTHENBERG. 2004. Evolutionary origins of lymphocytes: Ensembles of T cell and B cell transcriptional regulators in a cartilaginous fish. Journal of Immunology 172: 5851–5860. [ Links ]
3. ATKINSON, T.P., C.G. HALL, J. GOLDSMITH & P.M. KIRKHAM. 2003. Splice variant in TCR z links T cell receptor signaling to a G-protein-related signaling pathway. Biochemical Biophysics Research Communication 310: 761–766. [ Links ]
4. BARTL, S., M. BAISH, I.L. WEISSMAN & M. DIAZ. 2003. Did the molecules of adaptive immunity evolve from the innate immune system? Integrative and Comparative Biology 43: 338–346. [ Links ]
5. BOLLIGER, L., B. JOHANSSON & E. PALMER. 1997. The short extracellular domain of the T cell receptor zeta chain is involved in assembly and signal transduction. Molecular Immunology 34: 819-827. [ Links ]
6. CALL, M.E. & K.W. WUCHERPFENNIG. 2005. The T cell receptor: Critical role of the membrane environment in receptor assembly. Annual Reviews Immunology 23: 101–125. [ Links ]
7. CALL, M.E., J.R. SCHNELL, C. XU, R.A. LUTZ, J.J. CHOU & K.W. WUCHERPFENNIG. 2006. The structure of the zz transmembrane dimer reveals features essential for its assembly with the T cell receptor. Cell 127: 355–368. [ Links ]
8. CANTRELL, D. 1996. T Cell antigen receptor signal transduction pathways. Annual Reviews Immunology 14: 259–74. [ Links ]
9. CHAE, W.J. H.K. LEE, J.H. HAN, S.W.V. KIM, A.L.M. BOTHWELL, T. MORIO & S.K. LEE. 2004. Qualitatively differential regulation of T cell activation and apoptosis by T cell receptor z chain ITAMs and their tyrosine residues. International Immunology 16: 1225–1236. [ Links ]
10. COOPER, M. & M. ALDER. 2006. The evolution of adaptive immune systems. Cell 124: 815–822. [ Links ]
11. D'ORO, U., I. MUNITIC, G. CHACKO, T. KARPOVA, J. MCNALLY & J.D. ASHWELL. 2002. Regulation of constitutive TCR internalization by the z-chain. Journal of Immunology 169: 6269–6278. [ Links ]
12. FLAJNIK, M.F. & L. DU PASQUIER. 2004. Evolution of innate and adaptive immunity: can we draw a line? Trends in Immunology 25: 640-644. [ Links ]
13. GERMAIN, R.N. & I. STEFANOVÁ. 1999. The dynamics of T cell receptor signaling: Complex orchestration and the key roles of tempo and cooperation. Annual Reviews Immunology 17: 467–522. [ Links ]
14. GOUAILLARD, C., A. HUCHENQ-CHAMPAGNE, J. ARNAUD, C.H. CHEN & B. RUBIN. 2001. Evolution of T cell receptor (TCR) aß heterodimer assembly with the CD3 complex. European Journal of Immunology 31: 3798–3805. [ Links ]
15. HOEBE, K., E. JANSSEN & B. BEUTLER. 2004. The interface between innate and adaptive immunity. Nature Immunology 5: 971-974. [ Links ]
16. HUMPHREY, M.B., L.L. LANIER & M.C. NAKAMURA. 2005. Role of ITAM-containing adapter proteins and their receptors in the immune system and bone. Immunological Reviews 208: 50–65. [ Links ]
17. JANEWAY, C.A. JR. & R. MEDZHITOV. 2002. Innate immune recognition. Annual Reviews Immunology 20: 197–216. [ Links ]
18. KASAHARA, M., T. SUZUKI & L. DU PASQUIER. 2004. On the origins of the adaptive immune system: novel insights from invertebrates and cold-blooded vertebrates. Trends in Immunology 25: 105-111. [ Links ]
19. KERSH, E.N., A.S. SHAW & P.M. ALLEN. 1998. Fidelity of T cell activation through multistep T cell receptor z phosphorylation. Science 281: 572-575. [ Links ]
20. KERSH, E.N., G.J. KERSH & P.M. ALLEN. 1999. Partially phosphorylated T cell receptor z molecules can inhibit T cell activation. Journal of Experimental Medicine 190: 1627–1636. [ Links ]
21. KROGSGAARD, M. & M.M. DAVIS. 2005. How T cells 'see' antigen. Nature Immunology 6: 239-245. [ Links ]
22. LA GRUTA, N.L., H. LIU, S. DILIOGLOU, M. RHODES, D.L. WIEST & D. VIGNALI. 2004. Architectural changes in the TCR:CD3 complex induced by MHC:peptide ligation. Journal of Immunology 172: 3662–3669. [ Links ]
23. LAURITSEN, J.P.H., M.C. BONEFELD, M. VON ESSEN, M.W. NIELSEN, A.B. RASMUSSEN, N. ØDUM, J. DIETRIC & C. GEISLER. 2004. Masking of the CD3y di-leucine-based motif by z is required for efficient T-cell receptor expression. Traffic 5: 672–684. [ Links ]
24. LIU, Y.Y., Z. WANG, J. THOMAS, K.J. GOODWIN, S. STAVROU & D.M. NEVILLE JR. 2007. Polymorphisms of CD3e in cynomolgus and rhesus monkeys and their relevance to anti-CD3 antibodies and immunotoxins. Immunology Cell Biology 85:357-362. [ Links ]
25. MARCHALONIS, J.J., S. KAVERI, S. LACROIX-DESMAZES & M. KAZATCHKINE. 2002. Natural recognition repertoire and the evolutionary emergence of the combinatorial immune system. The FASEB Journal 16: 842-848. [ Links ]
26. MONTOYA, G.E., J.P. VERNOT & M.E. PATARROYO. 2002. Partial characterization of the CD45 phosphatase cDNA in the owl monkey (A. vociferans). American Journal of Primatology 57: 1-11. [ Links ]
27. MONTOYA, G.E., J.P. VERNOT & M.E. PATARROYO. 2004. Comparative analysis of CD45 proteins in primate context: owl monkeys vs humans. Tissue Antigens 64: 165-172. [ Links ]
28. MYERS, M.D., L.L. DRAGONE & A. WEISS. 2005. Src-like adaptor protein down-regulates T cell receptor (TCR)–CD3 expression by targeting TCRz for degradation. Journal of Cell Biology 170: 285–294. [ Links ]
29. ORLOFF, D.G., S.J. FRANK, F.A. ROBEY, A.M. WEISSMAN & R.D. KLAUSNER. 1989. Biochemical characterization of the z chain of the T-cell receptor. Journal of Biological Chemistry 264: 14812-14817. [ Links ]
30. PITCHER, L.A., J.A. YOUNG, M.A. MATHIS, P.C. WRAGE, B. BARTÓK & N.S.C. VAN OERS. 2003a. The formation and functions of the 21- and 23-kDa tyrosine phosphorylated TCRz subunits. Immunological Reviews 191: 47–61. [ Links ]
31. PITCHER, L.A. & N.S.C. VAN OERS. 2003b. T-cell receptor signal transmission: who gives an ITAM? Trends in Immunology 24: 554-560. [ Links ]
32. PITCHER, L.A., P.S. OHASHI & N.S.C. VAN OERS. 2003c. T Cell antagonism is functionally uncoupled from the 21- and 23-kDa tyrosine-phosphorylated TCRz subunits. Journal of Immunology 171: 845–852. [ Links ]
33. PITCHER, L.A., M.A. MATHIS, S. SUBRAMANIAN, J.A. YOUNG, E.K. WAKELAND, P.E. LOVE & N.S.C. VAN OERS. 2005. Selective Expression of the 21-kilodalton tyrosine-phosphorylated form of TCR z promotes the emergence of T cells with autoreactive potential. Journal of Immunology 174: 6071–6079. [ Links ]
34. RETH, M. & T. BRUMMER. 2004. Feedback regulation of lymphocyte signalling. Nature Reviews Immunology 4: 269-277. [ Links ]
35. SCHULENBURG, H., C.L. KURZ & J.J. EWBANK. 2004. Evolution of the innate immune system: the worm perspective. Immunological Reviews 198: 36–58. [ Links ]
36. SIGALOV, A., D. AIVAZIAN & L. STERN. 2004. Homooligomerization of the cytoplasmic domain of the T cell receptor z chain and of other proteins containing the immunoreceptor tyrosine-based activation motif. Biochemistry 43: 2049-2061. [ Links ]
37. STEFANOVÁ, I., B. HEMMER, M. VERGELLI, R. MARTIN, W.E. BIDDISON & R.N. GERMAIN. 2003. TCR ligand discrimination is enforced by competing ERK positive and SHP-1 negative feedback pathways. Nature Immunology 4: 248-254. [ Links ]
38. THOMASSEN, E.A.J., E.H.A. DEKKING, A. THOMPSON, K.L. FRANKEN, Ö. SANAL, J.P. ABRAHAMS, M.J.D. VAN TOL & F. KONING. 2006. The impact of single amino acid substitutions in CD3y on the CD3ey interaction and T-cell receptor–CD3 complex formation. Human Immunology 67: 579–588. [ Links ]
39. UDA, A., K. TANABAYASHI, R. MUKAI, M. YACHI, K.H. NAM & A. YAMADA. 2001. CD3 polymorphism in cynomolgus monkeys (Macaca fascicularis). Journal of Medical Primatology 30: 141-147. [ Links ]
40. WHITESIDE, T.L. 2004. Down-regulation of z-chain expression in T cells: a biomarker of prognosis in cancer? Cancer Immunology Immunotherapeutics 53: 865–878. [ Links ]
41. YAMAMOTO, R., T. ISOBE, T. EGUCHI, W.R. TANG, N. KIYOKAWA, H. AMEMIYA, J. FUJIMOTO, E. SATO, Y. TAKAGAKI & H. YASUE. 2005. Porcine TCR CD3z-chain and n-chain. Molecular Immunology 42: 1485–1493. [ Links ]
Recibido: 10/09/2007
Aceptado: 29/08/2008














