INTRODUCCIÓN
El Bosque Seco Tropical (bs-T) es definido como un ecosistema que se produce en climas cálidos durante todo el año, que incluye periodos de abundante precipitación y largas estaciones secas (Constantino c2012). Está presente en casi todos los continentes, pero más de la mitad de la cobertura remanente de este ecosistema se encuentra en Suramérica (Sánchez-Azofeifa et al. 2005, Miles et al. 2006). En el caso de Colombia, el bs-T ocupa un área intermedia, entre los bosques de Centroamérica y los de Suramérica (Etter 1993, Sánchez-Azofeifa et al. 2013), presentando también niveles intermedios de endemismo y diversidad beta (Linares-Palomino et al. 2011). No obstante, igual que los bosques regionales, el bs-T en Colombia se encuentra gravemente degradado y distribuido en fragmentos aislados de diversos tamaños desde la región Caribe hasta el enclave del río Patía al Suroccidente del país (Pizano y García 2014). Estas características lo convierten en un ecosistema de gran prioridad para la conservación (Dirzo y Raven 2003).
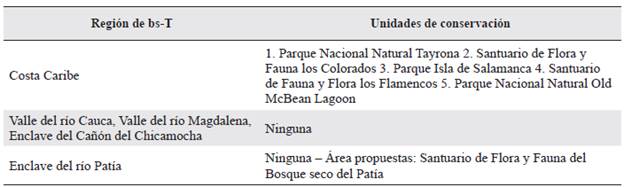
Sin embargo, la baja representación del bs-T en el sistema nacional colombiano de áreas protegidas (SINAP) (Tabla 1)y la escasa información a escala nacional de los componentes de su biodiversidad han generado fuertes limitaciones para establecer estrategias para su recuperación y conservación. Cabe mencionar que en las últimas dos décadas se ha incrementado el interés por el estudio de este ecosistema, no obstante, las investigaciones en su mayoría corresponden a caracterizaciones florísticas y ecológicas a escala local (Marulanda et al. 2003, Barreto et al. 2007, Linares y Fandiño 2009, Díaz-Pérez et al. 2011, Vargas 2012, Fernández-Méndez et al. 2013, Camargo 2015, Castellanos-Castro y Newton 2015, Ramírez-Padilla et al. 2015, Vitola et al. 2017).
Tabla 1 Regiones de bosque seco tropical (bs-T) en Colombia y unidades de conservación del Sistema Nacional de áreas Protegidas (SINAP) asociadas.
La conservación de ecosistemas a largo plazo no solo depende de la preservación de la riqueza de especies en las comunidades, sino también depende de la diversidad genética de las poblaciones de especies clave dentro del paisaje (Hughes et al. 1997, Luck et al. 2003). Conservar poblaciones genéticamente diversas permite maximizar el potencial evolutivo, mejorar las respuestas a disturbios, reducir al mínimo los riesgos a largo plazo de extinción y perpetuar los servicios que prestan las especies de plantas a la sociedad (Allendorf y Luikart 2009). A pesar de que el entendimiento de la diversidad y estructura genética de las poblaciones y su nivel de conexión entre los fragmentos es imprescindible para implementar estrategias de restauración y conservación efectivas (Cascante et al. 2002, Mckay et al. 2005, Allendorf y Luikart 2009), en Colombia existen muy pocos estudios enfocados a especies claves del bs-T (Thomas et al. 2017a, Bocanegra-González et al. 2018, Bocanegra-González y Guillemin 2018). Dicha información es indispensable en el contexto actual de cambio climático y de aumento de las presiones antrópicas sobre las especies tropicales maderables (Blaser et al. 2011), ya que el potencial efecto de la fragmentación del bs-T sobre el componente genético de las especies genera una gran preocupación.
La fragmentación puede estar teniendo varias consecuencias en las especies arbóreas como la reducción del tamaño poblacional, la restricción del flujo génico entre fragmentos aislados, el aumento de la endogamia, la reducción del vigor de las plántulas y en casos extremos la extinción local (Kageyama et al. 1998, Aguilar et al. 2006). De hecho, estos efectos ya han sido registrados en especies nativas de los bs-T de Centro y Sur América como Cordia alliodora (Ruiz & Pav.) Oken (Chase et al. 1995), Samanea saman (Jacq.) Merr. (Cascante et al. 2002), Tabebuia ochracea (Cham.) Standl. (Moreira et al. 2009), Caryocar brasiliense Cambess. (de Melo Jr et al. 2012) y Dipteryx alata Vogel (Tambarussi et al. 2017). En Colombia, la ausencia de información genética sobre especies locales hace difícil definir los efectos potenciales de este fenómeno. No obstante, los primeros estudios demuestran que la fragmentación del bs-T ha tenido un fuerte impacto en la diversidad genética de poblaciones naturales de especies arbóreas como Enterolobium ciclocarpum (Jacq.) Griseb. (Thomas et al. 2017a), Anacardium excelsum L. (Bocanegra-González y Guillemin 2018) y Ceiba pentandra L. Gaertn (Bocanegra-González et al. 2018) localizadas en las áreas más aisladas y con mayores presiones antropogénicas como los enclaves del río Patía, el Cañón del Chicamocha y algunas zonas al norte del Valle del Río Cauca (Arcila-Cardona et al. 2012, Valencia-Duarte et al. 2012, Vergara 2015).
Un elemento adicional a destacar, es que las poblaciones de estas especies han mostrado un patrón de estructuración genética corroborando la separación histórica entre las áreas de bs-T del Caribe y de los valles interandinos (Espinal 1964, Munoz-Uribe 2012). Particularmente, el grado de estructuración genética de las especies es un elemento sumamente importante al momento de definir fuentes de germoplasma para programas de restauración (Mckay et al. 2005, Thomas et al. 2014, 2017b).
Esta investigación estimó el nivel de diversidad y estructura genética de cuatro especies clave del bs-T mediante marcadores moleculares ISSR (Inter Simple Sequence Repeat), los cuales ofrecen una ventaja en especies no modelo, ya que no necesitan conocimiento previo del genoma, y han dado resultados valiosos en otras especies arbóreas del bs-T (de Abreu Moreira et al. 2015, Jiménez et al. 2015, dos Santos et al. 2016).
Bursera simaruba (L.) Sarg. (Burseraceae), Platymiscium pinnatum (Jacq.) Dugand (Fabaceae) y Hura crepitans L. (Euphorbiaceae) son especies distribuidas sobre una amplia área del continente americano, todas presentando estados de conservación bajo la categoría no evaluado (NE) (Klitgaard 2005, Daly c2015, Murillo-A. c2015). No obstante, se ha reportado que la pérdida de hábitat ha afectado poblaciones de B. simaruba en Puerto Rico (Dunphy y Hamrick 2007) y México (Bravo-Bolaños et al. 2016). Del mismo modo, para H. crepitans se reportó que las poblaciones con los mayores niveles de diversidad genética se encuentran en áreas ya fragmentadas de bs-T en Venezuela, las cuales pueden presentar problemas de conservación en el futuro (Nassar et al. 2011). Por otro lado, Caesalpinia ebano H.Karst. (Fabaceae), es un árbol endémico de Colombia, que se distribuye desde el norte de la región del Valle del Río Cauca hasta el Caribe y su estado de conservación está en la categoría en peligro (EN) dada las pocas poblaciones que se mantienen hoy día (Cárdenas y Salinas 2007, Gradstein c2015). Nuestra hipótesis sugiere que las cuatro especies tienen niveles reducidos de diversidad genética dada la fragmentación del hábitat y además siguen un patrón de estructuración genética que indica una separación histórica entre las áreas de bs-T del Caribe y los valles interandinos. Finalmente, esperamos que este estudio contribuya directamente a los procesos de restauración y conservación de estas especies en los distintos fragmentos de bs-T en Colombia, delimitando potenciales fuentes de germoplasma y poblaciones prioritarias para la conservación.
MATERIALES Y MÉTODOS
Área de estudio
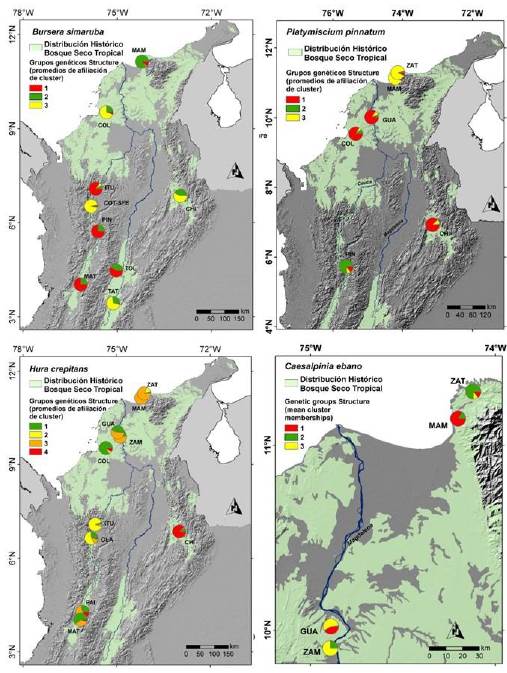
Se recolectaron muestras de tejido en catorce localidades de bosque seco tropical (bs-T) en Colombia (Tabla 2, Fig. 1). Las cuatro especies estudiadas no fueron halladas en cada una de las localidades: B. simaruba fue muestreada en nueve sitios, P. pinnatum en seis sitios, H. crepitans en diez sitios y C. ebano en cuatro sitios (Tabla 2, Fig. 1).
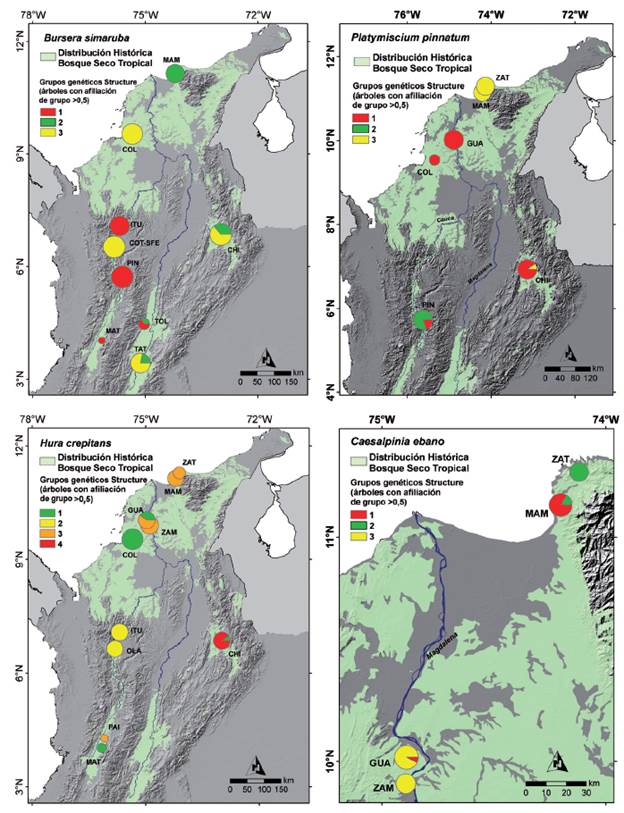
Figura 1 Localidades muestreadas por especie, grupos genéticos y afiliación de individuos a cada grupo. *El tamaño de los círculos representa el tamaño de la muestra por sitio.
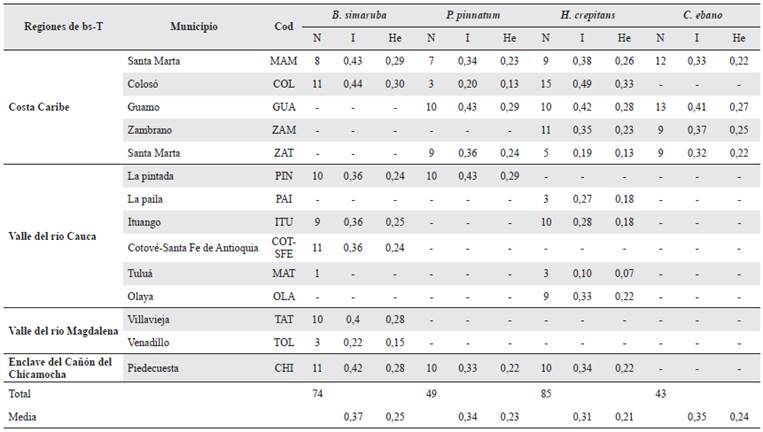
Tabla 2 Regiones de bosque seco tropical (bs-T) y distribución de la diversidad genética. Código de poblaciones estudiadas (Cod), Número de muestras (N), Índice de Shannon (I), Heterocigosidad esperada (He).
En cada localidad se recolectaron hojas jóvenes y sanas de individuos reproductivos de las especies disponibles. El número de individuos muestreados dependió de la abundancia en terreno (Tabla 2). Para cada especie se consideró una distancia mínima de 50 metros entre individuos para evitar en el muestreo altos valores de parentesco (González y Arenas-Castro 2017). El muestreo en campo fue guiado por mapas de idoneidad basados en datos de ocurrencia de las especies a lo largo del bs-T en Colombia obtenidos a partir de registros de colecciones botánicas. Todo el material biológico fue recolectado según el Decreto 302 de 2003 de Colombia.
Extracción de ADN y amplificación con marcadores ISSR
La extracción de ADN se realizó usando el procedimiento descrito por Doyle y Doyle (1987) y utilizando 500 mg de hojas secas. Se comprobó la amplificación de doce partidores de ISSR en cada especie estudiada (Material suplementario - Anexo 1). Para cada ISSR el programa de PCR contó con 37 ciclos de amplificación que consistió en 2 minutos de iniciación a 94 °C, desnaturalización a 94 °C 7 minutos. En cada reacción se usó un volumen durante 15 segundos, emparejamiento a 42 °C de 13 μ1, compuesto por 3 μ de ADN y 10 μ durante 30 segundos y extensión de 72 °C de mezcla [1,5 μ de PCR 10X (integrado por durante 1 minuto y un paso final a 72 °C durante 500 mM de Tris-HCl pH 8,0; 200 mM de KCl; 2,5 mg/ml de BSA; 200 mM de Tartrazina y 1 % de Ficoll) 1,5 μ de dNTP + MgCl2 (dNTP a 2,5 mM; MgCl2 a 25mM), 0,25 μ de Taq polimerasa, 2 μ! de primer (2 y el volumen restante se completó con agua pura].
A los productos de amplificación se añadieron 2μl de GelRed™, como marcador de peso molecular fue usado GeneRuler 100 pb (Thermo Scientific, Massachusetts, EE. UU.), y se separaron mediante electroforesis horizontal (80V, 3:30) en gel de agarosa al 1,5 % (p/v). Después de la electroforesis, el gel fue expuesto a la luz ultravioleta en el equipo UVP Doc-It-LS y fotografiado.
Análisis de datos
Los fragmentos amplificados de ISSR fueron incluidos en una matriz como ausentes (0), presentes (1) y en el caso de los datos faltantes como (-1). Se asumió que cada banda representaba el fenotipo en un solo locus bialélico (Crema et al. 2009). Los parámetros de diversidad genética de índice de Shannon (I) y heterocigosidad esperada (He) se calcularon utilizando el software GenAlEx 6.5 (Peakall y Smouse 2012) al igual que la estimación del número bandas privadas (Pa) en cada población.
Para evaluar la estructura genética de cada especie se realizaron análisis de conglomerados Bayesianos con STRUCTURE 2.2 (Pritchard et al. 2000). Los análisis se realizaron usando un modelo de mezcla (i.e., admixture) asumiendo que las frecuencias alélicas están correlacionadas entre poblaciones, con un quemado de 500 000 iteraciones y 1 000 000 iteraciones posteriores. Para B. simaruba, H. crepitans y C. ebano se tomaron en cuenta las localidades de muestreo para obtener resultados más estables y coherentes. Se usaron siete repeticiones independientes para cada número de grupos genéticos putativos (i.e. K) evaluado. Para cada especie en estudio, los K máximos evaluados correspondían al número de sitios muestreados con datos de ISSRs más uno. Se analizaron entonces para B. simaruba K = 1-10, P. pinnatum K = 1-7, H. crepitans, K = 1-11, C. ebano K = 1-5. Para estimar el número de K más probable en cada conjunto de datos, se calculó el ΔΚ de Evanno. De acuerdo con Evanno et al. (2005) el uso del valor ΔΚ permite encontrar el número real de grupo genéticos presentes en un conjunto de datos obtenido con marcadores dominantes, a pesar de existencia de divergencia genética leve. Utilizamos CLUMPP (Jakobsson y Rosenberg 2007) para unir los resultados de las siete ejecuciones independientes para cada valor de K. CLUMPP permite corroborar las similitudes entre los resultados entregados por las ejecuciones independientes realizadas en STRUCTURE y aliviar los efectos estocásticos siempre presentes en análisis de conglomerados Bayesianos.
RESULTADOS
Para cada una de las especies se generó un conjunto de datos de "fingerprinting" usando los genotipos únicamente para los partidores de ISSRs en los cuales se obtuvo una amplificación (i.e. al menos una banda visible en el gel) para el 90 % o más de los individuos, esto con el objetivo de evitar el exceso de datos faltantes. Para B. simaruba y P. pinnatum se usaron ocho partidores de ISSRs, para H. crepitans cinco partidores de ISSRs y para C. ebano nueve partidores de ISSRs (ver Material suplementario -Anexo 1 para más detalles). Además, para cada especie fueron eliminados previo a los análisis los individuos que presentaron fallas en más del 50 % de las amplificaciones. Se analizaron entonces 73 individuos de B. simaruba, 48 de P. pinnatum, 85 de H. crepitans y 41 de C. ebano (Tabla 2).
Diversidad genética
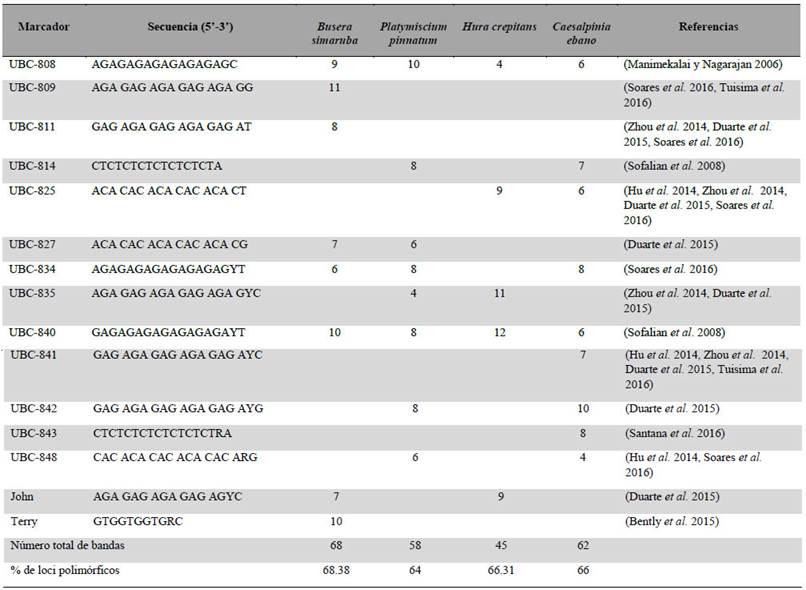
Los marcadores generaron 68 bandas para B. simaruba, 58 bandas para P. pinnatum, 45 bandas para H. crepitans y 62 bandas para C. ebano, el número total de bandas por partidor osciló de cuatro a doce, dependiendo de la especie y del partidor ISSR (Material suplementario - Anexo 1). El promedio de loci polimórficos fue muy similar para las cuatro especies y varió de 61,31 % en H. crepitans a 68,38 % en B. simaruba (Material suplementario - Anexo 1).
Para las especies B. simaruba y H. crepitans la diversidad genética fue más baja en los sitios del Valle del río Cauca respecto a las regiones del Cañón del Chicamocha y el Caribe, siendo esta última región la que presentó mayores valores para ambas especies (Tabla 2). Los sitios localizados en los municipios de Venadillo (TOL) y Tuluá (MAT) ubicados en los Valles de los ríos Magdalena y Cauca presentaron los índices más bajos para B. simaruba y H. crepitans (Tabla 2). En el caso de P. pinnatum, en la región Caribe se encontraron los sitios con la diversidad genética más baja en el municipio de Colosó (COL) así como la más alta en el municipio del Guamo (GUA) (Tabla 2). Finalmente, para C. ebano no se evidenciaron muchas diferencias entre los cuatros sitios muestreados en la región Caribe, aunque las localidades de la ciudad de Santa Marta, MAM y ZAT, mostraron ser ligeramente menos diversas (Tabla 2).
Estructura genética
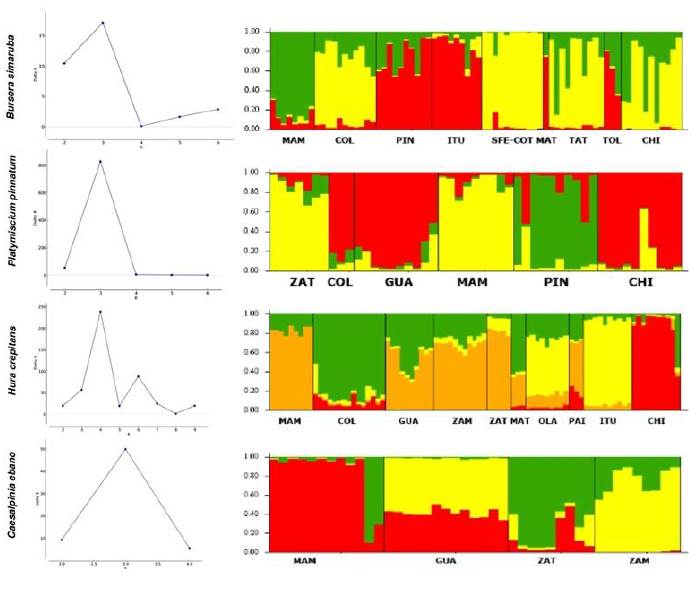
Se observó estructuración genética en las cuatro especies (Fig. 1 y Material suplementario -Anexo 2). Los patrones de estructuración variaron de la siguiente manera:
El ΔΚ de Evanno (Evanno et al. 2005) soportó la existencia de tres grupos genéticos en B. simaruba (Material suplementario - Anexo 2). Para K = 3,la distribución geográfica de los grupos es coherente para la mayoría de los lugares, excepto en los municipios de Cotové y Santa Fe de Antioquia (COT-SFE), y están distribuidos entre las poblaciones de la siguiente manera: el grupo uno tiene mayor representación en las poblaciones de los municipios de Ituango (ITU), La pintada (PIN), Venadillo (TOL) y Tuluá (MAT), el grupo genético dos si bien está distribuido a lo largo del país su mayor representación está en una de las localidades de la ciudad de Santa Marta (MAM). El grupo tres está presente en los sitios de los municipios de Colosó (COL), Cotové, Santa Fe de Antioquia (COT-SFE), Piedecuesta (CHI) y Villa Vieja TAT (Fig. 1).
En el caso de P. pinnatum el resultado de ΔΚ soportó un Κ = 3 como número de grupos genéticos (Material suplementario -Anexo 2). Los resultados para esta especie muestran una separación geográfica definida entre los tres grupos. El grupo genético uno se encuentra en el centro norte del país en los municipios de Piedecuesta (CHI), Colosó (COL) y Guamo (GUA) pertenecientes a la región del enclave del Cañón del Chicamocha y sur de la región Caribe, el grupo dos está presente en la localidad del municipio de La pintada (PIN) en el Valle del río Cauca y el grupo genético tres se encuentra en la región más septentrional en las dos localidades de la ciudad de Santa Marta (ZAT y MAM) (Fig. 1).
En el caso de H. crepitans el ΔΚ de Evanno soportó la existencia de cuatro grupos genéticos (Material suplementario 2). El grupo genético uno se encuentra mayormente distribuido en las localidades de los municipios de Colosó (COL) y Tuluá (MAT), el grupo dos está principalmente distribuido en las localidades de los municipios de Ituango (ITU) y Olaya (OLA), el grupo genético tres está al norte del país en los sitios de los municipios de Santa Marta (ZAT, MAM), Guamo (GUA) y Zambrano (ZAM) con una mínima representación en el sur en la localidad del municipio de La paila (PAI); finalmente, el grupo cuatro es mayormente representado en la región del Cañón del Chicamocha (Fig. 1).
Aunque C. ebano presenta una distribución limitada al norte de la región del rio Cauca y al Caribe, también se soportó la existencia de tres grupos genéticos (Material suplementario - Anexo 2). Las agrupaciones obtenidas separan un primer grupo genético localizado en una de las localidades del municipio de Santa Marta (ZAT), el grupo 2 también en el mismo municipio en otra localidad (MAM) y finalmente el tercer grupo está presente en las localidades de los municipios de Zambrano (ZAM) y el Guamo (GUA) (Fig. 1).
DISCUSIÓN
Las especies evaluadas mostraron valores de diversidad genética cercanos entre ellas. No obstante, los niveles de diversidad hallados en los cuatros modelos de estudio son menores a los registrados para otras especies del bosque seco tropical (bs-T) en Suramérica analizadas con marcadores moleculares ISSRs, como Ficus bonijesulapensis R.M.Castro (He: 0,30) (Duarte et al. 2015) y Caryocar brasiliense Cambess. (He: 0,49) (de Melo et al. 2012). De acuerdo con Kageyama y Gandara (1998) la fragmentación del paisaje es la principal causa de la pérdida de diversidad genética, cuanto mayor es la fragmentación menor es la variabilidad genética (Hall et al. 1996, Cascante et al. 2002, Pither et al. 2003) debido a que en este proceso se reduce el tamaño efectivo poblacional y el flujo de genes incrementando el efecto de la deriva. Como consecuencia, las poblaciones muy fragmentadas presentan una reducción de la diversidad de alelos contenidos en la población (Nassar et al. 2011). Los árboles tropicales son especialmente vulnerables a este proceso debido a su baja densidad, su sistema de auto-incompatibilidad y altas tasas de exogamia (Cascante et al. 2002). El efecto de la deriva genética tiende a incrementar en conjunto con el tiempo y poblaciones pequeñas aisladas desde hace muchas generaciones, y estas pueden presentar diversidad genética extremadamente reducida (Amos y Harwood 1998, Lowe et al. 2015).
Dado que B. simaruba es una especie dominante y de establecimiento rápido podría estar menos afectada, al menos en la inmediatez, a los procesos de pérdida del hábitat del bs-T (Trainer y Will 1984, Gillespie et al. 2000, Verea y Solórzano 2001). De forma contraria, P. pinnatum y H. crepitans, caracterizadas por su difícil establecimiento y crecimiento lento (Swaine y Beer 1977, Cordero y Boshier 2003), evidencian una diversidad genética más baja en localidades de las regiones del Caribe y el Valle del Río Cauca en comparación con B. simaruba de acuerdo con los resultados. Los procesos recientes de transformación del paisaje en estas áreas (Arcila-Cardona et al. 2012, Pizano y García 2014) podrían actuar sobre los tamaños poblacionales de estas dos especies, limitando el flujo génico entre parches ubicados en matrices no favorables.
Cabe destacar que los relictos más conservados y conectados de bs-T en Colombia corresponden a los localizados en la región Caribe, que además son los más extensos (Pizano y García 2014). Por el contrario, los fragmentos correspondientes a las otras regiones tienden a presentar disturbios más pronunciados y aislamiento debido a la expansión agrícola y ganadera (Arcila-Cardona et al. 2012, Valencia-Duarte et al. 2012, Pizano y García 2014). Estos grandes fragmentos de bs-T preservados de la región Caribe podrían explicar porque la mayor diversidad genética regional para las especies B. simaruba y H. crepitans e intermedia para P. pinnatum se encuentran allí. Contrario a lo esperado debido a los altos niveles de fragmentación para los relictos del bs-T ubicados en el enclave del Cañón del Chicamocha (Valencia-Duarte et al. 2012, Pizano y García 2014, Rojas 2017), se hallaron niveles intermedios de diversidad genética para B. simaruba, P. pinnatum y H. crepitans,
Por otra parte, se encontró estructura genética geográficamente coherente en las cuatro especies estudiadas. En el caso de P. pinnatum se detectó una distinción genética clara entre las localidades del sur del Valle del Río Cauca, el Cañón del Chicamocha con las del sur de la región Caribe y el norte del Caribe, y en el caso de H. crepitans se muestra una divergencia entre las localidades de las regiones del sur del Valle del Río Cauca, Cañón del Chicamocha respecto al norte del Valle del Río Cauca con el Caribe. Estas señales podrían estar relacionadas con el aislamiento histórico entre los remanentes de bs-T de los Valles interandinos y el Caribe (Prado y Gibbs 1993, Pennington et al. 2000), y la posible conexión histórica entre el Caribe y el Cañón de Chicamocha durante la última glaciación como se ha sugerido para otras especies del mismo ecosistema como Enterolobium cyclocarpum (Jacq.) Griseb. (Thomas et al. 2017a), Ceibapentandra (L.) Gaertn. (Bocanegra-González et al. 2018) y Anacardium excelsum L. (Bocanegra-González y Guillemin 2018). Para estas especies se ha propuesto que los patrones estructurales actuales se deben primero a la vicarianza histórica durante tiempos pre-glaciales y posterior expansión de la vegetación bs-T durante el último período glacial, y segundo, a una contracción de ámbito geográfico más reciente, es decir desde el Holoceno. De otro lado, los resultados genéticos sugieren que algunas poblaciones de Hura crepitans y Bursera simaruba en valle de Cauca (PAI y MAT, y SFE-COT, respectivamente) podrían ser de origen antropogénico, requiriendo más investigación.
Para garantizar la debida conservación de la diversidad y estructura genética de las cuatro especies de estudio en el bosque seco colombiano se debería asegurar la protección de las poblaciones más diversas de cada uno de los grupos genéticos. Se identificaron como áreas potenciales para la conservación in-situ y fuentes de germoplasma debido a su particularidad genética y niveles de diversidad a las localidades de ITU/PIN, COL y MAM para B. simaruba, PIN, GUA y MAM/ZAT para P pinnatum, COL, OLA, CHI y MAM para H. crepitans, y ZAT, MAM y GUA/ZAM para C. ebano. Destacan las localidades ubicadas en el norte Caribe y enclave del Cañón del Chicamocha como nodos importantes para la conservación de las especies del bs-T por lo que la conservación de estos relictos debería considerarse una prioridad.
Felizmente Mamancana (MAM) representa un área de conservación privada y este estudio ha confirmado su gran importancia. Finalmente, dado que se ha aconsejado evitar el uso de material no local para restauraciones debido a la probabilidad de depresión exogámica (Mckay et al. 2005), recomendamos el uso de material regional recolectado del mismo grupo genético con el objetivo de conservar las características genéticas históricas locales.