Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista de la Academia Colombiana de Ciencias Exactas, Físicas y Naturales
Print version ISSN 0370-3908
Rev. acad. colomb. cienc. exact. fis. nat. vol.37 no.143 Bogotá Apr./June 2013
FITOPATOLOGÍA
* Magister en Fitopatología, Correo electrónico: carlosgermand@hotmail.com
** Ph.D. Profesor Titular. Departamento de Producción Agropecuaria. Facultad de Ciencias Agropecuarias, Universidad de Caldas. Correo electrónico: jairo.castano_z@ucaldas.edu.co
***Â M.Sc. Profesor Asistente. Departamento de Producción Agropecuaria. Facultad de Ciencias Agropecuarias, Universidad de Caldas. Correo electrónico: bernardo.villegas@ucaldas.edu.co
RESUMEN
Los cultivos de maracuyá y otras pasifloras en Colombia regularmente son afectados por la Roña, una enfermedad degenerativa que afecta diferentes tejidos de la planta, sin embargo la etiología de la enfermedad no es bien conocida, lo que motivó esta investigación con el objetivo de establecer la verdadera etiología de la enfermedad a partir de pruebas de patogenicidad y caracterización morfológica del agente causante, complementadas con la caracterización molecular del patógeno utilizando la amplificaron de las regiones ITS del ADNr y del gen Actin, enzimas de restricción y secuenciación; concluyendo que la especie del hongo Cladosporium cladosporioides, es la causante de la Roña del maracuyá en Colombia.
Palabras clave: patogenicidad, morfología, RFLP, secuenciación, Cladosporium cladosporioides.
ABSTRACT
The passion fruit and other passion flower crops in Colombia regularly are affected by scab, a degenerative disease that attacks different parts of the plant, however the etiology of the disease is not completely known, which led to do this research to establish the etiology of the disease through pathogenicity tests and morphological characteristics of the causal agent, complemented with molecular characterization using the amplified rDNA ITS region and Actin region gen, restriction enzymes and sequencing, concluding that Cladosporium cladosporioides is the causal agent of passion fruit scab in Colombia.
Key words: pathogenicity, morphology, RFLP, sequencing, Cladosporium cladosporioides.
Introducción
En Colombia, las pasifloras que se cultivan comercialmente son: maracuyá (Passiflora edulis f. flavicarpa Degener), granadilla (P. ligularis Juss) y gulupa (P. edulis f. edulis Sims); y de manera artesanal, badea (P. quadrangularis L.) y curuba (P. mollissima Bailey) (Jaramillo, 1993). Dada la gran demanda de estos productos en el ámbito internacional, se han ido extendiendo las áreas de cultivo y se ha mejorado el manejo técnico debido a investigaciones acerca de la fisiología y control de plagas y enfermedades (Jaramillo, 1993).
Dentro de los problemas fitosanitarios del cultivo de maracuyá, se destaca una enfermedad conocida como Roña, de la cual se han escrito reportes que muestran un escaso conocimiento respecto a la identidad del agente causante de la enfermedad. Esta enfermedad fue descrita por primera vez en Australia sobre frutos de maracuyá (Simmonds, 1932). La enfermedad se describe como "Verrugosis" debido a los hinchamientos sobre el fruto, y se destaca su importancia a causa del deterioro de su apariencia y por consiguiente la disminución del valor comercial de la fruta (Figura 1). Simmonds (1932), en Australia identificó a Cladosporium herbarum, sin embargo, existen muchas discrepancias entre investigadores acerca del patógeno causante de esta enfermedad (Goes, 1998).

Cladosporium oxysporum fue reportado como el agente causante en Zimbabwe y Australia y C. cladosporioides y C. herbarum en Brasil (Bates, 1954; Persley, 1993; Barreto et al., 1996).
Los hongos del género Cladosporium Link, pertenecen a la clase Deuteromycetes, orden Moniliales, familia Dematiaceae. Son clasificados como hongos imperfectos y la especie tipo del género Cladosporium es C. herbarum, su fase perfecta o teleomorfo es Mycosphaerella tassiana Johans (Ellis, 1971), y según Kirk et al. (2001), pertenece a la clase Ascomycetes, subclase Dothydeomycetidade, orden Mycosphaerellales, familia Mycosphaerellaceae. Esta especie presenta gran abundancia de esporas asexuales, llamadas conidios. Los conidios son producidos en conidióforos grandes y oscuros que pueden ramificar en el ápice, y siempre están dispuestos sobre conidióforos en grupos ramificados.
C. oxysporum, produce conidióforos, rectos o ligeramente flexuosos, de color marrón pálido y hasta 500 μm de largo y 3-5 μm de ancho, con hinchazones intercalares de 6-8 μm de diámetro (Ellis, 1971). Los conidios son cilíndricos y con los extremos redondeados, elipsoidales, o subesféricos, de color marrón pálido o subhialinos de 5-30 x 3-6 μm, y se producen solos o en cadenas ramificadas (Ellis, 1971).
Los conidióforos de C. cladosporioides y C. herbarum son de color marrón claro y producen conidios desde la parte central hasta la parte superior del conidióforo (Domsch et al., 1980). Los conidióforos de C. cladosporioides son de 2-6 μm de ancho y hasta 350 μm de largo, y producen conidios unicelulares elipsoidales color marrón de 3-7 Ã 2-4 μm. Los conidióforos de C. herbarum son de 3-6 μm de ancho y hasta 250 μm de largo, y producen conidios color marrón amarillentos de 5,5-13 Ã 3,8-6,0 μm, que normalmente son unicelulares.
Cladosporium, también es un hongo saprófito de plantas en descomposición, textiles, cuero, caucho y madera, además es parásito de Gramíneas fundamentalmente, sus esporas en general son de tamaño aproximado de 10 μm de largo y 4 μm de ancho, de forma elipsoidal y de color hialino a marrón amarillento (Agrios, 2005). También produce conidióforos largos y oscuros que crecen en posición vertical, sus conidios son ovalados, irregulares o cilíndricos de color marrón oscuro o negro, y puede consistir de una a tres células, y dan al hongo una apariencia aterciopelada oscura. La germinación de los conidios es favorecida por una alta humedad ambiental y pueden causar lesiones en hojas, ramas o frutos (Agrios, 2005).
Las técnicas de caracterización morfológica y molecular, constituyen herramientas importantes que permiten una identificación completa de microorganismos. Debido a que en Colombia, no existe un trabajo de investigación que establezca la verdadera identidad de aislamientos patogénicos de Cladosporium spp. causando la Roña en plantaciones de maracuyá, esta investigación tuvo como objetivo identificar morfológica y molecularmente el agente causante de esta enfermedad.
Materiales y métodos
Obtención de cultivos monospóricos
Se colectaron 40 muestras de frutos enfermos de maracuyá, granadilla y gulupa con síntomas típicos de Roña en los departamentos de Huila (cinco de gulupa, cuatro de granadilla, cuatro de maracuyá), Caldas (diez de maracuyá), Valle del Cauca (tres de granadilla, tres de maracuyá, uno de gulupa), Boyacá (cuatro de gulupa), Cundinamarca (tres de maracuyá, uno de gulupa) y Tolima (uno de granadilla, uno de gulupa).
Las muestras se codificaron de acuerdo con el hospedante y lugar de origen. Se procesaron individualmente cortando fragmentos de tejido sano y enfermo de aproximadamente 2 mm2, se lavaron con agua destilada estéril (ADE) durante 10 min, y luego se desinfectaron con hipoclorito de sodio al 1% durante 1 min. Luego se enjuagaron con ADE y fueron sumergidos en etanol al 70% durante 1 min, finalmente se enjuagaron con ADE (Álvarez et al., 2004). Posteriormente se realizó siembra directa sobre cajas Petri conteniendo papa-dextrosa-agar, PDA (39 g/L), agar nutritivo, AN (42 g/L), y agar-jugo V8, AV8 (200 mL de jugo V8/ L). Finalmente, las cajas Petri se marcaron y sellaron con papel vinipel y fueron incubadas a una temperatura de 23°C y luz continua.
Para obtener cultivos monospóricos se sembró por agotamiento una asada de esporas del aislamiento puro, en cajas Petri con agar-dextrosa, AD (agar 15 g, dextrosa 20 g/L), incubándolas a 27°C en oscuridad por 24 h. Los aislamientos se observaron al estereoscopio ubicando esporas individuales germinadas, se marcó el sitio con un micro gancho sobre el medio y luego se verificó en el microscopio la presencia de esporas individuales. Se transfirió un fragmento del medio con la espora seleccionada a una caja Petri con PDA + estreptomicina (300 mg/L), teniendo la precaución de que la espora quedara en contacto con el medio. Se obtuvieron 40 aislamientos, Los cuales fueron conservados en papel filtro a 4°C y -20°C, utilizando sobres de papel kraft, para evitar la contaminación y conservar su viabilidad y patogenicidad. Luego de ser almacenados, se reactivaron los aislamientos cada dos meses para verificar su viabilidad y utilizarlos en las pruebas de patogenicidad (Aricapa & Correa, 1994).
Caracterización morfológica
Se registró tasa de crecimiento, textura y color de la colonia, y forma y tamaño de los conidios. De cada aislamiento desarrollado en medio PDA + estreptomicina, se tomaron discos de aproximadamente 3 mm de diámetro de crecimiento micelial del hongo y se transfirieron a cajas Petri con PDA + Acido láctico al 25%. Finalmente las cajas se incubaron a 24°C durante 15 días.
La tasa de crecimiento micelial (cm. día-1) se determinó mediante la fórmula propuesta por Mead et al. (1993):
Cf = Crecimiento final
Ci= Crecimiento inicial
Tf= Tiempo final
Ti= Tiempo inicial
La caracterización morfológica de las colonias se inició a los 8 días de la siembra, se evaluó el diámetro, textura y color de las colonias durante 8 días (Manzo et al., 2001; Jiménez, 2008). Para tal fin, los conidios, se montaron en portaobjetos y se observaron mediante un microscopio de luz, marca Revelation III LW Scientific, a través de los objetivos 40X y 100X. La determinación del tamaño de las esporas se realizó mediante un micrómetro marca B&L, midiendo el largo y ancho de 20 conidios elegidos al azar de cada aislamiento (Castaño-Zapata, 1998).
Se realizó un análisis de varianza para cada uno de los tratamientos (aislamientos) previa comprobación de la normalidad, y la separación de medias según la prueba de Tukey a nivel del 5%.
Pruebas de patogenicidad
Las pruebas fueron ejecutadas bajo condiciones de campo en la granja Luker, utilizando hojas y frutos sanos de maracuyá variedad Luker, de aproximadamente dos semanas de edad. Las condiciones agroecológicas de la granja son: temperatura media 22,5°C, altitud 1.100 msnm, precipitación anual 2.074 mm y brillo solar 1.791,4 h. Las inoculaciones se realizaron el mismo día para que los aislamientos tuvieran las mismas condiciones ambientales. Se inocularon 10 hojas y 10 frutos con cada aislamiento. las hojas y frutos antes de la inoculación, se desinfestaron con hipoclorito de sodio al 1%, luego se les produjeron heridas pequeñas mediante una aguja estéril, e inmediatamente después se les asperjó una suspensión de 1 x 106 conidios/mL de agua, cubriendo completamente el área herida (Figura 2). Para tal efecto se usó un aspersor marca "Plastihogar" de 200 mL de capacidad. Los frutos y hojas inoculados se mantuvieron adheridos a las plantas y fueron cubiertos con una bolsa de tul, que fue sellada con plastilina para evitar la entrada de insectos que pudieran alterar el ensayo. El testigo consistió en hacer heridas pequeñas mediante una aguja estéril y asperjar agua destilada estéril a las hojas y los frutos, cubriendo por completo los tejidos (Saldarriaga-Cardona et al., 2008; Dhingra & Sinclair, 1985; FAO, 1985; French & Hebert, 1982). Las variables registradas fueron periodo de incubación e incidencia de la enfermedad.

Los 40 aislamientos obtenidos de tejidos de maracuyá, granadilla y gulupa fueron inoculados siguiendo los postulados de Robert Koch.
Caracterización molecular
La caracterización molecular se realizó en el laboratorio de patología de yuca y frutas tropicales del Centro Internacional de Agricultura Tropical-CIAT, Palmira, donde se aplicaron tres diferentes técnicas como son: amplificación de los espaciadores internos transcritos (ITS, por sus siglas en inglés), amplificación del gen Actin y análisis del Polimorfismo en la Longitud de los Fragmentos de Restricción (RFLP, por sus siglas en inglés) para determinar una o varias especies de Cladosporium como causantes de la Roña.
Obtención del micelio
Los aislamientos se sembraron en Erlenmeyer de 250 mL con 100 mL de medio papa-dextrosa, PD (extracto de papa 200 g, dextrosa 20 g, agua destilada estéril 800 mL), se incubaron a 23°C en una incubadora marca WTB binder, durante 12 días. Se hizo un filtrado del hongo colocando el micelio sobre papel filtro Whatman N°2 dentro de un embudo de cerámica (Buchner funnel) unido a un Erlenmeyer y a una bomba de vacío, haciendo un lavado con agua destilada estéril.
Se tomó el micelio del papel filtro y se pasó a cajas Petri, las cuales fueron conservadas en una incubadora marca Thermo scientific, a 37°C por 8 h para extraer la humedad, posteriormente el micelio se maceró con nitrógeno líquido y se depositó en tubos eppendorf estériles de 2 mL de capacidad, los cuales se conservaron a -20°C.
Extracción y cuantificación de ADN
La extracción se realizó siguiendo el protocolo descrito por Álvarez et al. (2004), modificado por Delgado (2011). Se cuantificó la concentración del ADN en un espectrofotómetro NanoDrop ND-1000 (Nanodrop Technologies, Wilmington, DE) y se utilizó a una concentración de 5 ng/μL.
Amplificación de las regiones ITS del ADNr
Para la amplificación de las regiones ITS del ADN ribosomal (ADNr) se utilizaron los cebadores universales ITS1/ ITS4 e ITS4/ITS5 ITS1: (5'-TCCGTAGGTGAACCTGCGG-'3), ITS4: (5'TCCTCCGCTTATTGATATGC-'3), ITS5: (5'GGAAGTAAAAGTCGTAACAAGG-'3) (White, 1990). Las reacciones de PCR fueron calculadas para 30 μL; se utilizó las condiciones de amplificación reportadas por Wirsel et al. (2002).
Los resultados se evaluaron mediante electroforesis en geles de agarosa al 1,2% teñidos con el reactivo Gel red sobre Buffer TAE 1X y visualizados bajo luz UV. En la electroforesis se utilizó un marcador molecular de ADN HyperLadder II (Bioline; Batch #: H2K2-1008).
Amplificación del gen Actin
Para la amplificación del gen Actin de los 30 aislamientos, se utilizaron los cebadores específicos para el género Cladosporium CladActF1-CladActR1 y CladActF3-CladActR3. El coctel de la reacción de PCR contenía 0,5 μL de una solución de ADN fúngico en 50 μL de mezcla de reacción (1 X PCR buffer (MBI Fermentas GmbH, St. Leon-Roth, Alemania), 1,5 mM MgCl2, 0,2 mM dNTPs, 0,5 mg/mL BSA bovine serum albumin, 0,3 μL de cada primer, y 0,04 U/μL de Taq Polimerasa (MBI Fermentas). Las condiciones de amplificaciones se hicieron de acuerdo con las reportadas por Wirsel et al. (2002), ligeramente modificadas por Delgado (2011).
RFLP
Se utilizaron las enzimas de restricción HaeIII, HpaII, RsaI, AluI, y TaqI con el fin de encontrar diferencias entre la secuencia de nucleótidos de los 30 aislamientos de Cladosporium seleccionados.
Posteriormente se hizo una electroforesis en gel de agarosa al 3,0%, con Buffer TAE 1X y se visualizaron los resultados bajo luz UV. Para la observación de los fragmentos se utilizó un marcador molecular de ADN HyperLadder II (Bioline; Batch #: H2K2-1008).
Secuenciación
El ADN amplificado para las regiones ITS1, ITS4 e ITS5 del ADNr de 13 aislamientos y para el gen Actin de 10 aislamientos fue secuenciado en la Universidad del estado de Iowa, Estados Unidos. Se limpiaron y se ensamblaron las secuencias con el programa ChromasPro versión 1,5 (Technilysium Pty Ltd.) donde se desplegaron los electroferogramas. Posteriormente, con el programa Blast se hizo la anotación y se buscó la identidad del patógeno en la base de datos del Centro Nacional para la Información Biotecnológica (NCBI, por sus siglas en inglés).
Adicionalmente, se construyó un árbol filogenético con las secuencias, utilizando la herramienta Geneious Tree Builder, empleando el modelo de distancia genética de Jukes â Cantor mediante el método de Evolución mínima y análisis estadístico de probabilidad (bootstrap) con 500 réplicas.
Análisis filogenético
El análisis filogenético fue hecho usando las secuencias amplificadas de las regiones ITS1, ITS4, ITS5 y Actin del ADNr de los aislamientos amplificados en el paso anterior. las secuencias forward y reverse, fueron limpiadas y ensambladas en el programa ChromasPro versión 1,49; una vez completas las bases, los nucleótidos fueron alineados usando el programa CLUSTALW. Luego se utilizó el programa MEGA versión 4,0 de donde se obtuvo una secuencia completa y se realizó el análisis filogenético. Los grupos de múltiples datos fueron generados a través de la opción bootstrap y analizados por el método de parsimonia de ADN.
El método de neighbor-joining fue usado para analizar la distancia de las matrices producida de los grupos de múltiples datos y para generar los arboles filogenéticos. Después del alineamiento se generaron mapas de sitios de restricción usando el programa pDRAW32 versión 1,1.110 y los mapas fueron alineados manualmente para su comparación, también fueron examinados por concordancia con sitios de restricción previamente identificados por la digestión enzimática y comparados con las de las regiones ITS y del gen Actin de C. herbarum, C. oxysporum, C. cladosporioides, C. tenuissimum, Cladosporium sp., Mycosphaerella graminicola y Septoria tritici seleccionadas de la base de datos del Gen-Bank. Por último se realizaron cortes virtuales con las enzimas de restricción BsiEI, BtgZI, TifI y TspDTI a todas las secuencias incluidas en el ensayo para verificar la similitud de las secuencias de nuestros aislamientos con las obtenidas en el Gen-Bank de las otras especies de Cladosporium para evidenciar su similitud o diferencia.
Resultados
Colección y obtención de los aislamientos
De cada muestra colectada se obtuvo un aislamiento para una colección de 40, 30 pertenecientes al género Cladosporium y 10 al género Colletotrichum. El cultivo de los aislamientos en PDA incubados a 24°C en ausencia de luz por 12 días fue la más efectiva para la obtención y esporulación de los hongos.
Pruebas de patogenicidad
Se reprodujeron los síntomas de la enfermedad en hojas y frutos de maracuyá en condiciones de campo con los 10 aislamientos de Cladosporium spp. procedentes del departamento de Caldas.
Las hojas empezaron a manifestar síntomas de la enfermedad (periodo de incubación) después de 4-6 días. Los primeros síntomas consistieron en manchas pequeñas, de aproximadamente 1mm de diámetro, redondeadas, de color marrón oscuro (Figura 3A). La esporulación del hongo (periodo de latencia), se observó después de 6 días de la aparición de los primeros síntomas (Figura 3B).
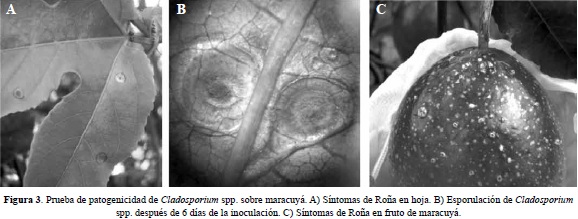
Los síntomas en frutos se manifestaron a través de lesiones pequeñas de color blanco o gris abultados, de forma circular que a través del tiempo se convirtieron en costras erupentes amorfas (Figura 3C). Los síntomas primarios fueron evidentes después del sexto día de la inoculación. Las lesiones fueron aumentando su tamaño y afectaron varias partes del fruto, sin embargo ésta es una enfermedad que no afecta la pulpa, presentándose sólo superficialmente. Las lesiones variaron en forma y tamaño, y las costras aumentaron de tamaño a medida que el fruto creció. Los frutos que se emplearon como controles permanecieron sanos.
Caracterización morfológica de los aislamientos
Los aislamientos seleccionados para la caracterización morfológica fueron del género Cladosporium, debido a que fueron los únicos que produjeron patogenicidad
Dichos aislamientos después de 8 días de sembrados en PDA + AL y conservados a 24°C, presentaron diferentes tonalidades de color, en el 67% de los cuales predominó consistentemente el color verde oliváceo como fondo del aislamiento y algunas veces rodeado por un color gris claro (Figura 4A). El 20% de los aislamientos tuvo un color verde claro amarillento (Figura 4B) y el 13% restante mostró un color gris claro con círculos concéntricos (Figura 4C).
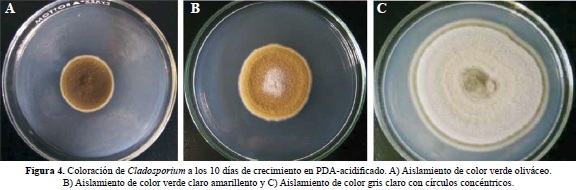
La textura de los aislamientos varió desde aterciopelada y corrugada en el centro, que con el tiempo presentó algún tipo de masa algodonosa en el centro, hasta una textura aterciopelada.
Tasa de crecimiento in vitro
El análisis de varianza mostró diferencia altamente significativa entre los aislamientos. La mayor tasa de crecimiento la tuvo el aislamiento GRLUK-MA-9 con 0,370833 cm/día y la menor, el aislamiento HUGA-GU-1 con 0,270833 cm/ día. En general todos los aislamientos que tuvieron patogenicidad presentaron una tasa de crecimiento diaria alta, entre 0,33 y 0,37 cm/día.
Tamaño y forma de los conidios
El análisis de varianza mostró diferencias significativas entre la longitud de los conidios de los aislamientos analizados.
Los valores fluctuaron entre 6,46µm y 7,595 µm. El ancho de los conidios presentó valores significativamente diferentes y en promedio fue de 3,035µm. Los 30 aislamientos de Cladosporium produjeron conidios en conidióforos grandes y oscuros, dispuestos sobre conidióforos en grupos ramificados, los conidios tuvieron forma ovalada, irregular o cilíndrica de color marrón oscuro o negro. Estos conidios típicamente mostraron una cicatriz pequeña en sus extremos formada por la gemación sucesiva del conidio anterior (Figura 5).
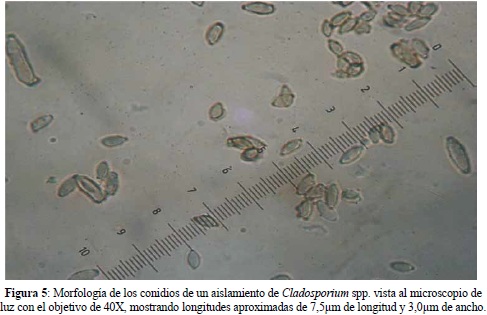
Caracterización molecular
Los 30 aislamientos de Cladosporium crecieron en medio líquido PD y se obtuvo un crecimiento puro al cabo de 12 días.
Extracción y cuantificación de ADN
El procedimiento de extracción del ADN de los 30 aislamientos permitió obtener un ADN limpio en cantidades entre 280 y 700 ng/µL de ADN. Al final, para el proceso de amplificación con cebadores para ITS y Actin todos los aislamientos se utilizaron a una concentración de 5 ng/µL.
Amplificación de las regiones ITS y Actin
La amplificación mediante PCR con los cebadores ITS1/ ITS4 e ITS4/ITS5 mostró fragmentos de ADN de 560 bp, mientras que la amplificación del gen Actin con cebadores específicos para el género Cladosporium: CladActF1-CladActR1 y CladActF3-CladActR3, mostró fragmentos de ADN de 440 bp, en los 30 aislamientos, lo cual indicó que todos los aislamientos pertenecen al género Cladosporium. (Figura 6).
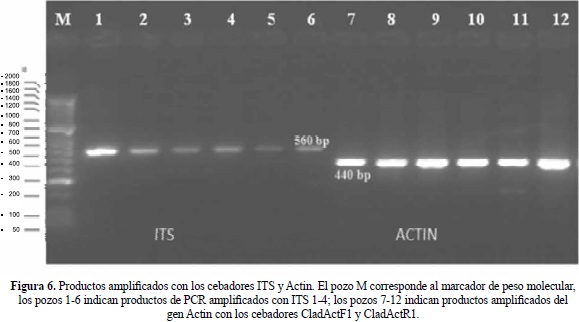
RFLP's
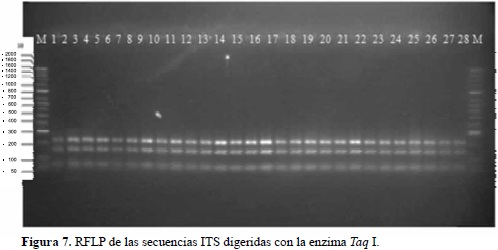
La digestión de las secuencias ITS y Actin indicaron patrones idénticos para los 30 aislamientos de Cladosporium.
Los patrones de restricción invariables entre los aislamientos evidenciaron que no hubo una diferencia molecular notoria entre estos con cualquiera de las cinco enzimas utilizadas, lo que sugiere que los aislamientos de Cladosporium obtenidos pertenecen a una misma especie (Figura 7).
Secuenciación de las regiones ITS y Actin
La secuencia de nucleótidos de las regiones ITS del ADNr editadas con el programa ChromasPro produjo un alineamiento general de 539 pb. La secuencia de nucleótidos del gen Actin con los cebadores CladActF3-CladActR3 generó un alineamiento de 401 pb. El alineamiento de las secuencias de las regiones ITS y Actin indicó que no había diferencias entre los aislamientos de Cladosporium, las secuencias fueron idénticas para los 13 aislamientos amplificados con ITS, ITS4 e ITS5, el mismo resultado se obtuvo para las secuencias del gen Actin de los 10 aislamientos seleccionados.
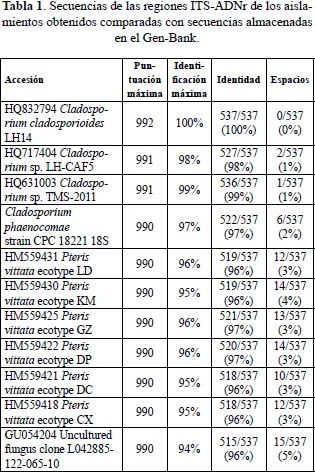
El análisis Blast en el Gen-Bank de una única secuencia de nucleotidos representativa de las regiones ITS de los 13 aislamientos secuenciados indicó que los aislamientos de Cladosporium obtenidos en el muestreo de campo, pertenecen a la especie C. cladosporioides, con una similitud entre secuencias de nucleótidos entre 94 y 100% (Tabla 1).
Análisis filogenético
El análisis filogenético y la utilización de cladogramas permitió realizar un agrupamiento específico entre las especies de Cladosporium obtenidas a partir de la secuenciación y la base de datos del Gen-Bank. Este agrupamiento refleja la similitud filogenética que tienen los aislamientos obtenidos en este ensayo con la especie C. cladosporioides al mismo tiempo que se diferencian de especies como C. oxysporum, C. tenuissimum, C. herbarum y Cladosporium sp.
Los resultados del análisis filogenético entre las secuencias de los aislamientos seleccionados de regiones ITS y gen Actin con las accesiones obtenidas del Gen-Bank mostraron varias metodologías para agrupar los aislamientos de Cladosporium, y en este caso el método de evolución mínima permitió obtener un cladograma de mejor definición (Figura 8). Las secuencias de la región ITS de los aislamientos obtenidos en este estudio y de las especies de Cladosporium obtenidas del Gen-Bank comparadas con la secuencia de M. graminicola formaron cladogramas completamente aparte y sin ninguna relación entre estos (Figura 8A), al igual que los resultados de las secuencias de la región del gen Actin comparados con S. tritici (Figura 8B). Para el caso de los aislamientos obtenidos en este trabajo, el agrupamiento los llevó hacia la especie C. cladosporioides (Figura 8).

Los aislamientos obtenidos en este estudio se agrupan en el árbol con el código de cada aislamiento; en cada nodo los valores representan el respectivo valor de "bootstrap", obtenido a partir del análisis con 500 repeticiones.
El análisis de la secuencia obtenida de las regiones ITS y Actin imitando cortes con enzimas de restricción y posteriores electroforesis simuladas por computador mostraron patrones específicos de corte cuando se realizó la comparación de la secuencia obtenida de Cladosporium con secuencias de otras especies de Cladosporium obtenidas del Gen-Bank.
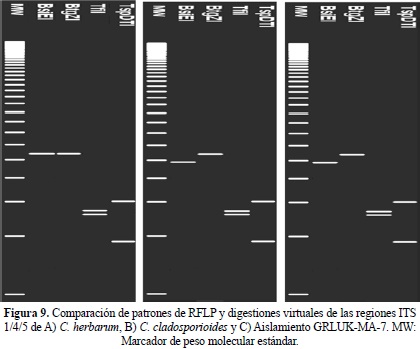
Las digestiones virtuales se realizaron con la secuencia de la región ITS de uno de los 30 aislamientos de Cladosporium obtenidos en este estudio codificado como GRLUK-MA-7 (Figura 9C). La comparación de esta secuencia con las de cinco especies de Cladosporium como son C. herbarum (Figura 9A), C. oxysporum, C. tenuissimum, Cladosporium sp. y C. cladosporioides (Figura 9B) obtenidas del Gen-Bank, permitió determinar que los cortes hechos a la secuencia de GRLUK-MA-7 fueron similares a los encontrados en la secuencia de C. cladosporioides, lo cual confirmó que nuestros aislamientos pertenecen a esta especie (Figura 9).
Discusión
Diversas especies del género Cladosporium han sido descritas, las cuales van desde micoparásitas, saprófitas y fitopatógenas hasta patógenas de humanos (Ellis, 1971; David, 1997; Moricca et al., 1999). En este trabajo se obtuvo un cepario conformado por 30 aislamientos de Cladosporium obtenidos a partir de frutos enfermos de pasifloras (17 de maracuyá, 11 de gulupa y 2 de granadilla), procedentes de seis departamentos de Colombia con diferente ambiente climático y características edafológicas, lo que confirma que este género está ampliamente distribuido en el medio ambiente y que la abundancia de sus esporas se debe a su tamaño promedio de 7,02µm de largo por 2,92µm de ancho y a la habilidad para crecer en un rango amplio de sustratos como caucho, suelo, y alimentos (David, 1997).
La diseminación de esporas de Cladosporium en los cultivos de pasifloras se debe principalmente a plantas infectadas, aire y a salpicaduras de agua, la semilla de las pasifloras no parece ser un medio útil de transporte del hongo para producir la Roña. Manicom et al. (2003), reportan que aunque los conidios de Cladosporium son encontrados frecuentemente sobre semillas de maracuyá no hay evidencia de diseminación conidial por este medio.
Los conidios obtenidos en este estudio a partir de los aislamientos de C. cladosporioides, tuvieron un tamaño que fluctuó entre 7,595µm y 6,46µm de largo y en promedio tuvieron un ancho de 3,035µm, lo cual coincide con lo descripción hecha por Domsch et al. (1980). La obtención de síntomas de Roña en frutos de maracuyá inoculados con esta especie, es uno de los más grandes aportes de este trabajo. Las condiciones ambientales en el campo y el empleo de una alta concentración conidial, permitieron establecer las mejores condiciones para presentarse la enfermedad. Bueno et al. (2002), destacan la dificultad para reproducir los síntomas de Roña en maracuyá. Nuestros resultados son importantes debido a que confirman la patogenicidad de Cladosporium en hojas y frutos sanos de maracuyá. Resultados similares obtuvieron Barreto et al. (1996) cuando a partir de plantas de Passiflora edulis, afectadas por necrosis en hojas y "corchos" en frutos obtuvieron aislamientos que fueron identificados como C. cladosporioides, que posteriormente utilizaron para inocular plantas sanas mantenidas en invernadero que después de dos semanas presentaron síntomas de necrosis en hojas y frutos.
La Roña de las pasifloras es una enfermedad ampliamente distribuida en el mundo y muchos investigadores han destacado sus trabajos con el fin de definir la etiología del agente causante de la enfermedad. Se ha planteado que C. oxysporum es el responsable de la enfermedad en Zimbabwe y Australia, mientras que C. cladosporioides y C. herbarum en Brasil (Bates, 1954; Persley, 1993; Barreto et al., 1996).
Rocha y Menezes (1997), indican que plantas sanas de maracuyá inoculadas con C. herbarum reprodujeron síntomas de "Cladosporiosis" y que los síntomas en frutos aparecieron después de siete días de la inoculación, lo cual concuerda con nuestros resultados donde en promedio, al séptimo día se presentaron los síntomas de la enfermedad en frutos. Sin embargo, los mismos autores reportan que en hojas los síntomas tardaron 12 días en manifestarse, mientras que en nuestro estudio se presentaron los síntomas en hojas a los 4 días. Por otra parte, Santos (2008), reporta que los primeros síntomas en hojas de plantas sanas inoculadas con Cladosporium spp., aparecieron después de 10 días, que corresponde al periodo de incubación, y que los primeros signos fueron observados al decimo segundo día, correspondiente al periodo de latencia.
La diferencia de tiempo en ocurrir la enfermedad se debe a varios factores como: manera de inoculación, concentración conidial, factores edafoclimáticos y microorganismo inoculado, lo que haría pensar que la especie C. herbarum necesita de condiciones distintas a las de C. cladosporioides.
La temperatura es sin lugar a dudas uno de los factores más importantes para lograr el establecimiento y la infección de un patógeno en un cultivo, por esta razón se han realizado trabajos para definir este factor en diferentes patógenos reportados como causantes de la enfermedad. Es así como Domsch et al. (1980), concluyeron que la temperatura óptima para que se presente la enfermedad para C. oxysporum es: 19,5-24°C; C. cladosporioides: 20-28°C; y C. herbarum: 28-30°C.
En esta investigación los aislamientos obtenidos a partir de muestras de maracuyá correspondientes a la especie C. cladosporioides fueron patogénicos en maracuyá, pero ningún aislamiento obtenido a partir de granadilla o gulupa fue patogénico en maracuyá. Estos resultados muestran la incapacidad de aislamientos que aunque fueron identificados también como C. cladosporioides no pudieron producir la enfermedad en otro hospedante, lo que llevaría a pensar en un posible movimiento de cepas regido por las condiciones agroclimáticas y edafológicas de cultivos de maracuyá, granadilla y gulupa.
Los síntomas de alteración del tejido, pueden presentar una considerable variación debido al comportamiento de las plantas infectadas por un hongo, la afección del tejido también varía dependiendo de la especie del hongo, e incluso pueden presentarse varias razas con un grado de patogenicidad distinto dentro de cada una de las especies del hongo (Agrios, 1991).
Considerando el hecho de que los aislamientos identificados molecularmente como C. cladosporioides, presentaron diferencias culturales y patogénicas, se sugiere la existencia de razas del hongo en cultivos de maracuyá, granadilla y gulupa. Esta opinión se ha comprobado en otros cultivos como el tomate de árbol, donde según Corpoica (1999), tres materiales de tomate de árbol (rojo común, tamarillo y amarillo), presentan una reacción diferente a cada cepa de Colletotrichum o aislamiento del hongo causante de la Antracnosis; y cada material en particular responde también de manera diferente a todos los aislamientos. Dodd et al. (1991), consideran que las diferencias en la patogenicidad de una cepa pueden ser consecuencia de las interacciones del medio ambiente, la secreción de enzimas del hongo y el genotipo del hospedante.
En la identificación molecular, el método de extracción de ADN reportado por Álvarez et al. (2004) permitió extraer el ADN total de 30 aislamientos obtenidos a partir de material vegetal de pasifloras. La calidad y el método de ajuste de concentración del ADN obtenido fueron adecuadas para la amplificación de la región ITS âADNr y su posterior digestión con enzimas de restricción. Muy buenos resultados también obtuvieron otros investigadores que reportan el método de extracción como exitoso para otros hongos, como Mycosphaerella fijiensis, Colletotrichum acutatum y Rhizoctonia spp. (Ospina, 2002; Álvarez et al., 2004).
Por otra parte, la utilización de la técnica de PCR con cebadores ITS del ADNr y del gen Actin mostró excelentes resultados para confirmar el género y la especie de aislamientos de Cladosporium. También se podría pensar en establecer cebadores específicos para diferenciar posibles razas de C. cladosporioides, con la amplificación de la región ITS (Álvarez et al., 2001; Freeman et al., 2000; Freeman et al., 1996; Freeman et al., 1998; Sreenivasaprasad et al., 1996).
Además Zeng et al. (2006), utilizaron una técnica muy sensible, la técnica de qPCR o PCR en tiempo real donde pudieron identificar especies de Cladosporium patógenas de humanos, obtenidas a partir de cajas de Petri expuestas al ambiente, lo que describe la posibilidad de implementar esta técnica para usos fitopatológicos y construir cebadores para qPCR.
En la literatura se encuentran pocos estudios como los de Oliveira et al. (1994) y Santos (2008), que hacen referencia a estudios de resistencia en pasifloras a diferentes especies de Cladosporium, los cuales solo pueden realizarse partiendo del conocimiento pleno del agente involucrado en la enfermedad y bajo condiciones controladas de invernadero, donde se limita la influencia ambiental así como los efectos de las interacciones con insectos que jugarían un papel muy importante cuando de producir heridas en frutos se trate, dejando las condiciones apropiadas para la entrada de conidios de Cladosporium.
Conclusiones
Las metodologías de diagnóstico morfológico y molecular utilizadas en este trabajo permitieron establecer la especie de los aislamientos obtenidos y definir que el agente causante de la Roña del maracuyá es Cladosporium cladosporioides, lo cual constituye un aporte importante para la fitopatología.
Primer reporte con pruebas de patogenicidad en maracuyá, bajo condiciones de campo, y pruebas morfológicas y moleculares para identificar a Cladosporium cladosporioides como causante de la Roña.
Ciertos caracteres morfológicos entre los aislamientos de Cladosporium como la longitud de los conidios, la tasa de crecimiento y la coloración de las cepas, variaron en gran medida, lo que hizo pensar, en la presencia de varias especies del género Cladosporium dentro de la colección de aislamientos, sin embargo, todos los aislamientos se identificaron molecularmente como C. cladosporioides, lo cual pone en evidencia la dificultad de establecer la especie del hongo mediante caracteres morfológicos.
Referencias
Agrios, N., G. 1991. Fitopatología. México: Limusa. Pp. 324337, 726. [ Links ]
Álvarez, E., Ospina, C.A., Mejía, J. F. & Llano, G.A. 2004. Caracterización morfológica, patogénica y genética del agente causal de la Antracnosis (Colletotrichum gloeosporioides) en guanábano (Annona muricata) en el Valle del Cauca. Fitopatología Colombiana 28 (1): 1-8. [ Links ]
Álvarez, E. C., Claroz, J. L., Loke, J. B. & Echeverri, C. 2001. Diversidad genética y patogénica de Sphaerotheca pannosa var. rosae, el hongo causante del Mildeo Polvoso en la rosa en Colombia. Revista Asocolflores, Bogotá. No. 58. (Enero Junio). Pp. 36-44. [ Links ]
Aricapa, M. G. & Correa, F. 1994. Almacenamiento de hongos sobre papel filtro. Programa Patología de Arroz. CIAT. Ascolfi Informa 20(3): 29-30. [ Links ]
Ávila, A., Benali A. & Trapero, C. 2004. Variabilidad morfológica y cultural de Pseudocercospora cladosporioides, agente del emplomado del olivo. Bol. San. Veg. Plagas 30: 369-384. [ Links ]
Barreto, R.W., Requia, A.C. & Casa, R.T. 1996. Queima de mudas do maracujazeiro Passiflora edulis causada por Cladosporium cladosporioides. Fitopatología Brasileira 21(87): 348. [ Links ]
Bates, G. R. 1954. Report of the chief botanist and plant pathologist for the year ending 30th September. Rhodesia Agricultural Journal 51(1): 359-370. [ Links ]
Bueno, P., Peixoto, J., Junqueira, N. & Mattos, J. 2002. Incidencia e severidade de septoriose (Septoria passiflorae Sydow) em mudas de 48 genotipos de maracujazeiro azedo, sob casa de vegetacao no distrito federal. Bioscience Journal 23(2): 88-95. [ Links ]
Castaño-Zapata, J. 1998. Prácticas de laboratorio de fitopatología. 2ª Edición. Centro Editorial Universidad de Caldas-Zamorano Academic Press. 103p. [ Links ]
Corpoica. 1999. Estudios biológicos y epidemiológicos de la Antracnosis del tomate de árbol y generación de alternativas para su manejo integrado en Colombia: Informe Técnico Final. Santafé de Bogotá, D.C.: CORPOICA. 47p. [ Links ]
David, J. C. 1997. A Contribution to the systematic of Cladosporium: Revision of the fungi previously referred to Heterosporium. CAB Int., Oxon, U.K. Pp. 1-157. [ Links ]
Dhingra, O. & Sinclair, J. 1985. Basic plant pathology methods. Boca Raton, Florida: CRC Press, Inc. Corporate Blvd. Pp. 15-61. [ Links ]
Dodd, J.C., Estrada A.B., Mateham J., Jeffries P. & Jeger, M.J. 1991. The effect of climatic factors on Colletotrichum gloesporioides, the causal agent of mango anthracnose, in The Philippines. Plant Pathology 40: 568-575. [ Links ]
Domsch, K., Gams, W. & Anderson, T. 1980. Compendium of soil fungi. Academic Press, New York, 120 p. [ Links ]
Ellis, M. 1971. Dematiaceous Hyphomycetes. Commonwealth Mycological Institute, Kew, UK. 608 p. [ Links ]
Freeman, S., Katan, T. & Shabli, E. 1996. Characterization of Colletotrichum gloeosporioides isolates from avocado and almond fruits with molecular and pathogenicity tests. Appl. Environ. Microbiol. 62: 1014-1020. [ Links ]
Freeman, S., Katan, T. & Shabi, E. 1998. Characterization of Colletotrichum species responsible for Anthracnose diseases of various fruits. Plant Disease 82(6): 596-604. [ Links ]
Freeman, S., Minz, D., Jurkevitch, E., Maymon, M. & Shabli, E. 2000. Molecular analyses of Colletotrichum species from almond and other fruits. Phytopathology 90:608-614. [ Links ]
French, E. & Hebert, T. 1982. Métodos de investigación fitopatológica. San José de Costa Rica: Instituto Interamericano de Cooperación para la Agricultura. Pp. 33-38, 47-55, 65-68, 151-152, 154-186. [ Links ]
FAO. 1985. Manual para patólogos vegetales. Lima: Pacific Press. 437 p. [ Links ]
Goes, A. 1998. Doencas fúngicas da parte aerea da cultura de maracuja. In: Simposio Brasileiro Sobre a Cultura o Maracujazeiro. Facultade de Ciencias Agrarias e Veterinarias, Universidade Estadual Paulista. Jaboticabal.. Pp. 208-216. [ Links ]
Jaramillo, E. 1993. Entomofauna causante de problemas en la curuba (P. mollisima H.B.K. Bailey) en el Gran Caldas. Agricultura tropicalFrutales. Colombia. 90 p. [ Links ]
Jiménez, I. 2008. Effect of the nutritional status of banana (Musa spp.) on leaf disease infestation by Mycosphaerella fijiensis Morelet in Ecuador. Tesis postgrado. Katholieke Universiteit Leuven, Division of Crop Biotechnics. Bélgica. 149 p. [ Links ]
Kirk, P. M., Cannon, P. f., David, J. C. & Stalpers, J. 2001. Ainsworth and Bisby's dictionary of the fungi. 9th ed. CAB International, Wallingford, UK. 771 p. [ Links ]
Manicom, B., Ruggiero, C., Ploetz, R.C. & Goes, A. 2003. Diseases of passion fruit. Ploetz RC (Ed) Diseases of Tropical Fruit Crops, CAB International. Wallingford. Pp. 413-441. [ Links ]
Manzo, S.G., Orozco, S.M. & Guzmán, G.S. 2001. Caracterización morfológica de Mycosphaerella fijiensis Morelet de la región del Pacífico centro de México y su desarrollo en medios de cultivo. Revista Mexica de Fitopatología 19(001): 66-71. [ Links ]
Mead, G., Hudson, M. & Hinton, M. 1993. Microbiological survey of five poultry processing plants in the UK. Br. Poult Sci. 34:497-503. [ Links ]
Moricca, S., Ragazzi, A. & Mitchelson, K.R. 1999. Molecular and conventional detection and identification of Cladosporium tenuissimum on two-needle pine rust aeciospores. Canadian Journal of Botany 77(3): 339â347. [ Links ]
Oliveira, J.C., Nakamura, K. & Mauro, A.O. 1994. Aspectos gerais do melhoramento do maracujazeiro. Em: Sao Jose AR (Ed) Maracujá. Produção e Mercado, UESB, Vitoria da Conquista, BA, Pp. 27-37. [ Links ]
Ospina, C. A. 2002. Caracterización de poblaciones de Colletotrichum spp. causando antracnosis en cítricos, en regiones productoras del oeste de Colombia. Universidad Nacional de Colombia, Bogotá. 87 p. [ Links ]
Persley, D. 1993. Diseases of fruit crops. Department of Primary Industries, Indooroopilly, Queensland. 114 p. [ Links ]
Rocha, C. & Menezes, M. 1997. Patogenicida de de Cladosporium herbarum em frutos e folhas de maracuja (Passiflora edulis f. flavicarpa) no Estado de Pernambuco. Fitopatologia Brasileira 22(1): 302 [ Links ]
Saldarriaga-Cardona, A., Castaño-Zapata, J. & Arango-Isaza, R. 2008. Caracterización del agente causante de la Antracnosis en tomate de árbol, manzano y mora. Academia Colombiana de Ciencias Exactas, Físicas y Naturales 32(123):145-156. [ Links ]
Santos, F. M. 2008. Seleção de maracujazeiro (Passiflora edulis) para resistência à cladosporiose (Cladosporium herbarum). Universidade Federal de Viçosa. Programa de Pós-graduação em Genética e Melhoramento. 104 p. [ Links ]
Simmonds, J.H. 1932. Powdery spot and fruit scab of the passion vine. Queensland Plant Industry Pamphlets 38(1):143-152. [ Links ]
Sreenivasaprasad, S., Mills P. R., Meehan B. M. & Brown A. E. 1996. Phylogeny and systematics of 18 Colletotrichum species based on ribosomal DNA spacer sequences. Genome 39(12): 499-512. [ Links ]
Wirsel, S. G. R., Runge-Frobose, C., Ahrén, D. G., Kemen, E., Oliver, R. P. & Mendge, N. K. W. 2002. Four or more species of Cladosporium sympatrically colonize phragmites australis. Fungal Genetics and Biology 35(1): 99-113. [ Links ]
Zeng, Q., Westermark, S., Rasmuson-Lestanderb, A. & Wang, X. 2006. Detection and quantification of Cladosporium in aerosols by realtime PCR. Journal Environmental Monitoring 8(1): 153â160. [ Links ]
Recibido: 23 de febrero de 2013
Aceptado para su publicación: 4 de junio de 2013













