Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista de la Academia Colombiana de Ciencias Exactas, Físicas y Naturales
Print version ISSN 0370-3908
Rev. acad. colomb. cienc. exact. fis. nat. vol.39 no.152 Bogotá July/Sept. 2015
https://doi.org/10.18257/raccefyn.199
doi: http://dx.doi.org/10.18257/raccefyn.199
Unidad de Saneamiento y Biotecnología Ambiental (USBA), Departamento de Biología, Pontificia Universidad Javeriana, Bogotá, Colombia *Correspondencia: Sandra Baena, baena@javeriana.edu.co
Recibido: 21 de febrero de 2015. Aceptado: 15 de mayo de 2015
Resumen
Los manantiales salinos terrestres son hábitats con una gran diversidad de especies microbianas, pero el conocimiento de dicha diversidad en el país es escaso. Se seleccionaron tres manantiales salinos ubicados en el sistema montañoso de los Andes colombianos para su estudio en el marco del programa de investigación en ecología y diversidad de manantiales salinos y termales. Estos manantiales, con un contenido total de sólidos disueltos de 20 a 54 g/L, fluyen a partir de aguas subterráneas antiguas y su régimen hidrológico no está directamente relacionado con recargas pluviométricas recientes. Presentaron composiciones fisicoquímicas diferentes: en las muestras de agua provenientes de los manantiales Salado de Consotá y La Cristalina se registró un alto contenido de cloro, sodio y calcio, en tanto que las aguas provenientes del manantial Salpa se caracterizaron por una gran concentración de sodio, potasio y sulfato. La composición de las comunidades bacterianas se analizó con base en diferentes aproximaciones para generar inventarios de diversidad biológica. Los resultados indicaron que los tres manantiales albergan una comunidad bacteriana constituida en su mayoría por organismos Gram-negativos, móviles, heterotróficos y litotróficos, halotolerantes y halófilos, algunos de los cuales han sido reportados previamente en ambientes marinos. La mayoría de los organismos aislados pertenecía a la gamaproteobacteria y alfaproteobacteria, aunque las diferencias en la composición microbiana de cada manantial fueron evidentes. El estudio demostró que estos manantiales son hábitat de nuevas variedades taxonómicas como se deduce de las especies bacterianas caracterizadas.
Palabras clave: manantiales salinos, diversidad microbiana, bacterias halófilas, halotolerantes.
Abstract
Terrestrial saline springs are habitats of a high diversity of microbial species, but knowledge of this diversity is scarce in our country. We have chosen to study three saline springs as part of our research program on the ecology and diversity of halophilic and thermophilic microorganisms. The saline springs are located on the Andes mountain range, their content of total dissolved solids is 20-54 g/L, they flow from ancient groundwater, and their water regime is not directly related to recent rainfall recharges. They exhibited different physicochemical compositions: the water samples from the Salado de Consotá and La Cristalina springs showed a high content of chlorine, sodium and calcium, while those from the Salpa spring were characterized by high concentrations of sodium, potassium and sulfate. The composition of microbial communities was analyzed using different approaches to generate biodiversity inventories. The results indicated that these springs harbor a microbial community made up mostly of Gram-negative, motile, litotrophic, heterotrophic, halotolerant and halophilic microorganisms, some of which have been previously reported in marine environments. Most isolated microorganisms belonged to the Gamma and Alphaproteobacteria classes, but differences in the microbial composition of each spring were evident. Our study revealed that these springs are a source of new taxonomic diversity as shown by the newly characterized bacterial species.
Key words: Saline springs, microbial diversity, halophilic bacteria, halotolerant bacteria.
Introducción
Colombia es considerado uno de los países con más diversidad biológica, pues en una extensión continental de aproximadamente 114.000.000 hectáreas, que representa el 0,7 % de la superficie continental mundial, contiene cerca del 10 % de la biota total del planeta (Romero, et al., 2008). Los esfuerzos por generar inventarios de la diversidad biológica y su relación con la estructura y la función de los ecosistemas no han incluido la diversidad microbiana, entendida en este contexto como la diversidad de bacterias y arquea en ambientes naturales. Este hecho es decisivo si se analiza el papel clave de los microorganismos en el mantenimiento de la biosfera al regular los ciclos biogeoquímicos de los principales elementos y participar en los procesos de descomposición de la materia orgánica y de los compuestos contaminantes y en el ciclo de nutrientes. Además, los microorganismos son fuente promisoria de energía renovable o sostenible en forma de biogás, bioetanol, o células microbianas combustibles (Prakash, et al., 2013). En los trabajos clásicos de Torsvik, et al. (1990), se demostró que era posible encontrar miles de taxones bacterianos en un gramo de suelo, sin embargo, esta diversidad microbiana sigue a la espera de ser descubierta. Por otra parte, cálculos recientes han indicado que el número de nombres científicamente validados de las especies de bacterias y arquea cultivadas en laboratorio era de aproximadamente 12.000, lo que se consideró como una subestimación del valor real (Chun & Rainey, 2014), ya que la complejidad de la diversidad microbiana es más grande que la estimada con base en el cálculo del número de especies cultivadas en laboratorio.
En un reciente análisis de los estudios sobre biodiversidad en Colombia (Arbeláez-Cortés, 2013), se encontró que los microorganismos estaban poco representados, lo que indica el escaso conocimiento que se tiene sobre su diversidad. Sin embargo, sí existen estudios sobre la diversidad microbiana de diversos hábitats naturales en el país (Baena, et al., 1998; Díaz-Cárdenas, et al., 2010a, Díaz-Cárdenas, et al., 2010b; Baena, et al., 2011; Rubiano-Labrador, et al., 2013; Bohórquez, et al., 2011; Delgado, et al., 2014). En particular, los hábitats salinos pueden ser excelentes modelos para el estudio de la diversidad microbiana y la ecología de los microorganismos en diferentes concentraciones de sal (Ventosa, et al., 2014). Aunque el aislamiento en cultivo puro de organismos halófilos no es tarea fácil, es un reto que vale la pena porque con el organismo aislado es posible identificar las características fisiológicas, metabólicas y genéticas aprovechables biotecnológicamente (Andrei, et al., 2012). La alta demanda de biocatalizadores que se ajusten a las condiciones de los procesos industriales ha despertado el interés por la búsqueda de nuevas enzimas, cuyo punto de partida es la diversidad biológica, incluida la de los genes, las especies y los ecosistemas, y cabe señalar que los microorganismos concentran la mayor diversidad metabólica y filogenética en el planeta.
En este artículo se presentan los resultados de la exploración de la diversidad microbiana de manantiales salinos que se caracterizan por aflorar sobre o cerca de un trazo de falla geológica, a pocos metros de drenajes mayores (Cortés & Cardona, 2006), algunos de los cuales se utilizaron en épocas precolombinas para la extracción de sal con diferentes fines, desde el consumo humano hasta la orfebrería (Acevedo & Martínez, 2004; Tistl, 2004). En la región suroccidental de la Cordillera Central de Colombia se han reportado numerosos manantiales salinos que, por su estrecha relación con las aguas subterráneas, se consideran fuente de recursos minerales en concentraciones explotables y de recursos hídricos y cuya principal aplicación actual es la balneología recreativa y terapéutica (Alfaro, 2002). Sin embargo, el desconocimiento de su diversidad microbiana no ha permitido considerarlos fuente de recursos biológicos.
Materiales y métodos
Sitios de estudio
Se seleccionaron tres manantiales salinos, dos en la Cordillera Central, en el departamento de Risaralda, Colombia: el Salado de Consotá (SC) (4°47'18.9" N - 75°41'35.0" O), a 1.391 m.s.n.m, y el manantial La Cristalina (LC) (4°45'50.1" N - 75°43'55.1" O), a 1.288 m.s.n.m. El tercer manantial salino, denominado Salpa (SP) (05°46'09.2" N - 73°06'09.7" O), se localiza en la Cordillera Oriental, en el departamento de Boyacá, a 2.517 m.s.n.m (Figura 1S, http://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/199/1008).

Muestreos y análisis
Se hicieron muestreos puntuales en los tres manantiales entre 2006 y 2009, recolectando muestras de agua a diferentes profundidades: en el manantial SC la profundidad oscilaba entre 90 y 100 cm, en el manantial LC entre 70 y 80 cm y en el SP entre 100 y 120 cm. Las muestras se transportaron a 4 °C hasta el laboratorio de la Pontificia Universidad Javeriana y se procesaron en el curso de las siguientes 18 horas para posteriores análisis (fisicoquímicos, isotópicos, recuentos microbianos totales y aislamiento de microorganismos). La temperatura y el pH se tomaron en el sitio con un potenciómetro Hach® equipado con sondas para pH y temperatura. Se determinó la conductividad eléctrica, los sólidos disueltos totales (SDT), y las concentraciones de cloruro, potasio, calcio, sodio, magnesio, nitrato, sulfato, fósforo total y hierro, siguiendo las metodologías estándar descritas en los Standard Methods for the Examination of Water and Wastewater(APHA, 2005).
La determinación de los isotopos estables del oxígeno (O18) y del hidrógeno (deuterio -H2), y del isotopo radiactivo del hidrógeno tritio (H3), se hizo mediante espectrofotometría de masas (Stable Isotope-Ratio Mass-Spectrometer, IRMS) en el Laboratorio de Isotopía Ambiental de la Universidad de Waterloo, en Ontario, Canadá.
Análisis de las variables fisicoquímicas
Los parámetros fisicoquímicos se analizaron mediante un análisis de varianza (ANOVA), para lo cual previamente se verificó la distribución normal de los datos de cada variable con la prueba de bondad de ajuste a una distribución normal de Kolmogorov Smirnov. Posteriormente se comprobó la homogeneidad de varianzas de los datos de cada variable con la prueba de Levene. Cuando se cumplió el supuesto de normalidad, pero no el de homogeneidad de la varianza, se utilizó el T2 de Tamhane. Cuando el análisis de ANOVA mostró diferencias significativas entre los parámetros eva- luados, se aplicó la prueba de diferencia honestamente significativa (honestly significant difference, HSD) de Tukey y Sheffé para hacer las comparaciones múltiples de medias y determinar los sitios específicos entre los que se expresaba la diferencia. Los análisis se realizaron con el programa SPSS v 16.0 (IBM SPSS Data Collection).
Caracterización de la composición de las comunidades microbianas en los manantiales salinos
La caracterización de la composición de las comunidades microbianas se llevó a cabo determinando sus características morfológicas mediante microscopía electrónica de barrido en los manantiales SC y SP, registrando los recuentos totales de microrganismos mediante la técnica de tinción con 4,6-diamidino-2-fenilindol diclorhidrato (DAPI), determinando la abundancia de grupos microbianos ampliamente reportados en ambientes acuáticos mediante hibridación fluorescente in situ (fluorescent in situ hybridization, FISH), y generando inventarios biológicos mediante el aislamiento de organismos y la identificación taxonómica con el gen ARNr 16S como marcador molecular.
Características morfológicas de las comunidades microbianas presentes en los manantiales salinos Salado de Consotá (SC) y Salpa (SP)
Para observar la comunidad microbiana in situ se utilizó la técnica de microscopía electrónica de barrido en los manantiales SC y SP siguiendo el protocolo descrito por Díaz-Cárdenas (2011); la evaluación microscópica se hizo según el protocolo de Glauert (1975). Las muestras se cubrieron con oro con un sputter coater(BioRad SC500) antes de observarlas y obtener imágenes digitales con un microscopio electrónico de barrido FEI Quanta 200 operado a 100kV.
Determinación del número total de células microbianas y detección de grupos microbianos específicos
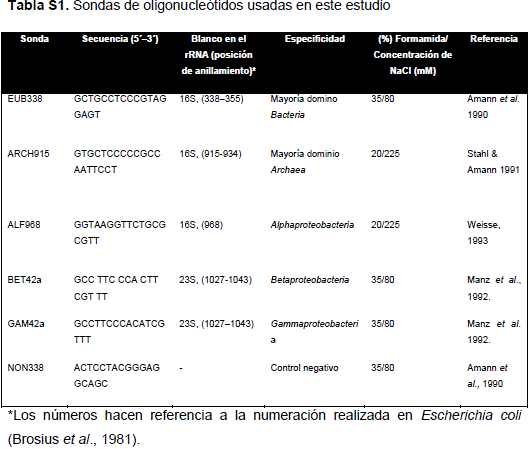
Se utilizó la técnica de tinción de las células con DAPI (Sigma) y se detectaron los grupos microbianos específicos con la técnica FISH siguiendo el protocolo de Pernthaler, et al. (2001) y analizando cada muestra dos veces. Con el fin de mantener la integridad de las células para los análisis con DAPI y FISH, se fijaron con formaldehido (Scharlau) en una concentración final de 2 % (v/v) en 500 mL de agua recién recolectada. Posteriormente, las células se recolectaron por filtración con membranas de policarbonato blanco (poro de 0,22 mm y 47 mm de diámetro, GTTP Millipore). El recuento total de células se hizo según el protocolo de Bohórquez, et al. (2011) en un microscopio de epifluorescencia (Nikon-Eclipse 50i) equipado con un objetivo de 100X Pan Fluor y un filtro de excitación triple DAPI-FITC-TRITC (Nikon). La detección de organismos del dominio Archaea se hizo usando la sonda ARCH915 y los del dominio Bacteria, con la sonda EUB338 mediante la técnica de FISH. Para el dominio Bacteria se detectaron organismos de las clases alphaproteobacteria con la sonda ALF968, betaproteobacteria, con la sonda BET42a y gammaproteobacteria, con la sonda GAM42a; estas sondas son complementarias de las regiones conservadas de los genes ARNr 16S o 23S (Tabla 1S, http://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/199/1010). Además, se utilizó la sonda NON338 como control negativo. Las sondas se sintetizaron, purificaron y conjugaron con los fluorocromos Alexa flour 488 o 546 de Invitrogen (USA).
Recuento de células
Se contaron 50 campos visuales por membrana cuando se observaba un número menor de 30 células por campo, y 20 campos visuales si se observaba un número mayor de células para contar 300 células por muestra como mínimo según las recomendaciones de Kirchman, et al. (1982). Se determinó si los recuentos presentaban diferencias significativas entre sitios y épocas de muestreo mediante el método estadístico no paramétrico de Kruskal-Wallis aplicado para el ANOVA de un factor (p<0,05) con el programa Statistix, versión 9.0.
Generación de inventarios biológicos a partir del aislamiento de microorganismos
Para el aislamiento de microorganismos se recolectaron en cada sitio de muestreo 100 mL de agua en botellas de vidrio estériles para ser usados como inóculos microbianos y cinco litros de agua en garrafas plásticas previamente purgadas con agua del manantial para los medios de cultivo utilizados en las diferentes series de aislamiento. Para favorecer el aislamiento de organismos halófilos o halotolerantes aerobios y anaerobios, se prepararon medios básicos (MB) aerobios (MB-AER) y anaerobios (MB-ANA). Los medios MB-AER y MB-ANA se prepararon con agua del manantial en estudio (previamente filtrada), suplementada con 1 mL/L de solución de oligoelementos (un litro de agua destilada contenía 0,06 g de ZnCl2, 0,1 g de MnCl2·4H2O, 0,06 g de H3BO3, 0,12â0,19 g de CoCl2·6H2O, 0,025 g de NiCl2·6H2O, 0,025-0,035 g de Na2MoO4·2H2O, 0,02 g de CuCl2·2H2O, 0,05 g de AlCl3 anhidro, 0,03 g de Na2SeO3 y 1,5 mL de FeCl2·4H2O diluido en HCl al 25 % (v/v)). Además, el MB-ANA fue suplementado con 0,5 g/L de monohidrato de L-cisteína hidroclorada (Sigma) y 1 mL/L de resazurina (Sigma) al 0,1 % (p/v). El pH de los medios se ajustó con una solución de hidróxido de sodio (NaOH) (Merck) al 10 % (p/v), al 6,5 para las series de los manantiales SC y SP y al 8,0 para las series de cultivo del manantial LC, teniendo en cuenta los datos de pH tomados en cada manantial. Después de ajustar el pH de los medios, se transfirió un volumen de 5 mL de medio de cultivo de la serie MB-AER a tubos de ensayo. El medio de cultivo MB-ANA se preparó siguiendo los protocolos descritos por Rubiano-Labrador, et al. (2013). Teniendo en cuenta que los microorganismos halófilos de los dominios Archaea y Bacteria tienen un amplio espectro metabólico, se utilizaron diferentes sustratos orgánicos comúnmente reportados como materia orgánica disuelta en sistemas acuáticos proveniente del medio ambiente circundante o de fuentes autóctonas como el fitoplancton, bacteriopláncton y perifiton, ya sea por la producción de exopolisacáridos, la muerte celular o la liberación de exudados (Gremm & Kaplan, 1997). Además, a algunas series se les agregó un suplemento de antibióticos para favorecer el aislamiento de organismos del dominio Archaea como se ha descrito anteriormente (Elshahed, et al., 2004). Después de la esterilización, los MB-AER fueron suplementados con 0,2 g/L de extracto de levadura (Sigma) y con diferentes fuentes de carbono como ácidos orgánicos, alcoholes, azúcares, aminoácidos, polisacáridos y lípidos (Díaz-Cárdenas, 2011). Además, a algunas series se les agregó un suplemento de 75 mg/mL de kanamicina (Sigma) o 75 mg/mL de ampicilina (Sigma). Los antibióticos se prepararon en agua desionizada y se esterilizaron filtrándolos en membranas de 0,22 mm de tamaño de poro (Millipore). A los MB-ANA se les agregó un suplemento de 0,05 mL de sulfuro de sodio (Na2 S·9H2O) (Sigma) al 2 % (p/v), 0,1 mL de bicarbonato de sodio (NaHCO3) (Merck) al 10 % (p/v), 0,2 g/L de extracto de levadura, y aceptores de electrones como sulfato de sodio (Na2 SO4) (Sigma), tiosulfato de sodio (Na2 SO3) (Sigma) y nitrato de potasio (KNO3) (Merck), todos en una concentración final de 10 mM. Tanto las fuentes de carbono como los aceptores de electrones se añadieron a cada tubo a partir de soluciones estériles anaerobias preparadas en agua reducida (Hungate, 1969). Las diferentes series de cultivo se inocularon con 1 mL del agua del manantial (como inóculo microbiano) por cada 5 mL de medio de cultivo y posteriormente se incubaron a 30 °C en oscuridad y sin agitación. Las series de cultivo se evaluaron periódicamente con un microscopio de contraste de fase (Nikon i50) y objetivo de 100X Pan Fluor.
Aislamiento de cepas microbianas
A partir de las series de cultivo con crecimiento positivo, se hizo una segunda siembra en las mismas condiciones, así como diluciones seriadas de 10-1 a 10-10, hasta obtener cultivos axénicos siguiendo los protocolos descritos por Díaz-Cárdenas, et al. (2010a). La pureza de las cepas se verificó por observación al microscopio y repicaje en medio enriquecido con extracto de levadura (2 g/L) y glucosa (20 mM) hasta observar un único morfotipo.
Identificación taxonómica de los organismos aislados
La identificación taxonómica de los organismos aislados se hizo usando como marcador el gen ARNr 16S. Se extrajo ADN de cada una de las cepas y se hizo la secuenciación del gen marcador (~ 1.300 pb). Se describieron las principales características fenotípicas (tinción de Gram, morfología y estructuras celulares, metabolismo y condiciones de salinidad, temperatura y pH óptimas para el crecimiento) según lo reportado por Díaz-Cárdenas (2011).
Para la extracción del ADN microbiano se siguió el método descrito por López, et al. (2014). La precipitación del ADN se hizo con etanol al 70 % (v/v) y la segunda suspensión en 50 ml de tampón TE (Tris-HCl 10 mM pH 7.5, EDTA 1 mM); finalmente se almacenó a -20 °C hasta su uso. Los métodos usados para la amplificación y secuenciación del gen ARNr 16S y para los análisis filogenéticos han sido previamente descritos por Díaz-Cárdenas, et al. (2010b) y López, et al. (2014). El gen que codifica para el ARNr 16S se amplificó con los iniciadores Fd1 (CCG AAT TCG TCG ACA ACA GAG TTT GAT CCT) y Rd1 (CCC GGG ATC CAA GCT TAA GGA GG TGA TCC AGC C) (Redburn & Patel, 1993). El producto de la reacción de secuenciación fue separado por electroforesis capilar en un analizador automático de biología molecular (Genetic Analyzer 3130xl, Applied Biosystems) usando el programa de análisis de secuenciación de ADN v5.2 (Applied Biosystems) (Servicio de Biología Molecular de la Universidad de Griffith, Brisbane, QLD, Australia). Todos los datos obtenidos a partir de los archivos de secuenciación automática del ADN se editaron manualmente para determinar nucleótidos codificados erróneamente usando el programa de edición Sequence Aligment Editor versión 7.0 de BioEdit (http://www.mbio.ncsu.edu/RNaseO/info/programs/BIOEDIT/bioedit.htmL).
Todas las secuencias del gen ARNr 16S obtenidas se compararon con las registradas en el GenBank usando una base de datos no redundante (nr) y el algoritmo BLAST (Basic Local Alignment Search Tool, http://www.ncbi.nlm.nih.gov/BLAST/) y con las de la base de datos del RDP, versión 11, usando la opción sequence match (http://redp.cme.msu.edu/seqmatch/seqmatch_intro.jsp).
Resultados y discusión
Caracterización de la composición fisicoquímica del agua de los manantiales salinos
Las características fisicoquímicas de estos manantiales se presentan en la Tabla 1. Los manantiales SC y SP registraron una temperatura promedio de 21 °C, mientras que la del manantial LC fue más alta, de 25 °C. La temperatura promedio del agua de los manantiales SP y LC que se registró fue superior a la temperatura ambiente promedio de la región: en Paipa la temperatura ambiental oscila entre 13 y 15 °C, mientras que en la zona del manantial LC oscila entre 18 y 25 °C. El pH registrado en el manantial SC oscilaba entre 6,1 y 7,4; en el manantial LC se mantuvo en alrededor de 8,5, y en el manantial SP fue cercano a 6,5. Según los rangos de conductividad eléctrica reportados por LaBaugh (1989), los manantiales SC y LC se clasifican como hábitats polisalinos (33.000-40.000 mS/cm), en tanto que el manatial SP como un hábitat eusalino (46.000-55.000 mS/cm). Los análisis químicos del agua de los manantiales SC y LC mostraron un alto contenido de cloro (Cl-) y calcio (Ca2+), y las aguas del manantial SP se caracterizaron por una alta concentración de sodio (Na+), potasio (K+) y sulfato (SO42-).
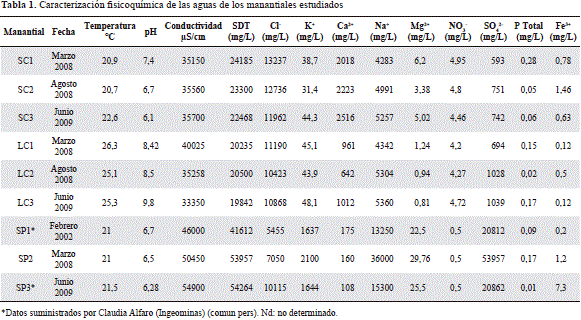
El análisis de varianza (ANOVA) de los datos fisicoquímicos mostró diferencias significativas (p<0,05) para los parámetros de temperatura, pH, conductividad, SDT, cloro, calcio, potasio, sodio, nitrato y sulfato. Para detectar diferencias significativas entre los sitios de muestreo de las variables mencionadas se hizo un análisis posterior (HDS de Tukey y Sheffé) en el que se determinó que los parámetros de SDT, calcio y nitrato diferían significativamente en los tres manantiales. Con respecto a la conductividad eléctrica y a las concentraciones de potasio, cloro, sodio y sulfato, no se registraron diferencias significativas entre los manantiales SC y LC, localizados en la Cordillera Central, pero sí entre estos dos manantiales y el manantial SP, localizado en la Cordillera Oriental. En cuanto a la temperatura y el pH, se observó que el manantial LC, que presentaba los rangos de temperatura y de pH más altos, difería significativamente de los manantiales SC y SP que, en cambio, no presentaron diferencias significativas entre ellos para estos dos parámetros (datos no mostrados). El análisis muestra diferencias en las características fisicoquímicas de cada uno de los sitios de muestreo, lo cual es indicativo de variaciones en las condiciones ambientales locales. Las variables que mejor describían estas diferencias fueron el contenido de cloro y calcio en el manantial SC, la conductividad y el contenido de sulfato, potasio y sodio en el manantial SP y las condiciones de pH y temperatura en el manantial LC. Los datos también mostraron que la composición fisicoquímica de los manantiales fue relativamente estable durante el tiempo de muestreo en cada uno de los manantiales.
Para la interpretación de los isotopos estables del agua, los resultados de oxígeno-18 (O18) y deuterio (H2) (Tabla 2S, http://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/199/1011) se graficaron con respecto a la línea meteórica mundial (Standard Mean Ocean Water, SMOW) (Figura 2S, http://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/199/1009. La representación gráfica indicó que las aguas provenientes de los manantiales no se ajustan a la línea SMOW y que, por lo tanto, su origen no está relacionado con la presencia de agua meteórica recientemente acumulada en los acuíferos. Por otra parte, los valores del contenido de tritio (H3) para las tres muestras fueron menores a 0,8 unidades de tritio (UT) (datos no mostrados), lo que indica que estos manantiales contienen agua antigua, infiltrada hace más de 50 años. Esta información isotópica indica que los tres manantiales fluyen a partir de aguas subterráneas antiguas y que su régimen hidrológico no está directamente relacionado con recargas pluviométricas recientes. Por otra parte, tal como lo describe Sigee (2005), la permanencia del agua en un acuífero, su interacción con la roca del reservorio y la presencia de minerales evaporíticos podrían ser las causas principales de las diferencias en las características fisicoquímicas de las aguas de los manantiales, lo que también demostraría que los manantiales SC y LC, ubicados en la misma región, se originan de acuíferos diferentes.

Caracterización de la composición de las comunidades microbianas
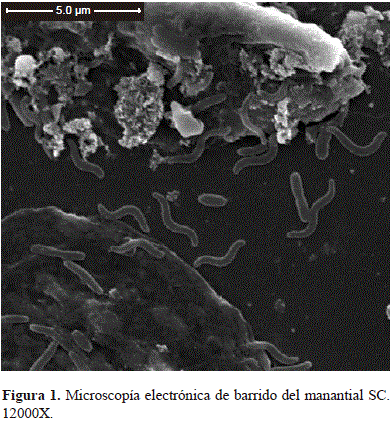
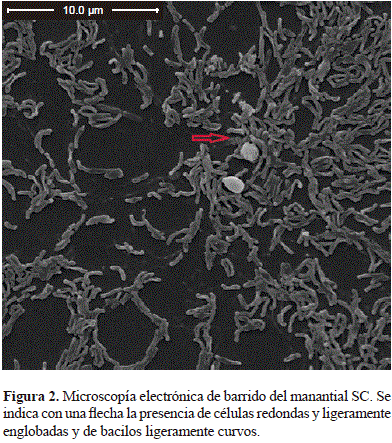
Las características morfológicas de los microorganismos predominantes en estos manantiales se describieron con base en la microscopía electrónica de barrido a partir de muestras de dos de los tres manantiales en estudio. Solamente se obtuvieron imágenes claras de las células microbianas en las láminas incubadas durante 24 horas, porque las láminas con menor tiempo de incubación no presentaron células microbianas, o estas eran escasas, y las láminas con 48 horas de incubación mostraron una alta precipitación de sales, lo que dificultó la observación de las células. En el manantial SC se observaron principalmente bacilos curvos con extremos puntiagudos, de 0,3 mm de ancho por 2,5 a 2,7 mm de largo, bacilos rectos de 0,5 a 0,7 mm de ancho por 1,5 a 2,0 mm de largo (Figura 1), bacilos rectos de extremos redondos de 0,6 mm de ancho por 2,5 mm de largo, solos o en pares, y bacilos rectos de extremo redondo de 0,6 mm de ancho por 2,0 mm de largo. También se observó el desarrollo de una colonia compuesta de bacilos rectos o ligeramente curvos de extremos redondos, de aproximadamente 0,4 a 0,5 mm de ancho por 1,25 a 3 mm de largo, adheridos entre sí posiblemente por la producción de exopolisacáridos (Figura 2). Además, se observó la presencia de células ligeramente englobadas de 3,5 a 4,0 mm de ancho por 5,5 mm de largo, con una pequeña elongación uno de sus extremos (Figura 2), que por su tamaño podrían catalogarse como protozoos según la clasificación de Sieburth, et al. (1978). Estos organismos fagotróficos y depredadores podrían tener influencia en la composición de la comunidad microbiana del manantial en estudio.
Los morfotipos dominantes en las muestras del manantial SP fueron los bacilos rectos o ligeramente curvos con terminación en punta, de 0,6 mm de ancho por 6 mm de largo, los bacilos ligeramente curvos con extremos redondos, de 0,4 mm de ancho por 1,5 a 2,0 mm de largo, los bacilos rectos de extremo redondo, de 0,15 mm de ancho por 1,2 mm de largo, los bacilos de 0,3 mm de ancho por 0,6 mm de largo, y los bacilos de 0,5 a 0,6 mm de ancho por 0,7 a 1,15 mm de largo en pares o individuales; los cocos estaban presentes en menor proporción. También se observaron células filamentosas de más de 0,2 a 0,3 mm de ancho por más de 20 mm de largo (Figura 3). La presencia de diatomeas, principalmente del género Nitzchia, fue evidente en este manantial, lo que se ha reportado comúnmente en ambientes marinos (Salm, et al., 2009) y lagos metasalinos (Gell & Gasse, 1990) (Figura 4).
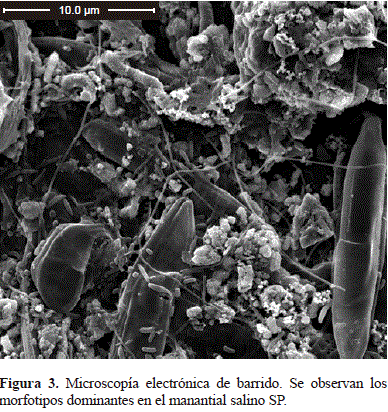

En general, mediante microscopía electrónica de barrido se detectaron diferencias en la morfología celular en los manantiales SC y SP. En el primero predominaban los bacilos curvos o espirilos, mientras que en el segundo predominaron los bacilos rectos y los cocos. Por otra parte, en el manantial SC se observaron células similares a las de los protozoos, posibles causantes de la mortalidad bacteriana en la mayoría de los ambientes acuáticos, lo que restringe su superpoblación. La presencia de diatomeas, que hacen parte del fitoplancton y participan en procesos de producción primaria, se observó únicamente en el manantial SP; los cambios en la composición de la comunidad fitoplanctónica pueden influenciar la composición de las comunidades bacterianas (Rooney-Varga, et al., 2005). El fitoplancton libera aproximadamente el 25 % del total del carbono orgánico fijado por fotosíntesis (DuRand, et al., 2001) y posteriormente la materia orgánica disuelta es rápidamente consumida y remineralizada por la comunidad bacteriana, por lo cual, los cambios en su composición se ven reflejados en la composición de la comunidad microbiana (Obernosterer & Herndle, 1995).
Los recuentos de células totales durante los tres muestreos variaron entre 1,8 y 2,8 x 106 células/mL/ en el manantial SC, entre 1,0 y 2,6 x 106 células/mL/ en el manantial LC y entre 1,7 y 4,8 x 106 células/mL en el manantial SP (Tabla 2). Estos valores son mayores a los reportados por Bohórquez, et al. (2011) en muestras de aguas de manantiales termales del Parque Nacional Natural Los Nevados en Colombia (2,35 (±0,7) x 105 células/mL), y se encuentran en los rangos de 105 a 106 células/mL reportados por Sogin, et al. (2006) para aguas superficiales oceánicas. El análisis estadístico no arrojó diferencias significativas entre los sitios de muestreo (p=0,7), pero sí entre las épocas de muestreo (p=0,025) en cada manantial, lo que sugiere cambios en la abundancia de las comunidades microbianas al interior de cada hábitat a lo largo del tiempo. Por otra parte, en los tres manantiales se observó el predominio de organismos del dominio Bacteria (detectados con la sonda EUB338) en comparación con los del dominio Archaea (detectados con la sonda ARCH915) (Figura 5). Los recuentos del dominio Bacteria no presenta- ron diferencias significativas entre los sitios de muestreo (p=0,7), mientras que los recuentos de Archaea sí (p=0,004). Por el contrario, al evaluar en cada manantial las diferencias entre los recuentos obtenidos a lo largo del tiempo de muestreo, se detectaron diferencias significativas para los organismos del dominio Bacteria (p=0,025), pero no para los organismos del dominio Archaea (p=0,13). Los resultados sugieren que al interior de cada comunidad en cada manantial, la abundancia de Bacteria cambia constantemente, mientras que la abundancia de las poblaciones de Archaea puede ser más estable a lo largo del tiempo. Estas variaciones podrían estar relacionadas principalmente con las características geológicas y con los factores ambientales locales, tal como se ha reportado en diferentes sistemas acuáticos (Pedersen, et al., 1996). Se requieren, sin embargo, estudios posteriores para identificar las variables ambientales que afectan la distribución y la abundancia de las poblaciones microbianas.
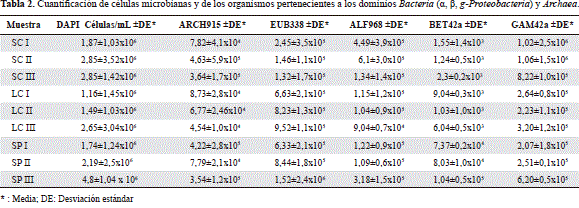
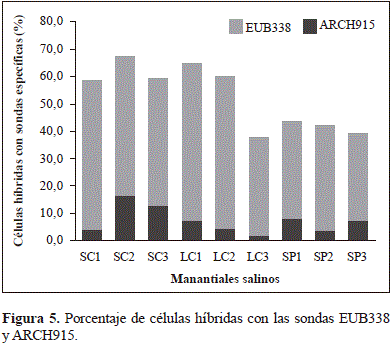
Por otra parte, en el recuento de organismos pertenecientes a las alfaproteobacterias, las betaproteobacterias y las gamaproteobacterias, se encontró que en los tres manantiales predominaban las gamaproteobacterias, como se ha reportado en otros ambientes salinos (Wu, et al., 2006). En el manantial SC el porcentaje de células del dominio Bacteria que hibridó con la sonda GAMM42a osciló entre el 41 y el 67 %, en LC, entre el 27 y el 39 % y en SP, entre el 30 y el 40 %. Los resultados con la sonda ALF968 para organismos de la clase de las alfaproteobacterias indicaron que estos representaban menos del 20 % del total de células del dominio Bacteria en los tres manantiales y durante los tres muestreos. Con respecto a los recuentos con la sonda BETA42a para organismos de la clase de las betaproteobacterias, se obtuvieron recuentos inferiores al 0,2 % del total de células de Bacteria en los manantiales SC y LC durante los tres muestreos, pero en el manantial SP estos recuentos fueron más altos (7 a 12 %), en tanto que el análisis estadístico indicó diferencias significativas entre los tres manantiales para esta sonda, lo cual evidencia variaciones en la composición de las comunidades microbianas a escala espacial.
Al evaluar en cada manantial la abundancia de las poblaciones de organismos de las tres clases de proteobacterias, se encontraron diferencias significativas para los recuentos obtenidos con las sondas ALF968 (p=0,02) y GAMM42a (p=0,02), lo cual confirma que la abundancia de poblaciones del dominio Bacteria cambia constantemente a lo largo del tiempo para cada sitio de muestreo.
La técnica de FISH es una herramienta útil para cuantificar microorganismos en ambientes naturales, sin embargo, como cualquier otra técnica también tiene limitaciones asociadas con la dificultad de detectar células microbianas con bajo contenido de ribosomas, ya que generan poca intensidad en la señal de fluorescencia, lo que dificulta su detección, así como en la penetrabilidad y especificidad de la sonda (Yeates, et al., 2003). Es posible que debido a estas limitaciones las sondas utilizadas para la detección de organismos de los dominios Bacteria y Archaea hayan permitido detectar solamente entre el 40 y el 70 % del total de células identificadas con el DAPI.
Nuestros resultados sugieren que en la comunidad microbiana de los tres manantiales predominan más los organismos del dominio Bacteria que los del dominio Archaea, lo cual se ha reportado en ambientes con salinidad de hasta 16 % (p/v) (Dong, et al., 2006; Perreault, et al., 2007; Clementino, et al., 2008); por otra parte, Swan, et al. (2010) encontraron que en ambientes moderadamente hipersalinos (5 % (p/v) los organismos del dominio Archaea eran más que los del dominio Bacteria. Con respecto a la predominancia de Bacteria sobre Archaea, se ha discutido la razón de que en un mismo ambiente se tienda a encontrar menor diversidad en Archaea que en Bacteria, pese a que las arqueas contribuyen en gran proporción al total de los filotipos procariotas presentes en la mayoría de los ambientes en los que se han estudiado organismos de ambos dominios (Aller & Kemp, 2008). Tal como lo señalan estos autores, las arqueas pueden colonizar microambientes que son más restrictivos para la mayoría de las bacterias, y estas últimas pueden estar más dispersas en los diferentes microambientes de un mismo macroambiente, lo cual dificulta establecer la razón de los bajos recuentos de Archaea frente a Bacteria cuando se analiza la diversidad procariota a partir de muestras que no discriminan entre estos posibles microambientes. Aunque los recuentos de Archaea hayan sido bajos, su presencia podría indicar interacciones metabólicas con Bacteria necesarias para el establecimiento de comunidades microbianas funcionales en estos ambientes.
Generación de inventarios biológicos
Se observó crecimiento microbiano en todas las series de cultivo, pero no se obtuvieron aislamientos en los cultivos puros de todas ellas. Una de las limitantes del aislamiento fue la purificación, así como la carencia de información para recrear las condiciones naturales de crecimiento en términos de, entre otros, los nutrientes y elementos traza presentes en el agua de los manantiales. Además, los medios de cultivo son a menudo poco eficientes para reproducir las condiciones endógenas bióticas y abióticas requeridas y, por otro lado, los microorganismos no se adaptan inmediatamente a estos cambios en las condiciones de crecimiento. Esto es lo que se ha llamado great plate count anomaly, que alude al fenómeno de que solo es posible recuperar un bajo porcentaje de células viables de comunidades microbianas en cultivo puro mediante los métodos tradicionales con agar o cualquier otro agente gelificante (Tanaka, et al., 2014), aunque los recuentos totales de microorganismos obtenidos mediante DAPI o FISH sean altos, como se observó en este estudio. El análisis de la secuencia del gen ARNr 16S permitió identificar estos aislamientos como pertenecientes al dominio Bacteria. Los medios selectivos y la utilización de kanamicina y ampicilina no fueron suficientes para favorecer la recuperación de arqueas. Los organismos aislados se identificaron taxonómicamente en los filos Proteobacteria (alfa, delta y gama), Bacteroidetes, Firmicutes, Synergistetes y Actinobacteria. La mayor parte de los géneros microbianos se aislaron de los grupos de alfaproteobacterias y gamaproteobacterias, datos que fueron sustentados con los recuentos bacterianos hechos con sondas de FISH.
A partir de las diferentes series de cultivo se aislaron y purificaron 62 cepas bacterianas. Del manantial SC se aislaron 31 cepas bacterianas, la mayoría previamente reportadas en ambientes salinos, principalmente marinos, relacionadas con los géneros Roseovarius, Thalassospira, Thalassobaculum, Labrenzia, Stappia, Thioclava, Dethiosulfovibrio, Vibrio y Prolixibacter. Del manantial LC se obtuvieron 14 cepas puras, cinco de las cuales han sido reportadas en ambientes marinos y se relacionan con los géneros Oceanibaculum, Dethiosulfovibrio y Vibrio; las nueve cepas restantes presentaron una distribución cosmopolita. Del manantial SP se aislaron 17 cepas, de las cuales cuatro, relacionadas con los géneros Desulfovibrio, Dethiosulfovibrio, Nitratireductory Shewanella, han sido previamente reportadas en ambientes salinos o marinos. Estas cepas se conservan en glicerol (20 % v/v) a -70 °C y están depositadas en la colección de microorganismos de la Pontificia Universidad Javeriana.
En la Tabla 3 se presentan las características identificadas en los aislamientos. Más del 70 % de las cepas bacterianas son móviles, lo cual puede ser relevante en manantiales donde la materia orgánica disuelta de bajo peso molecular (azúcares, fosfatos y aminoácidos) está disponible en un tiempo y espacio limitado en la columna de agua, por lo cual una conducta sensorial móvil proporciona a las células ventajas competitivas que favorecen su supervivencia (Alexandre, et al., 2004).
Las cepas USBA 369, USBA 355 y USBA 82 se destacaron por presentar rasgos fenotípicos y valores de similitud del gen ARNr 16S inferiores al 97 %, umbral usado corrientemente para la diferenciación de especies (Chun & Rainey, 2014).
Dichos datos indican que estas cepas podrían catalogarse como nuevas especies bacterianas. Se realizaron estudios taxonómicos usando el enfoque polifásico, en el que se consideraron múltiples dimensiones como las características fenotípicas, genotípicas y quimotaxonómicas (datos no mostrados).
La cepa USBA 369, aislada del manantial SC, registró un porcentaje de similitud del gen ARNr 16S con Aurantimonas altamirensisde 95 % (Mendes, et al., 2008); esta especie, aislada de la cueva Altamira en España, se reclasificó posteriormente como Aureimonas altamirensis(Rathsack, et al., 2011). Esta cepa es un organismo quimioheterotrófico, Gram-negativo, móvil, aerobio facultativo y halotolerante (Figura 6). Debido a sus características genotípicas, fenotípicas y quimotaxonómicas se propuso como una nueva especie del género Aurantimonas, denominada Aurantimonas salsifontis(Díaz-Cárdenas, et al., 2015, en preparación), aunque también presenta una gran similitud con miembros de los géneros Aureimonas (Rathsack, et al., 2011) y Rhizobium. El género Aurantimonas hace parte del orden Rhizobiales y, debido a las nuevas clasificaciones de las especies antes pertenecientes al género, a la fecha solo la especie tipo Aurantimonas coralicida(Denner, et al., 2003), aislada de muestras de corales, hace parte del género.

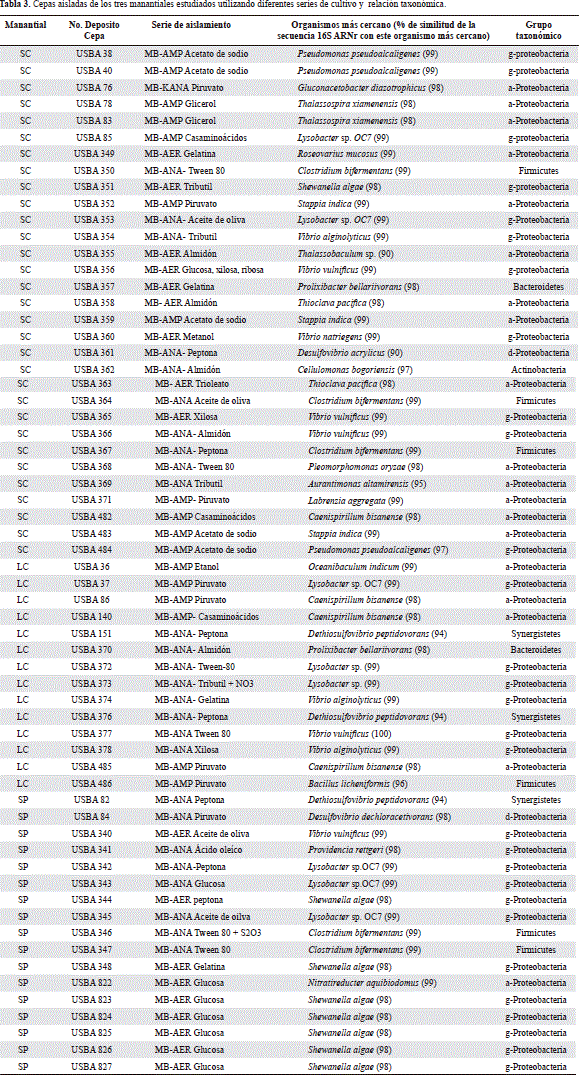
El bajo porcentaje de similitud de la secuencia del ARNr 16S de la cepa USBA 355 (Figura 7) con organismos del género Thalassobaculum (90 % de similitud) y las diferencias fenotípicas encontradas indican que la cepa USBA 355 podría catalogarse como un nuevo género de la familia Rhosdospirillaceae, a la cual pertenece el género Thalassobaculum, el más cercano a esta cepa. El análisis polifásico (datos no mostrados) mostró que, efectivamente, la cepa USBA 355 se identifica como un nuevo género de la familia Rhosdospirillaceae denominado Tistlia; la especie ha sido denominada Tistlia consotensis(Díaz-Cárdenas, et al., 2010b) por su relación con el sitio de aislamiento, el manantial SC. El análisis proteogenómico hecho para dilucidar los mecanismos de adaptación a condiciones de salinidad variables como las de este manantial mostró que hay una expresión diferencial de proteínas cuando el organismo crece en hábitats donde hay ausencia de cloruro de sodio o altas concentraciones de este compuesto comparadas con la concentración promedio (Rubiano- Labrador, et al., 2014).
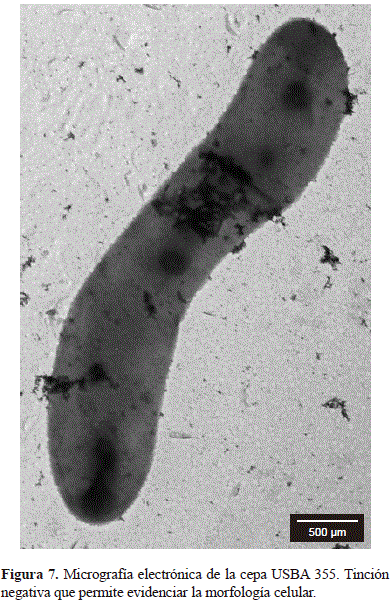
El análisis preliminar de la secuencia del ARNr 16S de la cepa USBA 82 indicó que está relacionada con Dethiosulfovibrio peptidovorans(94 % de similitud) del filo Synergistetes (Jumas-Bilak, et al., 2009), el cual incluye los géneros Dethiosulfovibrio, Synergistes, Anaerobaculum, Aminobacterium, Thermanaerovibrio, Aminomonas, Thermovirga, Aminiphilus, Jonquetella y Cloacibacillus (Rees, et al., 1997, Baena, et al., 1998, Baena, et al., 1999a; Baena, et al., 1999b; Dahle & Birkeland, 2006, Díaz, et al., 2007, Jumas-Bilak, et al., 2007, Ganesan, et al., 2008), cuya principal característica es su capacidad para degradar aminoácidos en condiciones anaerobias (Vartoukian, et al., 2007). El análisis polifásico demostró que la cepa aislada correspondía a una nueva especie del género Dethiosulfovibrio, que se denominó Dethiosulfovibrio salsuginis(Figura 8) (Díaz-Cárdenas, et al., 2010a).
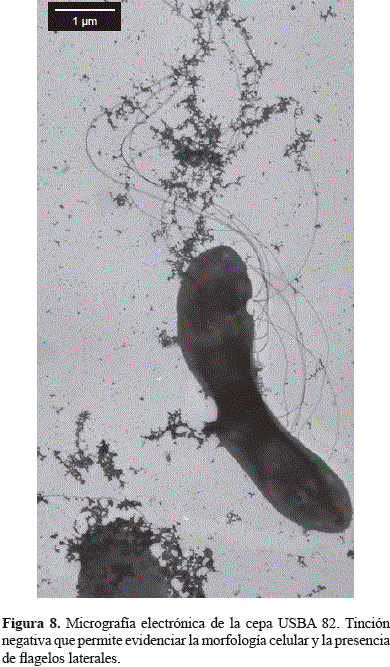
En términos generales, la caracterización fenotípica de los microorganismos aislados, que incluyó la determinación del rango óptimo de salinidad (medida como concentración de cloruro de sodio), demostró que aunque los tres manantiales presentaban una salinidad incluso más alta que la reportada para ambientes marinos, la mayoría de las cepas aisladas no tenía un requerimiento específico del ion Na+, ni siquiera aquellas estrechamente relacionadas con organismos de ambientes marinos que requieren de este ión, tales como Stappia (Lai, et al., 2010), Labrenzia (Biebl, et al., 2007), Roseovarius (Biebl, et al., 2005), Thioclava (Sorokin, et al., 2005) y Vibrio. También vale la pena resaltar que a pesar del elevado pH del manantial LC, que oscila entre 8,4 y 9,8 (lo cual lo diferencia de los otros dos manantiales), ninguno de los organismos aislados se relacionó con organismos alcalófilos. Estos resultados difieren de los obtenidos en otros ambientes salinos alcalinos, en los cuales se han aislado principalmente organismos del filo Firmicutes relacionados con bacterias Gram positivas de bajo contenido de G+C pertenecientes a los géneros Bacillus, Paenibacillus, Alkalibacillus, Exiguobacterium, Planococcus, Enterococcus y Vagococcus (Joshi, et al., 2008). Un elemento común en los tres manantiales fue el aislamiento de organismos relacionados con los géneros Vibrio y Lysobacter, mientras que los organismos del género Caenespirillum y Prolixibacter solo se aislaron de los manantiales SC y LC; los organismos de los géneros Desulfovibrio, Shewanellay Clostridium, de los manantiales SC y SP, y los organismos del género Dethiosulfovibrio, de los manantiales LC y SP (Figura 9).
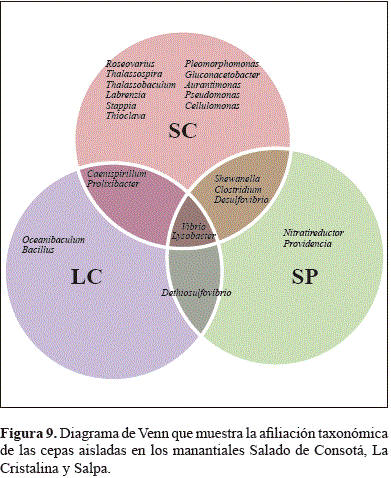
Este estudio constituye uno de los pocos desarrollados en Colombia sobre la composición de las comunidades microbianas de manantiales polisalinos y eusalinos que fluyen a partir de aguas subterráneas con más de 50 años de antiguedad. La biodiversidad microbiana de estos manantiales salinos incluye organismos halotolerantes de clase alfaproteobacteria, deltaproteobacteria y gama-proteobacteria Firmicutes, Bacteroidetes, Actinobacteria y Synergistetes y presentan una gran heterogeneidad fenotípica con respecto a los organismos más relacionados filogenéticamente. En los manantiales SC y LC se aislaron cepas bacterianas relacionadas principalmente con organismos halófilos de ambientes marinos. Por el contrario, la composición microbiana del manantial SP fue bastante heterogénea y se relacionó con organismos halotolerantes y halófilos de agua dulce, sedimentos de lagunas de cristalización, manantiales sulfurosos marinos, estuarios y suelo. Estos manantiales albergan una diversidad microbiana desconocida que representa un reservorio de diversidad genética inexplorada y podría ser fuente de enzimas, metabolitos y rutas metabólicas de interés ecológico y biotecnológico.
Agradecimientos
A Luisa Fernanda Bernal Aragón, por su participación en el aislamiento de microorganismos de los manantiales SC y LC. Esta investigación contó con el apoyo financiero de Colciencias y la Pontificia Universidad Javeriana (Proyecto de investigación 1203-405-20235, ID 2743), y con el permiso de acceso a recursos genéticos del Ministerio de Ambiente y Desarrollo Sostenible No. 56 de 2013.
Conflicto de intereses
Los autores declaran no tener conflicto de intereses
Bibliografía
Acevedo A, & Martínez S. (2004). La sal y las mercaderías en la provincia de Quimbaya. Primeras noticias y crónicas de los salados del río Consotá. En: C. López y M. Cano (Eds). Cambios ambientales en perspectiva histórica. Ecorregión del Eje Cafetero. Proyecto UTP-GTZ, Pereira. p. 167-187. [ Links ]
Alfaro C. (2002). Geoquímica del sistema geotérmico de Paipa. Ingeominas, informe inédito. Bogotá. p. 88 [ Links ]
Alexandre, G., Greer-Phillips, S., Zhulin, I.B. (2004). Ecological role of energy taxis in microorganism. FEMS Microbiol Reviews; 28 (1): 113-126. [ Links ]
Aller, J.Y. & Kemp, P.F. (2008). Are Archaea inherently less diverse than Bacteria in the same environments? FEMS Microbiol Ecol; 65 (1): 74-87. [ Links ]
Andrei, A.S., Banciu, H.L., Oren, A. (2012). Living with salt: Metabolic and phylogenetic diversity of Archaea inhabiting saline ecosystems. FEMS Microbiol Lett 330: 1-9. [ Links ]
American Public Health Association-American Water Works Association & Water Environment Federation. (2005). Standard Methods for the Examination of Water and Wastewater, 21st ed. American Public Health Association, Washington, D.C. [ Links ]
Arbeláez-Cortés, E. (2013). Knowledge of Colombian biodiversity: Published and indexed. Biodivers Conserv; 22 (12): 2875-2906. [ Links ]
Baena, S, Fardeau, M.L., Labat, M., Ollivier, B., García, J.L., Patel B.K.C. (1998). Aminobacterium colombiensegen. nov. sp. nov., an amino acid-degrading anaerobe isolated from anaerobic sludge. Anaerobe. 2: 241-250. [ Links ]
Baena, S., Fardeau, M.L., Woo, T.H.S., Ollivier, B., Labat, M., Patel, B.K.C. (1999a). Phylogenetic relationships of three amino-acid-utilizing anaerobes, Selenomonas acidaminovorans, "Selenomonas acidaminophila" and Eubacterium acidaminophilum, as inferred from partial 16S rDNA nucleotide sequences, and proposal of Thermanaerovibrio acidaminovoransgen. nov., comb. nov. and Anaeromusa acidaminophilagen. nov., comb. nov. Int J Syst Evol Microbiol. 49: 969-974. [ Links ]
Baena, S., Fardeau, M.L., Labat, M., Ollivier, B., Thomas, P., Patel, B.K.C. (1999b) Aminomonas paucivoransgen. nov., sp. nov., a mesophilic, anaerobic, amino-acid utilizing bacterium. Int J Syst Evol Microbiol. 49: 975-982. [ Links ]
Baena, S., Perdomo, N., Carvajal, C., Díaz, C., Patel, B.K.C. (2011). Desulfosoma caldarium gen. nov., sp. nov., a thermophilic sulfate-reducing bacterium from a terrestrial hot spring. Int J Syst Evol Microbiol. 61: 732-736. [ Links ]
Biebl, H., Allgaier, M., Lünsdorf, H., Pukall, R., Tindall, B.J., Wagner-Döbler, I. (2005). Roseovarius mucosussp. nov., a member of the Roseobacter clade with trace amounts of bacteriochlorophyll a. Int J Syst Evol Microbiol. 55: 2377-2383. [ Links ]
Biebl, H., Pukall, D., Lünsdorf, H., Schulz, S., Allgaier, M., Tindall, B., Wagner-Döbler, I. (2007). Description of Labrenzia alexandriigen. nov., sp. nov., a novel alphaproteobacterium containing bacteriochlorophyll a, and a proposal for reclassification of Stappia aggregataas Labrenzia aggregatacomb. nov., of Stappia marina as Labrenzia marina comb. nov. and of Stappia alba as Labrenzia alba comb. nov., and emended descriptions of the genera Pannonibacter, Stappiaand Roseibium, and of the species Roseibium denhamenseand Roseibium hamelinense. Int J Syst Evol Microbiol. 57: 1095-1107. [ Links ]
Bohórquez, L.C., Delgado-Serrano, L., López, G., Osorio- Forero, C., Klepac-Ceraj, V., Kolter, R., Junca, H., Baena, S., Zambrano, M.M. (2012). In-depth characterization via complementing culture-independent approaches of the microbial community in an acidic hot spring of the Colombian Andes. Microb Ecol. 63: 103-115. [ Links ]
Chun, J. & Rainey, F.A. (2014). Integrating genomics into taxonomic and systematics of the Bacteria and Archaea. Int J Syst Evol Microbiol. 64: 316-324. [ Links ]
Clementino, M.M., Vieira, R.P., Cardoso, A.M., Nascimento, A.P.A, Silveira C.B., Riva, T.C., Gonzalez A.S.M., Paranhos, R., Albano, R.M., Ventosa, A., Martins, O.B. (2008). Prokaryotic diversity in one of the largest hypersaline coastal lagoons in the world. Extremophiles. 12: 595-604. [ Links ]
Cortés, L. & Cardona, J.J. (2006). Cartografía geológica detallada para el Salado de Consotá y sus alrededores. En: López, M. Cano y D. Rodríguez (Compiladores). Cambios ambientales en perspectiva histórica. Ecología histórica y cultura ambiental. Universidad Tecnológica de Pereira-Sociedad Colombiana de Arqueología, Pereira. p. 93-109. [ Links ]
Dahle, H., & Birkeland, N.K. (2006). Thermovirga lieniigen. nov., sp. nov., a novel moderately thermophilic, anaerobic, amino-acid-degrading bacterium isolated from a North Sea oil well. Int J Syst Evol Microbiol. 56: 1539-1545. [ Links ]
Delgado-Serrano, L., López, G., Bohórquez, L., Bustos, J.R., Rubiano, C., Osorio-Forero, C., Junca, H., Baena, S., Zambrano, M.M. (2014). Neotropical Andes hot springs harbor diverse and distinct planktonic microbial communities. FEMS Microbiol Ecol. 89: 56-66. [ Links ]
Denner, E.B.M, Smith, G.W., Busse, H.J., Schumann, P., Narzt, T., Polson, S.W., Lubitz, W., Richardson, L.L. (2003). Aurantimonas coralicidagen. nov., sp. nov., the causative agent of white plague type II on Caribbean scleractinian corals. Int J Syst Evol Microbiol. 53: 1115-1122. [ Links ]
Díaz, C., Baena, S., Fardeau, M.L., Patel, B.K.C. (2007). Aminiphilus circumscriptusgen. nov., sp. nov., an anaerobic amino-acid-degrading bacterium from an upflow anaerobic sludge reactor. Int J Syst Evol Microbiol. 57: 1914-1918. [ Links ]
Díaz-Cárdenas, C., López, G., Patel, B.K.C., Baena, S. (2010a). Dethiosulfovibrio salsuginissp. nov., an anaerobic, slightly halophilic bacterium isolated from a saline spring. Int J Syst Evol Microbiol. 60: 850-853. [ Links ]
Díaz-Cárdenas, C., Patel, B.K.C., Baena, S. (2010b) Tistlia consotensisgen. nov., sp. nov., a novel aerobic chemohet- erotrophic free-living nitrogen-fixing α-Proteobacteria, isolated from a Colombian saline spring. Int J Syst Evol Microbiol. 60: 1437-1444. [ Links ]
Díaz-Cárdenas, C. (2011) Estudio de la composición de la comunidad microbiana de manantiales salinos ubicados en los departamentos de Risaralda y Boyacá. Pontificia Universidad Javeriana. Bogotá. p. 255 [ Links ]
Dong, H., Zhang, G., Jiang, H., Yu, B., Chapman, L., Lucas, C., Fields, M. (2006). Microbial diversity in sediments of saline Qinghai Lake, China: Linking geochemical controls to microbial ecology. Microbial Ecol. 51: 65-82. [ Links ]
DuRand, M.D., Olson, R.J., Chisholm, S.W. (2001). Phytoplankton population dynamics at the Bermuda Atlantic Time-series station in the Sargasso Sea. Deep Sea Res Part II. 48: 1983-2003. [ Links ]
Elshahed, M., Najar, F., Roe, B., Oren, A., Dewers, T., Krumholz, L. (2004). Survey of Archaeal diversity reveals an abundance of halophilic Archaea in a low-salt, sulfide and sulfur-rich spring. Appl Environ Microbiol. 70 (4): 2230-2239. [ Links ]
Gell, P. & Gasse, F. (1990). Relationships between salinity and diatom flora from some Australian saline lakes. 11th International Diatom Symposium. California Academy of Sciences, San Francisco, USA. p. 631-641. [ Links ]
Ganesan, A., Chaussonnerie, S., Tarrade, A., Dauga, C., Bouchez, T., Pelletier, E., Lepaslier, D., Sghir, A. (2008). Cloacibacillus evryensisgen. nov., sp. nov., a novel asaccharolytic, mesophilic, amino-acid-degrading bacterium within the phylum 'Synergistetes', isolated from an anaerobic sludge digester. Int J Syst Evol Microbiol. 58: 2003-2012. [ Links ]
Glauert, A.M. (1975). Fixation, dehydration and embedding of biological specimens. En Glauert, A.M., (Ed.): Practical methods in electron microscopy. Vol. 3. Part I. North- Holland, Amsterdam. p. 1-207. [ Links ]
Gremm, T., & Kaplan, L. (1997). Dissolved carbohydrates in streamwater determined by HPLC and pulsed amperometric detection. Limnology and Oceanography. 42 (2): 385-393. [ Links ]
Hungate, R.E. (1969). A roll tube method for cultivation of strict anaerobes. En: Norris, J.R. and Ribbons, D.W. (Eds.). Methods in microbiology, Vol. 3B. Academic Press, London. p. 117-132. [ Links ]
Joshi, A.A., Kanekar, P.P., Kelkar, A.S., Shouche, Y.S., Vani, A.A., Borgave, S.B., Sarnaik, S.S. (2008). Cultivable bacterial diversity of alkaline Lonar Lake, India. Microbial Ecology. 55: 163-172. [ Links ]
Jumas-Bilak, E., Carlier, J.P., Jean-Pierre, H., Citron, D., Bernard, K., Damay, A., Gay, B., Teyssier, C., Campos, J., Marchandin, H. (2007). Jonquetella anthropigen. nov,. sp. nov., the first member of the candidate phylum 'Synergistetes' isolated from man. Int J Syst Evol Microbiol. 57: 2743-2748. [ Links ]
Jumas-Bilak, E., Roudière, L., Marchandin, H. (2009). Description of 'Synergistetes' phyl. nov. and emended description of the phylum 'Deferribacteres' and of the family Syntrophomonadaceae, phylum 'Firmicutes'. Int J Syst Evol Microbiol. 59 (5): 1028-35. [ Links ]
Kirchman, D., Sigda, J., Kapuscinski, R., Mitchell, R. (1982). Statistical analysis of the direct count method for enumerating bacteria. Appl Environm Microbiol. 44: 376-382. [ Links ]
LaBaugh, JW. (1989). Chemical characteristics of water in northern prairie wetlands. En: Valk AV. Ames (Eds). Northern Prairie Wetlands. Iowa University Press. 57-90. [ Links ]
Lai, Q., Qiao, N., Wu, C., Sun, F., Yuan, J., Shao, Z. (2010). Stappia indicasp. nov., isolated from deep seawater of the Indian Ocean. Int J Syst Evol Microbiol. 60: 733-736. [ Links ]
López, G., Chow, J., Bongen, P., Lauinger, B., Pietruszka, J., Streit, W.R., Baena, S. (2014). A novel thermoalkalostable esterase from Acidicaldus sp. strain USBA-GBX-499 with enantioselectivity isolated from an acidic hot springs of Colombian Andes. Appl Microbiol Biotechnol. 98 (20): 8603-16. [ Links ]
Obernosterer, I. & Herndle, G.J. (1995). Phytoplankton extracellular release and bacterial growth: Dependence on the inorganic N:P ratio. Marine Ecology Progress Series. 116: 247-257. [ Links ]
Pedersen, K., Arlinger, J., Hallbeck, L., Pettersson, C. (1996). Diversity and distribution of subterranean bacteria in groundwater at Oklo in Gabon, Africa, as determined by 16S rRNA gene sequencing. Molecular Ecology. 5: 427-436. [ Links ]
Pernthaler, J., Glöckner, F.O., Schönhuber, W., Amann, R. (2001). Fluorescence in situ hybridization with rRNA- targeted oligonucleotide probes. En: J. Paul (Ed.). Methods in microbiology: Marine microbiology. Academic Press Ltd, San Diego, USA. vol. 30. p. 207-226. [ Links ]
Perreault, N.N., Andersen, D.T., Pollard, W.H., Greer, C.W., Whyte, L.G. (2007). Characterization of the prokaryotic diversity in cold saline perennial springs of the Canadian high Arctic. Appl Environ Microbiol. 73 (5): 1532-1543. [ Links ]
Prakash, O., Shouche, Y., Jangid, K., Kostka, J.E. (2013). Microbial cultivation and the role of microbial resource centers in the omics era. Appl Microbiol Biotechnol. 97: 51-62. [ Links ]
Rathsack, K., Reitner, J., Stackebrandt, E., Tindall, B.J. (2011). Reclassification of Aurantimonas altamirensis (Jurado, et al., 2006), Aurantimonas ureilytica (Weon, et al., 2007) and Aurantimonas frigidaquae (Kim, et al., 2008) as members of a new genus, Aureimonas gen. nov., as Aureimonas altamirensisgen. nov., comb. nov., Aureimonas ureilytica comb. nov. and Aureimonas frigidaquaecomb. nov., and emended descriptions of the genera Aurantimonas and Fulvimarina. Int J Syst Evol Microbiol. 61: 2722-2728. [ Links ]
Redburn, A.C. & Patel, B.K.C. (1993). Phylogenetic analysis of Desulfotomaculum thermobenzoicumusing polymerase chain reaction-amplified 16S rRNA-specific DNA. FEMS Microbiol Letters. 113: 81-86. [ Links ]
Rees, G.N, Patel, B.K.C., Grassia, G.S., Sheehy, A.J. (1997). Anaerobaculum thermoterrenumgen. nov., sp. nov., a novel thermophilic bacterium which ferments citrate. Int J Syst Bacteriol. 47: 150-154. [ Links ]
Romero, M., Cabrera, E., Ortiz, N. (2008). Informe sobre el estado de la biodiversidad en Colombia 2006-2007. Instituto de Investigación de Recursos Biológicos Alexander von Humboldt. Bogotá D. C., Colombia. p. 181. http://www.humboldt.org.co/download/INSEB_2006-2007.pdf. [ Links ]
Rooney-Varga, J.N., Giewat, M.W., Savin, M.C., Sood, S., LeGresley, M., Martin, J.L. (2005). Links between phytoplankton and bacterial community dynamics in a coastal marine Environment. Microbial Ecology. 49: 163-175. [ Links ]
Rubiano-Labrador, C., Baena, S., Díaz-Cárdenas C., Patel, B.K.C. (2013). Caloramator quimbayensissp. nov., an anaerobic, moderately thermophilic bacterium isolated from a terrestrial hot spring. Int J Syst Evol Microbiol. 63: 1396-1402. [ Links ]
Rubiano-Labrador, C., Bland, C., Miotello, G., Guerin, P., Pible, O., Baena, S., Armengaud, J. (2014). Proteogenomic insights into salt tolerance by a halotolerant alpha-proteobacterium isolated from an Andean saline spring. Journal of Proteomics. 97: 36-47. [ Links ]
Salm C.R., Saros, J.E., Martin, C.S., Erickson, J.M. (2009). Patterns of seasonal phytoplankton distribution in prairie saline lakes of the northern Great Plains (U.S.A.). Saline Systems. 5 (1): 1-13. [ Links ]
Sieburth, J.M., Smetacek, V., Lenz, J. (1978). Pelagic ecosystem structure-heterotrophic compartments of plankton and their relationship to plankton size fractions. Limnology and Oceanography. 23: 1256-1263. [ Links ]
Sigee, D.C. (2005). Freshwater Microbiology: Biodiversity and dynamic interactions of microorganisms in the freshwater environment. Primera edición. John Wiley & Sons Ltd., Inglaterra. p. 524. [ Links ]
Sogin, M., Morrison, H., Huber, J., Welch, D.M., Huse, S., Neal, P.R., Arrieta, J., Herndl, G. (2006). Microbial diversity in the deep sea and the underexplored ''rare biosphere''. PNAS; 103 (32): 12115-12120. [ Links ]
Sorokin, D.Y., Tourova, T.P., Spiridonova, E.M., Rainey, F.A., Muyzer, G. (2005). Thioclava pacificagen. nov., sp. nov., a novel facultatively autotrophic, marine, sulfur-oxidizing bacterium from a near-shore sulfidic hydrothermal area. Int J Syst Evol Microbiol; 55: 1069-1075. [ Links ]
Swan, B.K., Ehrhardt, C.J., Reifel, K.M., Moreno, L.I., Valentine, D. (2010). Archaeal and Bacterial communities respond differently to environmental gradients in anoxic sediments of a California hypersaline lake, the Salton Sea. App Environ Microbiol. 76 (3): 757-768. [ Links ]
Tanaka, T., Kawasaki, K., Daimon, S., Kitagawa, W., Yamamoto, K., Tamaki, H., Tanaka, M., Nakatsu, C.H., Kamagata, Y. (2014). A hidden pitfall in the preparation of agar media undermines microorganism cultivability. App Environ Microbiol. 80 (24): 7659-7666. [ Links ]
Tistl M. (2004). Sal, cobre y oro en el Consotá. En: López, C, Cano, M. (Eds.) Cambios ambientales en perspectiva histórica. Ecorregión del Eje Cafetero. Universidad Tecnológica de Pereira, Programa Ambiental GTZ. Pereira, Colombia. p. 41-53. [ Links ]
Torsvik, V., Salte, K., Sørheim, R., Goksøyr, J. (1990). Comparison of phenotypic diversity and DNA heterogeneity in a population of soil bacteria. Appl Environ Microbiol. 56 (3): 776-781. [ Links ]
Vartoukian, S., Palmer, R., Wade, W. (2007). The division "Synersgistes". Anaerobe. 13 (4): 99-106. [ Links ]
Ventosa, A., Fernández, A.B., León, M.J., Sánchez-Porro, C., Rodríguez-Varela F. (2014). The Santa Pola saltern as a model for studying the microbiota of hypersaline environments. Extremophiles. 18: 811-824. [ Links ]
Wu, Q.L., Zwart, G., Schauer, M., Kamst-van Agterveld M.P., Hahn, M.W. (2006). Bacterioplankton community composition along a salinity gradient of sixteen high- mountain lakes located on the Tibetan plateau, China. Appl Environ Microbiol. 72: 5478-5485. [ Links ]
Yeates, C., Saunders, A.M., Crocetti, G.R., Blackall, L.L. (2003). Limitations of the widely used GAM42a and BET42a probes targeting bacteria in the Gammaproteobacteria radiation. Microbiology. 149: 1239-1247. [ Links ]













