Introducción
Una de las enfermedades más limitantes del cultivo de la soya es causada por Phytophthora sojae Kaufm. & Gerd, un patógeno del suelo que pudre el tallo y la raíz de la planta. Entre 1996 y 2009 se la consideró como la segunda enfermedad más limitante de la producción de soya a nivel mundial y causante de pérdidas que fluctuaban entre 4 y 100 %, es decir, cifras anuales por encima del billón de dólares (Tyler, et al., 2007; Sugimoto, et al., 2012; Schneider, et al., 2016). En Colombia se reportó por primera vez en el Valle del Cauca en 1985 (Granada & Varon de Agudelo, 1986) y posteriormente en los Llanos Orientales, en 1991 (Montoya, 1991). Desde entonces, la interacción entre las características de los suelos ácidos y con problemas de drenaje, la introducción de la variedad de soya Orinoquia 3, altamente susceptible a la enfermedad, la popularización de la siembra directa y la aparición de condiciones ambientales muy favorables, han sido motivo de alarma sobre el impacto económico que la enfermedad puede tener en los cultivos de soya de la Orinoquia colombiana (Tapiero, 2001).
Dada la importancia de la enfermedad y al hecho de que en la agricultura moderna la práctica más eficiente para la reducción de pérdidas en las cosechas ocasionadas por P. sojae ha sido la utilización de variedades con genes de resistencia, las investigaciones recientes se han enfocado en identificar ocho alelos de genes Rps con la habilidad diferencial para reconocer la virulencia de poco más de las 45 razas del patógeno encontradas hasta ahora en el mundo (Schneider, et al ., 2016). La caracterización de la población de P. sojae según los ecosistemas es indispensable para identificar los genes de resistencia más apropiados para el mejoramiento genético de la soya.
Las poblaciones de este oomicete están constituidas por numerosas razas fisiológicas o patogénicas, las cuales se clasifican con base en su fórmula de virulencia en un conjunto de variedades de soya diferenciales (Keeling, 1979; Layton & Khun, 1990; Schmitthenner, et al ., 1994; Wagner & Wilkinson, 1992). Catorce genes Rps se han asignado a ocho loci en el genoma de la soya, con una serie alélica en dos de ellos: Rps1 (1a, 1b, 1c, 1d y 1k), Rps2, Rps3 (3a, 3b y 3c), Rps4, Rps5, Rps6, Rps7 (Dorrance, et al ., 2004) y Rps8 (Burnham, et al ., 2003; Gordon, et al ., 2006). Recientemente se describieron dos nuevos genes, uno de los cuales se ha designado provisionalmente como RpsYu25 (Sun, et al ., 2011); el otro se halló en el cultivar japonés Waseshiroge. Los dos son alelos de Rps1, o se ubican en un locus estrechamente vinculado con un grupo de genes (Sugimoto, et al ., 2012). Entre estos genes Rps solo cinco se han utilizado ampliamente en cultivares de soya: Rps1a, Rps1c, Rps1k, Rps3a y Rps6 (Abney, et al ., 1997; Dorrance, et al ., 2003). Con el fin de estandarizar los diversos aspectos de tales hallazgos y facilitar la notificación de resultados, la comunidad científica ha optado por la caracterización del patógeno en términos de patotipos, o fórmulas de virulencia, en lugar de razas, y se ha adoptado el uso de códigos octales para comparar los resultados (Dorrance, et al ., 2003; Nelson, et al ., 2008).
En el 2001, Agrosavia logró los primeros avances en la metodología de aislamiento y conservación del patógeno en el Centro de Investigación La Libertad. Posteriormente, se aplicaron las técnicas desarrolladas por el laboratorio de Scot Abney de la Universidad de Purdue (EE.UU.) (Tapiero, 2001). En los estudios de Tapiero (2004) y Tapiero, et al. (2006), se sugiere que los aislamientos colombianos de P. sojae se clasifican en cuatro grupos según su virulencia: la fórmula más común de virulencia es 3a 3c 5 6 7, correspondiente a 34 % de los aislamientos, seguida por 2 3c 5 6 7, con el 25 %, por 2 3c 6 7, con el 22 % y por 2 3a 3c 5 6 7 con el 9 %. Los trabajos de Garnica, et al. (2007) confirmaron que la población colombiana de P. sojae es muy poco diversa, no presenta polimorfismos, y registra valores relativamente bajos de frecuencia de heterocigotos, así como en el índice de Shannon, indicadores que establecen la diversidad genética dentro de una población o entre poblaciones.
Actualmente, se desconoce en el país la resistencia a P. sojae de las variedades y líneas promisorias de soya que hacen parte de la oferta genética de Agrosavia, por lo cual se desarrolló este trabajo de investigación.
Materiales y métodos
Inóculo de P. sojae. Se emplearon dos aislamientos puros, con los códigos 5057301 y 5057302, para determinar la resistencia en los cultivares de soya con edades entre 10 y 15 días en medio agar V8. Los aislamientos se obtuvieron de la colección de oomicetes recolectados en el 2016 en el municipio de Puerto López, vereda Carubare, en la Estación Experimental Taluma en el Meta. Las cepas pertenecen a zonas representativas de la producción de soya de la altillanura del Meta, en la que previamente se habían hecho muestreos y estudios que demuestran la ausencia de diversidad genética en la población colombiana de P. sojae (Garnica, et al., 2007). En la selección de las cepas fue determinante la alta virulencia en las variedades Orinoquia 3 y Williams, consideradas como altamente susceptibles al patógeno.
Las cepas se identificaron por secuenciación de productos de la reacción en cadena de la polimerasa (PCR) con el uso de los cebadores universales ITS4/ITS5 de la región ITS del ADN nuclear ribosómico (White, et al ., 1990). Se hizo el análisis y la edición de las secuencias de la región ITS empleando el programa Geneious 8.1 (Geneious biologics). Las secuencias de consenso generadas a partir del ensamblaje de las reacciones 5´ y 3´ secuenciadas se analizaron con la herramienta BLAST® en la base de datos del National Center for Biotechnology Information. Los aislamientos reportados mostraron una homología de 97 y 99 % con P. sojae.
Fórmulas de virulencia. La inoculación de los aislamientos 5057301 y 5057302 se hizo con la metodología de palillos colonizados desarrollada por Keeling (1982) y modificada por Yorinori (1996). Se inocularon los aislamientos 5057301 y 5057302 en 14 líneas diferenciales de soya: Unión (Rps1a), Haro 13 (Rps1b), L 75 - 3735 (Rps1c), Haro 16 (Rps 1d), Williams 82 (Rps 1k), L 76 - 1988 (Rps 2), PI - 171442 (Rps 3a), L 91 - 8347 (Rps 3b), L 92 - 7857 (Rps3c), L 85 - 3059 (Rps 5), Altona (Rps 6), Harosoy (Rps 7), Williams (Rps), y Sloan (Rps). La caracterización se hizo en términos de fórmula de virulencia en lugar de razas, empleando códigos octales para que los resultados fueran comparables y con el fin de estandarizar y facilitar la notificación de resultados (Dorrance, et al ., 2003; Nelson, et al ., 2008). Bajo las condiciones del Centro de Investigación La Libertad (altitud de 336 msnm, 04° 03’ latitud norte y 73° 29’ longitud oeste, temperatura máxima promedio de 30,9 °C, temperatura mínima promedio de 22,01 °C, y humedad relativa promedio de 81,45 %), y utilizando casa de malla, se sembraron 12 semillas de las 14 variedades diferenciales en macetas que contenían suelo encalado con corrección de pH extraído del mismo centro de investigación. Estas se inocularon con dos aislamientos de P. sojae (cepas 5057301 y 5057302) a los 10 a 12 días de edad, y ya inoculadas se mantuvieron en condiciones de cámara húmeda durante 48 horas y se evaluaron diariamente hasta la aparición de síntomas. La clasificación de las reacciones a cada tratamiento se basó en la metodología descrita por Yorinori (1996) y Embrapa (2016). Las plantas se inocularon tres a cuatro veces para confirmar el resultado.
Cultivares de soya. Se evaluaron 18 líneas de soya: tres de la nueva oferta genética del programa de mejoramiento de Agrosavia desarrolladas entre el 2012 y el 2017, las líneas 105, 224 y 233. Seis variedades desarrolladas por Embrapa introducidas de Brasil para adaptarlas a las condiciones de la altillanura y el piedemonte del Meta: los cultivares BRS_Gralha, BRS_252, BRS_Serena, BRS_Corisco, BRS 314 y BRS_Barreiras. Nueve variedades liberadas de Agrosavia: Obando, Orinoquia 3, Superior 6, Sabana 7, Taluma 5, Libertad 4, Achagua 8, Guayuriba 9 e Iraca 10, las cuales conforman actualmente la oferta genética de soya de Agrosavia, y la Panorama 29 como testigo comercial. Se sembraron 15 semillas de todos los materiales de soya en macetas que contenían aproximadamente 2 kg de suelo, suficientes para obtener 10 a 15 plantas, en promedio, para la inoculación. De 11 a 15 días después de la siembra, las plántulas se inocularon mediante el método de palillos colonizados a 1 cm por debajo del cotiledón, aproximadamente. Después de la inoculación las plantas se mantuvieron en cámara húmeda durante 48 horas y se evaluaron diariamente hasta la aparición de síntomas. Las plantas se inocularon entre 3 y 4 veces para confirmar el resultado.
Calificación de los materiales. Las lecturas de las reacciones se hicieron diariamente hasta la aparición de síntomas siguiendo los criterios de clasificación de Yorinori (1996) y Embrapa (2016): resistente (R) si había entre 0 y 25 % de plantas muertas; moderadamente resistente (MR), entre 26 y 50 % de plantas muertas, y susceptible (S), más de 50 % de plantas muertas.
Análisis estadístico. Se empleó un diseño completamente al azar con dos aislamientos de P. sojae, 18 materiales de soya evaluados (cada uno con 8 a 12 unidades experimentales) y tres repeticiones. Los datos se analizaron con el programa estadístico JMP® SAS, versión 13.1.0.
Resultados y discusión
Determinación de las fórmulas de virulencia. Además de los testigos susceptibles Williams y Sloan (genes Rps), todos los aislamientos infectaron por lo menos a ocho variedades diferenciales de soya. Ningún aislamiento logró infectar la totalidad de variedades ni la portadora de los genes 1a, 1b, 1c, 1d, 1k y 3b. Las variedades con los alelos Rps 2, 3a, 3c, 5, 6 y 7, fueron sensibles a los dos aislamientos de P. sojae, los cuales correspondían a la fórmula de virulencia 2 3a 3c 5 6 7 (Tabla 1).
Tabla 1 Fórmula de virulencia asignada a los aislamientos inoculados de Phytophthora sojae en variedades diferenciales de soya con presencia de genes Rps
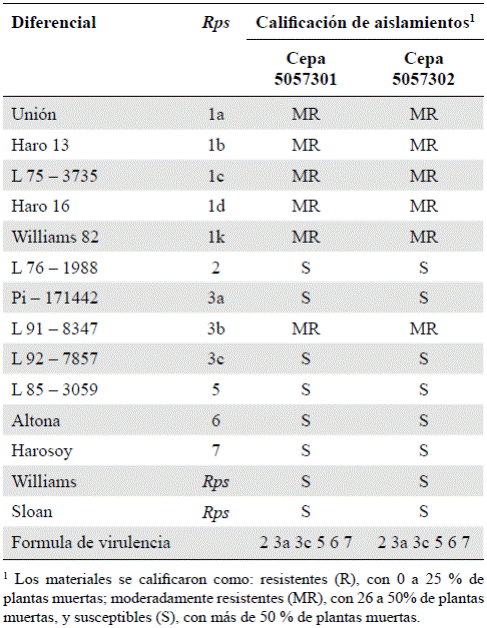
Según Tapiero (2004), la patogenicidad de una colección de 40 aislamientos colombianos de P. sojae obtenidos de las zonas más representativas del cultivo (Valle del Cauca y Llanos Orientales), caracterizada mediante inserción en el hipocótilo en 14 variedades diferenciales, indicó que los aislamientos del Valle presentaban dos fórmulas de virulencia: una patogénica frente a Rps 2 3c 5 6 7 y otra que, además, lo era a Rps 1b 3a 3b. En los Llanos, los aislamientos provenientes de Villavicencio se diferencian de los de Granada porque, además de ser patogénicos frente a Rps 2 3c 6 7, también lo son frente a Rps 3a. Los dos aislamientos evaluados fueron patogénicos en variedades diferenciales que contenían el gen 3a y constituían buenos representantes de la población de P. sojae en los Llanos Orientales. El conocimiento sobre la composición de la virulencia de P. sojae es fundamental para determinar el programa de manejo apropiado de la resistencia genética a este oomycete. Por lo menos seis variedades diferenciales, además de los testigos, fueron sensibles al ataque del patógeno, lo que indica que la capacidad de virulencia de P. sojae en la altillanura del Meta superpone individualmente la resistencia combinada de por lo menos seis genes de resistencia específicos para este patógeno, los cuales deben tenerse en cuenta en el programa de mejoramiento genético de soya en Agrosavia.
Generalmente, cuando una enfermedad introducida en una región o país, se encuentra una raza predominante. Por ejemplo, en Estado Unidos y en Canadá prevaleció la fórmula Rps7 (raza 1) hasta la década de 1990 (Anderson & Buzzell, 1992; Laviolette & Athow, 1981; Schmitthenner, et al ., 1994). Tanto en Argentina como en Brasil, los primeros aislamientos de P. sojae encontrados en esa misma década pertenecieron exclusivamente a la fórmula Rps 7 (Barreto, et al ., 1995;Costamilan & Bonato, 1996). Sin embargo, hay evidencia de que aparecieron nuevos patotipos a medida que la enfermedad adquirió importancia en el cultivo y se fueron liberando variedades resistentes con genes Rps. Es así como en la zafra 2013-2014 en el sudeste bonaerense de Argentina, se determinaron 12 patotipos distintos en 18 aislamientos, ninguno de los cuales correspondió a la raza 1 (Grijalba, et al ., 2015).
En Heilongjiang, China, con la caracterización fisio lógica de P. sojae, se identificaron ocho fórmulas de virulencia, incluidas las 1, 3, 4, 5, 9, 13, 44 y 54. De la fórmula Rps 7 (antes raza 1) se identificaron 56 aislamientos que representaban el 60 % de la población del patógeno. La fórmula Rps 1a, Rps 7 (raza 3) abarcó 14 aislamientos correspondientes al 7 % de la población del patógeno. Las fórmulas 4, 5, 44 y 54 se identificaron por primera vez en Heilongjiang, cada una representada por dos o tres aislamientos solamente (Zhang, et al ., 2010). En cuanto a la reacción diferencial a las ocho fórmulas encontradas de los 62 materiales sembrados comúnmente en la provincia, 44 cultivares fueron resistentes (<30 % de mortalidad) a por lo menos una raza (Zhang, et al ., 2010), lo que demostró la resistencia parcial de los cultivares. Por otra parte, en Uruguay el 57 % de los aislamientos de P. sojae pertenecían a un único patotipo (fórmula 1b, 3a, 3b, 5) (Sans, et al ., 2017). El conocimiento de los patotipos de P. sojae predominantes en las regiones productoras de soya resulta fundamental en los programas de mejoramiento para la selección según la resistencia, así como para determinar su compatibilidad con las variedades comerciales.
Evaluación de cultivares de soya por su reacción a Phytophthora sojae . En la Tabla 2 se presentan los resultados de significación de la prueba F para las variables de plan- tas muertas (%) y de líneas de soya evaluadas. Se observaron diferencias significativas (p<0,01) entre los diferentes mate- riales de soya, la mayoría de los cuales fueron sensibles a P. sojae, con valores por encima del 50 % de plantas muertas después de la inoculación. Solamente la variedad Corpoica La Libertad 4 fue moderadamente resistente a los dos aisla- mientos de P. sojae evaluados con las fórmulas de virulencia 2 3a 3c 5 6 7 (Figura 1, Tabla 3).
Tabla 2 Cuadrados medios del análisis de varianza y comparación de las medias del porcentaje de plantas muertas por cultivar de soya evaluado
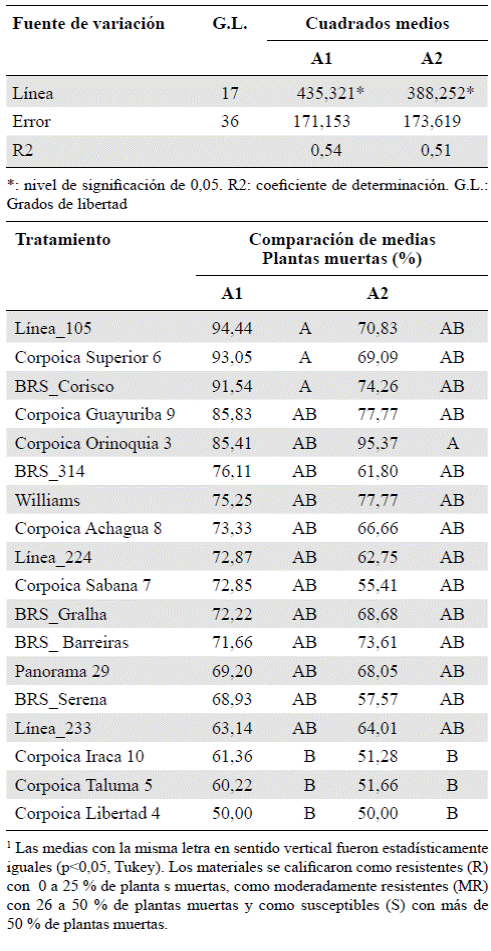
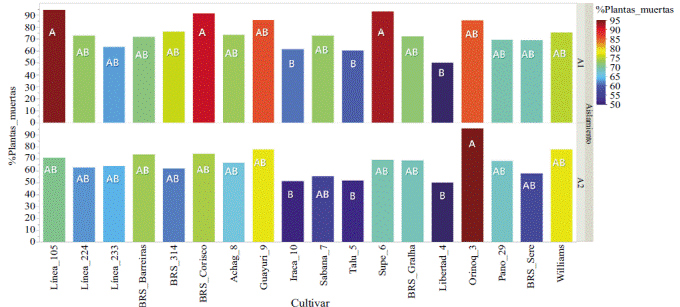
Figura 1 Porcentaje de plantas muertas por aislamiento de P. sojae inoculado y cultivares de soya evaluados. Las letras diferentes denotan diferencias estadísticamente significativas (p<0,05 en el test de Tukey).
Tabla 3 Reacción de patogenicidad de los aislamientos de Phytophthora sojae del departamento del Meta en los cultivares de soya evaluados
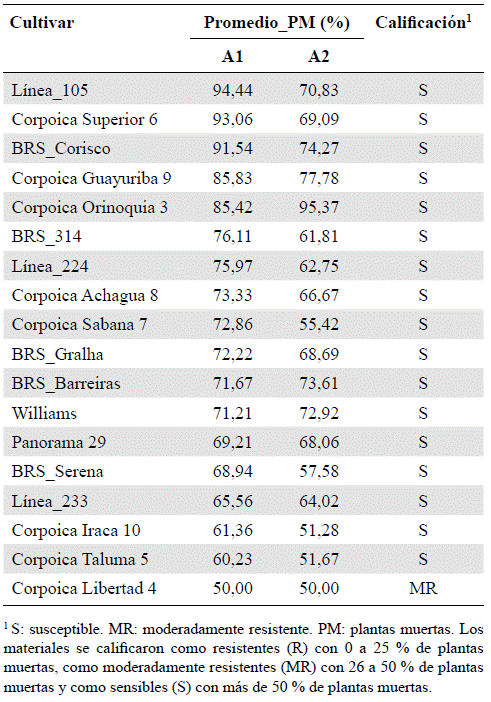
Estos resultados coinciden con lo reportado por Tapiero (2004), quien encontró que la variedad Corpoica La Libertad 4 era resistente al patógeno cuando se la inoculaba con aislamientos de las fórmulas de virulencia 2 3c 6 7, 3a 3c 5 6 7 y 2 3c 5 6 7. El autor menciona, asimismo, que la variedad Taluma 5 fue resistente a los aislamientos con las fórmulas 2 3c 6 7 y 3a 3c 5 6 7.
Según Tapiero (2004), la prueba de patogenia con las variedades de soya colombianas evidenció las posibilidades de selección de fuentes de resistencia en la variedad Corpoica La Libertad 4. Las variedades Corpoica Iraca 10 (% de plantas muertas: 61,36) y Corpoica Taluma 5 (% de plantas muertas: 60,22), aunque se comportaron como susceptibles , presentaron valores relativamente bajos de plantas muertas (Tabla 2), razón por la cual deberían ser inoculadas nuevamente con el fin de confirmar su verdadera reacción a P. sojae. Cabe resaltar la reacción de los materiales Corpoica Orinoquia 3, la Línea_105, Corpoica Superior 6 y BRS_Corisco, los cuales resultaron altamente sensibles al patógeno con valores promedio de plantas muertas más altos: 95,37 94,44, 93,05 y 91,54 %, respectivamente, cuando fueron inoculados con los aislamientos de P. sojae con la fórmula de virulencia 2 3a 3c 5 6 7. En el caso de la Línea_105 y de BRS_Corisco, no se recomienda su liberación como posible oferta genética de Agrosavia debido a su gran sensibilidad al patógeno. Se recomienda no sembrar las variedades Orinoquia 3 y Corpoica Superior 6 en suelos compactados y con problemas de drenaje, ya que dichas condiciones aumentan la predisposición a la presencia de la enfermedad.
Conclusiones
Las pruebas de patogenicidad en los cultivares de soya que conforman la oferta genética de Agrosavia evidenciaron las posibilidades de selección de fuentes de resistencia en el germoplasma de soya, en especial con la variedad Corpoica La Libertad 4, la cual demostró resistencia moderada específica frente a la virulencia de los aislamientos utilizados.
La Línea_105, desarrollada en el esquema de mejoramiento del 2016, y las variedades Corpoica Orinoquia 3, Superior 6 y BRS_Corisco fueron muy susceptibles a P. sojae. En el caso de la Línea_105 y de BRS_Corisco, no se recomienda su liberación como nueva oferta genética debido a su gran sensibilidad al patógeno.














