Introducción
Los tripanosomátidos son parásitos protozoos flagelados de importancia médica que se transmiten al hombre y a los ani males a través de insectos vectores y causan diversas enfermedades de reconocido impacto en salud pública como la enfermedad de Chagas, la enfermedad del sueño y la leishmaniasis. Estos microorganismos eucariotas se caracterizan por haber divergido tempranamente de la línea evolutiva, de tal manera que poseen mecanismos de regulación génica tremendamente eficaces que pueden calificarse como sui generis, y que les han permitido enfrentar los sistemas de defensa de sus huéspedes y completar su complejo ciclo biológico tras millones de años. Así, para lograr adaptarse a ambientes tan disímiles como los de sus huéspedes invertebrados y mamíferos, adoptan a lo largo de su ciclo de vida diversas formas que son el resultado de una orquestada modulación de la expresión de sus genes, lo cual redunda no solo en la adquisición de una morfología propia, sino de un potencial bioquímico y una capacidad de adaptación diferentes (Puerta, 2007). A diferencia de lo que ocurre en la mayoría de eucariotas, la expresión génica en los tripanosomátidos se regula después de la transcripción gracias, precisamente, a la organización génica peculiar que presentan (Clayton, 2016). Sus cromosomas contienen grandes agrupaciones génicas, algunas conformadas por más de un centenar de genes, a menudo sin ninguna relación funcional, pero sí con la misma orientación de la transcripción. Estas agrupaciones se denominan unidades de grupos de genes policistrónicos (polycistronic gene clusters, PGC) y están diseñadas para ser transcritas como grandes policistrones (Berriman, et al., 2005; El-Sayed, et al., 2005; Ivens, et al., 2005). En cada cromosoma existen varias unidades PGC; las unidades de transcripción vecinas tienen una orientación convergente o divergente y están separadas por las denominadas regiones de cambio de hebra. Una vez se genera el transcrito policistrónico, debe ser procesado y convertido a ARNm monocistrónicos antes de ser traducido, lo cual es mediado por dos procesos íntimamente acoplados: el procesamiento en trans o trans-splicing y la poliadenilación (Requena, 2011). En el primero, se adiciona en el extremo 5’ una secuencia conocida como spliced leader (SL) de 39 nucleótidos, la cual ya porta la caperuza o estructura cap (cap-4 en tripanosomátidos) propia de los ARN mensajeros, cuya función, al igual que sucede en el resto de eucariotas, es proteger los ARNm y facilitar su interacción con la maquinaria de traducción (Agabian, 1990; Bangs, et al., 1992). Por otra parte, en el proceso de poliadenilación se adiciona una cola de adeninas en el extremo 3’ de los mensajeros tras el corte endonucleotídico, la cual, en general, le otorga estabilidad a la molécula (LeBowitz, et al., 1993).
La peculiar organización génica y la transcripción policistrónica de estos organismos explican por qué la tasa de transcripción es similar en la mayoría de sus genes. En consecuencia, dada la pérdida de este punto de control, los tripanosomátidos han apostado por regular la expresión de sus genes a través de mecanismos posteriores a la transcripción, entre ellos el procesamiento del pre-ARNm y el transporte, la localización, la estabilidad y la traducción de los mensajeros monocistrónicos maduros (Araujo & Texeira, 2011). Todos estos procesos están mediados por la acción de proteínas de unión a ARN (RNA binding protein, RBP), que reconocen su mensajero blanco gracias a la presencia de elementos cis localizados en las sus regiones 5´ y 3´no traducidas (untranslated región, UTR) y se asocian con dicho ARN formando un complejo ribonucleoproteico cuya composición va a dictar el destino de los mensajeros (Cassola & Frash, 2009; Romaniuk, et al., 2016). De esta manera, las RBP desempeñan un papel central en la regulación de la expresión génica, y ya que no puede haber una RBP específica para cada transcrito, las células han establecido redes reguladoras en las que una misma RBP puede actuar sobre centenares de ARN mensajeros (regulones u operones de ARN), en tanto que el destino de cada ARN es dictado por la combinación de RBP con las que interactúa (Lapointe, et al., 2015;Muller-McNicoll & Neugebauer, 2013).
Si bien ya se conocen cerca de un centenar de proteínas con capacidad de unión al ARN en tripanosomátidos (Kramer & Carrington, 2011; Romaniuk, et al., 2016), sin duda son muchas las que están aún por descubrir. Dada su divergencia evolutiva, la mayoría de las proteínas de estos parásitos carecen de ortólogos reconocibles en otros eucariotas. Además, muchas RBP carecen de dominios de unión a ARN típicos. En este artículo, se presentan las estrategias seguidas para la identificación de proteínas reguladoras de la expresión génica en tripanosomátidos durante la última década en el Laboratorio de Parasitología Molecular del Departamento de Microbiología de la Facultad de Ciencias de la Pontificia Universidad Javeriana, trabajos que se han desarrollado en colaboración con el Laboratorio de Regulación de la Expresión Génica en Leishmania del Centro de Biología Molecular “Severo Ochoa” de la Universidad Autónoma de Madrid. Entre los estudios llevados a cabo, se describen con mayor detalle y consignando resultados ilustrativos aquellos dedicados a la identificación y caracterización de proteínas RBP directamente implicadas en la regulación posterior a la transcripción de los genes HSP70 de Leishmania braziliensis.
Concepto y punto de partida
Aunque la investigación propuesta es de carácter básico y su objetivo es aumentar nuestro conocimiento sobre los mecanismos de control de la expresión génica en tripanosomátidos desentrañando las redes de interacción entre las RBP y sus mensajeros blancos, no podemos olvidar que estos organismos son patógenos que actúan en humanos. Por ello, como punto de partida se propuso el estudio de la expresión de proteínas críticas para el parásito o de factores de virulencia, cuyos mensajeros (más concretamente, las regiones UTR) se usaron como cebos para la captura de las RBP. En este sentido, el primer paso del estudio consistió en la elección del gen y del parásito a estudiar. Si bien en nuestro grupo se ha trabajado con otros tripanosomátidos, se escogió Leishmania como el tripanosomátido objeto de estudio por las siguientes razones: Leishmania es el agente causal de la leishmaniasis, parasitosis asociada a la pobreza con un amplio espectro de manifestaciones clínicas que van desde lesiones cutáneas autocurativas (LC), pasando por lesiones mucocutáneas (LMC) causantes de desfiguración hasta afectaciones viscerales (LV), siendo estas últimas fatales si no se las trata adecuadamente (Bañuls, et al., 2007). Se estima que aproximadamente 12 millones de personas sufren actualmente leishmaniasis en un centenar de países de todo el mundo, y al menos 500.000 nuevos casos de LV y 1,5 millones de casos de LC grave se reportan anualmente (Alvar, et al., 2012). En Latinoamérica, la leishmaniasis es endémica en 22 países, y Leishmania (Viannia) braziliensis es el principal agente causante de la forma mucocutánea. En Colombia, la leishmaniasis es endémica en casi todo el territorio nacional, y L. braziliensis es una de las seis principales especies causantes de leishmaniasis cutánea y mucocutánea en el país (Instituto Nacional de Salud - INS, 2011).
Una vez elegida L. braziliensis, se pensó en estudiar un factor clave como es la proteína de choque térmico de 70 kDa (HSP70) con base a las siguientes características: (i) durante la transición de promastigote (insecto-vector) a amastigote (huésped mamífero), se requiere de la actividad chaperona de las proteínas de choque térmico, fundamentalmente de la HSP70 (Requena, 2012); (ii) a partir de un determinado gen del locus HSP70 (el HSP70-II) de Leishmania infantum la expresión de dicha proteína es necesaria para la supervivencia de los promastigotes en fase estacionaria y de los amastigotes tanto in vitro como in vivo (Folgueira, et al., 2008); (iii) la depleción del gen HSP70-II disminuye la capacidad infectiva del parásito y, por ende, la enfermedad que ocasiona según diferentes modelos animales (Carrion, et al., 2011; Folgueira, et al., 2008), y (iv) induce una fuerte respuesta inmunitaria humoral y celular (Requena, et al., 2012).
El factor de virulencia
Dado que las RBP gobiernan la regulación posterior a la transcripción a través de su interacción con elementos en cis, localizados principalmente en las regiones 5´ y 3´ UTR de los mensajeros blanco (Kramer & Carrington, 2011), en la primera etapa del desarrollo de la propuesta experimental, es necesario conocer la arquitectura de los genes y de los mensajeros del factor de virulencia seleccionado.
En este caso, se encontró que L. braziliensis, al igual que otras especies del género Leishmania, posee dos tipos de genes HSP70, el HSP70-I y el HSP70-II, los cuales se organizan en el cromosoma 28 en una agrupación conformada por seis copias de los genes HSP70-I y una de los HSP70-II (Ramírez, et al., 2011). Cabe mencionar que estos genes se diferencian solamente en la secuencia y la longitud de su región 3´ UTR (Ramírez, et al., 2011), pero dichas diferencias han resultado fundamentales en los mecanismos moleculares de adaptación del parásito, pues le permiten tolerar el estrés térmico al que es sometido durante su ciclo de vida: 26 °C en el insecto vector y 35 a 37 °C en el huésped mamífero (Folgueira, et al., 2005). Para definir y caracterizar la región 5´ UTR, común a ambos mensajeros, así como la 3´ UTR específica para cada uno de ellos, se hicieron ensayos de RT-PCR a partir del ARN de formas promastigotas del parásito; a continuación, los ADN complementarios (ADNc) obtenidos se clonaron y secuenciaron (Ramírez, 2012). Para el caso de la región 5´ UTR (número de acceso al GenBank JF449363, posiciones 1-198), se encontró que, además de la secuencia SL de 39 nucleótidos (nt), esta comprende los 198 nt corriente arriba del codón de inicio de la traducción. Se observó que las regiones 3´ UTR abarcan 936 (UTR-I, número de acceso al GenBank JF449364, posiciones 143-1078) o 932 (UTR-II, número de acceso al GenBank JF449365, posiciones 143-1074) nucleótidos corriente abajo del codón de parada de los genes respectivos (Figura 1). Teniendo en cuenta el papel de los elementos cis presentes en las UTR como mediadores de la interacción ARN-proteína, se procedió a la identificación de secuencias que potencialmente pudieran funcionar como tales elementos. Para ello, se implementó una aproximación que combinó hallazgos previos consignados en la literatura con el análisis computacional de secuencias conservadas y la predicción de la estructura secundaria del ARN mediante la cual se identificaron dos secuencias ricas en pirimidinas sobre la 3´ UTR-I (Ramírez, 2012). Una de ellas, ubicada en la posición 808-842 (5´ GCGCTCCCCCTCCTGCCGCCTGTCTTCCTCCCCTT 3´), contiene tramos de cadena sencilla y tiene un 53,8 % de identidad con un elemento rico en pirimidinas de la región 3´ UTR del gen HSP83 de L. amazonensis relacionado con la traducción preferencial de estos genes en condiciones de choque térmico (David, et al., 2010). Otro elemento cis putativo (5´ ACCGACTCTCTTTCCTCCCCCTACCCGTA 3´) se encontró en una región altamente conservada entre los nt 489 y 605 de la 3´ UTR-I, posición que concuerda con la localización de una secuencia reguladora identificada en la región 3´ UTR de los genes HSP70-I de L. infantum, asociada con la acumulación de estos mensajeros en respuesta al choque térmico (Quijada, et al., 2000). Al hacer la predicción de la estructura secundaria de estas regiones en L. braziliensis, se observó que el tramo rico en pirimidinas presentó una identidad del 89,7 % con la misma región en L. infantum y se ubicó en una zona estructurada en forma de cadena sencilla (Ramírez, 2012). La conformación del ARN en cadena sencilla es una condición importante para que las proteínas de unión a ARN puedan interactuar con este, dado que cuando el ARN adopta una estructura de doble hélice, el surco mayor es demasiado estrecho e impide el establecimiento de puentes de hidrógeno entre las bases nitrogenadas del ARN y los residuos de aminoácidos de la proteína implicada (Baumeister, 1997). Por su parte, en la 3´ UTR-II del mensajero de la HSP70 se encontraron dos motivos, ambos en regiones de cadena sencilla (Nocua, et al., 2017; Ramírez, 2012). Uno de ellos, localizado en la posición 252-302, tiene forma de horquilla y se relaciona con los elementos ARE, conocidos por desestabilizar a los ARNm que los contienen (Di Noia, et al., 2000): 5´ ATGTCTTTTATTTTTTTGTGTG TGTTTTATATTTTTCTCCTTTCGTACTAA 3´. El otro motivo es una región rica en citosinas en las posiciones 338-361 (5´ CCCCTCCTCCCCTCTCCCCCTCCC 3´). Según la literatura, las proteínas que se unen a secuencias ricas en citosinas participan en el control posterior a la transcripción de la expresión génica y tienen un amplio rango de funciones que van desde la estabilización del ARNm y activación de la traducción hasta su silenciamiento (Makeyev & Liebhaber, 2002).
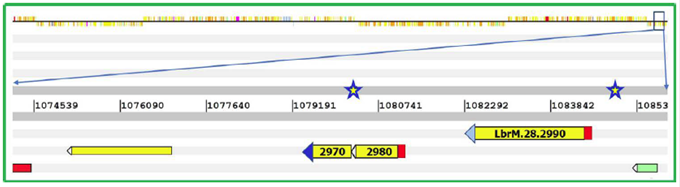
Figura 1 Ubicación de las regiones UTR determinadas para los genes HSP70 sobre el cromosoma 28 en el genoma publicado de L. braziliensis. El gen LbrM.28.2990 (2990), codificante para la proteína HSP70, es portador de la región 3´ UTR-I (triángulo azul claro) mientras que el gen LbrM.28.2970 (2970), también codificante para la proteína HSP70, tiene la región 3´ UTR-II (triángulo azul oscuro). Los genes LbrM.28.2990 y LbrM.28.2980 (2980) comparten la región 5´ UTR. Cada estrella simboliza un hueco en la secuencia genómica actualmente disponible en la base de datos (GeneDB.org).
Detección de las proteínas
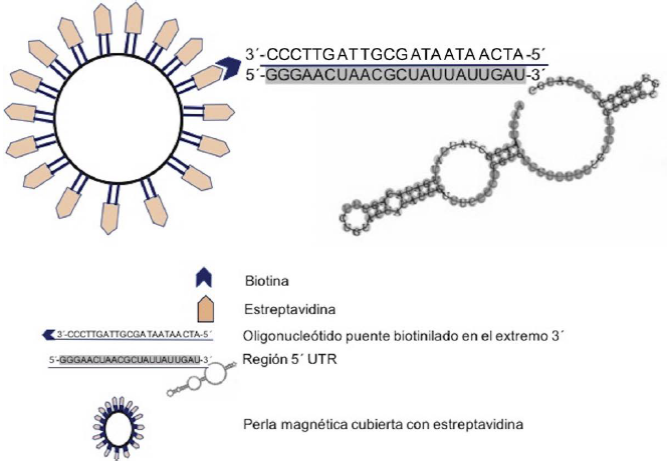
Para identificar las RBP asociadas con la regulación de la expresión del gen seleccionado, se utiliza un ensayo de pull-down o pesca en el cual las regiones UTR de los mensajeros de interés, en este caso los que codifican para la proteína HSP70, se usan como cebos para capturar las proteínas RBP (Ramírez, et al., 2013). Para ello, las regiones UTR previamente identificadas deben clonarse en un vector apropiado para dirigir la transcripción in vitro y así obtener una cantidad suficiente de moléculas de ARN que servirán como cebos en el ensayo. Como molde para la transcripción in vitro también se pueden utilizar las regiones UTR amplificadas con un oligonucleótido quimérico hacia adelante que contenga en su extremo 5´ la secuencia del promotor, el cual se usará seguido de una secuencia complementaria de la UTR. Una vez transcritas las UTR, deben ser unidas a un soporte sólido antes de proceder con el siguiente paso experimental. Para ello, se utilizan perlas magnéticas recubiertas de estreptavidina, a las cuales se une el cebo de ARN mediante un oligonucleótido puente marcado con biotina cuya secuencia es complementaria de uno de los extremos del cebo. En este caso, en el experimento de captura con la región 5´ UTR se utilizó la secuencia complementaria de la región SL marcada con biotina en el extremo 3´ como oligonucleótido puente (Figura 2) y, para las regiones 3´ UTR, un oligo d(T) marcado en su extremo 5´. Paralelamente, se prepararon extractos de proteínas totales del parásito a partir de los cuales se pescaron las RBP de interés. En este estudio se usaron extractos proteicos provenientes de lisados de las formas promastigotas de la cepa MHOM/BR/75/M2904 de L. braziliensis cuyo contenido proteico se verificó cualitativamente mediante electroforesis en gel de poliacrilamida con dodecil sulfato de sodio (sodium dodecyl sulfate polyacrylamide gel electrophoresis, SDS-PAGE), y cuantitativamente con el método de Bradford, antes de usarlas. A continuación, cada cebo de ARN del complejo de unión se incubó con la mezcla de proteínas del parásito; en el medio de incubación es preciso añadir inhibidores tanto de ARNasas como de proteasas para evitar la degradación de los componentes de la mezcla, y de ARN competidores (como ARNt de levadura, ARN de Escherichia coli o de células Vero), para disminuir uniones inespecíficas de proteínas a las moléculas cebo. Posteriormente, los complejos de proteína y ARN cebo se recuperaron mediante atracción magnética ejercida sobre las perlas, y se lavaron varias veces para retirar las proteínas de unión inespecífica. Por último, la resolución de los complejos obtenidos y la identificación de las proteínas que los conforman se hicieron mediante técnicas de separación como la electroforesis en geles bidimensionales o la cromatografía líquida de alta eficiciencia (high performance liquid chromatography, HPLC), seguidas de análisis de espectometría de masas (EM). Los datos sobre las huellas peptídicas obtenidas y sus masas se buscan en las bases de datos de proteínas de la secuencia genómica de L. braziliensis para identificar las proteínas presentes en los complejos obtenidos. En el caso de la electroforesis en geles bidimensionales se requiere de un paso adicional: la disgregación de las proteínas del complejo, las cuales son luego separadas mediante electroforesis en gel, primero en función de su punto isoeléctrico (pI) y, después, por su tamaño molecular. Una vez obtenidas las bandas correspondientes a las proteínas, estas se analizan mediante espectometría de masas, lo que en este caso se hizo con las técnicas de MALDI/TOF o MALDI/TOF/TOF. En este procedimiento es importante incluir un cebo de control, es decir, una secuencia irrelevante para los propósitos del ensayo que, por comparación con los resultados de los cebos problema, garantice su especificidad. También es primordial hacer réplicas biológicas de cada cebo de ARN, con el fin de obtener resultados más sólidos y confiables y configurar todos los parámetros para la identificación según los análisis de separación y espectrometría de masas llevados a cabo. En el ejemplo que nos ocupa, se utilizaron como cebo de control las regiones UTR de los mensajeros de la proteína estructural del citoesqueleto de la tubulina alfa y se hicieron dos réplicas biólogicas. Los parámetros utilizados para el análisis de las imágenes de los geles y de los espectros de los péptidos se detallan en Ramírez, et al. (2013). Además, dado que se ha demostrado que en L. infantum los mensajeros del HSP70-I se traducen tanto a 26 como a 35 °C, en tanto que los mensajeros del HSP70-II se traducen preferencialmente bajo choque térmico (Folgueira, et al., 2005), los ensayos de pesca o pull-down se realizaron para cada cebo usando lisados proteicos de parásitos que habían crecido tanto a 26°C como a 35 °C. De esta forma, se obtuvieron 52 proteínas distintas usando los cebos de las regiones UTR de los mensajeros de la proteína HSP70, todas ellas ausentes en los ensayos de pesca con el cebo de control. De estas, 26 se asociaron exclusivamente con la región 5´ UTR, cinco con la región 3´ UTR-I y seis con la 3´ UTR-II. Además, tan solo cuatro proteínas interactuaron con todos los tres cebos y 11 lo hicieron con dos de ellos: cuatro proteínas con la 5´ UTR y la 3´ UTR-I, cinco con las 3´ UTR-I y II y dos con la 5´ UTR y la 3´ UTR-I. Estos resultados muestran la especificidad de la unión ARN-proteína, lo cual se explica, muy seguramente, tanto por la secuencia como por la estructura secundaria particular de cada una de las UTR usadas como cebo (De Gaudenzi, et al., 2005). Ahora bien, teniendo en cuenta la longitud de los cebos, fue un tanto sorpresivo que la región con mayor número de interacciones fuera la 5´ UTR, ya que su longitud es casi cinco veces menor que las de la 3´ UTR. Sin embargo, estos hallazgos podrían indicar que la regulación sobre los transcritos de HSP70 opera fundamentalmente a nivel de la iniciación de la traducción. De hecho, entre las proteínas asociadas con la región 5´ UTR se encontraron proteínas que participan tanto en el inicio como en la elongación de la traducción. Por otra parte, en concordancia con la temperatura de crecimiento del parásito, la gran mayoría de proteínas (41) se identificaron a los 26 °C (Ramírez, et al., 2013), lo cual podría obedecer, en parte, a un sesgo de la metodología in vitro, la cual solo opera para la interacción de las proteínas libres presentes en el lisado y descarta aquellas que participan en los complejos que se forman durante el choque térmico entre las proteínas reguladoras y los mensajeros endógenos de HSP70.
Análisis de las proteínas
Una vez obtenido el listado de proteínas que interactúan con los distintos cebos, es preciso ponderar el valor de su presencia en el proteoma obtenido. Así, las proteínas identificadas inicialmente se analizan según su categoría funcional utilizando la Kyoto Encyclopedia of Genes and Genomes (KEGG: http://www.genome.jp/tools/blast/) (Kanehisa, et al., 2006) y la Protein ANalysis THrough Evolutionary Relationships, version 7 (Panther: http://www.pantherdb.org/pathway/) (Mi, et al., 2010), así como datos previamente reportados en la literatura y en las bases de datos de tripanosomátidos. En nuestro ejemplo, las categorías funcionales más representadas en las 52 proteínas identificadas fueron, como se esperaba, las de metabolismo del ARN (27 %) y las de traducción (7 %), para un total de 34 %, seguidas por las de proteínas citoplasmáticas relacionadas con el metabolismo energético (21 %) y el metabolismo de proteínas (19 %) que, en principio, no guardan relación con los procesos de regulación génica. La presencia de estas proteínas podría explicarse como un ruido de fondo de la metodología, que arrastra proteínas irrelevantes para el estudio gracias a que se encuentran en mayor cantidad en la célula, o a que la condición in vitro posibilita interacciones que in vivo se encuentran limitadas por factores como la compartimentación subcelular. Sin embargo, es importante no perder de vista que en numerosos estudios en organismos distintos y alejados a lo largo de la escala evolutiva, se ha descrito la existencia de proteínas pluriempleadas (moonlight) que, siendo enzimas metabólicas, son capaces de realizar otras funciones bioquímicas, como unirse al ARN y cumplir un papel en la regulación de la expresión génica, entre otros (Jeffery, 2018). Ahora bien, para efectos de la metodología aquí propuesta, se recomienda que mientras se amplían las bases de proteínas multitarea o moonlight (Multitasking Proteins DataBase: http://wallace. uab.es/multitask/), debe enfocarse la atención en aquellas relacionadas con el metabolismo del ARN y la traducción. En la Tabla 1 se presentan las 19 proteínas identificadas en estas categorías en nuestro estudio. De estas, es importante descartar las que tienen localización mitocondrial (3), ya que muy seguramente fueron detectadas gracias a la naturaleza in vitro del estudio y no a las condiciones fisiológicas in vivo. En cuanto a las restantes, en este estudio se detectaron tres grupos de proteínas: las previamente identificadas en el parásito con un rol ya establecido (2); las identificadas en el parásito con funciones putativas asignadas (7), y las hipotéticas, cuya existencia y función en el parásito se desconocen (7). En el presente caso, en el primer grupo se identificaron dos componentes del proceso de elongación de la traducción: el factor de elongación 1 α (LbEF1α, LbrM.35.0270), asociado a las regiones 5´ UTR y 3´ UTR-I a 26°C y, el factor de elongación 2 (LbEF2, LbrM.35.0270), aislado por su unión tanto a la 5´ UTR como a la 3´ UTR-II a 26°C. La LbEF1α es una proteína abundante que participa en otros procesos celulares como el transporte nuclear, el control de calidad de las proteínas y la degradación concomitante con la traducción, entre otros (Mateyak & Kinzy, 2010; Mittal, et al., 2013). La LbEF2, con un 61 % de identidad con la proteína ortóloga humana y con la de Saccharomyces cerevisiae, se conoce por su función en la translocación de los ribosomas a lo largo del ARNm durante el proceso de traducción (Justice, et al., 1998; Noble & Song, 2008). Acerca del segundo y tercer grupo de proteínas, primero debe comprobarse tanto in vitro como in vivo su interacción con el ARN, para luego confirmar o elucidar su función específica. Por ello, en el presente estudio la atención se centró en estas proteínas, especialmente las hipotéticas.
Tabla 1 Lista de proteínas identificadas con los cebos de las regiones UTR de los mensajeros de la proteína HSP70 pertenecientes a las categorías funcionales del metabolismo del ARN y la traducción
Teniendo en cuenta que las herramientas bioinformáticas pueden predecir la presencia de dominios o motivos secuenciales o estructurales relacionados con la interacción entre ARN y proteína, estas fueron útiles para seleccionar con cuáles de las siete proteínas hipotéticas continuar el estudio (Nocua, et al., 2014). Para ello, se usaron programas como el InterProScan (http://www.ebi.ac.uk/Tools/pfa/iprscan/), el Phyre2 (http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index) y el Modeller (http://salilab.org/modeller/), y se hizo una revisión de la literatura. En el ejemplo que se presenta, gracias a la presencia de los dominios Lsm y NTF2 presentes en familias de proteínas relacionadas con la represión de la traducción, o la acumulación de mensajeros en los cuerpos P y los gránulos de estrés (Lsm) y en el transporte de ARN (NTF2), se seleccionaron las proteínas codificadas por los genes LbrM.25.2210 y LbrM.30.3080 (Nocua, et al., 2017).
Análisis estructural y modelamiento de las proteínas de interés
Con el ánimo de ahondar en la estructura de las proteínas codificadas por los genes LbrM.25.2210 y LbrM.30.3080, se recurrió a diversas predicciones bioinformáticas que, en primer lugar, mostraron que tenían ortólogos tanto en otras especies de leishmanias como de tripanosomas y de otros organismos evolutivamente mucho más distantes, como S. cerevisiae, Xenopus laevis y Homo sapiens (De Gaudenzi, et al., 2005; Kruger, et al., 2013; Nocua, 2017).
Tras estos análisis de homología de secuencia, se consideró conveniente cambiar el nombre de las proteínas codificadas por los genes LbrM.25.2210 y LbrM.30.3080 a LbSCD6 y LbRBP42, respectivamente. Para el caso de LbSCD6, la predicción de dominios permitió localizar en los primeros 79 aminoácidos de la proteína un dominio Lsm, que ha sido implicado en la formación de cuerpos P y gránulos de estrés (Kruger, et al., 2013). En la parte central de la molécula se identificó el dominio DFDF (DFDF-x(6)-F), caracterizado por una alternancia de aminoácidos no polares y cargados, responsables de la interacción con el ARN (Anantharaman & Aravind, 2004). Además, en el extremo carboxilo terminal, se identificaron las cajas FFD (Y-x-K-x(3)-FFD-x-[IL]-S) y TFG ([RKH]-x(2-5)-Ex(0-2)-[RK]-x(3-4)-[DE]-TFG), y, flanqueando el dominio DFDF y la caja TFG, se encontró un motivo de aminoácidos RGD, el cual ha sido implicado en la mediación de interacciones entre ARN y proteína y entre proteína y proteína, así como en la formación de gránulos de estrés (Kruger, et al., 2013; Nocua, et al., 2017; Rajyaguru & Parker, 2012). En el modelamiento tridimensional de la proteína, se encontró que estos dominios adoptaron las estructuras y plegamientos previamente descritos en la literatura, con excepción de la estructura de hélice α presente en el extremo N-terminal del dominio Lsm (Nocua, et al., 2017). Sin embargo, al comparar la estructura de LbSCD6 con la EDC3 humana y la TraI de Drosophila melanogaster, se encontró que la LbSCD6 encajaba perfectamente en los segmentos conservados Sm1 y Sm2 que hacen parte de la estructura típica de barril beta de este dominio (Nocua, et al., 2017). En cuanto a la RBP42, se identificó el dominio NTF2 (Ribbeck, et al., 1998), relacionado con procesos de transporte entre núcleo y citoplasma de ARN, y que en esta proteína está conformado por los primeros 114 aminoácidos del extremo N-terminal y también adopta el plegamiento típico consistente en tres hélices α alineadas contra una hoja β conformada por cinco cadenas. Así mismo, los 73 aminoácidos del extremo C-terminal de RBP42 conforman un dominio RRM (RNA recognition motif), que es uno de los dominios de unión a ARN más conservados a lo largo de la escala filogenética. No obstante, a diferencia de la mayoría de estos dominios, la LbRPB42 solo posee una de las dos marcas conservadas, RNP1, consistente en ocho residuos (con abundancia de aminoacidos aromáticos o cargados), al parecer responsable de la unión al ARN (Maris, et al., 2005). Cabe destacar que la proteína ortóloga de humanos, la G3BP, la cual, además de poseer ambos dominios como la proteína LbRPB42, también presenta tres repeticiones del motivo PxxP y ha sido implicada en la localización de esta proteína en los gránulos de estrés (Tourriere, et al., 2003).
Clonación, expresión y obtención de anticuerpos contra las proteínas de interés
Una vez conocidos los dominios y la estructura tridimensional de las proteínas promisorias, se procede a su clonación y expresión como proteínas recombinantes, con el fin de obtenerlas en estado nativo y producir sus anticuerpos. Dichas proteínas constituyen herramientas muy útiles en los experimentos encaminados a descifrar su funcionalidad. En nuestro ejemplo, las proteínas se clonaron en el sistema de expresión pQE30 y, tras ensayar diversas condiciones de solubilización y renaturación, finalmente se purificaron y se obtuvieron en su estado nativo, en tanto que los anticuerpos se obtuvieron mediante inmunización en conejos (Nocua, et al., 2017).
Confirmación de la capacidad de unión a ARN tanto in vitro como in vivo de las proteínas de interés
Teniendo en cuenta que la técnica de pesca o pull-down empleada es una tamización del lisado de proteínas totales del parásito, el siguiente paso metodológico son los ensayos confirmatorios, los cuales combinan los ensayos de pull-down y visualización tras el dot blotting, utilizando exclusivamente las proteínas específicas de interés y los cebos de ARN, con el fin de confirmar su capacidad de unión a este. En el presente caso, con esta metodología se corroboró que tanto la LbSCD6 como la LbRBP42 son proteínas capaces de unirse al ARN e interactuar de forma específica con la región 3´ UTR-II de los mensajeros de la HSP70 (Nocua, et al., 2017).
Después de demostrar la interacción in vitro de ambas proteínas (LbSCD6 y LbRPB42) con la región 3´ UTR de los genes HSP70-II, se hicieron ensayos para delimitar posibles elementos en cis involucrados en dicha interacción. Para lograr este objetivo, se sintetizaron los dos elementos en cis previamente identificados en la región 3´ UTR-II. El primero de ellos, correspondiente a la secuencia rica en adeninas y uridinas relacionada con los elementos ARE, se denominó 3´ UTR-II.1 y, el segundo, correspondiente a la region rica en citosinas, se denominó 3´ UTR-II.2. Para analizar la posible interacción entre las proteínas LbSCD6 y LbRBP42 con los motivos seleccionados, se hicieron ensayos de retraso en la movilidad electroforética (electrophoretic mobility shift assay, EMSA) utilizando las proteínas recombinantes, así como los oligorribonucleótidos marcados con digoxigenina en su extremo 5´. Los resultados mostraron una fuerte interacción entre el motive 3´ UTR-II.1 y cada una de las proteínas recombinants rLbSCD6 (Figura 3A) y rLbRBP42 (Figura 3B) (Nocua , 2017). En cambio, en el ensayo con el motivo 3´ UTR-II.2 no se observó interacción con las proteínas recombinantes (Nocua, et al., 2017). Los ensayos adicionales de EMSA de competición confirmaron los resultados y evidenciaron que la interacción entre las proteínas recombinantes y el motivo 3´ UTR-II.1 es específica y depende tanto de sus características estructurales como de su secuencia (Nocua, et al., 2017).
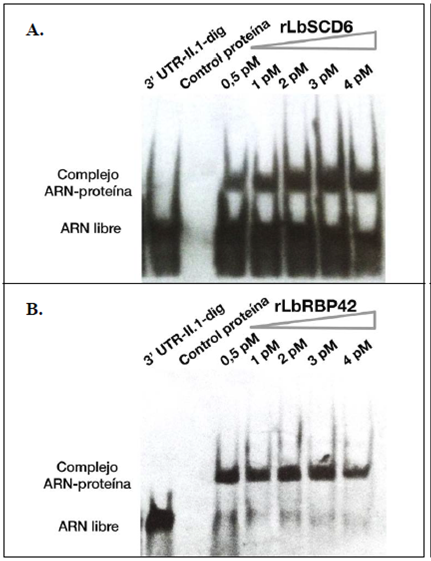
Figura 3 Interacción de las proteínas rLbSCD6 y rLbRBP42 con el motivo de ARN 3´ UTR-II.1 detectada mediante ensayos EMSA. (A) Análisis de la capacidad de interacción de rLbSCD6 con el motivo 3´ UTR-II.1 utilizando diferentes concentraciones de la proteína. (B) Análisis de la capacidad de interacción de rLbRBP42 con el motivo 3´ UTR-II.1 utilizando diferentes concentraciones de la proteína
Confirmación in vivo de la capacidad de unión al ARN de las proteínas de interés
Una vez confirmada la capacidad de estas proteínas de interactuar directamente con moléculas de ARN in vitro, se determinó esta misma capacidad in vivo, con el objetivo adicional de conocer los transcritos de ARN que pueden unirse a ellas en promastigotes de L. braziliensis cultivados a 26 ºC. Para ello, se hicieron ensayos de coinmunoprecipitación utilizando anticuerpos específicos para cada una de las proteínas, con el fin de obtener los complejos LbSCD6-ARN y LbRBP42-ARN. Estos mismos ensayos se realizaron utilizando el suero preinmunización correspondiente a cada anticuerpo (preSCD6 y preRBP42) y la información obtenida se utilizó como control de uniones no específicas, las cuales se descartaron de las lecturas obtenidas en las dos réplicas biológicas hechas para cada una de las proteínas estudiadas (Nocua, 2017). Los resultados en las dos proteínas confirmaron que estas interactúan in vivo con distintos transcritos del parásito, principalmente a través de sus regiones UTR (Nocua, 2017). Específicamente, para proteínas LbSCD6 se encontraron 155 transcritos que codifican, siendo la interacción con las regiones 3’ UTR la más frecuente (66 %). Con el fin de categorizar estos genes según el proceso biológico en el que están implicados, se hizo un análisis de enriquecimiento con la herramienta Gene Ontology (http://geneontology.org/page/go-enrichment-analysis), y se encontró que 98 (63 %) correspondían a genes anotados en la base de datos TriTryp con una función conocida (Aslett, et al., 2010), en tanto que el 37 % restante correspondió a genes hipotéticos. En cuanto a la adscripción de los 98 transcritos a procesos biológicos, se encontró que, en su mayoría, estos pertenecen a proteínas del metabolismo celular (30,61 %), seguidos por proteínas asociadas a procesos celulares (11,28 %), al transporte celular (8,16 %) y a la proteólisis celular (7,14 %). Resultó interesante observar la interacción de la LbSCD6 con transcritos que codifican para la proteína DNAJ-like, indispensable en la maquinaria de activación y funcionamiento de la proteína HSP70 (Kampinga & Craig, 2010). Se encontraron 192 (74,13 % del total) transcritos que codifican asociados a la proteína LbRBP42 (Nocua, 2017). En cuanto a la localización de la interacción ARNm-proteína, se encontraron principalmente las interacciones con las regiones 3´ UTR de los transcritos (57,8 %). De los 192 transcritos sometidos a coinmunoprecipitación, el 68,04 % correspondió a proteínas con anotación funcional en la base de datos TriTryp, en tanto que el 31,96 % correspondió a proteínas hipotéticas. Según su clasificación por asociación a procesos biológicos, se observó una gran proporción de proteínas asociadas al metabolismo celular (18,18 %), así como a diferentes procesos celulares, entre las cuales cabe resaltar las adscritas al ciclo celular (13,64 %). Asimismo, se encontraron proteínas asociadas a la respuesta a estímulos (7,58 %), entre ellas las asociadas a la respuesta al estrés. Cabe destacar que el transcrito correspondiente al gen HSP70- II se encontró asociado a la proteína LbRBP42, interacción localizada en la región 3´ UTR de este transcrito.
Teniendo en cuenta que tanto la LbSCD6 como la LbRBP42 se encontraron interactuando in vivo con la región 3´ UTR de más de cien transcritos y que in vitro se demostró la interacción de ambas proteínas con el motive 3´ UTR-II.1, relacionado con el elemento ARE, se hizo una búsqueda preliminar de motivos en la región 3´ UTR de todos los transcritos asociados. Así, se encontró un motivo estructural asociado a las regiones que interactúan tanto con la proteína LbSCD6 (Figura 4A) como con la proteína LbRBP42 (Figura 4B). Puede observarse que, si bien la secuencia del motivo no corresponde a la de un elemento ARE típico, su estructura presenta una horquilla semejante a la observada en el motivo 3´ UTR-II.1, lo que sugiere que la interacción entre los ARNm y las proteínas LbSCD6 y LbRBP42 puede estar guiada principalmente por motivos estructurales más que por una secuencia específica.
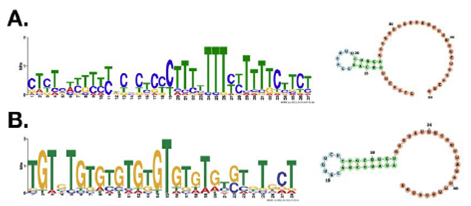
Figura 4 Motivos estructurales presentes en los transcritos asociados in vivo con las proteínas SCD6 y RBP42. Los posibles motivos asociados con las proteínas (A) SCD6 y (B) RBP42, se encontraron con la herramienta MEME, y las estructuras secun- darias de un motivo representativo para cada proteína se obtuvieron con la herramienta RNAfold.
Estudios de aproximación a la función biológica de las proteínas de interés
Con el propósito de dilucidar los posibles procesos biológicos en los cuales podrían estar involucradas estas proteínas, se determinó, en pirmer lugar, el interactoma de cada una. Los resultados evidenciaron que los interactomas de LbSCD6 y LbRBP42 comparten una alta proporción de proteínas, lo que sugiere algún tipo de relación funcional entre ellas; de hecho, las dos proteínas forman parte de los interactomas recíprocos. Es de resaltar que ambos interactomas están enriquecidos en proteínas asociadas a los procesos de traducción, al metabolismo del ARN y a la regulación de la expresión génica. Además, se encontraron varias proteínas cuyos homólogos se han reportado como constituyentes de gránulos de ARN, principalmente gránulos de estrés y cuerpos de procesamiento (Nocua, 2017); ambos son estructuras macromoleculares carentes de membrana en las que se acumulan ARN y proteínas. En eucariotas superiores los gránulos de estrés se forman transitoriamente en respuesta al estrés celular y están asociados con la traducción y estabilidad del ARNm. Bajo las condiciones apropiadas, los ARNm pueden salir de estos gránulos y asociarse de nuevo con polisomas (Buchan & Parker, 2009). En cuanto a los cuerpos de procesamiento, estos son estructuras constitutivas, las cuales contienen generalmente maquinaria relacionada con la degradación del ARNm (Parker & Sheth, 2007). Tanto los gránulos de estrés como los cuerpos de procesamiento interactúan entre sí y están en una estrecha asociación con el proceso de traducción (Decker & Parker, 2012).
Por último, para analizar la relevancia celular y funcional de LbSCD6 y LbRBP42 se generaron líneas mutantes de parásitos que sobreexpresan las proteínas fusionadas a la proteína fluorescente mCherry, y que denominamos LbSCD6-mCherry y LbRBP42-mCherry. La línea que sobreexpresó control (expresión de la proteína mCherry) de L. braziliensis pudo establecerse, pero luego de una semana de crecimiento, las líneas de sobreexpresión para cada una de estas proteínas perdieron viabilidad. Si bien estos resultados no son concluyentes, sugieren que la sobreexpresión de LbSCD6 y LbRBP42 en L. braziliensis afecta negativamente el desarrollo y la supervivencia del parásito. Como alternativa, se intentó con éxito la generación de estas líneas de sobreexpresión utilizando la especie L. major, lo que ha permitido vislumbrar importantes implicaciones de estas proteínas en el desarrollo de los parásitos, observándose alteraciones morfológicas significativas y el retardo del crecimiento de los parásitos en condiciones fisiológicas de crecimiento, siendo el efecto mucho más pronunciado en condiciones de estrés térmico. Por otra parte, la capacidad infectiva in vitro de estos parásitos mutantes resultó también significativamente afectada en relación con la cepa silvestre (Nocua, 2017). Teniendo en cuenta todos los hallazgos encontrados, en la figura 5 se presenta un modelo que refleja el papel de las proteínas LbSCD6 y LbRBP42 en el parásito.
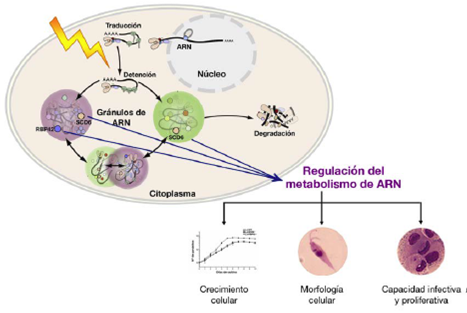
Figura 5 Modelo del rol de de las proteínas LbSCD6 y LbRBP42 en la regulación de la expresión génica a nivel del ARN en el parásito. Teniendo en cuenta todos los hallazgos derivados de este trabajo, se sugiere que las proteínas SCD6 y RBP42 están implicadas en la regulación de transcritos de ARN debido a su capacidad de unión con este tipo de moléculas y a la asociación que presentan con constituyentes de gránulos ribonucleoproteicos, como los cuerpos de procesamiento y los gránulos de estrés, localizados en el citoplasma. La localización citoplasmática granular se observa claramente cuando los parásitos están sometidos a condiciones de estrés, particularmente de estrés térmico (Datos no mostrados). Los cambios en la expresión de estas proteínas afectan la regulación de la estabilidad y su capacidad de traducción de los ARN mensajeros (metabolismo de ARN), lo cual desencadena un efecto pleitrópico en el parásito, observándose cambios en el crecimiento y morfología celular, así como en la capacidad infectiva y proliferativa del parásito.
Conclusiones
Se concluyó que la LbSCD6 y la LbRBP42 son proteínas de unión al ARN implicadas en la regulación de la estabilidad o degradación no solo del transcrito de HSP70-II sino de un amplio grupo de transcritos en Leishmania.
En cuanto a la estrategia aquí propuesta, la cual se resume en la figura 6, los resultados evidencian su bondad y su potencial para la identificación de proteínas reguladoras de la expresión génica en tripanosomátidos.
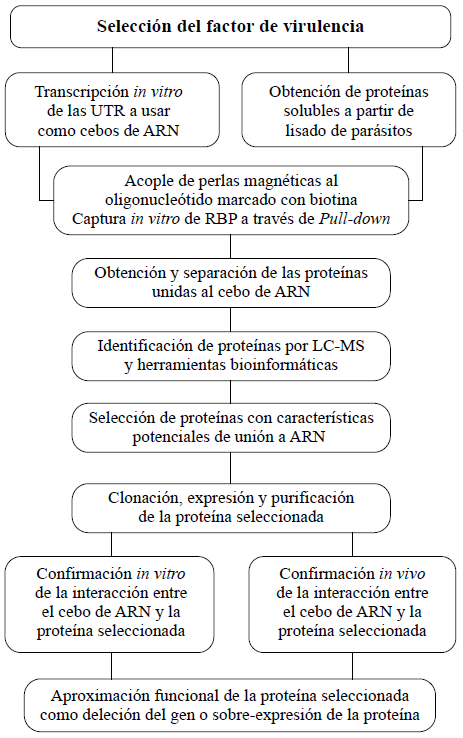
Figura 6 Esquema de resumen de la estrategia con el conjunto de pasos para la identificación de proteínas reguladoras de la expresión génica en tripanosomátidos.
Perspectivas de la estrategia. La metodología empleada comprende varias modificaciones y mejoras que pueden implementarse para subsanar algunas falencias detectadas. Por ejemplo, para simular las condiciones in vivo, es posible someter a los parásitos a la acción de la luz ultravioleta antes de la obtención del lisado proteico, con el fin de inmovilizar los complejos de ARN y proteínas formados bajo condiciones fisiológicas. Asimismo, se puede trabajar con los componentes subcelulares de interés, así como usar técnicas más finas de separación de proteínas que en una misma reacción permitan elucidar las proteínas diferenciales en relación con el control no relevante utilizado.
Teniendo en cuenta que la estrategia aquí propuesta se focaliza en la búsqueda de RBP que modulen la expresión de factores de virulencia de los tripanosomátidos, que como se vio en el caso de las proteínas LbSCD6 y LbRBP42, no solo gobiernan la expresión de dicho factor, en este caso la proteína HSP70, sino de muchas otras proteínas; en consecuencia, se proponen estas RBP como nuevos blancos parasitarios para el desarrollo de medicamentos. En este sentido, no resulta extraño que, dado su papel preponderante en la desregulación asociada al desarrollo de las células cancerosas, hayan sido propuestas también como nuevos blancos para el desarrollo de alternativas terapéuticas frente a los procesos tumorales (Turner & Díaz-Muñoz, 2018).