Introducción
La paloma doméstica Columba livia (Gmelin, 1789) pertenece a la familia Columbidae, la cual probablemente evolucionó mediante procesos de selección a partir de especies de aves antecesoras. A menudo denominada paloma bravía de Castilla o zuro, se la considera una especie invasora (González, et al., 2018) que se asienta en hogares y se mantiene como un ave de ornato (Mancera, et al., 2013); tiene un elevado crecimiento poblacional debido a su capacidad adaptativa en los entornos de gran incidencia humana (Mondocorre, et al., 2014), lo que le permite desarrollarse bajo diversas condiciones urbanas (Vargas, 2016) y, probablemente, responde a cierto tipo de selección artificial (Jacquin, et al., 2011).
El plumaje de la paloma doméstica presenta variaciones (Lesiss, et al., 1999a), y las bases genéticas de la coloración y el plumaje se codifican mediante un sistema múltiple de alelos (Dunmore, 1968). Asimismo, existen 60 factores hereditarios que influyen en la coloración de la paloma doméstica (Gibson, 1995). Los genes que codifican la pigmentación de la melanina tienen efectos pleiotrópicos sobre otros procesos biológicos de las aves (Gasparini, et al., 2009). Los procesos adaptativos bajo los cuales se fija la coloración y la melanización resultan de factores de la historia natural de las aves, por lo que se cree que las palomas de diferentes colores están adaptadas a ambientes alternativos cuya abundancia de alimentos (Roulin, 2004) y la exposición a los parásitos varían (Miranda, 2006).
Los marcadores fenotípicos asociados con la coloración son herramientas útiles para analizar los perfiles genéticos de las poblaciones naturales; la información obtenida permite inferir las relaciones genealógicas entre los individuos de una población, la valoración del flujo genético entre poblaciones, estimar el tamaño efectivo de las poblaciones y minimizar los efectos negativos de la endogamia (Eiroa, 2016).
Varios autores han reportado estudios genético-poblacionales de palomas domésticas en Colombia. En Bogotá se ha hecho la caracterización genética de la población de palomas domésticas (Pardo, et al., 2015): los marcadores Checker y Spread fueron los de mayor frecuencia alélica y, asimismo, se evidenció un elevado flujo génico y bajas distancias genéticas. En Lorica, Córdoba, se evaluó la diversidad genética de las palomas utilizando genes asociados con el color (Causil, et al., 2016), registrándose la escasa diferenciación genética entre las poblaciones y la posible selección natural para el marcador Spread. La diversidad genética implica aspectos importantes en términos de la adaptación y el mejoramiento de las especies (Díaz, et al., 2004). En ese contexto, el propósito de este estudio fue analizar la diversidad genética de la paloma doméstica (C. livia) a partir de los genes asociados con el color del plumaje en San Antero, Córdoba.
Materiales y métodos
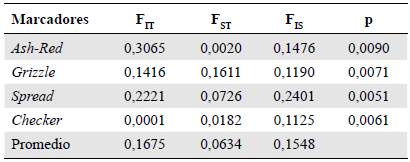
Area de estudio. El estudio se llevó a cabo en San Antero, municipio ubicado en la costa norte de Colombia a 9° 22' 28,711" N y 75° 45' 30,661" O, cuya temperatura promedio es de 28 °C. Se muestrearon cuatro subpoblaciones ubicadas en los sitios de Calle Abajo, Calle Central, Parque Central e Iglesia Central.
Recolección de los datos. Entre marzo y abril del 2017, se hicieron muestreos aleatorios en cuatro subpoblaciones del municipio durante excursiones urbanas, mediante la observación directa y con ayuda de registros fotográficos en horas de la mañana (de 6:00 am a 9:00 am). Se hizo una clasificación fenotípica de cada uno de los individuos adultos encontrados (n=235), atendiendo a la presencia o ausencia de los marcadores autosómicos (Tabla 1) Grizzle (G), Spread (S), Checker (C) y Ash-Red (B).
Tabla 1 Descripción de los cuatro genes estudiados
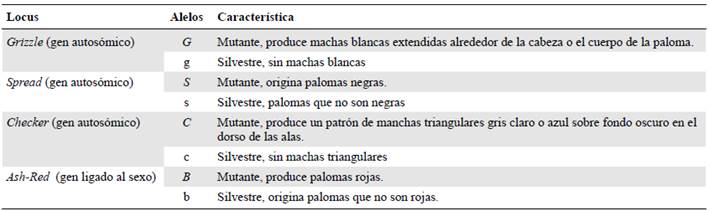
Grizzle (G); Spread (S); Checker (C) y Ash-Red (B)
Diseño estadístico. Los perfiles genéticos de las poblaciones se calcularon con los siguientes índices genético-poblacionales: las frecuencias alélicas de cada marcador a nivel poblacional y global; las medidas de diversidad genética establecidas por Nei (1973) correspondientes a la heterocigocidad esperada (He), la heterocigocidad esperada de la población total (HT), el coeficiente de diferenciación genética (Gst), el flujo génico (Nm), y las distancias genéticas de Nei (1972), de las poblaciones, las cuales se estimaron utilizando el programa PopGene 1.31 (Yeh, et al., 1999). La estructura genética de las poblaciones según los índices de fijación propuestos por Wright (Fis, Fit y Fst), se calculó mediante el programa FSTAT v 2.9.3.2 (Goudet, 2002).
Resultados
Se muestrearon 235 individuos distribuidos en cuatro sub-poblaciones (Figura 1): Calle Abajo (n=42), Calle Central (n=63), Parque Central (n=83) e Iglesia Central (n=47).
Frecuencias alélicas. Las frecuencias alélicas para cada población (Tabla 2) evidenciaron que el marcador Checker (p=0,4293) fue el más frecuente, seguido de Spread (p=0,3683) y de Ash-Red, con los valores más bajos (p=0,0729).
Tabla 2 Frecuencias alélicas de cada marcador en las poblaciones estudiadas
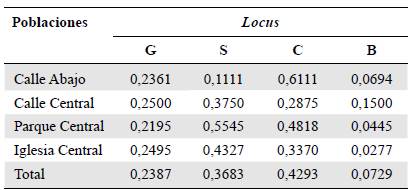
G: Grizzle; S: Spread; C: Checker; B: Ash-Red
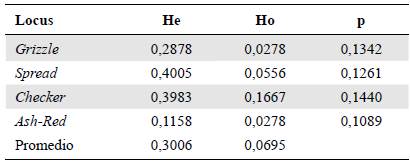
Diversidad genética. La mayor heterocigocidad esperada se encontró en el marcador Spread, con un valor de 0,4005, y la más baja, en el marcador Ash-Red, con un valor de 0,1158 (Tabla 3). Los valores de la heterocigocidad observada fueron bajos: oscilaron entre un máximo de 0,1667 para el marcador Checker y un mínimo de 0,0278 para el Ash-Red. En cada marcador los valores de la variabilidad genética fueron relativamente bajos.
Tabla 3 Índice de diversidad genética de Nei (1973) en cada marcador a través de la heterocigocidad esperada (He), la hetero-cigocidad observada (Ho) y el valor de p para cada marcador
Coeficiente de diferenciación genética. El nivel de diferenciación genética (Tabla 4) entre las distintas subpoblaciones de palomas domésticas (C. livia) en la población de San Antero fue relativamente bajo según el valor promedio de Fst, el cual fue de 0,0634, con un +inidce de heterogeneidad entre poblaciones (Gst) igual a 0,037, lo cual indica que aproximadamente el 3,7 % de la variación revelada se debe a la diferencia entre las poblaciones y, por lo tanto, las cuatro subpoblaciones no fueron significativamente diferentes en cuanto a los marcadores estudiados.
El valor del flujo génico (Nm=2.870) evidenció que las poblaciones mantenían el intercambio genético, asumiéndose un total aproximado de tres migrantes por generación.
Cabe señalar que el valor obtenido está por encima de 1, lo que indica que el flujo génico supera los efectos de la deriva génica en las subpoblaciones y previene la diferenciación local. Ello significa que las subpoblaciones de palomas domésticas se comportaron como una sola y sugiere que para un mejor análisis y una comprensión espacio-temporal dinámica, no se debería establecer una subestructura de la población.
Estructura poblacional. Los valores positivos del Fis para todos los marcadores (Tabla 5) evidenciaron un exceso de homocigotos entre los individuos comparados con cada subpoblación, lo que lleva a admitir la presencia de consanguinidad dentro de las subpoblaciones, con valores que oscilan entre 0,2401 para el marcador Spread y 0,1125 para el marcador Checker. El valor promedio del Fst fue de 0,0634, lo que podría indicar una diferenciación genética significativa entre las poblaciones. Por último, el estadístico Fit evidenció un exceso de homocigotos de los individuos con respecto a la población total.
Distancias genéticas. La distancia genética (Tabla 6) entre las subpoblaciones fue baja, siendo más cercanas las subpoblaciones de Calle Central e Iglesia Central, en tanto que las subpoblaciones de Calle Abajo y Calle Central resultaron ser las de mayor diferencia genética. La subpoblación de Calle Abajo registró el valor más alto de distancia génica en comparación con el resto de las subpoblaciones.
Tabla 6 Matriz de distancia genética (Nei, 1972) entre las poblaciones
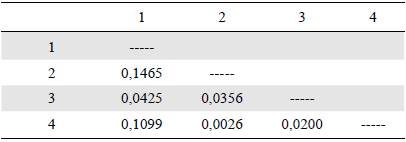
1: Calle Abajo; 2: Calle Central.; 3: Parque Central; 4: Iglesia Central
Discusión
Los elevados valores de las frecuencias alélicas de los marcadores Checker y Spread en este estudio concuerdan con los resultados descritos en otras investigaciones (Causil, et al., 2016; Pardo, et al, 2015). Los genes asociados con los colores melanóticos en palomas domésticas son más frecuentes en los centros urbanos, ya que, al parecer, las gónadas son más activas en épocas de menos lluvias, de manera que pueden reproducirse en mayor número en una temporada del año, en contraposición con otras aves que tienen diferentes patrones del pelaje (Hetmansky, 2008).
La mayor frecuencia de individuos melanóticos podría obedecer a cierta ventaja selectiva que tienen las palomas domésticas de coloraciones oscuras, ya que se ha reportado que son menos susceptibles de ser atacadas por parásitos hemosporidios; asimismo, serían más resistentes a ciertos patógenos (Lei, et al., 2013). Las coloraciones claras de las plumas en palomas domésticas podrían hacerlas fisiológicamente propensas a los parásitos cuando las ciudades son relativamente grandes; se cree que estos individuos más claros tienen una menor capacidad inmunitaria; por el contrario, los individuos más oscuros tienen una gran actividad inmunológica. Esta relación entre la inmunidad y la coloración con melanina podría deberse a efectos pleiotrópicos resultado de los altos niveles de la melanocortina (Jacquin, et al., 2013). Otra posible explicación se relacionaría con las expresiones fenotípicas oscuras, ya que estas tienen mayor resistencia al estrés y es posible que estas palomas tengan exposición perinatal a la corticosterona, lo cual depende de la condición materna, de las características del nido y de las provisiones de alimento, aspectos que, al parecer, tienen efectos organizativos y permiten que los individuos oscuros soporten mejor el estrés en los centros urbanos (Corbel, et al., 2016)
Otro factor asociado a la selectividad de colores oscuros en las palomas domésticas podría deberse a la gran producción de huevos por parte de las palomas más oscuras, hecho que sería atribuible a posibles ventajas selectivas de los individuos con mayor masa corporal, lo que ocasiona un incremento en la actividad reproductiva de las palomas y, probablemente, causaría efectos selectivos de las condiciones ambientales en los genes melanóticos del plumaje (Causil, et al., 2016)
El uso del hábitat puede tener un papel importante en el momento de fijar los genes melanóticos, siendo este el caso de los marcadores polimórficos. Específicamente, es posible que las plumas melanóticas tengan una mayor eficacia de desintoxicación por la intensa actividad de muda de estas aves, lo cual permite suponer que las palomas más oscuras podrían habitar zonas más contaminadas que las palomas con patrones más claros, rasgo que representaría una posible fuerza selectiva a favor de los fenotipos melanóticos entre las palomas domésticas en los centros urbanos (Chatelain, et al. 2014). Era de esperarse que las frecuencias alélicas se acentuaran en los marcadores más oscuros como el Checker y el Spread, pues el estudio se llevó a cabo en un municipio urbano, lo que influiría en que los genes melanóticos exhibieran patrones selectivos como la inmunidad a los parásitos, así como la ventaja fisiológica de la acción de la mela-nocortina y la gran producción de huevos y el consecuente aumento de estos marcadores, hecho coherente con el grado de urbanización de esta localidad.
El grado de flujo genético permite inferir que existe una fuerza que mantiene integrada la población, lo que condicionaría la persistencia de un perfil genético en la población local, de manera que los rasgos de distribución geográficas evolucionaron de forma conjunta (Cárdenas, 2012).
La reducción de heterocigotos obtenida al aplicar los distintos índices de fijación (Fis y Fit) en cada una de las sub-poblaciones estudiadas evidenció que se comportan como una población, en tanto que los bajos índices de diferenciación genética revelan una débil estructuración de la diversidad genética (Vergara, 2015), hecho que sería atribuible a que las poblaciones naturales y comunicadas entre sí exhiben una menor diferenciación genética (Matallanas, 2015). El grado de diferenciación genética entre las poblaciones fue bajo (Gst=0,037). Un estudio en aves demostró que los machos se aparean con varias generaciones de su descendencia provocando una creciente similitud entre ellas (Acuña, 2016). Las palomas tienden a elegir parejas genéticamente similares, lo que ocasiona altos niveles de endogamia en la población (Jacob, et al., 2016); por otra parte, es posible que exista una correlación entre las distancias genéticas y las geográficas, pues la diferenciación genética aumenta proporcionalmente con las distancias geográficas, lo que podría implicar un número significativo de inmigrantes (Zuleta, et al., 2012). Esto sugiere que los eventos selectivos conllevaron un intercambio genético que superó los efectos de la deriva genética y la distinción local (Causil, et al., 2016).
Las distancias genéticas entre las cuatro subpoblacio-nes del estudio, especialmente la cercanía geográfica entre la subpoblación de Calle Central e Iglesia Central, se explicarían por el alto flujo genético entre ellas, ya que, al parecer, las palomas domésticas presentan mayor probabilidad de vuelo de una población cercana a otra, lo que genera en los individuos un mayor grado de confianza y probablemente es producto de su adaptación en zonas urbanas (Skandrani, et al., 2016). Esto podría explicar el parecido genético de las subpoblaciones estudiados y permite concluir que la población de palomas domésticas de San Antero tendría un mismo perfil genético-poblacional, por lo que se deberían estudiar como una sola población.
Conclusiones
Los marcadores más frecuentes fueron el Checker y el Spread, ya que, al parecer, la selección natural está favoreciendo a los individuos más oscuros por su mayor eficacia reproductiva, cierta resistencia a los parásitos y las ventajas inmunológicas producto de efectos pleiotrópicos. La población total mostró un alto porcentaje de homocigotos y a nivel de las subpoblaciones la diferenciación génica fue baja, lo cual evidencia un parentesco genético entre las palomas de los sitios estudiados debido al intercambio de genes entre estas subpoblaciones y refleja la existencia de similitudes genéticas locales.