Comentario sobre el artículo
Hedlund B.P., Chuvochina M., Hugenholtz P., Konstantinidis K.T., Murray A.E., Palmer M., Parks D.H., Probst A.J., Reysenbach A.L., Rodriguez-R L.M., Rossello-Mora R., Sutcliffe I.C., Venter S.N., Whitman W.B. (2022). SeqCode: a nomenclatural code for prokaryotes described from sequence data. Nature Microbiology, 7, 1702-1708. https://doi.org/10.1038/s41564-022-01214-9
What's in a name? That which we call a rose By any other name would smell as sweet.
Romeo y Julieta. William Shakespeare, 1597
Esa pregunta la puso el conocido dramaturgo William Shakespeare en boca de Julieta, para quien Romeo seguiría siendo su amante aun si llevara otro nombre, con lo que sugiere que los nombres serían una convención sin mayor significado individual.
La necesidad de darle un nombre a lo que nos rodea, desde objetos a personas e, incluso, sentimientos y emociones, es un atributo que nos resulta esencial para distinguir, clasificar y comunicarnos. Y aunque es cierto que en algunas ocasiones el nombre de un objeto puede no ser tan importante, este no es el caso para las ciencias. La comunicación efectiva y el intercambio de ideas científicas requieren de nomenclaturas precisas. En las ciencias de la vida esta necesidad es particularmente evidente al hablar de las diversas especies de organismos que habitan nuestro planeta.
Las especies se nombran siguiendo códigos de nomenclatura para garantizar que cada una tenga un nombre único que ayude a entender su relación con otros organismos en el contexto taxonómico. Este nombre único consta de dos partes y sigue el sistema de nomenclatura "binomial" introducido hace casi 300 años por Carl Linnaeus, científico sueco reconocido como el padre de la taxonomía moderna (Venter, 2022). En este sistema, la primera parte identifica el género y la segunda es un nombre específico que distingue la especie de otras en el mismo género. Es así como nuestra especie Homo sapiens pertenece al género Homo y se distingue de especies ya extintas pero del mismo género, como Homo erectus y Homo neanderthalensis.
La labor de la taxonomía, es decir, el proceso de clasificación ordenada y jerárquica de los organismos, no está exenta de dificultades. En el caso de los microorganismos procariotas (bacterias y arqueas), la clasificación y la nomenclatura de las especies se complican por varios factores. Para comenzar, es difícil definir lo que constituye una especie. Los microorganismos pertenecientes a la misma especie varían en cuanto a sus funciones y el tamaño de sus genomas debido a su plasticidad y al hecho de que pueden intercambiar genes con otras especies mediante un mecanismo denominado transferencia horizontal de genes. Por otro lado, su tamaño microscópico dificulta la identificación de rasgos distintivos, razón por la cual hoy se utilizan varios parámetros para identificar especies, entre ellos, características morfológicas, metabólicas y genómicas. En los últimos años la secuenciación de genes conservados, como el gen 16S rRNA que codifica para la subunidad pequeña del ribosoma, y de genomas completos, se ha convertido en una ayuda fundamental para la clasificación de microorganismos. El gran número de genomas bacterianos que están en las bases de datos sirve como un recurso de información sólida para la comparación de organismos y la descripción de especies.
Por último, bajo el código de nomenclatura actual (International Code of Nomenclature of Prokaryotes, ICNP), cada especie procariota debe tener un representante "tipo" almacenado en alguna colección biológica que sirva como evidencia de su existencia y referencia para estudios posteriores. Este punto es particularmente polémico, ya que la gran mayoría de especímenes microbianos en nuestro planeta no se puede cultivar y se conocen únicamente a través de información genómica obtenida por secuenciación.
No sabemos cuántas especies procariotas hay en el planeta, pero se estima que solo una pequeña proporción del total (<0,2 %) cuenta con nombres asignados en la actualidad (Sutcliffe et al., 2021). El resto, la gran mayoría de los microorganismos no cultivados e identificados solo por secuenciación, permanece sin nomenclatura, pues no cumple con los criterios consignados en el código (Nayfach et al., 2021; Hedlund et al., 2022). Esta situación está causando un embotellamiento de la información dada la expansión de registros microbianos ambientales gracias a los avances en tecnologías de secuenciación para estudiar las comunidades microbianas sin necesidad de realizar cultivos. A partir de estas secuencias de ADN ambiental, o metagenómica, es posible ensamblar genomas procariotes, los denominados metagenome-assembled genomes (MAG). Con estos estudios, se ha expandido notablemente el repertorio y el conocimiento sobre la diversidad y la ecología de estas comunidades microbianas (Nayfach et al., 2021).
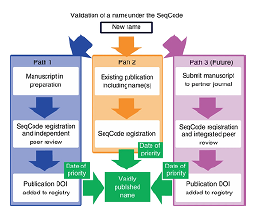
Con el fin de incluir esta mayoría aún no cultivada de microorganismos en el árbol de la vida y, de paso, también aquellos que se consideran fastidiosos por ser difíciles de cultivar y mantener en una colección, varios investigadores han propuesto un sistema de nomenclatura basado en la secuenciación genómica. Este nuevo código de nomenclatura se llama Code of Nomenclature of Prokaryotes Described from Sequence Data, o SeqCode, y utiliza genomas "tipo" para darle nombre a especies procariotas (Whitman et al., 2022). Este protocolo se desarrolló ante la negativa del comité internacional de sistemática de procariotes (International Committee on Systematics of Prokaryotes) a admitir secuencias genómicas como material tipo en su código.
SeqCode busca simplificar el proceso de nomenclatura y unificar la clasificación incorporando microorganismos cultivados y no cultivados. Este sistema permite registrar y validar un nombre utilizando un sistema en línea que sigue guías de nomenclatura y estándares claros para identificar genomas tipo (Hedlund et al., 2022). Se incluyen también aquellos registros que se designan como "candidatus", nombre que se otorga a microorganismos que aún no han sido cultivados, con lo cual se aumenta el registro de especies procariotas y se facilita el intercambio de información en la comunidad científica. Más importante aun es el hecho de incorporar al proceso de clasificación la información genómica y utilizar genomas "tipo", muchos de ellos obtenidos por secuenciación metagenómica.
La utilidad de SeqCode como sistema de clasificación y su aceptación por parte de la comunidad científica es aún incierta (Pennisi, 2022). Para algunos investigadores es difícil aceptar que una especie se pueda definir exclusivamente con base en un genoma, en particular a partir de los MAG. Para otros, sin embargo, puede ser una herramienta necesaria para clasificar el gran número de especies que se descubren en las exploraciones ambientales. Solo con el tiempo y el uso se podrá validar su utilidad en la taxonomía microbiana. En cualquier caso, este esfuerzo reconoce la relevancia que tiene la información genómica para la clasificación de microorganismos, y abre la posibilidad de organizar, conocer y explorar más a fondo la diversidad microbiana del planeta.