Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Colombia Médica
versión On-line ISSN 1657-9534
Colomb. Med. vol.50 no.3 Cali jul./set. 2019
https://doi.org/10.25100/cm.v50i3.2385
Articulo original
Mutaciones en los genes BRCA1 y BRCA2 en una muestra de familias con cáncer de mama y/u ovario del pacífico colombiano.
1 Universidad del Valle, Departamento de Biología, Human Molecular Genetics Lab, Cali, Colombia
Introducción:
El cáncer de mama es la neoplasia más común en mujeres de todo el mundo, y, también de Colombia. 5% a 10% de todos los casos son causados por factores hereditarios; 25% de estos casos tienen mutaciones en los genes BRCA1/BRCA2.
Objetivo:
El propósito de este estudio fue el de identificar mutaciones asociadas con riesgo de cáncer de mama y/u ovario familiar en pacientes del pacífico colombiano.
Métodos:
Fueron revisados para mutaciones en BRCA1 y BRCA2 de línea germinal mediante SSCP y secuenciación 58 familias de alto riesgo para cáncer de mama y/u ovario y 20 controles
Resultados:
cuatro familias (6.9%) presentaron mutaciones en BRCA1 y ocho familias (13.8%) en BRCA2. En BRCA1, encontramos tres variantes de significado clínico desconocido (VUS), de las cuales concluimos, usando herramientas bioinformáticas, que c.8112C>G y c.3119G>A (p.Ser1040Asn) son probablemente deletéreas, y c.3083G>A (p.Arg1028His) es probablemente neutral. En BRCA2, encontramos tres VUS: una mutación nueva y dos previamente descritas, usando análisis bioinformáticos, concluimos que c.865A>G (p.Asn289Asp) y c.6427T>C (p.Ser2143Pro) son probablemente deletéreas y c.125A>G (p.Tyr42Cys) es probablemente neutral. Solo una de ellas ha sido reportada previamente en Colombia. También identificamos 13 polimorfismos (4 en BRCA1 y 9 en BRCA2), dos de ellos asociados con un moderado incremento del riesgo para cáncer de mama (BRCA2 c.1114A>C and c.875566T>C).
Conclusión:
de acuerdo con nuestros resultados, la población del suroccidente colombiano presenta un espectro mutacional diverso para los genes BRCA que difiere de lo encontrado en otras regiones del país.
Palabras clave: Síndrome de Cáncer de Mama y Ovario Hereditario; BRCA1; BRCA2; Mutación de Línea Germinal; Colombia
Introduction:
Breast cancer is the most common neoplasia of women from all over the world especially women from Colombia. 5%10% of all cases are caused by hereditary factors, 25% of those cases have mutations in the BRCA1/BRCA2 genes.
Objective:
The purpose of this study was to identify the mutations associated with the risk of familial breast and/or ovarian cancer in a population of Colombian pacific.
Methods:
58 high-risk breast and/or ovarian cancer families and 20 controls were screened for germline mutations in BRCA1 and BRCA2, by Single Strand Conformation Polymorphism (SSCP) and sequencing.
Results:
Four families (6.9%) were found to carry BRCA1 mutations and eight families (13.8%) had mutations in BRCA2. In BRCA1, we found three Variants of Uncertain Significance (VUS), of which we concluded, using in silico tools, that c.8112C>G and c.3119G>A (p.Ser1040Asn) are probably deleterious, and c.3083G>A (p.Arg1028His) is probably neutral. In BRCA2, we found three variants of uncertain significance: two were previously described and one novel mutation. Using in silico analysis, we concluded that c.865A>G (p.Asn289Asp) and c.6427T>C (p.Ser2143Pro) are probably deleterious and c.125A>G (p.Tyr42Cys) is probably neutral. Only one of them has previously been reported in Colombia. We also identified 13 polymorphisms (4 in BRCA1 and 9 in BRCA2), two of them are associated with a moderate increase in breast cancer risk (BRCA2 c.1114A>C and c.875566T>C).
Conclusion:
According to our results, the Colombian pacific population presents diverse mutational spectrum for BRCA genes that differs from the findings in other regions in the country.
Keywords: Hereditary Breast and Ovarian Cancer Syndrome; BRCA1; BRCA2; Germ-line mutations; Colombia
Contribución del estudio
| 1) Por que se hizo este estudio? |
| La prevalencia y el tipo de mutaciones de la línea germinal BRCA1 o BRCA2, varía considerablemente entre diversos grupos étnicos y áreas geográficas. La población del Pacífico colombiano tiene un trasfondo genómico especial con alto grado de mezcla poblacional entre amerindios, afrodescendientes y europeos. Los datos sobre la contribución de las mutaciones de la línea germinal BRCA1 / BRCA2 para cáncer de mama en la población colombiana son escasos. El propósito de este estudio fue identificar las mutaciones asociadas con el riesgo de cáncer de mama y/o ovario familiar en una población del Pacífico colombiano. |
| 2) Cuales fueron los resultados mas relevantes? |
| Se realizó un tamizaje mutacional de los genes BRCA1 y BRCA2 para toda la región de codificación y los límites exón-intrón en familias colombianas de alto riesgo, seleccionadas en función de sus antecedentes familiares de cáncer de mama y de ovario. Fueron examinadas 58 familias con alto riesgo y con antecedentes de cáncer de mama y/u ovario y 20 controles, para detectar mutaciones de la línea germinal en BRCA1 y BRCA2, mediante polimorfismo de conformación de cadena simple (SSCP) y secuenciación. Se encontró que cuatro familias portaban mutaciones BRCA1 y ocho familias tenían mutaciones en BRCA2. En BRCA1, encontramos tres variantes de significancia incierta (VUS), de las cuales concluimos, utilizando herramientas in silico, que c.8112C> G y c.3119G> A (p.Ser1040Asn) son probablemente perjudiciales, y c.3083G > A (p.Arg1028His) es probablemente neutrales. En BRCA2, encontramos tres variantes de significado incierto, utilizando el análisis in silico, concluimos que c.865A> G (p.Asn289Asp) y c.6427T> C (p.Ser2143Pro) son probablemente perjudiciales y c.125A> G ( p.Tyr42Cys) es probablemente neutrales. También identificamos 13 polimorfismos (4 en BRCA1 y 9 en BRCA2), dos de ellos están asociados con un aumento moderado en el riesgo de cáncer de mama (BRCA2 c.1114A> C y c.875566T> C). |
| 3) Que significan los hallazgos? |
| La población del Pacífico colombiano presenta un espectro mutacional diverso para genes BRCA que difiere de los hallazgos en otras regiones del país. Por lo tanto, la extrapolación de resultados de una región al resto de la población colombiana es arriesgada; Estos hallazgos indican que la población colombiana tiene un espectro heterogéneo de mutaciones BRCA, por lo que es de suma importancia generar estudios en los que estén representadas las diferentes regiones del país, con el fin de hacer un enfoque real del espectro mutacional colombiano |
Introducción
El cáncer de mama (BC) es el tipo de cáncer más común entre las mujeres, con un valor estimado de 1.67 millones de nuevos casos diagnosticados en el año 2012 1. En Colombia, esta neoplasia es la principal causa de muerte por cáncer en mujeres 1 y para Santiago de Cali, que es el principal centro urbano del Sur Occidente de Colombia, el cáncer de mama ocupa el primer lugar en incidencia y mortalidad en la población femenina 2.
De todos los casos de cáncer de mama, alrededor del 5%-10% tienen un fuerte componente hereditario, de los cuales 25% es explicado por mutaciones germinales en los genes de predisposición a cáncer de mama de alta penetrancia, BRCA1 (3 y BRCA2 (4. El riesgo de cáncer de mama para portadores de mutaciones en BRCA1 a los 70 años, ha sido estimado en un rango de 40%-87% y para cáncer de ovario de 16%-68%; el riesgo correspondiente para portadores de mutaciones en BRCA2 fue estimado en 40%-84% para cáncer de mama y 11-27% para cáncer de ovario (5. El riesgo no explicado por los genes de alta penetrancia se debe a genes de baja y moderada penetrancia 6,7, cada uno teniendo un pequeño efecto sobre el riesgo de cáncer de mama 8.
La prevalencia y tipo de mutaciones germinales en BRCA1 y BRCA2 varían considerablemente entre diversos grupos étnicos y áreas geográficas. Se han descrito mutaciones específicas y recurrentes en poblaciones, entre los judíos Ashkenazi, Islandia, Países Bajos, Suecia, Noruega, Alemania, Francia, España, Canadá, países del este y sur de Europa 9 y en poblaciones Latinoamericanas, como Chile, México, Brasil y Costa Rica (10. Miles de mutaciones encontradas en los genes BRCA, en familias con cáncer de mama ovario se describen ahora en varios sitios web, como The Breast Cancer Information Core (BIC) (https://research.nhgri.nih.gov/bic/), The Leiden Open Variation Database (LOVD) (http://www.lovd.nl/3.0/home) y The Universal Mutation Database (UMD) website (http://www.umd.be/).
La población del pacífico colombiano tiene un background genómico especial con un alto grado de mezcla entre población Amerindia, Afrodescendiente y Europea. (11. Los datos sobre la contribución de mutaciones germinales en BRCA1/BRCA2 al cáncer de mama en la población colombiana son escasos; Torres et al. (12, evaluaron ambos genes utilizando las metodologías SSCP (Single Strand Conformation Polymorphism), DHPLC (Denaturing High Performance Liquid Chromatography) y PTT (Protein Truncation Test), y reportaron cinco mutaciones deletéreas para la región central de Colombia.
Los trabajos adicionales conducidos en el país se han enfocado en la búsqueda de mutaciones específicas (13,14 o en la evaluación de paneles mutacionales 15,16. En este estudio, un barrido mutacional fue realizado para toda la región codificante y los limites exón-intrón de los genes BRCA1 y BRCA2 en un grupo de 58 familias de alto riesgo seleccionadas con base en su historia familiar de cáncer de mama ovario.
Materiales y Métodos
Familias de alto riesgo para BC/OC (Cáncer de Mama/Cáncer de Ovario) del pacífico colombiano fueron seleccionadas de las historias clínicas de cuatro instituciones que ofrecen atención a la población de la región pacífica. Las familias seleccionadas cumplieron al menos uno de los siguientes criterios: al menos tres miembros de la familia con cáncer de mama u ovario a cualquier edad; dos miembros de la familia de primer grado afectados, al menos uno, con cáncer de mama antes de los 41 años o con cáncer de ovario a cualquier edad; un caso de cáncer de mama diagnosticado antes de los 35 años o un caso de cáncer de ovario diagnosticado antes de los 32 años.
Los pedigríes fueron construidos con base en un caso índice; ninguna de las familias cumplió con los criterios estrictos para otros síndromes conocidos que involucran cáncer de seno, como el síndrome de Li Fraumeni, la ataxia telangiectasia o la enfermedad de Cowden
Población control
La muestra de controles colombianos sanos fue reclutada de la comunidad de Cali. Muestras de ADN fueron tomadas de individuos no relacionados, sin antecedentes personales o familiares de cáncer. Estos individuos fueron entrevistados e informados acerca de los objetivos del estudio. La muestra de control fue apareada por edad, sexo y estado socioeconómico con respecto a los casos.
Las muestras de ADN de pacientes y controles fueron obtenidas de acuerdo a las consideraciones de requisitos éticos y legales de la Universidad del Valle y la ley colombiana para la investigación en humanos.
Análisis mutacional BRCA1 y BRCA2
DNA Genómico fue extraído a partir de linfocitos de sangre periférica, basado en el método descrito por Miller et al17. Toda la región codificante y los límites exón-intrón de los genes BRCA1 y BRCA2 fueron amplificados por la reacción en cadena de la polimerasa usando los cebadores descritos por Barker 18 y por base de datos BIC. Los fragmentos obtenidos fueron analizados usando SSCP 19,20. Los fragmentos que mostraron diferencias de movilidad fueron directamente secuenciados en ambas direcciones. La secuenciación fue realizada en un analizador genético ABI 3130 (Applied Biosystems, United States).
Las secuencias obtenidas fueron analizadas con el software ChromasPro versión 1.7.6 (http://technelysium.com.au/wp/chromaspro/). La presencia y localización de mutaciones fue obtenida por alineamiento de secuencias usando BLAST (Basic Local Alignment Search Tool) (http://blast.ncbi.nlm.nih.gov/Blast.cgi),
y las secuencias de referencia usadas fueron para BRCA1: ADN NG_005905.2, ARN NM_007294.3 y para BRCA2: ADN NG_012772.3 and ARN MN_000059.3. Dos tipos de nomenclatura fueron usados para verificar si las variantes detectadas habían sido reportadas en bases de datos o en la literatura; el sistema tradicional de la bases de datos BIC y la nomenclatura de la Human Genetic Variation Society (HGVS) (http://www.hgvs.org/).
Análisis computacional
Con el fin de determinar los posibles efectos de alteraciones de secuencia de significancia clínica desconocida, los siguientes software de predicción fueron corridos, para sustituciones missense: AlignGVGD (http://agvgd.iarc.fr/index.php), PANTHER (http://pantherdb.org/), PolyPhen2 (http://genetics.bwh.harvard.edu/pph2/), SIFT (http://sift.bii.a-star.edu.sg/), PROVEAN (http://provean.jcvi.org/index.php), MutPred (http://mutpred.mutdb.org/) y SNPs&GO (http://snps.biofold.org/snps-and-go/snps-and-go.html). Alineamientos nativos de cada algoritmo fueron usados. Para mutaciones intrónicas, predicciones de splicing fueron realizadas con: SSF, MaxEntScan, NNSplice y HSF algorithms, a través de Alamut Visual Software v.2.7 (http://www.interactive-biosoftware.com/). Condiciones predeterminadas fueron usadas para todos los análisis. Para la interpretación de las predicciones de estos cinco algoritmos, utilizamos los criterios descritos por Thery et al21.
Resultados
Un total de 72 casos índices pertenecientes a 58 familias de alto riesgo para BC/OC, fueron examinados para BRCA1 y BRCA2. En la muestra de familias seleccionadas, 57% (33/58) presentaban solo BC, 24% (14/58) presentaban BC y OC, 7% (4/58) presentaban BC y cáncer. Las características clínicas de las familias incluidas en este estudio se muestran en la Tabla 1. El diagnóstico de cáncer se certificó solicitando a todos los casos índice sus informes de patología originales. El promedio de edad de los pacientes al diagnóstico fue 41.7 años (rango entre 30-66 años). El 46.6% de los pacientes fueron diagnosticados antes de los 40 años, y el 87.9% fueron diagnosticados antes de los 50 años. El 31 % de las familias presentaban 3 o más o más parientes de primer grado con cáncer de mama y/o de ovario.
Tabla 1 Criterios de selección y características clínicas de las familias incluidas en este estudio
| Criterio de Selección | Familia n (%) |
|---|---|
| ≥3 miembros de la familia con cáncer de mama y al menos un cáncer de ovario. | 5 (8.6%) |
| ≥3 miembros de la familia con cáncer de mama. | 13 (22.4%) |
| 2 miembros de la familia con cáncer de mama y al menos un cáncer de ovario | 8 (13.8%) |
| 2 miembros de la familia con cáncer de ovario. | 18 (31.0%) |
| 1 miembro de la familia con cáncer de mama y al menos un cáncer de ovario | 7 (12.1%) |
| Un individuo afectado con cáncer de mama < 35 años | 6 (10.4%) |
| Un individuo afectado con cáncer de ovario < 31 años | 1 (1.7%) |
| Total | 58 (100.0%) |
Mutaciones germinales en BRCA1 o BRCA2 fueron encontradas en 12 (20,6%) de las 58 familias de la región pacifica colombiana estudiadas. Nosotros detectamos 6 variantes de significancia clínica desconocida (3 en BRCA1 y 3 en BRCA2) (Table 2), solo una de ellas reportada previamente para Colombia y también encontramos 13 polimorfismos (4 en BRCA1 y 9 en BRCA2) (Tabla 3).
Tabla 2 Mutaciones germinales en BRCA1 y BRCA2 detectadas en familias de alto riesgo de cáncer de mama/ovario del pacifico colombiano
| E/I no. | cDNA | Cambio Proteína | Tipo de Mutación | Familia | Familia Tipo | Caso índice (edad) | Historia Familiar (Br/Ov Ca) | Otros cánceres |
|---|---|---|---|---|---|---|---|---|
| BRCA1 | ||||||||
| I2 | c.81-12C>G | NA | Intrónica | F43 | Br/Ov | BrCa (42) Ov Ca (42, 44) | 7 Br Ca 4 Ov Ca | Próstata, cérvix, pulmón |
| E11 | c.3083G>A | p.Arg1028His | Missense | F52 F57 | Br Br | BrCa (45) BrCa (59) | 2 Br Ca 3 Br Ca | Columna Próstata Gástrico Linfático |
| E11 | c.3119G>A | p.Ser1040Asn | Missense | F56 | Br | BrCa (50) | 2 Br Ca | - |
| BRCA2 | ||||||||
| E3 | c.125A>G | p.Tyr42Cys | Missense | F44 | Br | BrCa (39) | 4 Br Ca | Pulmón, estomago |
| E10 | c.865A>G | p.Asn289Asp | Missense | F9 | Br | BrCa (41) | 3 BrCa | - |
| F37 | Br | BrCa (47) | 4 BrCa | Pulmón, Colon | ||||
| F38 | Br/Ov | BrCa (49) | 1 BrCa 1 OvCa | Próstata Ovario | ||||
| F49 | Br | BrCa (65) | 3 BrCa | - | ||||
| F52 | Br | BrCa (45) | 2 BrCa | Columna | ||||
| F53 | Br | BrCa (51) | 2 BrCa | Colon | ||||
| E11 | c.6427T>C | p.Ser2143Pro | Missense | F34 | Br | BrCa (40) | 2 Br Ca | próstata, melanoma |
E/I= exón/intrón, Br and BrCa: Cancer de mama, Ov y OvCa: Cancer de Ovario.
Tabla 3 Polimorfismos detectados en BRCA1 y BRCA2 en familias de alto riesgo de cáncer de mama/ovario.
| E/I no. | Cambio Nucleótido | Cambio Proteína | dbSNP | Tipo de Mutación | pacientes n=72 | controles n=20 | |
|---|---|---|---|---|---|---|---|
| BRCA1 | |||||||
| I7 | c.442-34C>T | NA | rs799923 | Intrónica | 3 | 2 | |
| E11 | c.2311T>C | p.Leu771= | rs16940 | S | 7 | 6 | |
| E11 | c.3113A>G | p.Glu1038Gly | rs16941 | M | 32 | 12 | |
| E16 | c.4837A>T | p.Ser1613Cys | rs1799966 | M | 48 | 12 | |
| BRCA2 | |||||||
| E2 | c.-26G>A | NA | rs1799943 | 5’UTR | 23 | 2 | |
| I4 | c.426-89T>C | NA | rs3783265 | Intrónica | 2 | 0 | |
| E10 | c.1114C>A | p.Asn372His | rs144848 | M | 25 | 2 | |
| E11 | c.2971A>G | p.Asn991Asp | rs1799944 | M | 1 | 0 | |
| E11 | c.3396A>G | p.Lys1132= | rs1801406 | S | 34 | 9 | |
| E11 | c.6513C>G | p.Val2171= | rs206076 | S | 4 | 4 | |
| I11 | c.6841+80_6841+83delTTAA | NA | rs3783265 | Intrónica | 4 | 1 | |
| I21 | c.8755-66T>C | NA | rs4942486 | Intrónica | 40 | 9 | |
| E22 | c.8851G>A | p.Ala2951Thr | rs11571769 | M | 23 | 6 | |
E/I: exón/intrón, S: sinónima, M: missense, NA: No aplica, 5’UTR: 5’ Región no traducida
Individuos sin antecedentes personales o familiares de cáncer de mama fueron invitados a participar como controles en el estudio. Reclutamos 20 controles de la ciudad de Cali (Colombia) que cumplieron con los criterios de inclusión como se indica en los métodos.
Mutaciones BRCA1
Para BRCA1 encontramos 3 VUS, 2 corresponder al tipo missense y la otra es de tipo intrónica. Las mutaciones encontradas fueron identificadas en 4 familias (6.9%) de nuestra Cohorte, tres de estas familias presentaban cáncer de mama y una familia presentaba cáncer de mama/ovario.
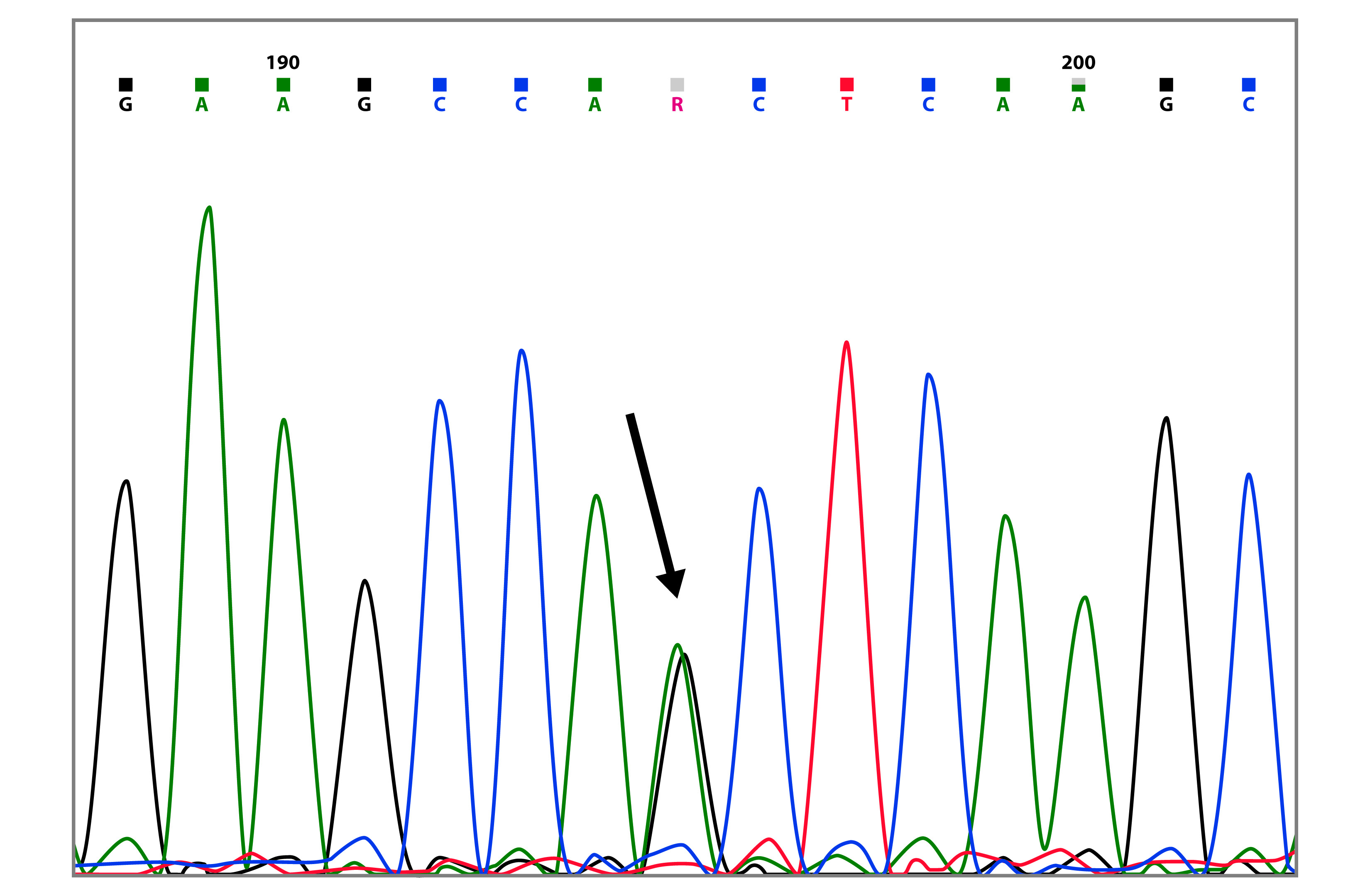
La mutación c.8112C>G, fue encontrada en 3 hermanas, 2 con cáncer de ovario (42 y 44 años, respectivamente) y una con cáncer de mama and (42 años) (Tabla 2 y Fig. 1). Esta mutación ha sido reportada 9 veces en el BIC y se ha encontrado principalmente en descendientes de Afroamericanos. A la fecha, en Sur América, no ha sido reportada previamente esta mutación. Además de esta mutación intrónica, las tres pacientes también presentaron los polimorfismos c.3113A>G y c.4837A>T en BRCA1.
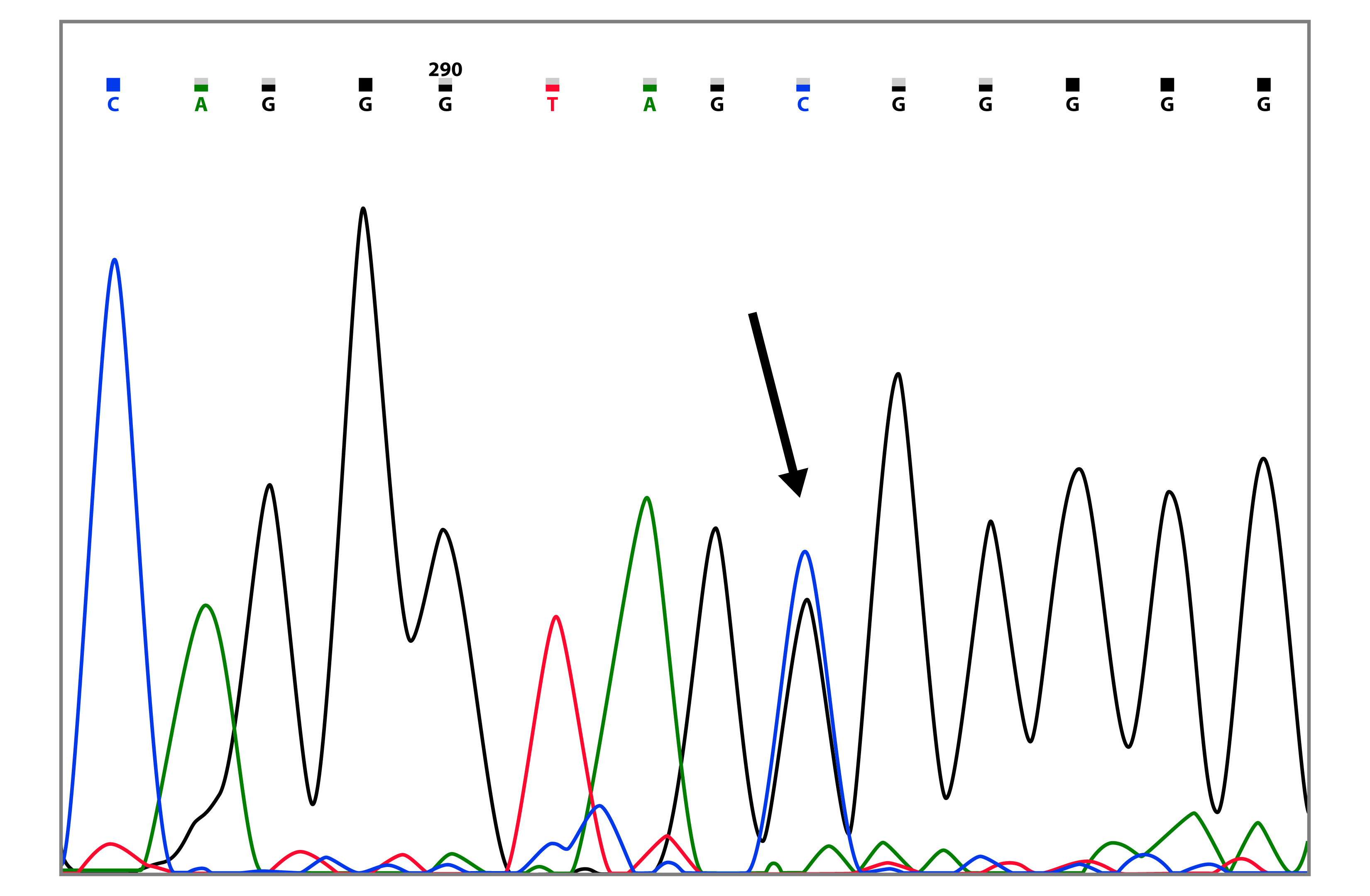
Figura 1 Cromatograma de secuenciación capilar que muestra la variante BRCA1 c.8112C>G (marcada con la flecha).
La mutación c.3083G>A (p.Arg1028His) fue encontrada en 2 familias, los casos índices fueron diagnosticados a los 45 y 49 años (Table 2 y Fig. 2). Esta mutación ha sido reportada 13 veces en la base de datos BIC, 6 de los reportes son de Latino-América. Adicionalmente, Arias-Blanco et al., reportaron esta mutación en la población colombiana 22.
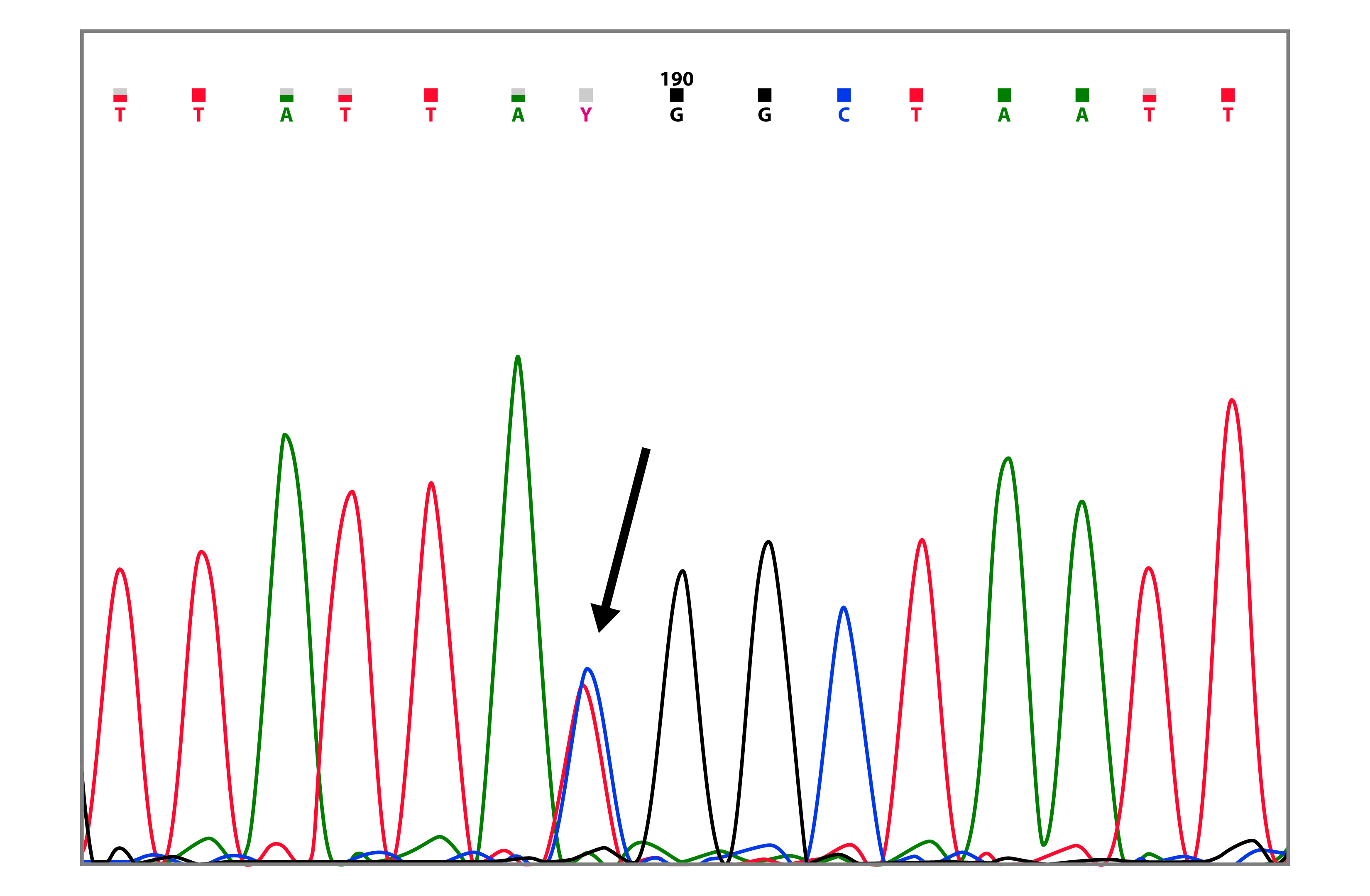
Figura 2 Cromatograma de secuenciación capilar que muestra la variante BRCA1 c.3083G>A (p.Arg1028His) (marcada con la flecha).
La mutación BRCA1 c.3119G>A (p.Ser1040Asn), fue encontrada en una familia con 2 casos de cáncer de mama (Table 2 y Fig. 3). En la base de datos BIC, ha sido reportada 68 veces, en Latino-América ha sido reportada en Brasil 23, Argentina 24 y Venezuela 25. Las mutaciones missense c.3119G>A y c.3083G>A, fueron siempre encontradas junto con el polimorfismo BRCA1 c.3113A>G.
Mutaciones BRCA2
En BRCA2 nosotros detectamos tres variantes missense de significado incierto
Una nueva mutación no descrita previamente fue encontrada en el gen BRCA2, la transición c.6427T>C que genera un cambio de serina por prolina (p.Ser2143Pro), esta variante no ha sido reportada en las base de datos BIC, ni en otras bases de datos consultadas. Debido a que es una mutación missense, la consideramos como una VUS. Esta mutación fue encontrada en una familia con dos casos de cáncer de mama y un caso de cáncer de próstata (Tabla 2). Ninguno de los controles presentó la mutación.
La mutación más prevalente fue c.865A>G, se encontró en seis (10.3%) de las familias analizadas, en cinco de ellas la edad de diagnóstico de los casos índice fue entre 41 a 51 años (Tabla 2). Esta mutación genera un cambio de asparagina por aspartato (p.Asn289Asp) en la proteína BRCA2 y ha sido reportada 13 veces en la base de datos BIC, donde es clasificada como una VUS; sin embargo, este es el primer reporte en Sur América. En este trabajo, está mutación fue encontrada en todos los casos acompañada por el polimorfismo BRCA2 c.8755-66T>C.
La mutación c.125A>G (p.Tyr42Cys) fue identificada en un paciente diagnosticado con cáncer de mama a los 39 años, cuya madre y dos tías también presentaron la neoplasia (Tabla 2 and Fig. 4). De los 144 reportes en la base de datos BIC, solo dos de ellos son de Latino-América, adicionalmente, Ruiz-Flores et al. (26, reportaron esta mutación en población mexicana.
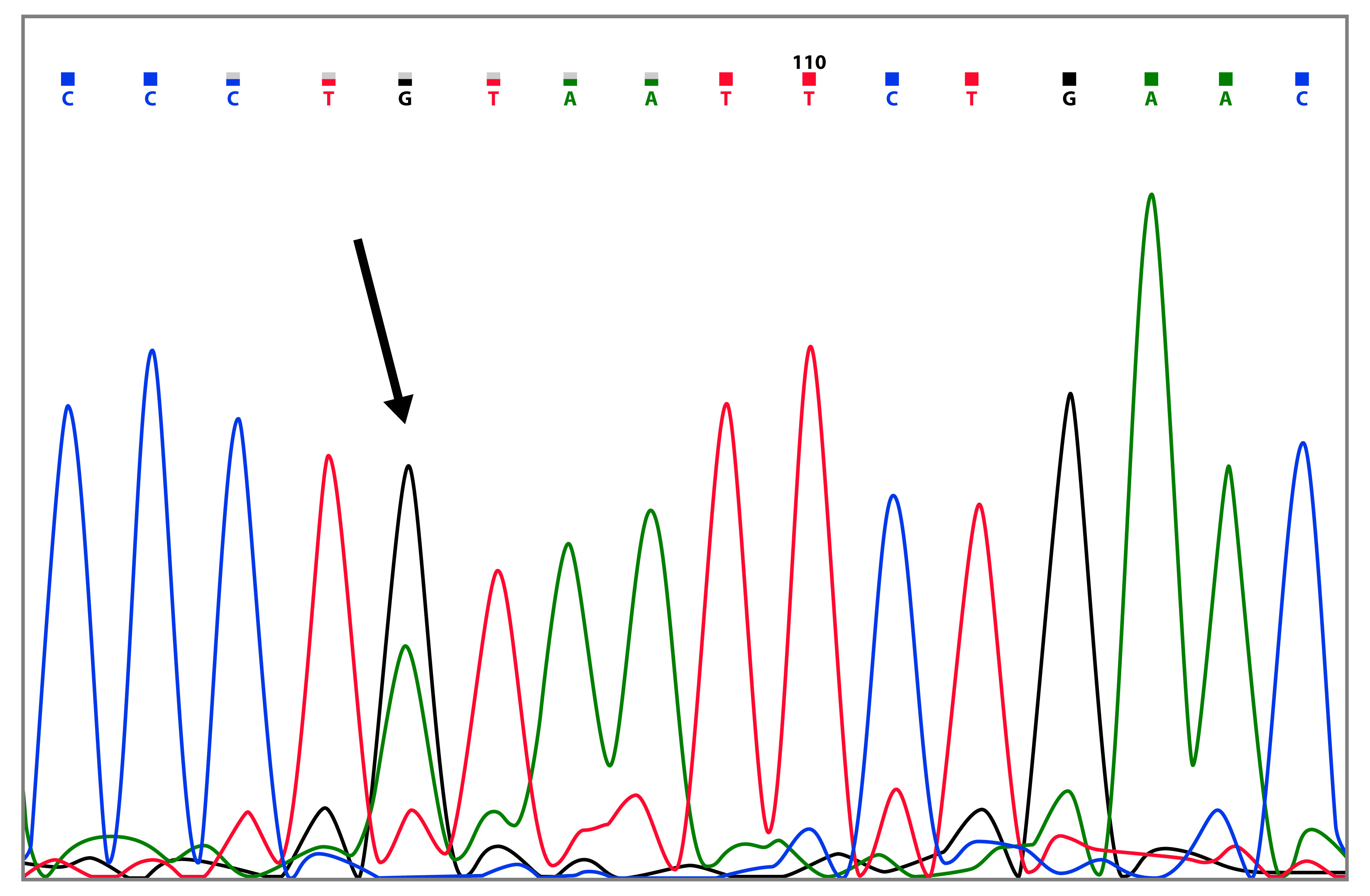
Figura 4 Cromatograma de secuenciación capilar que muestra la variante BRCA2 c.125A>G (p.Tyr42Cys) (marcada con la flecha).
Ninguna de las mutaciones identificadas fue encontrada en la muestra control. Todas las familias con mutaciones en los genes BRCA, también presentaron polimorfismos. Con respecto a los polimorfismos encontrados, es importante señalar que la mayoría de estos se detectaron tanto en pacientes como en controles, a excepción de c.426-89T> C y c.2971A> G, observados solo en pacientes (Tabla 3).
BRCA2 c.6841+80_6841+83delTTAA ha sido reportada como una VUS, 223 veces en la base de datos BIC. En este estudio, esta alteración fue encontrada en estado homocigoto, teniendo en cuenta la presencia de la mutación en doble dosis, y dado que, esta fue encontrada en pacientes y controles, concluimos que esta variante no tiene importancia clínica.
Análisis in silico
Para predecir la posible importancia clínica de VUS en BRCA1 y BRCA2, se utilizaron herramientas bioinformáticas. Estos análisis in silico fueron realizados para 4 variantes missense (2 en el gen BRCA1 y 3 en el gen BRCA2) y para una variante intrónica. A partir de estos resultados las mutaciones c.8112C>G, c.3119G>A (p.Ser1040Asn) en BRCA1 and c.865A>G (p.Asn289Asp), c.6427T>C (p.Ser2143Pro) en BRCA2, fueron clasificadas como probablemente deletéreas y las mutaciones BRCA1 c.3083G>A (p.Arg1028His) y BRCA2 c.125A>G (p.Tyr42Cys), como probablemente neutrales. Los detalles de los resultados de los análisis in silico son mostrados en (Tablas 4 and 5).
Tabla 4 Resultados de los análisis bioinformáticos para mutaciones en BRCA1 y BRCA2
| Gen | cDNA | Cambio Proteína | Align-GVGD | PANTHER (SubSPEC) | SIFT (Score) | PROVEAN (Score) | PolyPhen-2 (Prob.) | MutPred (Prob.) | SNP&GO (RI; EA) |
|---|---|---|---|---|---|---|---|---|---|
| BRCA1 | c.3083G>A | p.Arg1028His | C0 | Neutral (-2.90) | Tolerated (0.23) | Neutral (0.12) | Benign (0) | Neutral (0.391) | Disease (4; 72%) |
| c.3119G>A | p.Ser1040Asn | C0 | Damaging (-3.46) | Tolerated (1) | Neutral (-1.69) | Probably Damaging (0.974) | Neutral (0.123) | Disease (7; 85%) | |
| BRCA2 | c.125A>G | p.Tyr42Cys | C0 | NA | Tolerated (0.113) | Neutral (-1.32) | Benign (0.09) | Neutral (0.243) | Disease (6; 78%) |
| c.865A>G | p.Asn289Asp | C0 | NA | Damaging (0.008) | Neutral (-1.14) | Benign (0.055) | Neutral (0.165) | Disease (9; 96%) | |
| c.6427T>C | p.Ser2143Pro | C0 | Damaging (-3.38) | Damaging (0.015) | Neutral (-1.22) | Probably Damaging (0.999) | Neutral (0.179) | Disease (8; 88%) |
Tabla 5 Resultados del análisis bioinformático para la variante intrónica de BRCA1
| Gen | cDNA | 5' o 3' score modificación | (% variación) | |||
|---|---|---|---|---|---|---|
| BRCA1 | c.81-12C>G | c.81N | SSF: | 70.09 → | - | (-100.0%) |
| c.81N | MaxEnt: | 7.05 → | 5.37 | (-23.8%) | ||
| c.81N | NNSPLICE: | 0.52 → | - | (-100.0%) | ||
| c.81N | GS: | 4.46 → | 3.40 | (-23.8%) | ||
| c.81N | HSF: | 77.26 → | 75.36 | (-2.5%) | ||
Discusión
Un gran número de mutaciones han sido caracterizadas en los genes BRCA1 y BRCA2. A excepción de algunos grupos étnicos específicos, no existe una mutación predominante que explique la mayoría de los casos de cáncer de mama hereditario 27. A pesar de la alta prevalencia de cáncer de mama en Colombia, los datos para esta población son escasos 12,16. Una extrapolación de los resultados obtenidos en otras poblaciones es riesgosa debido a que la población colombiana es el resultado de un complejo proceso de mezcla 28.
En este estudio, examinamos 72 pacientes (58 familias) con cáncer de mama y/o ovario familiar de la región Suroccidental de Colombia, con el propósito de identificar el espectro mutacional de los genes BRCA1 y BRCA2. Para el gen BRCA1, encontramos 7 variantes de secuencia, de las cuales 3 habían sido clasificadas previamente como de significancia clínica desconocida y 4 como polimorfismos. Para el gen BRCA2, encontramos 12 variantes, de las cuales 2 habían sido clasificadas previamente como de significancia clínica desconocida y una mutación nueva la cual no ha sido descrita previamente, y 9 polimorfismos.
La mutación más recurrente en este estudio fue BRCA2 c.865A>G (p.Asn289Asp). El análisis con SIFT clasifica esta variante como probablemente dañina (score: 0.008), así como SNP&GO (RI: 9) (Tabla 4). c.865A>G está localizada en una región de activación de activación transcripcional. 29, y en la región de interacción con la proteína ALIX (aminoácidos 203300) 30, esta proteína es parte de la maquinaria que media el evento final de abscisión de membrana 31. El posible papel de BRCA2 en este proceso es controversial, pero es probable que reclute y entregue ALIX y otras proteínas, facilitando la finalización de la abscisión 30. Considerando esta evidencia y los resultados de nuestros análisis in silico, sugerimos que la mutación p.Asn289Asp es probablemente deletérea.
Interesantemente, p. Asn289Asp fue encontrada en todos los casos acompañada por el polimorfismo c.875566T>C, el cual ha sido asociado con un incremento en riesgo de 1.79 veces para cáncer de mama (95% CI: 1.16-2.78, p= 0.009) 32, por lo tanto, es probable que la convergencia de p.Asn289Asp con el polimorfismo c.875566T>C incremente aún más el riesgo de mal funcionamiento de la proteína.
Por lo tanto, se sugieren, estas variantes como marcadores a considerar en el diagnóstico de la predisposición al cáncer de mama y de ovario en pacientes colombianos, teniendo en cuenta la alta frecuencia en que se encontró, ya que el 10,3% de las familias exhibieron esta alteración
También en el gen BRCA2, reportamos una nueva mutación la c.6427T>C (p.Ser2143Pro), esta mutación fue encontrada en un caso índice que presento BC a los 40 años c.6427T>C (p.Ser2143Pro). Para esta mutación missense PANTHER (3.38), SIFT (0.015) y Polyphen2 (score: 0.999) (Tabla 4) predicen que esta variante es dañina, debido a que la Ser2143 está en una posición altamente conservada, adicionalmente, SNP&GO predice que es patológica (RI 8), indicando una alta probabilidad de que se afecte la estructura tridimensional y probablemente su función. Por lo tanto, proponemos BRCA2 c.6427T> C como una variante a ser considerada en el diagnóstico de predisposición a cáncer de mama, a la fecha, exclusiva de la población de Colombia, considerando que no ha sido reportada previamente.
La otra VUS encontrada en el gen BRCA2 fue la alteración c.125A>G (p.Tyr42Cys), clasificada como no patogénica por la mayoría de las herramientas in silico (Table 4). Esta variante ha sido clasificada como una variante neutral 33. Wu et al. (34 y Kuznetsov et al. (35, usando diferentes ensayos funcionales compararon las formas mutantes y silvestres de BRCA2 y concluyeron que la variante p.Tyr42Cys no afecta la función de BRCA2 Estos resultados son consistentes con los resultados obtenidos en nuestro análisis in silico, por lo que sugerimos que la variante BRCA2 p.Tyr42Cys es probablemente neutral.
Para la variante intrónica BRCA1 c.8112C>G la herramienta integrada Alamut predijo que el sitio natural de empalme en el exón 3 (c.81N) podría ser alterado, como se muestra en la Tabla 5, donde cuatro de las cinco herramientas de empalme mostraron un porcentaje de variación superior al 10%.
Para entender mejor el efecto de esta mutación en el sitio aceptor del exón 3, usamos el programa NNsplice, el programa predice que con la mutación el sitio aceptor normal podría ser alterado y un nuevo sitio alternativo posible podría ser usado, el programa otorga a este sitio una puntuación de 0,97, el cual corresponde al sitio aceptor del exón 4 (tag//ATTTTGC), esto llevaría a la pérdida del exón 3 y la generación de un codón de parada prematuro. Por lo tanto, sugerimos que la mutación BRCA1 c.8112C>G es probablemente deletérea debido a la generación de cambios significativos en la proteína BRCA1; esta mutación fue encontrada en tres hermanas, que pertenecen a una familia con una alta incidencia de BC (7 casos) y OC (4 casos).
Otra mutación missense identificada en BRCA1 fue c.3119G>A (p.Ser1040Asn), el algoritmo PolyPhen-2 predice este cambio como probablemente dañino (0,974), tal como PANTHER (3.46) y SNP&GO (7. 85%) (Tabla 4). La región en la cual está localizada esta variante, se ha sugerido es una región de interacción directa de BRCA1con la proteína RAD51, esta unión directa con RAD51 se ha propuesto que ocurre en los aminoácidos 758-1064 de BRCA1 36; sin embargo, a la fecha, todavía no hay consenso sobre si esta interacción es directa o indirecta 37, la controversia sobre el rol de esta variante necesita ser clarificada puesto que algunos autores la clasifican como neutral 38-43. Teniendo en cuenta la interacción con RAD51 y el hecho de que tres de los programas muestran esta posición como altamente conservada, sugerimos que esta variante es probablemente deletérea. Para la mutación missense BRCA1 c.3083G>A (p.Arg1028His), la mayoría de las herramientas in silico utilizadas predicen que esta variante es neutral o tolerada (Tabla 4), Solo el algoritmo SNP&GO predice esta variante como inductora de la enfermedad; sin embargo, el índice RI carece de suficiente confianza para la predicción.
Estos resultados son consistentes con estudios previos, entre ellos se encuentra un análisis de variación de secuencia interespecífica en el que la variante p.Arg1028His es clasificada como probablemente neutral o de poca importancia clínica 44. Teniendo en cuenta la evidencia previa, Podemos concluir que esta variante es probablemente neutral.
A pesar de que BRCA2 c.1114A>C (p.Asn372His) es considerado como un polimorfismo, esta variante está localizada dentro de una región que interactúa con el dominio acetil transferasa de la proteína factor asociado a P300/CBP (PCAF) 45 y ha sido asociada con un incremento en riesgo de 2.29 veces (95% CI: 1.164.49; p= 0.016).
En familias sin mutaciones en BRCA1/2 con alto riesgo de BC 32. Wen-Qiong et al. (46, realizaron un análisis agrupado, donde la variante BRCA2 p.Asn372His se asoció significativamente con un mayor riesgo de cáncer general (modelo dominante: OR= 1.07, 95% CI: 1.01-1.13; modelo recesivo: OR= 1.12, 95% CI: 1.02-1.23). Por lo tanto, sugerimos que esta variante podría ser considerada como un alelo de baja penetrancia para cáncer de mama.
Adicionalmente, el polimorfismo BRCA2 c.426-89T>C fue encontrado en 2 familias, en ambos casos acompañado por la mutación c.865A>G (p.Asn289Asp) y los polimorfismos 8755-66T>C and c.8851G>A. La base de datos BIC reportó esta variante 37 veces y es una variante alélica normal que no juega un papel directo en la generación de tumores en cáncer de mama 47.
Encontramos, en la región del pacifico colombiano, que 11 (18.9%) de las familias estudiadas presentaron mutaciones germinales en BRCA1 o BRCA2 y 9 de ellas fueron probablemente patogénicas. Por lo tanto, estas mutaciones explican el 15.5% de los casos de cáncer de mama/ovario en nuestra muestra. Adicionalmente, todos los individuos analizados tenían, al menos, un polimorfismo y ninguno de los controles presentó las mutaciones clasificadas como probablemente deletéreas en este estudio.
El barrido mutacional fue realizado usando una combinación de SSCP (polimorfismos conformacionales de cadena sencilla) y secuenciación. Teniendo en cuenta que el método SSCP tiene una sensibilidad del 94% 48 es posible que alrededor del 6% de las mutaciones en la región codificante de los genes BRCA1 y BRCA2 no hayan sido detectadas. Adicionalmente, este trabajo no incluyó la búsqueda de grandes deleciones ni reordenamientos genómicos que explican aproximadamente el 10% de todas las mutaciones BRCA112.
El espectro de mutaciones encontradas en este estudio es diferente de las reportadas en estudios previos, los cuales evaluaron pacientes de la región central de Colombia 12,22,49,50. Sugerimos que la razón principal de estas diferencias entre estudios es que la población colombiana es el resultado de un complejo proceso de mezcla entre Europeos, Africanos y Nativos-americanos, en diferentes grados dependiendo de la región 28.
Por lo tanto, la extrapolación de los resultados de una región al resto de la población colombiana resulta riesgosa, por ejemplo, la población del Suroccidente de Colombia tiene una dinámica poblacional específica y está es diferente a la de la región central 51.
Estos hallazgos indican que la población colombiana tiene un espectro heterogéneo de mutaciones BRCA, por lo cual es de suma importancia generar estudios donde estén representadas las diferentes regiones del país, con el fin de hacer un acercamiento real al espectro mutacional Colombiano.
Conclusiones
Encontramos que 11 de las familias estudiadas presentaron mutaciones germinales en BRCA1 o BRCA2 y 9 de ellas fueron probablemente deletéreas. Estas mutaciones explican el 15.5% de los casos de cáncer de mama/ovario de nuestra muestra.
La mutación más recurrente en este estudio BRCA2 c.865A>G, sugerimos que la mutación p.Asn289Asp es probablemente deletérea. También, en el gen BRCA2 reportamos una nueva mutación, c.6427T>C (p.Ser2143Pro), proponemos esta mutación como una variante a ser considerada como exclusiva de la población colombiana.
Sugerimos que la variante intrónica BRCA1 c.81 12C>G es probablemente deletérea; esta mutación fue encontrada en una familia con alta incidencia de BC y OC. En el gen BRCA1, también reportamos la variante c.3119G>A (p.Ser1040Asn) y sugerimos que es probablemente deletérea.
Consideramos entonces, que actualmente, el uso de pruebas genéticas basadas en paneles mutacionales para BRCA1 y BRCA2 en pacientes colombianos, no sería suficientemente informativo, debido al espectro mutacional diverso de esta población, el cual es evidenciado en este trabajo. Por lo tanto, el barrido mutacional de toda la región codificante de los genes BRCA1 y BRCA2 es necesario para el diagnóstico genético molecular de pacientes colombianas con alto riesgo de cáncer de mama.
Agradecimientos
Los autores agradecen a las familias que participaron en los estudios de investigación descritos en este artículo. También reconocemos a las instituciones participantes en el estudio, Hospital Universitario del Valle, Fundación Fondo de Droga Contra el Cáncer (FUNCANCER), Clínica Universitaria Rafael Uribe Uribe de Cali y Hospital Universitario “San José” de Popayán.
REFERENCIAS
1. Ferlay J, Soerjomataram I, Ervik M, Dikshit R, Eser S, Mathers C, Rebelo M et al. Cancer incidence and mortality worldwide: Sources, methods and major patterns in GLOBOCAN 2012. Int J Cancer. 2015; 136(5):E359-86. DOI: 10.1002/ijc.29210. [ Links ]
2. Bravo LE, Collazos T, Collazos P, García LS, Correa P. Trends of cancer incidence and mortality in Cali, Colombia 50 years experience. Colomb Med (Col). 2012; 43(4):246-55. DOI: 10.25100/cm.v43i4.1266. [ Links ]
3. Miki Y, Swensen J, Shattuck-Eidens D, Futreal PA, Harshman K, Tavtigian S, et al. A strong candidate for the breast and ovarian cancer susceptibility gene BRCA1. Science. 1994; 266(5182):66-71. DOI: 10.1126/science.7545954. [ Links ]
4. Wooster R, Bignell G, Lancaster J, Swift S, Seal S, Mangion J, et al. Identification of the breast cancer susceptibility gene BRCA2. Nature. 1995; 378(6559):789-92. DOI: 10.1038/378789a0. [ Links ]
5. Barnes DR, Antoniou AC. Unravelling modifiers of breast and ovarian cancer risk for BRCA1 and BRCA2 mutation carriers: update on genetic modifiers. J Intern Med. 2012; 271:331-43. DOI: 10.1111/j.1365-2796.2011.02502.x. [ Links ]
6. Thacker J. The RAD51 gene family, genetic instability and cancer. Cancer Lett. 2005; 219(2):125-35. DOI: 10.1016/j.canlet.2004.08.018. [ Links ]
7. Levy-Lahad E, Lahad A, Eisenberg SA. A Single nucleotide polymorphism in the RAD51 gene modifies cancer risk in BRCA2 but not BRCA1 carriers. Proc Natl Acad Sci USA. 2001; 98(6):3232-6. DOI: 10.1073/pnas.051624098. [ Links ]
8. Didraga MA, van Beers EH, Joosse SA, Brandwijk KI, Oldenburg RA, Wessels LF, et al. A non-BRCA1/2 hereditary breast cancer sub-group defined by aCGH profiling of genetically related patients. Breast Cancer Res Treat. 2011;130(2):425-36. DOI: 10.1007/s10549-011-1357-x [ Links ]
9. Balmaña J, Díez O, Rubio IT, Cardoso F. BRCA in breast cancer: ESMO Clinical Practice Guidelines. Ann Oncol. 2011;22(Suppl 6): vi31-vi34. DOI: 10.1093/annonc/mdr373 [ Links ]
10. Ashton-prolla P, Vargas FR. Prevalence and impact of founder mutations in hereditary breast cancer in Latin America. Genet Mol Biol. 2014;37(1 Suppl):234-40. DOI: 10.1590/S1415-47572014000200009 [ Links ]
11. Rojas W, Parra MV, Campo O, Caro MA, Lopera JG, Arias W, et al. Genetic make up and structure of Colombian populations by means of uniparental and biparental DNA markers. Am J Phys Anthropol. 2010;143(1):13-20. DOI: 10.1002/ajpa.21270 [ Links ]
12. Torres D, Rashid MU, Gil F, Umana A, Ramelli G, Robledo JF, et al. High proportion of BRCA1/2 founder mutations in Hispanic breast/ovarian cancer families from Colombia. Breast Cancer Res Treat. 2007;103(2):225-32. DOI: 10.1007/s10549-006-9370-1 [ Links ]
13. Sanabria MC, Muñoz G, Vargas CI. Análisis de las mutaciones más frecuentes del gen BRCA1 (185delAG y 5382insC) en mujeres con cáncer de mama en Bucaramanga, Colombia. Biomédica. 2009;29:61-72. DOI: 10.7705/biomedica.v29i1.42 [ Links ]
14. Torres D, Umaña A, Robledo JF, Caicedo JJ, Quintero E, Orozco A, et al. Estudio de factores genéticos para cáncer de mama en Colombia. Universitas Medica. 2009;50(3):297-301. [ Links ]
15. Rodríguez AO, Llacuachaqui M, Pardo GG, Royer R, Larson G, Weitzel JN, et al. BRCA1 and BRCA2 mutations among ovarian cancer patients from Colombia. Gynecol Oncol. 2012;124:236-43. DOI: 10.1016/j.ygyno.2011.10.027. [ Links ]
16. Londoño-Hernández JE, Llacuachaqui M, Palacio GV, Figueroa JD, Madrid J, Lema M, et al. Prevalence of BRCA1 and BRCA2 mutations in unselected breast cancer patients from medellín, Colombia. Hered Cancer Clin Pract. 2014;12:11-15. DOI: 10.1186/1897-4287-12-11. [ Links ]
17. Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Res. 1988;16(3):1215. DOI: 10.1093/nar/16.3.1215. [ Links ]
18. Barker DF. Direct genomic multiplex PCR for BRCA1 and application to mutation detection by single-strand conformation and heteroduplex analysis. Hum Mutat. 2000;16(4):334-44. DOI: 10.1002/1098-1004(200010)16:4<334::AID-HUMU6>3.0.CO;2-6. [ Links ]
19. Orita M, Iwahana H, Kanazawat H, Hayashi K, Sekiya T. Detection of polymorphisms of human DNA by gel electrophoresis as single-strand conformation polymorphisms. Proc Natl Acad Sci USA. 1989;86:2766-70. DOI: 10.1073/pnas.86.8.2766. [ Links ]
20. Naranjo J, Posso A, Cárdenas H, Muñoz JE. Detección de variantes alélicas de la kappa-caseína en bovinos Hartón del Valle. Rev Acta Agronóm. 2007;56(1):43-8. [ Links ]
21. Théry JC, Krieger S, Gaildrat P, Révillion F, Buisine M-P, Killian A, et al. Contribution of bioinformatics predictions and functional splicing assays to the interpretation of unclassified variants of the BRCA genes. Eur J Hum Genet. 2011;19(10):1052-8. DOI :10.1038/ejhg.2011.100. [ Links ]
22. Arias-Blanco JF, Ospino-Durán EA, Restrepo-Fernández CM, Guzmán-AbiSaab L, Fonseca-Mendoza DJ, Ángel-Guevara DI, et al. Frecuencia de mutación y de variantes de secuencia para los genes BRCA1 y BRCA2 en una muestra de mujeres colombianas con sospecha de síndrome de cáncer de mama hereditario: serie de casos. Rev Colomb Obstet Ginecol. 2015;66(4):287-96. DOI: 10.18597/rcog.294. [ Links ]
23. de Oliveira ES, Soares BL, Lemos S, Rosa RCA, Rodrigues AN, Barbosa LA, dos Santos LL. Screening of the BRCA1 gene in Brazilian patients with breast and/or ovarian cancer via high-resolution melting reaction analysis. Fam Cancer. 2016; 15(2): 173-181. DOI: 10.1007/s10689-015-9858-0 [ Links ]
24. Solano A, Aceto G, Delettieres D, Veschi S, Neuman M, Alonso E, et al. BRCA1 and BRCA2 analysis of Argentinean breast/ovarian cancer patients selected for age and family history highlights a role for novel mutations of putative south-American origin. Springerplus. 2012;1:20. DOI: 10.1186/2193-1801-1-20. [ Links ]
25. Lara K, Consigliere N, Pérez J, Porco A. BRCA1 and BRCA2 mutations in breast cancer patients from Venezuela. Biol Res. 2012;45:117-30. DOI: 10.4067/S0716-97602012000200003 [ Links ]
26. Ruiz-Flores P, Sinilnikova OM, Badzioch M, Calderon-Garcidueñas a L, Chopin S, Fabrice O, et al. BRCA1 and BRCA2 mutation analysis of early-onset and familial breast cancer cases in Mexico. Hum Mutat. 2002;20(6):474-5. DOI: 10.1002/humu.9084 [ Links ]
27. Dufloth RM, Carvalho S, Heinrich J K, Shinzato J Y, dos Santos CC, Zeferino LC,et al. Analysis of BRCA1 and BRCA2 mutations in Brazilian breast cancer patients with positive family history. Sao Paulo Med J. 2005;123(4):192-7. DOI: 10.1590/S1516-31802005000400007. [ Links ]
28. Salas A, Acosta A, Alvarez-Iglesias V, Cerezo M, Phillips C, Lareu M V, et al. The mtDNA ancestry of admixed Colombian populations. Am J Hum Biol 2008;20(5): 584-91. DOI: 10.1002/ajhb.20783. [ Links ]
29. Tommasi S, Pilato B, Pinto R, Monaco A, Bruno M, Campana M, et al. Molecular and in silico analysis of BRCA1 and BRCA2 variants. Mutat Res Mol Mech. 2008;644(1-2):64-70. DOI: 10.1016/j.mrfmmm.2008.07.005. [ Links ]
30. Mondal G, Rowley M, Guidugli L, Wu J, Pankratz VS, Couch FJ. BRCA2 localization to the midbody by filamin a regulates CEP55 signaling and completion of cytokinesis. Dev Cell. 2012;23:137-52. DOI: 10.1016/j.devcel.2012.05.008. [ Links ]
31. Agromayor M, Martin-Serrano J. Knowing when to cut and run: mechanisms that control cytokinetic abscission. Trends Cell Biol. 2013;23(9):433-41. DOI: 10.1016/j.tcb.2013.04.006. [ Links ]
32. Seymour I J, Casadei S, Zampiga V, Rosato S, Danesi R, Falcini F, et al. Disease family history and modification of breast cancer risk in common BRCA2 variants. Oncol Rep. 2008;19:783-6. DOI: 10.3892/or.19.3.783. [ Links ]
33. Goldgar D E, Easton D F, Deffenbaugh A M, Monteiro ANA, Tavtigian S V, Couch FJ, et al. Integrated evaluation of DNA sequence variants of unknown clinical significance. Am J Hum Genet. 2004;75:535-44. DOI: 10.1086/424388 [ Links ]
34. Wu K, Hinson S R, Ohashi A, Farrugia D, Wendt P, Tavtigian S V, et al. functional evaluation and cancer risk assessment of BRCA2 unclassified variants. Cancer Res. 2005;65(2):417-426. [ Links ]
35. Kuznetsov S G, Liu P, Sharan S K. Mouse embryonic stem cell-based functional assay to evaluate mutations in BRCA2. Nat Med. 2008;14(8):875-81. DOI: 10.1038/nm.1719 [ Links ]
36. Scully R, Chen J, Plug A, Xiao Y, Weaver D, Feunteun J, et al. Association of BRCA1 with Rad51 in mitotic and meiotic cells. Cell. 1997;88:265-75. DOI: 10.1016/S0092-8674(00)81847-4. [ Links ]
37. Caestecker KW, Van de Walle GR. The role of BRCA1 in DNA double-strand repair: Past and present. Exp Cell Res. 2013;319(5):575-87. DOI: 10.1016/j.yexcr.2012.11.013. [ Links ]
38. Durocher F, Shattuck-eidens D, Mcclure M, Labrie F, Skolnick MH, Goldgar DE, et al. Comparison of BRCA1 polymorphisms, rare sequence variants and/or missense mutations in unaffected and breast/ovarian cancer populations. Hum Mol Genet. 1996;5(6):835-42. DOI: 10.1093/hmg/5.6.835. [ Links ]
39. Arnold N, Peper H, Bandick K, Kreikemeier M, Karow D, Teegen B, et al. Establishing a control population to screen for the occurrence of nineteen unclassified variants in the BRCA1 gene by denaturing high-performance liquid chromatography. J Chromatogr B. 2002;782(1-2):99-104. DOI: 10.1016/S1570-0232(02)00696-7. [ Links ]
40. De La Hoya M, Diaz-Rubio E, Calde T. Denaturing gradient gel electrophoresis-based analysis of loss of heterozygosity distinguishes nonobvious, deleterious BRCA1 variants from nonpathogenic polymorphisms. Clin Chem. 1999;45(11):2028-30. [ Links ]
41. Infante M, Duran M, Esteban-Cardeñosa E, Miner C, Velasco E. High proportion of novel mutations of BRCA1 and BRCA2 in breast/ovarian cancer patients from Castilla-León (central Spain). J Hum Genet. 2006;51:611-617. DOI: 10.1007/s10038-006-0404-7. [ Links ]
42. Claes K, Poppe B, Coene I, Paepe AD, Messiaen L. BRCA1 and BRCA2 germline mutation spectrum and frequencies in Belgian breast/ovarian cancer families. Br J Cancer. 2004;90(6):1244-51. DOI: 10.1038/sj.bjc.6601656. [ Links ]
43. Tavtigian S V, Deffenbaugh A M, Yin L, Judkins T, Scholl T, Samollow PB, et al. Comprehensive statistical study of 452 BRCA1 missense substitutions with classification of eight recurrent substitutions as neutral. J Med Genet. 2006;43(4):295-305. DOI: 10.1136/jmg.2005.033878. [ Links ]
44. Abkevich V, Zharkikh A, Deffenbaugh A M, Frank D, Chen Y, Shattuck D, et al. Analysis of missense variation in human BRCA1 in the context of interspecific sequence variation. J Med Genet 2004;41(7):492-507. DOI: 10.1136/jmg.2003.015867. [ Links ]
45. Teare MD, Cox A, Shorto J, Anderson C, Bishop DT, Cannings C. Heterozygote excess is repeatedly observed in females at the BRCA2 locus N372H. J Med Genet. 2004;41(7):523-28. DOI: 10.1136/jmg.2003.017293. [ Links ]
46. Wen-Qiong X, Yong-Qiao H, Jin-Hong Z, Jian-Qun M, Jing H, Wei-Hua J. Association of BRCA2 N372H polymorphism with cancer susceptibility: a comprehensive review and meta-analysis. Sci Rep. 2014;4:6791. DOI: 10.1038/srep06791. [ Links ]
47. Kawahara M, Sakayori M, Shiraishi K, Nomizu T, Takeda M, Abe R, et al. Identification and evaluation of 55 genetic variations in the BRCA1 and the BRCA2 genes of patients from 50 Japanese breast cancer families. J Hum Genet. 2004;49:391-5. DOI: 10.1007/s10038-004-0160-5. [ Links ]
48. Gross E, Arnold N, Goette J, Schwarz-Boeger U, Kiechle M. A comparison of BRCA1 mutation analysis by direct sequencing, SSCP and DHPLC. Hum Genet. 1999;105:72-8. DOI: 10.1007/s004399900092. [ Links ]
49. Briceño-Balcazar I, Gómez-Gutiérrez A, Díaz-Dussán NA, Noguera-Santamaría MC, Díaz-Rincón D, Casas-Gómez MC. Mutational spectrum in breast cancer associated BRCA1 and BRCA2 genes in Colombia. Colomb Med (Col). 2017;48(2):58-63. DOI: 10.25100/cm.v48i2.1867. [ Links ]
50. Cock-Rada AM, Ossa CA, Garcia HI, Gomez LR. A multi-gene panel study in hereditary breast and ovarian cancer in Colombia. Fam Cancer. 2018;17(1):23-30. DOI: 10.1007/s10689-017-0004-z. [ Links ]
51. Avila S, Briceño I, Gomez A. Genetic population analysis of 17 Y-chromosomal STRs in three states (Valle del Cauca, Cauca and Nariño) from Southwestern Colombia. J Forensic Leg Med. 2009;16(4):204-11. DOI: 10.1016/j.jflm.2008.12.002. [ Links ]
Recibido: 01 de Julio de 2016; Revisado: 20 de Junio de 2018; Aprobado: 25 de Enero de 2019











 texto en
texto en