Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Colombia Médica
versión On-line ISSN 1657-9534
Colomb. Med. vol.50 no.3 Cali jul./set. 2019
https://doi.org/10.25100/cm.v50i3.3969
Original article
Características clínicas, inmunológicas y genéticas de pacientes con el fenotipo asociado a deficiencia LRBA en Colombia
1 Universidad de Antioquia UdeA, Facultad de Medicina, Grupo de Inmunodeficiencias Primarias, Medellin, Colombia
2 Universidad de Antioquia, Grupo de Inmunología Celular e Inmunogenética, Medellin, Colombia
3 Universidad de Antioquia, Programa de Posgrado, Reumatología Pediátrica, Medellin, Colombia
Antecedentes:
la deficiencia de LRBA (del inglés, LPS-responsive beige -like anchor protein) es una inmunodeficiencia primaria causada por la pérdida de la expresión de la proteína LRBA, debido a mutaciones bialélicas en el gen LRBA. Los pacientes con deficiencia de LRBA exhiben un síndrome clínicamente heterogéneo. La principal complicación clínica de la deficiencia de LRBA es la desregulación inmune. Además, la hipogammaglobulinemia se encuentra en más de la mitad de los pacientes con deficiencia de LRBA. Hasta la fecha, no se han reportado pacientes con esta afección en Colombia
Objetivo:
Evaluar la expresión de la proteína LRBA en pacientes de Colombia con fenotipo clínico asociado a deficiencia de LRBA
Métodos:
En el presente estudio se evaluó la expresión de LRBA en pacientes de Colombia con fenotipo clínico asociado a deficiencia de LRBA. Después de eso, se evaluaron las características clínicas, inmunológicas y las posibles variantes genéticas en LRBA o en otros genes asociadados con el sistema inmune en pacientes que exhiben una disminución de la expresión de la proteína.
Resultados:
En total, se evaluaron 112 pacientes con diferentes manifestaciones clínicas asociadas al fenotipo clínico LRBA. La expresión de LRBA varía mucho entre diferentes donantes sanos y pacientes. A pesar de la gran variabilidad en la expresión de LRBA, se observaron seis pacientes con una disminución en la expresión de la proteína LRBA. Sin embargo, no se encontraron variantes bialélicas patógenas o posibles patógenas en LRBA, o en genes relacionados con el sistema inmune.
Conclusión:
La expresión de LRBA varía mucho entre diferentes donantes sanos y pacientes. Se observó reducción de la expresión de LRBA en 6 pacientes sin mutaciones homocigotas en LRBA o en genes asociados. Estos resultados sugieren los otros mecanismos genéticos, por ejemplo epigenéticos o ambientales, que podrían estar regulados por la expresión de LRBA
Palabras clave: LRBA; Inmunodeficiencia variable común; Gamaglobulinemia asociada-X; hipogamaglobulinemia; deficiencia de inmunoglobulina A; enteropatía; trombocitopenia purpura idiopática; lupus eritematoso sistémico; secuenciación total del exoma
Background:
LPS-responsive beige -like anchor protein (LRBA) deficiency is a primary immunodeficiency disease caused by loss of LRBA protein expression, due to biallelic mutations in LRBA gene. LRBA deficiency patients exhibit a clinically heterogeneous syndrome. The main clinical complication of LRBA deficiency is immune dysregulation. Furthermore, hypogammaglobulinemia is found in more than half of patients with LRBA-deficiency. To date, no patients with this condition have been reported in Colombia
Objective:
To evaluate the expression of the LRBA protein in patients from Colombia with clinical phenotype associated to LRBA-deficiency.
Methods:
In the present study the LRBA-expression in patients from Colombia with clinical phenotype associated to LRBA-deficiency was evaluated. After then, the clinical, the immunological characteristics and the possible genetic variants in LRBA or other genes associated with the immune system in patients that exhibit decrease protein expression was evaluated.
Results:
In total, 112 patients with different clinical manifestations associated to the clinical LRBA phenotype were evaluated. The LRBA expression varies greatly between different healthy donors and patients. Despite the great variability in the LRBA expression, six patients with a decrease in LRBA protein expression were observed. However, no pathogenic or possible pathogenic biallelic variants in LRBA, or in genes related with the immune system were found.
Conclusion:
LRBA expression varies greatly between different healthy donors and patients. Reduction LRBA-expression in 6 patients without homozygous mutations in LRBA or in associated genes with the immune system was observed. These results suggest the other genetic, epigenetic or environmental mechanisms, that might be regulated the LRBA-expression.
keywords: LRBA; Common variable immunodeficiency; X-linked agammaglobulinemia; Hypogammaglobulinemia; Immunoglobulin A deficiency; Enteropathy; Idiopathic Thrombocytopenic Purpura; Systemic Lupus Erythematosus; Whole Exome Sequencing
Contribución del estudio
| 1) Por que se hizo este estudio? |
| Los pacientes con deficiencia de LRBA exhiben un síndrome heterogéneo clínico, donde la principal complicación clínica es la desregulación inmune. La enteropatía, la anemia hemolítica autoinmune y púrpura trombocitopénica idiopática son las manifestaciones más comunes. Se encuentra en más de la mitad de los pacientes niveles reducidos de IgM e IgG o de IgA e IgG, de IgA solamente o de IgG. Hasta la fecha, no se han reportado pacientes con esta afección en Colombia. Se evaluaron mutaciones en LRBA en pacientes de Colombia con predominantemente deficiencias de anticuerpos como CVID, XLA, IHG y SIgAD, manifestaciones autoinmunes como ITP y SLE y/o enteropatías. |
| 2) Cuales fueron los resultados mas relevantes? |
| Seis pacientes (n= 112) mostraron disminución en la expresión de la proteína LRBA. Una reducción en la expresión de LRBA en dos pacientes con CVID, dos con XLA y en dos con IHG. Estos pacientes exhiben diferentes características fenotípicas e inmunológicas. Todos los pacientes presentaron una reducción en IgG e infecciones recurrentes como otitis y sinusitis, y cinco presentaron gastropatía. No encontramos variantes patogénicas o potencialmente patogénicas ni en LRBA ni en otros genes relacionados con el sistema inmune. |
| 3) Que significan los hallazgos? |
| La expresión de LRBA varía mucho en las CMSP entre diferentes DS y pacientes. Seis pacientes exhibieron una disminución de la expresión de esta proteína. No se detectaron genes asociados con la producción defectuosa de LRBA por estas células en estos pacientes. Las mutaciones homocigotas en LRBA causan deficiencia de LRBA que afecta la expresión de la proteína. Sin embargo, la reducción de la expresión de LRBA en pacientes sin mutaciones homocigotas o en genes asociados estaban presentes. Estos resultados sugieren los otros mecanismos genéticos, epigenéticos o ambientales, podrían estar regulando la expresión de LRBA. |
Introducción
Las inmunodeficiencias primarias (IDP) son un grupo heterogéneo de trastornos genéticos hereditarios que causan alteraciones cuantitativas y/o funcionales en diferentes mecanismos involucrados en la respuesta inmune 1. Las IDP se consideran enfermedades "raras", pero la mayoría de las estimaciones de prevalencia se han basado en poblaciones seleccionadas (por ejemplo, clínicas especializadas, registros de enfermedades) 2. Sin embargo, estas frecuencias son mucho más altas que las estimaciones reportadas basadas en los datos del registro, lo que sugiere sesgos de subregistro y de verificación. Por lo tanto, los datos obtenidos de los registros no proporcionan una cobertura adecuada para estimar la prevalencia o la distribución por edad de IDP 2.
La deficiencia de LRBA (del inglés: Lipopolysaccharide (LPS)-Responsive and Beige-like Anchor protein) es una IDP autosómica recesiva causada por la pérdida de expresión de la proteína LRBA debido a mutaciones homocigotas o heterocigotas compuestas en el gen LRBA3. La deficiencia de LRBA produce un síndrome clínicamente variable con un amplio espectro de manifestaciones clínicas. La principal complicación clínica de la deficiencia de LRBA es la disregulación inmune, seguida de organomegalia e infecciones recurrentes. Entre los pacientes con disregulación inmune, la enteropatía es la manifestación clínica más común, seguida de anemia hemolítica autoinmune y púrpura trombocitopénica idiopática 4. Además, la hipogammaglobulinemia se encuentra en el 57% de los pacientes. Entre los pacientes con hipogammaglobulinemia, el 17% de los pacientes tienen reducción de los niveles de IgM e IgG, el 17% presentan reducción los niveles de IgA e IgG, y el 34% muestran títulos bajos de los tres isotipos de inmunoglobulinas. Además, el 16% de los pacientes tenían solamente niveles reducidos de IgA y el 16% tenían niveles bajos de IgG4. Hasta la fecha, no se han reportado pacientes con esta afección en Colombia.
El objetivo del presente estudio fue evaluar la expresión de la proteína LRBA en pacientes colombianos con fenotipo clínico asociado a la deficiencia de LRBA. Posteriormente, evaluar las características clínicas, inmunológicas y las posibles variantes genéticas involucradas en la regulación de la expresión de esta proteína u otra asociada con el sistema inmune en pacientes que exhiben disminución de la expresión de LRBA.
Materiales y Métodos
El resumen de la metodología se muestra en la Figura S1
Sujetos humanos
Pacientes remitidos al Grupo de Inmunodeficiencias Primarias de la Universidad de Antioquia (Medellín, Colombia) que presentan alguna manifestación clínica asociado con el fenotipo de la deficiencia de LRBA (Disregulación inmune: enteropatía, anemia hemolítica autoinmune, púrpura trombocitopénica idiopática, enfermedad pulmonar intersticial linfocítica granulomatosa, diabetes melitus tipo I y/o hepatitis autoinmune crónica; organomegalia: esplenomegalia, linfadenopatía y / o hepatomegalia; hipogammaglobulinemia: niveles bajos de IgM, IgG o IgA de título, o los tres isotipos de inmunoglobulina reducidos) se reclutaron para evaluar la expresión de LRBA en células mononucleares de sangre periférica (CMSP) Se excluyeron pacientes con diagnóstico incompleto según los criterios del especialista. También se incluyeron controles sanos apareados por edad y género con los pacientes, padres y hermanos y se analizaron muestras en paralelo con las de los pacientes, esto se realizó para garantizar que la variabilidad de la expresión no fuera inherente a la edad o el sexo y para determinar si otro miembros de la familia también tenían afectada la expresión de la proteína. El estudio fue aprobado por el Comité de Ética Institucional de la Sede de Investigaciones Universitarias (SIU), Universidad de Antioquia, UdeA. Se obtuvo el consentimiento informado por escrito de todos los participantes involucrados en este estudio.
Evaluación de la expresión LRBA
Análisis de Western Blot (WB)
Las CMSP se obtuvieron de sangre periférica aislada por centrifugación en gradiente de densidad usando Ficoll-Hypaque (Sigma-Aldrich, St Louis, MO). Las CMSP se resuspendieron en medio RPMI 1640 con L-glutamina y bicarbonato de sodio (Sigma-Aldrich), suplementado con 10% de suero fetal bovino inactivado por calor (SFB, Gibco, MA, EE. UU.), penicilina/estreptomicina al 2% (Lonza, Walkersville, MD) (RPMI completo) y estimulado con fitohemaglutinina A (PHA, 10 mg/mL, Sigma-Aldrich). Las células se cosecharon y se lisaron usando buffer RIPA (Cell Signaling, MA) que contiene un cóctel inhibidor de proteasa (Amresco, GA) y fluoruro de fenilmetanosulfonia 0,1 M (PMSF, Sigma-Aldrich). Los niveles de proteína se midieron usando el Ensayo de proteína Quick Start® Bradford (Bio-Rad, CA) siguiendo las instrucciones del fabricante. Los lisados se resolvieron mediante SDS-PAGE (Poliacrilamida de Amresco, gel de gradiente al 10%). Las proteínas se transfirieron a membranas de fluoruro de polivinilideno (PVDF) (Bio-Rad) y se incubaron con anticuerpo policlonal de conejo anti-LRBA humano (HPA023597, Sigma-Aldrich), anti-GAPDH (Sigma-Aldrich) y anticuerpo anti-IgG-peroxidasa anti-conejo como secundario anticuerpo (Sigma-Aldrich). Las señales fueron detectadas por el Western Blotting Luminol Reagent (Santa Cruz Biotechnology, TX) usando el sistema de imágenes quimioluminiscentes y fluorescentes multicolores Syngene G: Box XR5 (Integrated Scientific Solutions, San Diego, CA). La cuantificación de la banda se realizó utilizando el software ImageJ 1.48v (National Institutes of Health, Bethesda, MA). Como control de la reducción de la expresión de LRBA, se usó el lisado celular del paciente CVID022, ya que exhibía una disminución en la expresión del primer WB que realizamos.
Análisis de citometría de flujo (FACS- Fluorescence-activated cell sorting)
Las células de sangre periférica ex vivo y las CMSP estimuladas sin PHA (10 µg/mL) se tiñeron intracelularmente con el anticuerpo policlonal anti-LRBA (HPA019366, Sigma-Aldrich) usando anti-conejo PE-F (ab) `2 Donkey IgG (Sigma -Aldrich) como anticuerpo secundario. La tinción celular intracelular se realizó con Cytofix/Cytoperm y las soluciones Perm/Wash de Becton Dickinson (BD) siguiendo las instrucciones del fabricante. Las células se resuspendieron en Dulbecco PBS 1X (dPBS Sigma-Aldrich) antes del análisis. FACS se realizó con Fortessa II (BD, Franklin Lakes, NJ) y se analizó con el software FlowJo V9.9.5 (Tree Star, Inc. Ashland, OR).
Además, para evaluar la especificidad de tinción de LRBA del anticuerpo policlonal por FACS, el péptido reconocido por del anticuerpo policlonal anti-LRBA (HPA019366, Sigma-Aldrich) fue sintetizado por ProteoGenix (Schiltigheim, Francia) y luego diluido en dPBS (Sigma-Aldrich ) a una concentración final de 4 mg/mL. Luego, se incubaron diferentes concentraciones de los péptidos (2, 0.2, 0.02, 0.002 mg/mL diluidos en dPBS) con el anticuerpo anti-LRBA, y posteriormente, se evaluó en células ex vivo CD3+ provenientes de CMSP la expresión intracelular de LRBA, luego de la adición de esta mezcla de anticuerpo anti-LRBA/peptido.
Estudios inmunológicos
La información clínica y el análisis inmunológico (niveles de inmunoglobulina, números de subpoblación de linfocitos en sangre periférica) de pacientes se obtuvieron de los registros del servicio de diagnóstico del Grupo de Inmunodeficiencias Primarias.
Secuenciación del exoma completo (WES- Whole-Exome Sequencing)
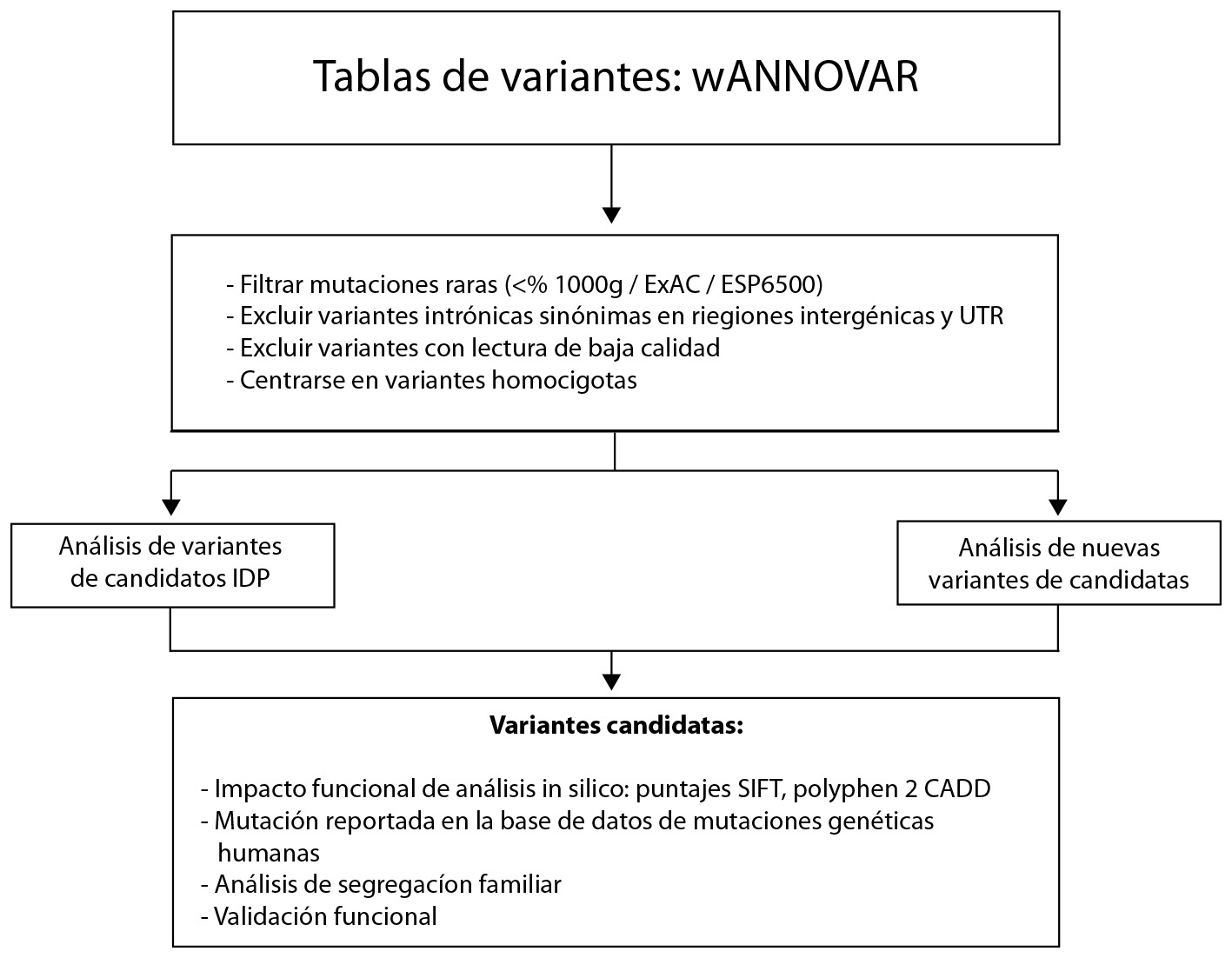
El ADN genómico (ADNg) se aisló de sanfre periferica obtenida de pacientes y sus padres utilizando el kit de aislamiento de ADN de Puregene ™ (Gentra Systems, Minneapolis, MN) siguiendo las instrucciones del fabricante. Los ADNg fueron sometidos a WES por Macrogen® (Seúl, Corea), utilizando el sistema de secuenciación Illumina HiSeq X 10. El ADNg se sometió a una preparación de biblioteca, que introdujo adaptadores en el ADN genómico, mientras lo fragmentaba en segmentos de 100 pb. La secuencia del adaptador se usó para amplificar el ADNg fragmentado en una reacción en cadena de la polimerasa de ciclo limitado. El ADN se enriqueció con fragmentos exónicos, que también se amplificaron 5. Las secuencias se alinearon para la variantes y se anotaron con la secuencia de referencia del genoma humano (hg19 build) utilizando el software BWA (http://bio-bwa.sourceforge.net). El procesamiento posterior se realizó con el software Samtools - Faidx y Picard - Dict, PICARD (http://broadinstitute.github.io/picard/) y el kit de herramientas de análisis del genoma (GATK), https://software.broadinstitute.org/gatk/best-practices/workflow?id=11145.
La línea de comando para seleccionar variantes en genes candidatos fue: variantes en genes previamente asociados con IDP, frecuencia de población <1% tanto en la base de batos de 1000 genomes como en el Consorcio de Agregación del Exoma (ExAC), no sinónimas, ubicadas en regiones exónicas o de splicing, efectos deletereos predicho por SIFT, Polyphen2, MutationTaster o CADD (Tabla S1). Las mutaciones homocigóticas se filtraron porque sospechábamos un patrón de herencia recesivo (Figura S2).
Para predecir el efecto de las variantes intrónicas, también se utilizó la herramienta In-silico EX-SKIP (http://ex-skip.img.cas.cz/). Además, se utilizaron predictores NetGene2 (http://www.cbs.dtu.dk/services/NetGene2/) y Fruitfly (http://www.fruitfly.org/seq_tools/promoter.html) para evaluar las variantes en sitios de empalme. Las secuencias de referencia NM_006726.4 se usaron para el análisis de las variantes de LRBA.
Análisis de BLAST
Se realizó un análisis BLAST (del inglés, Basic Local Alignment Search Tool, https://blast.ncbi.nlm.nih.gov) para evaluar si los péptidos reconocidos por los anticuerpos policlonales anti-LRBA utilizados en este estudio (HPA019366 y HPA023597, Sigma- Aldrich) reaccionaron de forma cruzada con otras proteínas humanas. Los péptidos de los anticuerpos policlonales anti-LRBA (HPA019366 y HPA023597, Sigma-Aldrich) se sometieron a un análisis de BLAST contra todas las proteínas de referencia utilizando los siguientes parámetros: Base de datos: proteína refseq; Organismo: Homo sapiens (taxid: 9606); Algoritmo: blatstp (proteína-proteína BLAST).
Análisis estadístico
Se realizó el coeficiente de correlación de Pearson utilizando GraphPad Prism 5 (https://www.graphpad.com/support/prism-5-updates/) para medir la correlación estadística entre la intensidad de la detección de LRBA por WB (intensidad de banda de LRBA) y el Tinción de LRBA por FACS (intensidad media de fluorescencia, IMF).
Resultados
Detección de LRBA en células de sangre periférica.
Para confirmar la sensibilidad de esta prueba y la precisión de dos diferentes anticuerpos anti-LRBA disponibles comercialmente, estandarizamos la evaluación de la expresión de LRBA en CMSP humanas por WB y FACS. Usando WB, se detectó la banda LRBA a 319 kDa en células CMSP estimuladas con PHA de donantes sanos (DS) (Figura 1A). Además, se detectó LRBA por FACS en células de sangre periférica ex vivo y en CMSP estimuladas y sin PHA. El LRBA se expresa constitutivamente en células CD3+ de sangre periférica ex vivo y en CMSP no estimuladas después de tres días de incubación, sin embargo, tras la estimulación con PHA, la expresión aumenta dos veces (Figura 1B). Se evaluó la correlación entre la capacidad de WB y FACS para detectar LRBA (Figura 1C), y se observó una correlación positiva (r: 0.4723, p 0.0112) entre el nivel de detección de LRBA entre ambas técnicas. Para asegurar la especificidad de nuestros anticuerpos, se realizó un análisis BLAST para evaluar si cada epítope reconocido por los anticuerpos anti-LRBA se estaba alineando con otras proteínas humanas (Figura 1D). Además del 100% de coincidencia con la proteína LRBA, estos epítopes también coincidieron con la proteína paraloga NBEA en 36% y 44%, respectivamente. Estos resultados indican que los anticuerpos policlonales anti-LRBA utilizados por nosotros también podrían reconocer NBEA, pero esta posibilidad se descarta porque esta proteína tiene una expresión muy limitada en el cerebro 6. Además, el anticuerpo HPA019366 mostró una unión específica a LRBA intracelular que se perdió al agregar un péptido de bloqueo específico de LRBA de una manera dosis dependiente (Figura 1E).
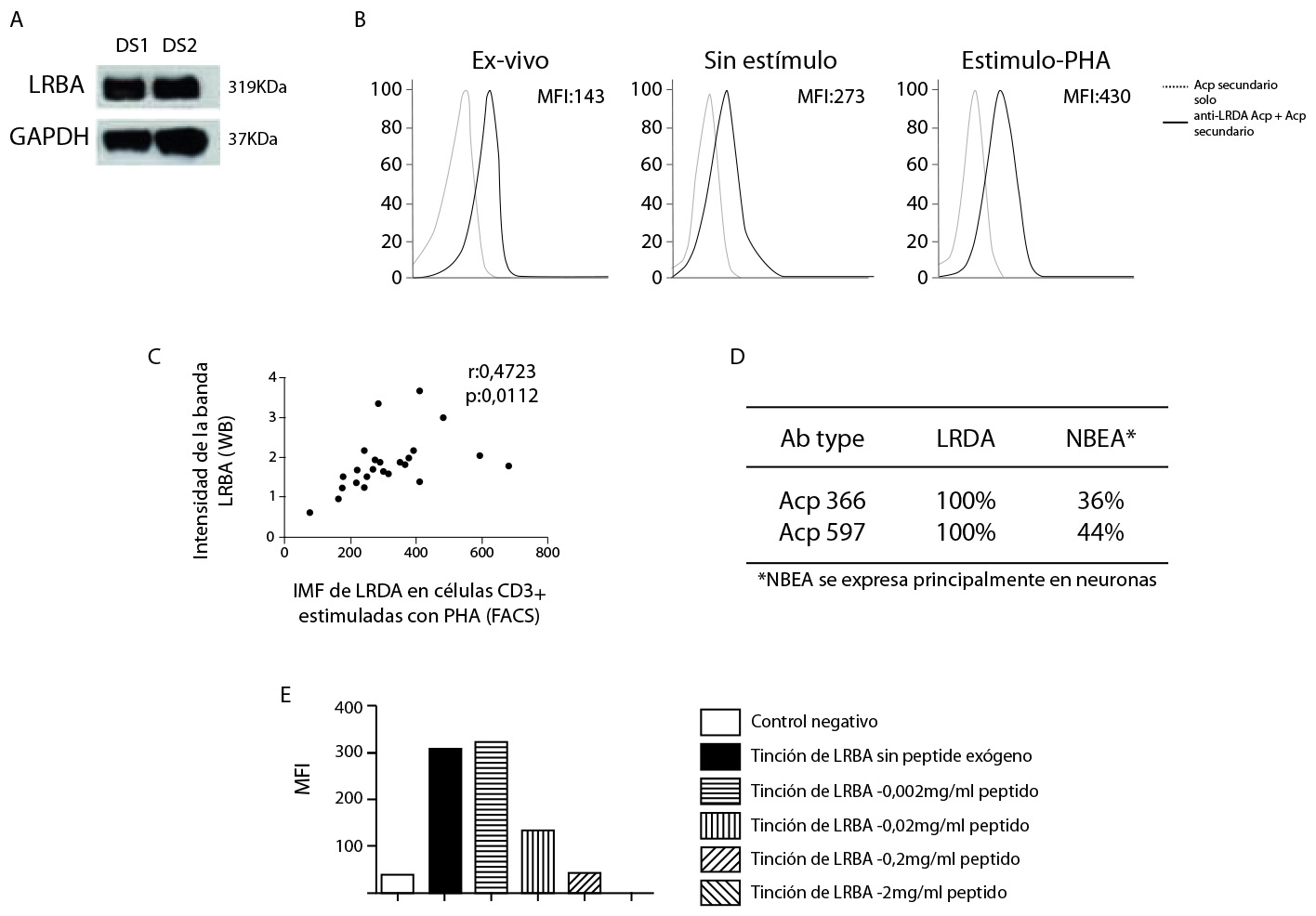
Figura 1 Estandarización de la tinción de LRBA en células de sangre periférica humana. Se obtuvieron células mononucleares de sangre periférica (CMSP) estimuladas con fitohemaglutinina A (PHA) de donantes sanos (DS) y se lisaron inmediatamente para extraer y cuantificar proteínas (40 ug) que se analizaron en una transferencia Western Blot (WB) con el anticuerpo anti-LRBA (HPA023597 , Sigma) usando GAPDH como una proteína expresada constitutivamente. Se detectaron bandas a 319 y 37 KDa, respectivamente (A). Consecutivamente, las células de sangre periférica ex vivo y las CMSP estimuladas sin PHA (1 µg / ml) se tiñeron intracelularmente con el anticuerpo anti-LRBA (HPA019366, Sigma) usando anti-conejo PE-F (ab) `2 Donkey IgG como anticuerpo secundario. Las líneas punteadas indican la tinción solo con el anticuerpo secundario. Las líneas continuas representan la tinción con el anticuerpo anti-LRBA junto con el anticuerpo secundario. Se muestra la tinción de LRBA solamente las células CD3+ activadas (B). Se realizó un análisis de correlación de Pearson comparando la intensidad de la detección de LRBA por WB (intensidad de banda de LRBA) y la tinción de LRBA por FACS (intensidad de fluorescencia media, IMF) (C). Además, se realizó un análisis BLAST (https://blast.ncbi.nlm.nih.gov) para evaluar si el péptido reconocido por los anticuerpos anti-LRBA utilizados en este estudio tenían reacción cruzada con NBEA (Secuencia de www.ncbi .nlm.nih.gov/nuccore/NC_000013.11) (NBEA es un paralog de LRBA) (D). En función de los anticuerpos anti-LRBA que se comercializan son policlonales, la especificidad de reconocimiento también se evaluó mediante el péptido LRBA SVLMVSKYRDILEPQNERHSQSCTETGSENENVSLSEITPAAFSTLTTASVEESESTSSARRRDSGIGEETATGLGSHVEVTPHTAPPGVSAGPDAISEVLSTLSLEVNKSPETKNDRGNDLDTKATPSVSV como un reactivo competitivo a diferentes concentraciones. Se calculó el porcentaje de reconocimiento de la proteína LRBA celular (E).
Expresión de LRBA en pacientes con fenotipo clínico asociado a deficiencia de LRBA
Acorde a los criterios de inclusión, se reclutaron ciento doce pacientes, 29 pacientes con inmunodeficiencia común variable (ICV), 14 con agamaglobulinemia ligada al cromosoma X (XLA, del inglés, X-linked agamaglobulinemia), 55 con hipogammaglobulinemia idiopática (IHG, del inglés, idiopathic hypogammaglobulinemia), 1 con deficiencia selectiva de IgA (SigAD, del inglés, selective IgA deficiency), 5 con enteropatías, 3 con púrpura trombocitopénica idiopática y 5 con lupus eritematoso sistémico (LES) (Figura 2A). En nuestra cohorte, los pacientes con ICV, IHG, XLA y SIgAD presentaron hipo o agammaglobulinemia, 9 pacientes con ICV exhibieron manifestaciones autoinmunes (Tabla S2). La expresión relativa de LRBA fue evaluada por WB en 25 DS y cuantificada usando densitometría, para normalizarla contra la expresión de la proteína constitutiva (TUBA o GAPDH) (Figura 2A). Se seleccionó el punto de corte óptimo de LRBA para distinguir entre "expresión disminuida de LRBA" y "expresión normal de LRBA " considerando el valor mínimo de la expresión de LRBA entre DS (corte del punto 0.5).
Como control negativo, se usó CVID022 ya que esta muestra siempre fue negativa para la expresión de LRBA (Figura 2A). Además, se evaluó la expresión de LRBA en células de sangre perférica ex-vivo y CMSP estimuladas y sin PHA por FACS (Figura S3). El LRBA exhibió una disminución de 2.3 veces en las células de pacientes ex vivo en comparación con las células DS. Por lo tanto, la reducción en la expresión de LRBA en linfocitos de CVID022 se observó no solo por WB sino también por FACS. Desafortunadamente, no fue posible evaluar la expresión de LRBA mediante citometría de flujo en los otros pacientes con disminución de la expresión de LRBA por WB. Se observaron resultados similares en CMSP sin estímulos de PHA. Sin embargo, el efecto fue más evidente en las CMSP estimuladas con PHA con una disminución de ocho veces en la expresión de LRBA en el paciente en comparación con una DS (Figura S3).
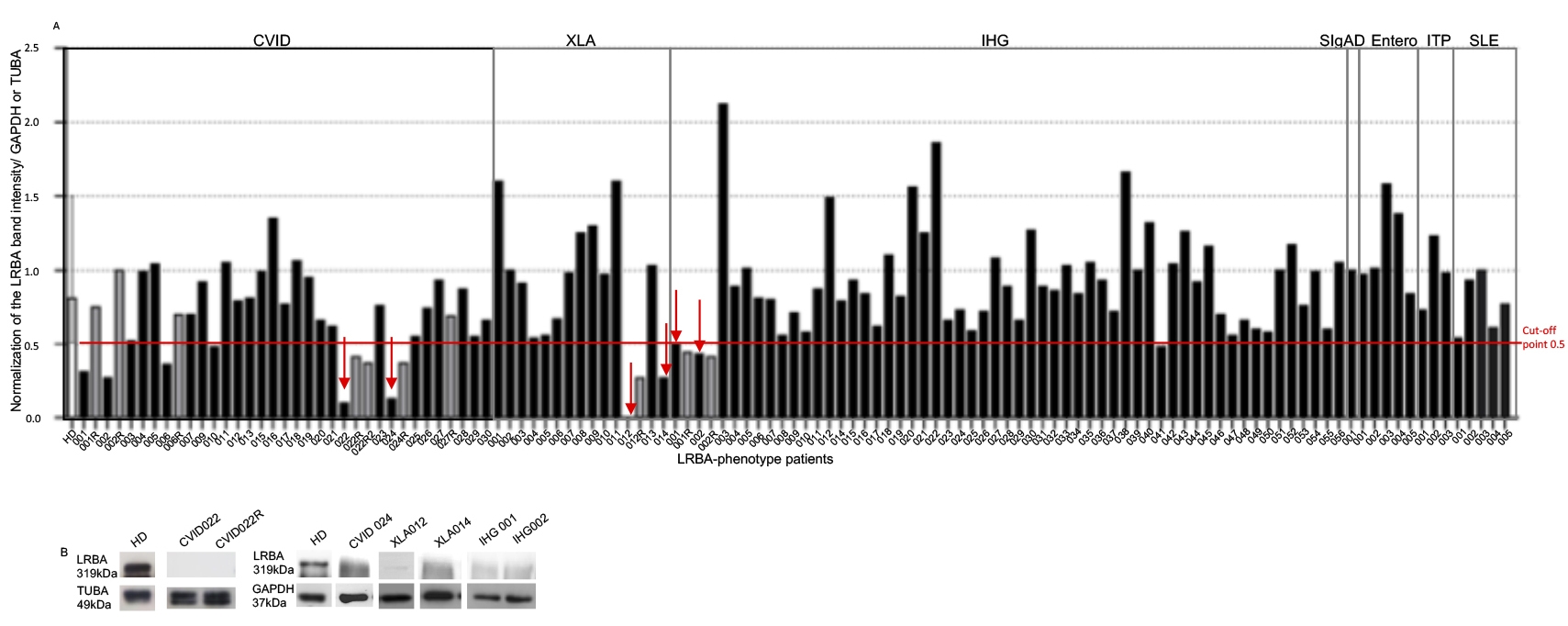
Figura 2 Expresión de LRBA en pacientes. Ciento doce pacientes fueron reclutados para evaluar la expresión de LRBA por westerm blot (WB). Se obtuvieron CMSP estimuladas con PHA de 25 donantes sanos para extraer y cuantificar proteínas (40 μg) que se analizaron en un WB con el anticuerpo anti-LRBA Ab HPA023597 (Sigma) usando GAPDH o TUBA como proteína expresada constitutivamente. Se detectaron bandas a 319 kDa para LRBA, 37 KDa para GAPDH y 49 KDa para TUBA. La normalización de la intensidad de la banda LRBA contra la proteína constitutiva (GAPDH o TUBA) se muestra en (A). Las intensidades de la banda WB de LRBA en células estimuladas con PHA de pacientes con expresión disminuida de LRBA se muestran en (B). ICV o CVID: inmunodeficiencia común variable, XLA: agammaglobulinemia ligada a X, IHG: hipogammaglobulinemia, SIgAD: deficiencia selectiva de IgA, PTI: púrpura trombocitopénica idiopática, LES: lupus eritematoso sistémico. Las barras grises representan las muestras repetidas. Las flechas rojas indican los pacientes con disminución de LRBA que continuaron en el estudio.
Se observó una disminución de la expresión de LRBA en nueve lisados de proteínas de CMSP estimuladas con PHA (Figura 2A, barras grises), pero solo se repitieron en ocho y se confirmaron en seis lisados por obtención de una segunda muestra de sangre: dos pacientes con CVID (CVID22 y CVID024), dos pacientes con XLA (XLA012 y XLA014) y dos pacientes con hipogammaglobulinemia (IHG001 e IHG002) (Figura 2 flecha roja). Curiosamente, los pacientes con hipogammaglobulinemia son hermanos. Es importante mencionar que CVID027 es hermana de estos hermanos, sin embargo, no mostró disminución de la expresión de LRBA en CMSP (Figura 2A).
No hubo diferencias en la expresión de LRBA por WB en células obtenidas de pacientes con las diferentes condiciones clínicas evaluadas en comparación con DS (media/DE de la intensidad de banda normalizada: DS: 0.86/0.25; CVID: 0.69/0.32; XLA: 0.86/0.47; IHG: 0.90/0.35; enteropatías: 1.10/0.31; ITP: 0.98/0.25 y SLE: 0.77/ 0.20).
Curiosamente, se observó un aumento en la expresión de LRBA en siete pacientes (XLA001, XLA011, IHG003, IHG022, IHG022, IHG038, Entero003). La Figura 2B muestra las intensidades de banda de WB de LRBA en células estimuladas con PHA de pacientes con expresión disminuida de LRBA.
Fenotipo clínico de pacientes con expresión disminuida de LRBA
Las características clínicas de los pacientes se muestran en la Tabla 1. La paciente CVID022 es una mujer nacida a término en 1982, producto del primer embarazo de padres sanos no relacionados de Medellín (Antioquia-Colombia). Aunque no se documentaron antecedentes familiares de infecciones recurrentes, autoinmunidad o neoplasias hematológicas, los miembros de la familia describieron rinitis, bronquitis, diabetes tipo II, asma y enfermedad pulmonar obstructiva crónica.
Tabla 1 Resumen de las características clínicas de pacientes con disminución de la expresión de LRBA
| Paciente | Edad actual | Edad del primer síntoma | Edad de diagnóstico | Infecciones recurrentes | Enfermedades gastrointestinales | Otras manifestaciones clínicas | |
|---|---|---|---|---|---|---|---|
| Alergias | Anemia | ||||||
| CVID022 | 36 | 10 | 13 | Amigdalitis, neumonía, otitis, sinusitis. | Diarrea con moco (giardiasis y amebiasis), gastritis y pólipos duodenales. | Rinitis | Yes |
| CVID024 | 19 | 2 | 35 | Amigdalitis, neumonía, otitis, parotitis, celulitis periorbitaria, sinusitis. | Apendicitis con sepsis abdominal, enfermedad del intestino irritable. | Conjuntivitis, Rinitis | - |
| XLA012 | 7 | 1 | 1 | Infección del tracto urinario, otitis, aspergilosis*, síndrome bronquial crónico, sinusitis. | Gastroenteritis aguda y enfermedad diarreica (Salmonelosis). | - | Yes |
| XLA014 | 4 | 1 | 1 | Infecciones del tracto respiratorio superior, otitis, sinusitis. | - | Rinitis | - |
| Hipo001 | 9 | 1 | 3 | Bronquiolitis, neumonía, otitis media, laringitis. | Enfermedad por reflujo gastroesofágico grado IV, alergia alimentaria. | Asma | - |
| Hipo002 | 11 | 1 | 4 | Otitis, amigdalitis. | Infección gastrointestinal, diarrea, dolor abdominal crónico. | - | - |
*. Esta infección fue sistémica pero no recurrente.
El paciente CVID024 es un hombre, nacido a término en 1998 de padres sanos no relacionados. Es el primer hijo y tiene un hermano más joven y saludable. Su padre es de Medellín y su madre de Bolívar (Antioquia-Colombia). Solo tenía antecedentes familiares de diabetes tipo II y asma en su abuela paterna. A los 8 años, se identificaron dos variantes en TACI en un patrón de herencia heterocigoto compuesto: una variante, c.121G> C (p.41D> H) en el exón 2 y la segunda variante c.297_299insT (p.Q99fsX6) en el exón 3. Sin embargo, ambas variantes también se identificaron en la madre y en el hermano del paciente, los cuales no están afectados, El padre portaba ambas variables tipo silvestre.
El paciente XLA012 es un hombre, nacido por cesárea en 2010 de padres sanos no relacionados de Cúcuta (Norte de Santander, Colombia). Él también tiene una hermana mayor sana. La diabetes y la hipertensión se informaron en su abuela materna. También se documentó la historia familiar de un tío fallecido a la edad de 5 años debido a "edema". El diagnóstico genético de XLA se realizó a los 2 años con la detección de la mutación c.1573C> T p.Arg525X en Btk. Durante los cinco años de seguimiento, se ha documentado un síndrome bronquial crónico, anemia y sinusitis crónica.
El paciente XLA014 es un hombre, nacido a término en 2013, producto del segundo embarazo de padres sanos no relacionados de Santa Rosa del Sur de Bolívar (Santander-Colombia). Su madre tuvo un aborto espontáneo previo y él también tiene un hermano menor saludable. Cáncer hematológico se informó en su abuela paterna.
IHG001 es un hombre, nacido en 2008 en Medellín - Colombia de padres sanos no relacionados. Fue el producto del tercer embarazo, nacido prematuramente a las 34 semanas de gestación por cesárea con enfermedad de membrana hialina. Su padre sufría de rinitis alérgica y su abuela materna documentó asma e hipotiroidismo. También tiene un primo con hipotiroidismo. Su hermana (IHG002) nació en 2006. Ella fue el producto del segundo embarazo, nacido a las 36 semanas de gestación por cesárea. Estos hermanos tienen una hermana mayor (CVID027) con inmunoglobulinas séricas normales y expresión de LRBA en CMSP.
Fenotipo inmunológico en pacientes con disminución de la expresión de LRBA
Un análisis inmunológico de los diferentes subgrupos de linfocitos en sangre periférica de todos los pacientes con disminución de LRBA en CMSP no mostró una característica fenotípica común y distintiva (Tabla 2). Sin embargo, las características inmunológicas de los diferentes pacientes se confirmaron según el grupo de enfermedad. Todos los pacientes con ICV exhibieron una disminución de IgG e IgA en suero. Las subpoblaciones de linfocitos T en sangre periférica fueron normales en los pacientes con ICV. Se observó una disminución en los linfocitos B de memoria (IgD+ / CD27+) en ambos pacientes, sin embargo, solo CVID022 tuvo una disminución en los linfocitos B de memoria con cambio de isotipo (IgD- / CD27+). Con respecto a los pacientes con XLA, la disminución en los tres isotipos de Ig en suero se observó en XLA012, mientras que no se encontraron datos de inmunoglobulinas (Ig) en suero previos a la terapia de reemplazo de inmunoglobulina intravenosa (IVIG, del inglés intravenous immunoglobulin) para XLA14. Fue notorio el muy bajo número de linfocitos B de sangre periferica observadas en estos pacientes. Además, se observaron algunas alteraciones menores de los linfocitos T CD3+ en los pacientes con XLA. Curiosamente, los dos hermanos con IHG exhibieron un fenotipo inmunológico diferente. IHG001 exhibió niveles muy bajos de IgG e IgA en suero, mientras que IHG002 presentó solo una disminución de IgG en suero. Sin embargo, ambos exhibieron un aumento en la subpoblación de los linfocitos B de memoria (Tabla 2).
Tabla 2 Caracterización inmunológica de los pacientes con disminución de la expresión de LRBA en CMSP estimuladas con PHA
| Pacientes | |||||||
|---|---|---|---|---|---|---|---|
| CVID022 | CVID024 | XLA012 | XLA014 | IHG001 | IHG002 | ||
| Niveles de inmunoglobulinas (mg/dL) | |||||||
| IgG | 0.0↓ | 19.2( | 111.8( | ND | 6.4↓ | 675.42↓ | |
| IgM | 5.7↓ | 55.1↑ | 53.1 | ND | 64.0 | 81.99 | |
| IgA | 2.1↓ | 2.0↓ | 5.2↓ | ND | <3.25↓ | 72.23 | |
| Linfocitos | Porcentajes/número absoluto(células/mm 3 ) | ||||||
| CD3+ | 64/1,169 | 74/2,963 | 90.5/2,222↑ | 88/4,738↑ | 64.4/2,655 | 74.0/2,776 | |
| CD3+ CD4+ | 44.8/524 | 23.4/937 | 56.7/1,393↑ | 25.6/1,366 | 35.2/1,452 | 35.8/1,346 | |
| CD3+ CD8+ | 52/606 | 40.2/1610 | 27.4/673 | 58.3/3111↑ | 23.3/961 | 31.8/1,196 | |
| CD19+ | 8.21/147↓ | 12.8/230 | 0.67/16( | 0.1/3↓ | 23.3/961↑ | 17.6/660 | |
| CD19+/CD21+ | 7.02/125 | 11.0/197 | 0.4/11 | 0.0/1 | 22.1/914 | 16.1/606 | |
| CD19+CD27+ | 14.75/22↓ | 16.0/37 | -- | -- | 24.77/238↑ | 30.6/202↑ | |
| IgD+/CD27- | 77.2/113↑ | 68.0/156 | -- | -- | 71.7/689↑ | 63.6/420↓ | |
| IgD+/CD27+ | 10.5/15↓ | 5.9/14↓ | -- | -- | 15.1/145↑ | 15.6/103↑ | |
| IgD-/CD27+ | 4.3/6↓ | 10.1/23 | -- | -- | 9.67/93↑ | 15/99↑ | |
| IgD+/CD38+ | 0.5/1 | 12.9/30 | -- | -- | 11.1/107 | -- | |
| IgD-/CD38++ | 0.0/0↓ | 3.3/2.1 | -- | -- | 0.93/9↑ | 1.53/10 | |
| CD24++/CD38++ | 0.5/1↓ | 12.8/29 | -- | -- | 11.0/106 | 13/86 | |
Ig: inmunoglobulina, ND: sin datos. Las flechas hacia abajo indican disminución y las flechas hacia arriba con respecto al valor de referencia.
Variantes de LRBA en los pacientes con disminución de la expresión de LRBA
WES se realizó en los pacientes con disminución de la expresión de LRBA. En general, se identificaron dieciocho variantes no sinónimas en exones o intrónes en LRBA, en estos pacientes (Tabla 3). Se encontraron cinco variantes con una frecuencia de población <1% (Tabla 3, barras grises), de las cuales, dos variantes, rs148385798 en el paciente CVID022 y el rs950337550 en el paciente CVID024 fueron posiblemente patógenas (Tabla 3, recuadro rojo). La variante rs148385798 es un cambio sin sentido heterocigoto c.T194C en el exón 2 que corresponde al cambio p.I65T en el dominio ConA de la proteína. El análisis in silico predijo ser "perjudicial" con SIFT (puntuación: 0.02); "Causante de enfermedad" con MutationTaster (valor p: 0.995) y "perjudicial" en PROVEAN (puntaje: -2.558) y el puntaje CADD fue 16.37. La segunda variante rs950337550 es una variante sin sentido heterocigota c.6892A> G en el exón 46 que confiere un cambio p.K2298E en el dominio BEACH. El análisis en silco predijo que sería "perjudicial" con SIFT (puntuación: 0.005); "Causante de enfermedad" con MutationTaster (valor p: 1) y "No perjudicial" con PROVEAN (puntaje: -1.44).
Tabla 3 Variantes de LRBA en los pacientes con disminución de la expresión de LRBA en CMSP estimuladas con PHA
| Pacientes | Localización | Ref | Alt | Exons/introns | SNP | Alelos | Frec* |
|---|---|---|---|---|---|---|---|
| CVID022 | 151199080 | G | A | Exon | rs2290846 | het | 0.15 |
| 151207127 | C | T | Exon | rs3749574 | het | 0.16 | |
| 151242312 | C | T | Intron | rs11737450 | het | 0.18 | |
| 151753134 | A | - | Intron | rs145452262 | het | 0.062 | |
| 151765243 | G | A | Intron | rs186080 | hom | 0.75 | |
| 151935601 | A | G | Exon | rs148385798 | het | 0.002 | |
| 151408800 | T | C | Intron | rs7674989 | hom | 0.2 | |
| 151656589 | C | T | Intron | rs1993109 | het | 0.4 | |
| 151829659 | T | C | Intron | rs1201207 | hom | 0.04 | |
| 151773593 | G | C | Exon | rs1782360 | hom | 0.29 | |
| XLA12 | 151207127 | C | T | Exon | rs3749574 | het | 0.16 |
| 151236393 | A | G | Intron | rs74459799 | het | 0.0062 | |
| 151242312 | C | T | Intron | rs11737450 | het | 0.18 | |
| 151357938 | G | A | Exon | rs950337550 | het | 0.000008 | |
| 151765243 | G | A | Intron | rs186080 | hom | 0.75 | |
| 151817507 | C | T | Intron | rs1813134 | het | 0.011 | |
| XLA14 | 151765243 | G | A | Intron | rs115139393 | hom | 0.75 |
| 151829659 | TC | - | Intron | 151829659 | hom | 0.96 | |
| 151231371 | T | C | Intron | rs1813134 | het | 0.52 | |
| 151187011 | - | GAGAT | Intron | rs201486611 | het | 0.69 | |
| IHG001 IHG002 | 151231371 | T | C | Intron | rs1813134 | hom | 0.52 |
| 151235987 | G | A | Intron | -- | het | -- | |
| 151753134 | A | - | Intron | rs145452262 | het | 0.062 | |
| 151762104 | G | A | Intron | rs893110507 | het | 0.00004 | |
| 151765243 | G | A | Intron | rs186080 | het | 0.75 |
Ref: alelo de referencia, Alt: alelo alterado, SNP: polimorfismo de un solo nucleótido, Frec*: frecuencia, hom: homocigoto, het: heterocigoto. En gris se destacan las variantes con una frecuencia <0.001 en el proyecto 1,000 genomes (http://www.internationalgenome.org). Las variables indicadas en rojo son patógenas o posiblemente patógenas según los predictores en silico.
Teniendo en cuenta que el patrón de herencia LRBA es autosómico recesivo y que no habíamos encontrado variantes bialélicas, continuamos con el análisis de variantes en otros genes involucrados en el sistema inmune.
Otras variantes relevantes encontradas secuenciación del exoma completo (WES- Whole-Exome Sequencing)
Se identificaron 50,300 variantes de alta calidad en el exoma CVID022. Entre ellos, 17,056 fueron homocigotos y 33,244 heterocigotos (Tabla 4). Las variantes en los genes IDP se analizaron primero y se encontraron variantes homocigóticas en tres genes (PLCG2, POLE, RLTPR). Sin embargo, estas variantes se localizaron en regiones intrónicas, muy distantes del exón proximal. Para proceder con el análisis de WES, siguiendo la hipótesis de un estado de herencia recesivo, se seleccionaron variantes homocigóticas o heterocigotas compuestas. Estas variantes deben cumplir también los criterios definidos. Se seleccionaron tres genes candidatos (KCNN3, TRPM3, ECT2L), sin embargo, KCNN3 y TRPM3 no se expresan en los tejidos linfoides. Además, el padre del paciente también portaba ambas variantes en ECT2L. Finalmente, evaluamos las variantes heterocigotas de novo. Se encontró una variante en la proteína L3 mitocondrial ribosómica (MRL3). Las enfermedades asociadas con MRPL3 incluyen la Deficiencia combinada de fosforilación oxidativa 9 7. sin embargo, esta es una condición severa que afecta principalmente la función neurológica y hepática.
Tabla 4 Resumen de resultados de secuenciación de exoma completo en pacientes con disminución de la expresión de LRBA
| Variantes | CVID022 | XLA012 | XLA014 | IHG001 | IHG002 |
|---|---|---|---|---|---|
| Número total | 50,300 | 53,002 | 52,821 | 53,215 | 53,215 |
| Número de homocigotos | 17,056 | 14,785 | 15,015 | 14,654 | 14,654 |
| Número de heterocigotos | 33,244 | 20,731 | 20,530 | 21,352 | 21,352 |
| Genes relacionados con IDP | PLCG2, POLE, RLTPR | BTK | BTK | --- | |
| Variantes candidatas homocigotas | KCNN3, TRPM3 | --- | --- | PLCL2, FDFT1, GPRIN2 | |
| Variantes candidatas heterocigotos | ECT2L | --- | --- | SEC22B | |
En XLA12, se identificaron 53,002 variantes, 14,785 homocigotas y 20,730 heterocigotas (Tabla 4). Dentro de los genes IDP, se identificaron 27 variantes. Entre ellos, se confirmó la variante c.1573C> T p.Arg525X en BTK. En otros genes, no se encontraron variantes candidatas patogénicas potenciales.
En XLA14, se identificaron 52,821 variantes de alta calidad en el exoma, 15,015 fueron homocigóticas y 20,530 heterocigotas (Tabla 4). Dentro de los genes relacionados con IDP, se encontraron 40 variantes y se identificó una variante potencialmente patógena en el gen BTK. Esto corresponde a c.622 + 1G> A (NM_001287344). 3. El análisis in silico (fruit fly y NetGene2) mostró que los efectos de la mutación BTK confieren un cambio en el sitio donante de empalme al final del intrón 6 dentro de este gen que afecta a la proteína en el dominio PH.
En los hermanos IHG001 e IHG002, se encontraron un total de 53,215 variantes en ambos. Los números de variantes homocigotos y heterocigotos se especifican en la Tabla 4. Sin embargo, no se identificaron heterocigotos homocigotos o compuestos comunes en los genes IDP. Sin embargo, las variantes homocigotas en PLCL2, FDFT1 y GPRIN2, su padre tenía las mismas variantes homocigotas en PLCL2 y FDFT1 y su madre tenía las mismas variantes homocigotas en GPRIN2. Además, se encontraron variantes heterocigóticas compuestas en SEC22B, sin embargo, su padre portaba las mismas variantes.
Discusión
En este trabajo, se evaluó la expresión de LRBA en 112 pacientes con manifestaciones clínicas asociadas con deficiencia de LRBA. Se observó concordancia interindividual e intraindividual en la detección de la expresión de LRBA en CMSP estimuladas con PHA. La expresión de LRBA detectada por WB fue muy heterogénea en DS. Gamez et al informaron que esta heterogeneidad también se encontró en FACS un rango de IMF entre 2.14 -10.00 (min - max) en la expresión relativa de LRBA en CMSP tras la estimulación con PHA durante cuatro días en DS 8. Entre estos pacientes, se observó una disminución de la expresión de LRBA en nueve lisados de proteínas de pacientes, después de repetidos, solo se observó una disminución en seis lisados de proteínas de pacientes. Todavía no sabemos por qué las células de algunos pacientes exhibieron niveles discordantes de expresión de LRBA en diferentes muestras de sangre y puntos de tiempo. Una posible explicación de estos resultados es una senescencia celular activada por el sistema inmune. Diferentes factores como las infecciones virales persistentes, el estrés y los síndromes inflamatorios inducen la acumulación de linfocitos T con características de senescencia 9. Es posible que estos pacientes hayan pasado por períodos de estrés o infección con la consecuencia de una senescencia inducida y alteraciones en la proliferación de las células. Dado que la detección de la expresión de LRBA en CMSP en nuestros entornos es un fenómeno dependiente de la activación, disminuir la activación de las células senescentes podría conducir a una disminución en la expresión de LRBA detectada por las células inmunes. Sin embargo, un marcador de activación, como CD69, debe incluirse en los controles de tinción en los estudios futuros. Por otro lado, también se observó un aumento en la expresión de LRBA en siete pacientes. Hasta la fecha, no se ha informado sobreexpresión de este gen asociado con alguna IDP, sin embargo, Wang et al. en 2004 informó por micromatrices y análisis de PCR en tiempo real que LRBA se sobreexpresa en varios tipos de cáncer en relación con sus controles de tejido normales 10. Además, la expresión de la activación celular debió haber sido monitoreada en las células de nuestros pacientes, ya que se ha demostrado previamente que la expresión de LRBA aumenta con la activación celular 4. Esto podría afirmarse como una de las limitaciones de nuestros resultados, ya que la disminución de la expresión de LRBA por WB podría haber sido causada por una pobre activación de las células inmunes. Sin embargo, siempre tuvimos una limitación de muestra debido a la enorme cantidad de proteína requerida para realizar el WB. Además, no ha habido correlación entre la intensidad de la expresión de LRBA por citometría de flujo y el índice de proliferación en linfocitos B en donantes sanos (datos no mostrados). Por otro lado, los pacientes con escasa proliferación de células T y B incluidas en esta cohorte, exhibieron una expresión de LRBA normal por WB (datos no mostrados).
Los pacientes con disminución de la expresión de LRBA exhibieron diferentes características fenotípicas e inmunológicas. Sin embargo, todos los pacientes presentaron una reducción en IgG e infecciones recurrentes y todos excepto uno presentaron gastropatía. No encontramos variantes bialélicas patógenas o potencialmente patógenas ni en LRBA ni en otros genes relacionados con el sistema inmune. La expresión de LRBA en CMSP puede disminuir debido a mecanismos epigenéticos 11. Los mecanismos epigenéticos determinan el fenotipo sin cambios en el genotipo, que operan a nivel transcripcional y postranscripcional de la actividad génica, así como a nivel de traducción de proteínas y modificaciones postraduccionales. Participan en procesos como la diferenciación celular, la morfogénesis, la variabilidad y la adaptabilidad de un organismo y pueden verse afectados por factores genéticos y ambientales 11. Además, se han propuesto cambios epigenéticos pueden contribuir a la patogénesis de ICV. Rodríguez-Cortez et al demostraron mediante análisis de metilación de ADN de alto rendimiento, en gemelos monocigóticos discordantes para ICV, ganancia predominante de metilación de ADN en linfocitos B en los pacientes con ICV con respecto a los de los hermanos sanos 12. En conjunto, se requiere la aplicación de estudios epigenéticos para identificar y caracterizar no solo las firmas moleculares de la deficiencia de LRBA, así como los mecanismos subyacentes a la patogénesis de la enfermedad y las respuestas a las nuevas modalidades terapéuticas. Otra posible explicación de la disminución en la expresión de LRBA en CMSP estimulada por PHA de pacientes sin mutaciones en LRBA u otros genes por WES, sería la presencia de mutaciones patógenas en elementos reguladores, incluidos promotores, potenciadores, aisladores y silenciadores en LRBA. Cualquier variante que se encuentre dentro de una región genómica funcional tiene la capacidad potencial de causar una disregulación en la expresión génica a través de la modificación de elementos reguladores, posiblemente resultando en la patogénesis de enfermedades 13. En los últimos años, los estudios de asociación de todo el genoma (GWAS) identificaron más de diez mil variantes asociadas con diversas enfermedades/rasgos, ~ 90% de los cuales se localizan fuera de las regiones de codificación de proteínas conocidas 14. Además se requieren estudios futuros para evaluar la variación en el número de copias y la tasa de cobertura de cada exón en LRBA, para excluir una segunda variante heterocigótica en pacientes con alta sospecha clínica de deficiencia de LRBA.
En el presente estudio, no se encontraron variables con significancia clínica en LRBA, luego, continuamos con el análisis de variables en otros genes en todo el exoma. Se encontró una nueva variante (c.622 + 1G> A NM_001287344) en BTK que probablemente confiere una pérdida en el sitio donante de empalme al final del intrón 6 en el paciente XLA14. Cuando un sitio de splicing natural se elimina por mutaciones, el splicesosoma suele utilizar el siguiente sitio legitimo disponible para el splicing (omisión de exón) o selecciona el siguiente mejor, aunque ilegítimo, sitio de splicing cercano (utilización críptica del splicing) 15. En este caso específico, la transcripción de ARNm podría estar afectada después del exón 6, afectando la proteína del aminoácido 25 y generando una nueva proteína truncada. Los ARNm aberrantes podrían eliminarse mediante la vía de degradación del ARN mensajero mediada por mutación terminadora, que se dirige a los ARNm que albergan codones de terminación prematura para la degradación 16. Por lo tanto, la proteína truncada podría no contener los dominios PH, SH3, SH2 y quinasa de la proteína y, por lo tanto, podría no ser funcional o estar sujeta a degradación por la vía Ubiquitina-Proteasoma. Sin embargo, el efecto exacto de la mutación debe verificarse en estudios funcionales.
En conjunto, los resultados de este estudio indican que la expresión de LRBA varía mucho en las CMSP entre diferentes DS y pacientes. A pesar de la gran variabilidad en la expresión de LRBA en las diferentes muestras, seis pacientes exhibieron una disminución de la expresión de esta proteína por WB en CMSP estimulada por PHA. No se detectaron genes asociados con la producción defectuosa de LRBA por estas células en estos pacientes. Las mutaciones homocigotas en LRBA causan deficiencia de LRBA que afecta la expresión de la proteína. Sin embargo, la reducción de la expresión de LRBA en pacientes sin mutaciones homocigotas o en genes asociados estaba presente. Estos resultados sugieren los otros mecanismos genéticos, epigenéticos o ambientales, podrían estar regulando la expresión de LRBA.
La especificidad de la detección de LRBA por WB y FACS se evaluó previamente. Se demostró una correlación positiva entre la capacidad de WB y FACS para detectar LRBA. También se realizaron experimentos FACS utilizando epítopos competitivos para validar la especificidad del reconocimiento de LRBA. Con estos resultados, demostramos que los anticuerpos mostraron una unión específica al LRBA intracelular que se perdió al agregar un péptido de bloqueo de LRBA específico de una manera dependiente de la dosis.
REFERENCIAS
1. Casanova JL, Conley ME, Seligman SJ, Abel L, Notarangelo LD. Guidelines for genetic studies in single patients: lessons from primary immunodeficiencies. J Exp Med. 2014; 211(11):2137-49. doi: 10.1084/jem.20140520 [ Links ]
2. Bousfiha AA, Jeddane L, Ailal F, Benhsaien I, Mahlaoui N, Casanova JL, et al. Primary immunodeficiency diseases worldwide: More common than generally thought. J Clin Immunol. 2013;33(1):1-7. doi: 10.1007/s10875-012-9751-7. [ Links ]
3. Lopez-Herrera G, Tampella G, Pan-Hammarström Q, Herholz P, Trujillo-Vargas CM, Phadwal K, et al. Deleterious mutations in LRBA are associated with a syndrome of immune deficiency and autoimmunity. Am J Hum Genet. 2012;90(6):986-1001. doi: 10.1016/j.ajhg.2012.04.015 [ Links ]
4. Gamez-Días L, August D, Stepensky P, Revel-Vilk S, Seidel MG, Noriko M, et al. The extended phenotype of LPS-responsive beige-like anchor protein (LRBA) deficiency. J Allergy Clin Immunol. 2016;137(1):223-230. doi: 10.1016/j.jaci.2015.09.025. [ Links ]
5. Pabinger S, Dander A, Fischer M, Snajder R, Sperk M, Efremova M, et al. A survey of tools for variant analysis of next-generation genome sequencing data. Brief Bioinform. 2014; 15: 256-78. doi: 10.1093/bib/bbs086. [ Links ]
6. Wang X, Herberg FW, Laue MM, Wullner C, Hu B, Petrasch-Parwez E, et al. Neurobeachin: A protein kinase A-anchoring, beige/Chediak-higashi protein homolog implicated in neuronal membrane traffic. J Neurosci. 2000; 1:20(23):8551-65. Doi: 10.1523/JNEUROSCI.20-23-08551.2000. [ Links ]
7. Galmiche L, Serre V, Beinat M, Assouline Z, Lebre AS, Chretien D, et al. Exome sequencing identifies MRPL3 mutation in mitochondrial cardiomyopathy. Hum Mutat. 2011;32(11):1225-31. doi: 10.1002/humu.21562. [ Links ]
8. Gámez-Díaz L, Sigmund EC, Reiser V, Vach W, Jung S, Grimbacher B. Rapid flow cytometry-based test for the diagnosis of lipopolysaccharide responsive beige-like anchor (LRBA) deficiency. Front Immunol. 2018; 23(9):720. doi: 10.3389/fimmu.2018.00720. [ Links ]
9. Di Mitri D, Azevedo RI., Henson SM, Libri V, Riddell NE, Macaulay R,et al. Reversible Senescence in Human CD4+CD45RA+CD27- Memory T Cells. J Immunol. 2011;187(5):2093-100. doi: 10.4049/jimmunol.1100978. [ Links ]
10. Wang J, Gamsby JJ, Highfill SL, Mora LB, Bloom GC, Yeatman TJ, et al. Deregulated expression of LRBA facilitates cancer cell growth. Oncogene. 2004; 23(23): 4089-4097. doi: 10.1038/sj.onc.1207567 [ Links ]
11. Santos-Rebouças CB, Pimentel MM. Implication of abnormal epigenetic patterns for human diseases. Eur J Hum Genet. 2007;15(1):10-7. doi: 10.1038/sj.ejhg.5201727 [ Links ]
12. Rodríguez-Cortez VC, Del Pino-Molina L, Rodríguez-Ubreva J, Ciudad L, Gómez-Cabrero D, Company C,et al. Monozygotic twins discordant for common variable immunodeficiency reveal impaired DNA demethylation during naïve-to-memory B-cell transition. Nat Commun. 2015; 17(6):7335. doi: 10.1038/ncomms8335. [ Links ]
13. Ma M, Ru Y, Chuang LS, Hsu NY, Shi LS, Hakenberg J,et al. Disease-associated variants in different categories of disease located in distinct regulatory elements. BMC Genomics. 2015;16(8): S3. doi: 10.1186/1471-2164-16-S8-S3 [ Links ]
14. Welter D, MacArthur J, Morales J, Burdett T, Hall P, Junkins H, et al. The NHGRI GWAS Catalog, a curated resource of SNP-trait associations. Nucleic Acids Res. 2014; 42(D1): 1001-1006. doi: 10.1093/nar/gkt1229. [ Links ]
15. Kurmangaliyev YZ, Gelfand MS. Computational analysis of splicing errors and mutations in human transcripts. BMC Genomics. 2008; 9: 1-10. doi: 10.1186/1471-2164-9-13. [ Links ]
16. Hug N, Longman D, Cáceres JF. Mechanism and regulation of the nonsense-mediated decay pathway. Nucleic Acids Res. 2016;29:44(4):1483-95. doi: 10.1093/nar/gkw010. [ Links ]
Financiación: Este estudio fue apoyado por el Departamento Administrativo de Ciencia, Tecnología e Innovación, COLCIENCIAS (Grant # 111556934430). Beca COLCIENCIAS 727.
Anexos
Tabla S1 Resumen de los métodos de predicción deletéreos utilizados en este estudio
| Name | Score | Information used |
|---|---|---|
| SIFT (Sorting Intolerant from tolerant) | El rango va de 0 a 1. Se predice que la sustitución de aminoácidos es dañina si el puntaje es ≤ 0.05 y se tolera si el puntaje es> 0.05. | Predice si una sustitución de aminoácidos afecta la función de la proteína según la homología de secuencia y las propiedades físicas de los aminoácidos. |
| PolyPhen-2 (Polymorphism Phenotyping v2) | Rangos de 0.0 (tolerado) a 1.0 (perjudicial) | Predice el posible impacto de una sustitución de aminoácidos en la estructura y función de una proteína humana utilizando consideraciones físicas y comparativas directas. |
| 0.0 a 0.15: benigno. | ||
| 0.15 a 0.85: posiblemente dañino. | ||
| 0.85 a 1.0: dañino. | ||
| MutationTaster | El valor prob es la probabilidad de la predicción, es decir, un valor cercano a 1 indica una alta 'seguridad' de la predicción. Tenga en cuenta que el valor prob utilizado aquí NO es el valor p (probabilidad de error) como se usa en las estadísticas de prueba t. | Conservación de la secuencia de ADN, predicción del sitio de empalme, predicción de estabilidad de ARNm y anotaciones de características de proteínas. |
| PROVEAN (Protein Variation Effect Analyzer) | Los puntajes se promedian dentro y entre los grupos para generar el puntaje PROVEAN final. Si el puntaje PROVEAN es igual o inferior a un umbral predefinido (por ejemplo, -2.5), se predice que la variante de proteína tendrá un efecto "perjudicial". | Predice si una sustitución de aminoácidos o indel tiene un impacto en la función biológica de una proteína. |
| CADD (Combined Annotation Dependent Depletion) | ( 15 | 63 anotaciones de variante distintas recuperadas del predictor de efecto de variante Ensembl (VEP), datos del proyecto ENCODE e información de las pistas del explorador del genoma UCSC. |
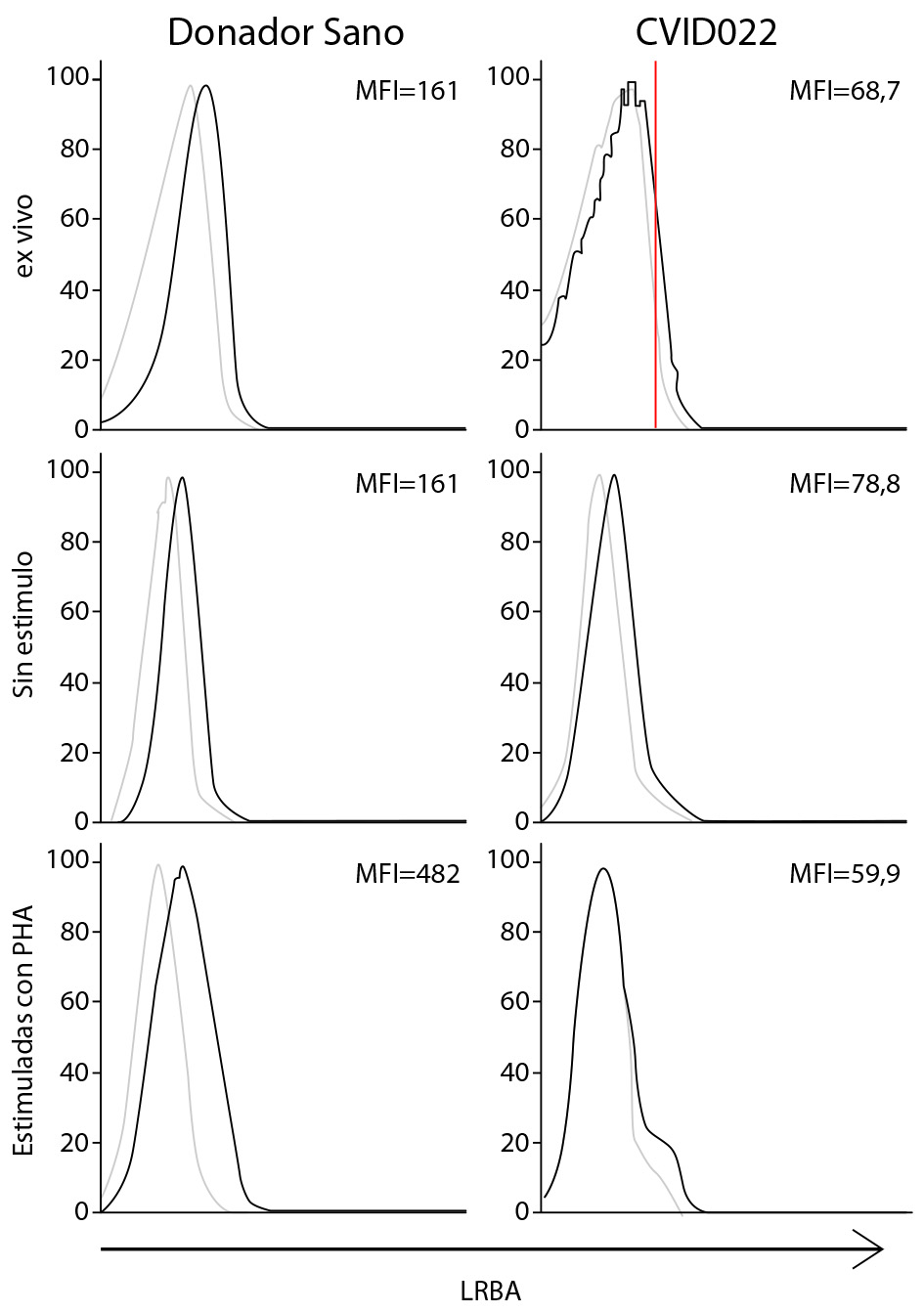
Figura S3 Expresión de LRBA en CMSP estimulada por PHA de CVID022 por FACS. Las células de sangre periférica ex-vivo y CMSP estimuladas sin PHA (1 μg / ml) se tiñeron intracelularmente con el anticuerpo anti-LRBA (HPA019366, Sigma) usando IgG de burro anti-conejo PE-F (ab) `2 como anticuerpo secundario. Las líneas punteadas indican la tinción solo con el anticuerpo secundario. Las líneas continuas representan la tinción con el anticuerpo anti-LRBA junto con el anticuerpo secundario. Se muestra la tinción de LRBA que activa solo las células CD3+. La línea roja representa la intensidad media de fluorescencia de LRBA (IMF) del experimento correspondiente en DS
Tabla S2 Resumen de manifestaciones autoinmunes en pacientes con CVID e IHG
| Enfermedad | Code | Manifestación autoinmune |
|---|---|---|
| ICV | 001 | Hipotiroidismo |
| 003 | Hipotiroidismo | |
| 004 | Lupus discoide, colitis ulcerosa | |
| 005 | Anemia hemolítica y hepatitis autoinmune | |
| 007 | Hypothyroidism, inflammatory bowel disease | |
| 011 | Hipotiroidismo, enfermedad inflamatoria intestinal. | |
| 017 | Hipotiroidismo | |
| 023 | Hipotiroidismo | |
| 036 | Trombocitopenia autoinmune | |
| IHG | 033 | Hipotiroidismo |
| 039 | Hipotiroidismo | |
| 049 | Hipotiroidismo | |
| 052 | Neutropenia autoinmune |
ICV: Inmunodeficiencia común variable, IHG Hipogammaglobulinemia
Recibido: 25 de Febrero de 2019; Revisado: 28 de Agosto de 2019; Aprobado: 16 de Septiembre de 2019











 texto en
texto en