Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Colombia Médica
versión On-line ISSN 1657-9534
Colomb. Med. vol.52 no.1 Cali ene./mar. 2021 Epub 16-Mar-2021
https://doi.org/10.25100/cm.v52i1.4196
Articulo original
Papel de las proteínas de choque térmico en la infección de la línea celular de adenocarcinoma humano MCF-7 por aislados de rotavirus adaptados a tumores
1 Universidad Nacional de Colombia, Faculty of Medicine, Department of Physiological Sciences, Bogota, D.C., Colombia.
Antecedentes:
Los virus se utilizan como herramientas alternativas y complementarias para el tratamiento del cáncer. Los virus oncolíticos exhiben tropismo por tumores, capacidad para intensificar la inmunidad antitumoral y la capacidad para utilizarse en combinación con quimioterapia y radioterapia convencionales. Recientemente, hemos seleccionado algunos aislamientos de rotavirus que están adaptados para infectar y eliminar de manera eficiente líneas de células tumorales.
Objetivo:
Se ensayaron cinco aislamientos de rotavirus adaptados a células tumorales para determinar su capacidad para infectar la línea celular de adenocarcinoma humano MCF-7.
Métodos:
Las proteínas asociadas a la membrana de la superficie celular que median la unión de partículas de virus se caracterizaron mediante ELISA, inmunoprecipitación, análisis FACS y bloqueo de anticuerpos.
Resultados:
Se encontró que las proteínas de choque térmico (HSPs) como Hsp90, Hsp70, Hsp60 y Hsp40 se expresan en la superficie celular formando complejos con la proteína disulfuro isomerasa (PDI), la integrina β3 y la proteína análoga de choque térmico 70 (Hsc70) en microdominios lipídicos (rafts). La interacción de los aislamientos de rotavirus con estas proteínas celulares se confirmó adicionalmente mediante un ensayo de competición y un ensayo de inhibición que incluía las HSP ensayadas.
Conclusión:
Nuestros hallazgos sugieren que los aislamientos de rotavirus adaptados a las células tumorales estudiados aquí ofrecen una herramienta prometedora para eliminar las células tumorales, lo que estimula más investigaciones sobre este tema, incluidos los modelos animales.
Palabras clave: Virus oncolíticos; viroterapia oncolítica; rotavirus; vacunas contra el cáncer; células MCF-7; proteínas disulfuro-isomerasas; proteínas protooncogénicas c-akt; proteínas de choque térmico; infecciones por rotavírus; integrinas; piroptosis; muerte celular autofágica
Background:
Viruses are being used as alternative and complementary tools for treating cancers. Oncolytic viruses exhibit tumor tropism, ability to enhance anti-tumor immunity and ability to be used in combination with conventional chemotherapy and radiotherapy. We have recently selected some rotavirus isolates which are adapted to efficiently infect and kill tumor cell lines.
Aim:
We tested five tumor cell-adapted rotavirus isolates for their ability to infect the human adenocarcinoma cell line MCF-7.
Methods:
Cell surface membrane-associated proteins mediating virus particle attachment were characterized using ELISA, immunoprecipitation, FACS analysis, and antibody blocking.
Results:
It was found that heat shock proteins (HSPs) such as Hsp90, Hsp70, Hsp60, and Hsp40 are expressed on the cell surface forming complexes with protein disulfide isomerase (PDI), integrin β3, and heat shock cognate protein 70 (Hsc70) in lipid raft microdomains. Interaction of rotavirus isolates with these cellular proteins was further confirmed by a competition assay and an inhibition assay involving the HSPs tested.
Conclusion:
Our findings suggest that the tumor cell-adapted rotavirus isolates studied here offer a promising tool for killing tumor cells, thus encouraging further research into this topic, including animal models.
Key words: Oncolytic viruses; oncolytic virotherapy; rotavirus; cancer vaccines; MCF-7 cells; protein disulfide-isomerases; proto-oncogene proteins c-akt; heat-shock proteins; rotavirus infections; integrins; pyroptosis; autophagic cell death
Contribución del estudio
| 1) ¿Por qué se realizó este estudio? |
| El presente estudio fue realizado considerando los estimulantes resultados previos consistentes en la generación de aislamientos de rotavirus capaces de infectar e inducir eficientemente la muerte de líneas celulares derivados de tumores malignos. Estos rotavirus adaptados a células tumorales ya se habían seleccionado mediante evolución in vitro. El propósito de este estudio fue ensayar la capacidad de estos rotavirus para infectar e inducir la muerte de la línea celular de adenocarcinoma humano MCF-7. También constituyó un motivo para llevar a cabo la presente investigación, la identificación de las moléculas receptoras de la superficie celular responsables del tropismo de los aislamientos rotavirales por estas células. Este estudio intentó producir conocimiento acerca de la dependencia de los virus oncolíticos de los receptores celulares para entrar a la célula tumoral, ya que los receptores parecen no estar presentes en todos los tumores o en todas las células de un mismo tumor. Por lo tanto, la identificación de receptores más universales, tales como las HSPs, es crucial para sugerir si los rotavirus pueden ser candidatos para el tratamiento de cáncer de seno. |
| 2) ¿Cuáles fueron los resultados más relevantes del estudio? |
| Los resultados más relevantes del presente estudio están representados por la identificación de las proteínas de choque térmico (HSPs: Hsp90, Hsp70, Hsp60 y Hsp40) en la superficie de la célula, las cuales fueron requeridas por los aislamientos de rotavirus para infectar exitosamente las células MCF-7. Las HSPs fueron encontradas formando complejos con la proteína disulfuro isomerasa (PDI), integrina β3, y la proteína análoga de choque térmico 70 (Hsc70) en microdominios lipídicos (rafts). |
| 3¿Qué aportan estos resultados? |
| Estos resultados contribuyen a fortalecer la senda de la investigación dirigida a encontrar estrategias alternativas y complementarias para el tratamiento del cáncer basadas en virus oncolíticos. Estos resultados estimulan la realización de investigación adicional de los aislamientos de rotavirus aquí ensayados como potenciales agentes oncolíticos en el marco de la viroterapia |
Introducción
El uso de virus para el tratamiento del cáncer data de principios del siglo XX 1,2. Sin embargo, en las últimas dos décadas se han vuelto a realizar estudios de viroterapia ya que los virus oncolíticos son una herramienta prometedora para ser utilizada como terapia alternativa y complementaria para el tratamiento de entidades tumorales 3. El uso de virus oncolíticos como agentes terapéuticos se basa en su replicación selectiva en las células tumorales sin ser nocivos para el paciente (4. Los virus oncolíticos se caracterizan por su tropismo tumoral, su capacidad para potenciar la inmunidad antitumoral y su capacidad para combinarse con quimioterapia y radioterapia convencionales, así como agentes inmunoterapéuticos 5. Los virus oncolíticos dentro de las células tumorales son capaces de estimular el sistema inmunológico facilitando así una respuesta eficaz y duradera contra el tumor 6.
Hasta la fecha, varios virus oncolíticos han demostrado ser eficaces contra algunas células tumorales, incluidos los modelos animales 7-10, los estudios preclínicos 11) y los ensayos clínicos 11-13. Ensayos clínicos recientes han demostrado la seguridad, tolerabilidad y eficacia de algunos virus oncolíticos utilizados para el tratamiento de cánceres 14. Los virus oncolíticos realizan múltiples interacciones con las células tumorales diana al funcionar como agentes citotóxicos directos y como vacunas terapéuticas contra el cáncer para inducir la muerte celular a través de varios mecanismos que incluyen apoptosis inmunogénica, piroptosis, necrosis y muerte celular autofágica 14-16. El tropismo de las células tumorales de los virus oncolíticos aprovecha los receptores de la superficie celular que facilitan la unión del virus y la penetración en las células tumorales para establecer un ciclo lítico en las células tumorales sin afectar a las células normales 17. Se han estudiado muchos virus sobre sus propiedades oncolíticas 17y aproximadamente 40 ensayos clínicos se están llevando a cabo con varios virus oncolíticos 18. IMLYGICTM (T-VEC/Talimogene Laherparepvec, un virus herpes simple modificado genéticamente indicado para el tratamiento local de lesiones cutáneas, subcutáneas y ganglionares no resecables en pacientes con melanoma es la primera terapia viral oncolítica aprobada por la FDA) 19.
Las proteínas celulares de choque térmico (HSPs) desempeñan un papel central en el mantenimiento de la homeostasis celular y en la formación de una red protectora contra desafíos estresantes, también en la facilitación del plegamiento de proteínas nacientes y el replegamiento de proteínas desnaturalizadas 20. Las infecciones por virus pueden inducir HSPs que a su vez apoyan la replicación de virus. Hsp70, que es particularmente inducida por infecciones virales y fiebre, apoya y mejora la expresión de los genes virales 21. Las HSPs se expresan en altos niveles en una amplia gama de cánceres humanos donde contribuyen al desarrollo del tumor al promover la proliferación celular, invasión, metástasis, muerte y reconocimiento por el sistema inmunológico 22,23. Hsp90 tiene un papel positivo en el proceso de infección por rotavirus en células MA104 modulando la señalización de Akt 24. La proteína choque térmico constitutiva (Hsc70) es un receptor utilizado para rotavirus durante la infección de células MA104 25) y está implicada en la inducción de cambios conformacionales en las partículas de rotavirus 26. La PDI de la superficie celular y la integrina β3 también se utilizan como receptores para el rotavirus 27,28.
El rotavirus se compone de tres capas concéntricas de proteína de la cápside que rodean un genoma compuesto por 11 segmentos de RNA de doble cadena. El tropismo de rotavirus depende fundamentalmente del uso de receptores y de sus niveles de expresión en la superficie celular. Se ha demostrado que el crecimiento de rotavirus en líneas celulares humanas y de monos se correlaciona con la expresión en la superficie celular de algunas integrinas 29. La interacción de las proteínas externas del rotavirus con las moléculas receptoras de la superficie celular posibilita su entrada en las células hospedadoras permisivas después de algunos cambios estructurales en proteínas virales de la capa externa, tales como VP4 (VP8 * y VP5 *) y VP7 30.
Recientemente se han reportado aislamientos de rotavirus adaptados a células tumorales que pueden inducir la muerte celular en algunas células tumorales 31. En el presente estudio, mostramos que estos aislamientos de rotavirus pueden infectar con éxito la línea celular de adenocarcinoma de glándula mamaria humana MCF-7 usando algunas HSPs de la superficie celular como receptores para entrar en la célula. Nuestros hallazgos sugieren que estos aislamientos de rotavirus podrían investigarse más a fondo como una posible estrategia terapéutica para tratar el cáncer.
Materiales y Métodos
Células y virus
La línea celular de adenocarcinoma de glándula mamaria humana MCF-7 (ATCC, Cat. No. HTB-22) se propagó en medio de Eagle Modificado de Dulbecco (DMEM) (Sigma-Aldrich, St. Louis, MO, EE. UU., Producto No. D6429) suplementado con SBF al 10%, estreptomicina 100 μg/mL y penicilina 100 μg/mL (Eurobio, Les Ulis, Francia) a 37º C en cámara húmeda con CO2 al 5%. Esta línea celular fue amablemente donada por el Dr. C. Parra (Departamento de Microbiología, Facultad de Medicina, Universidad Nacional de Colombia). Los aislamientos de rotavirus WT1-5, TRUYO, WWM, WTEW o ECwt-o se generaron como se describió anteriormente 31. Los virus se propagaron en células MCF-7.
Purificación de virus
Las partículas de rotavirus se purificaron como se informó anteriormente 32. Brevemente, las células infectadas en solución salina tamponada con Tris (TBS) (Tris-HCl 10 mM, pH 7.4, NaCl 150 mM, MgCl2 1 mM y CaCl2 5 mM) se rompieron congelando y descongelando dos veces. El lisado se emulsionó con 1,1,2-triclorotrifluoroetano (Freón) y se centrifugó a 12 g durante 10 min a 4º C. La fase orgánica se reextrajo con TBS y las fracciones acuosas se sometieron a centrifugación a 120,000 g durante 1.5 horas 4º C. El virus sedimentado se resuspendió en tampón TNC (Tris-HCl 20 mM, pH 8.0, NaCl 100 mM, CaCl2 1 mM) y luego se centrifugó a través de un gradiente discontinuo de sacarosa-cloruro de cesio a 210,000 g durante 1 h a 4º C. Las bandas virales (TLP y DLP) se recolectaron por separado y se sometieron a un segundo ciclo de sedimentación a alta velocidad y centrifugación en gradiente de sacarosa-cloruro de cesio. Las TLP y DLP purificadas en tampón TNC se almacenaron a -70º C hasta su uso.
Infección viral
Se infectaron células MCF-7 (5×104) en 50 µL de DMEM por pozo con aislamientos de rotavirus WT1-5, TRUYO, WWM, WTEW o ECwt-o (0.8 MOI) purificados en gradiente de cloruro de cesio, que se habían activado previamente con tripsina (1 µg/mL). Las placas de cultivo de 96 pozos que contenían las células se incubaron a 37 ºC y las células se analizaron mediante un ensayo de inmunocitoquímica a 12 h.p.i. Los títulos de virus se determinaron infectando células con MOI de 0.04 seguido de detección del porcentaje de células infectadas a 12 h.p.i usando un ensayo de inmunocitoquímica.
Ensayo de inmunocitoquímica
Las células se cultivaron en cubreobjetos lavados con xilol en placas de cultivo de 96 pozos y a las 12 h.p.i. se fijaron a temperatura ambiente (TA) con paraformaldehído (PFA) al 4% en PBS. Las células lavadas con PBS se hicieron reaccionar con anticuerpos policlonales de conejo, contra las proteínas estructurales del rotavirus (PE) y se incubaron a 37º C durante 1 hora. Las células se lavaron dos veces con PBS y se incubaron con anticuerpos anti-conejo de cabra conjugados con HRP (0.133 μg/mL, Santa Cruz Biotechnology Inc., Santa Cruz, CA, EE. UU.) a 37º C durante 1 hora. Después de tres lavados con PBS, las células positivas al antígeno de rotavirus se visualizaron usando sustrato de 3-amino-9-etilcarbazol (AEC) (0.64 mg/mL) en tampón de acetato de sodio 50 mM, pH 5.0, que contenía H2O2 al 0.04%. Las células de control consistían en células no infectadas o células infectadas tratadas con anticuerpos no relacionados. Se registraron fotográficamente diez imágenes representativas utilizando un microscopio óptico (VanGuard, Scottsdale, EE. UU.) y se determinó el porcentaje promedio de células infectadas.
Viabilidad celular
Las monocapas de células MCF-7 se trataron con una solución de azul de tripano al 0,25% durante 1 min a temperatura ambiente (TA). Se evaluó la exclusión de azul de tripano en las células lavadas con PBS. Este ensayo se utilizó para determinar las concentraciones máximas de los diferentes anticuerpos, proteínas y reactivos inhibidores que podrían usarse sin afectar la viabilidad celular. Las células se trataron por separado con proteínas recombinantes (rHsp90 y rHsp70) y anticuerpos contra HSP (90, 70, 60 y 40) y Hsc70, y también con los inhibidores de Hsp90 17AAG (33), NVP-AUY922 (34) y radicicol (35), y los inhibidores de Hsp70 VER-155008 (36) y quercetina (37). La incubación de las células con los tratamientos se realizó durante 12 horas 37º C y como control se utilizaron células no tratadas.
Aislamiento de la membrana celular
Las células previamente lavadas con PBS (1x108) se suspendieron en tampón hipotónico (HEPES 5 mM, pH 7.4, y sacarosa 50 mM) y se lisaron usando un homogeneizador Dounce de vidrio durante 5 min en hielo. Al homogeneizado se le añadió CaCl2 (concentración final 10 mM) y luego se centrifugó a 3,500 g durante 5 minutos a 4º C. El sobrenadante se volvió a centrifugar a 20,000 g durante 30 min a 4º C. El sedimento se resuspendió en PBS que contenía azida de sodio al 0.02% y se almacenó a 4º C hasta su uso en 24 horas.
Aislamiento de proteínas de la membrana celular
Las proteínas asociadas a la membrana se solubilizaron a partir de las fracciones de la membrana celular tratándolas con tampón RIPA (Tris-HCl 50 mM, pH 8.0, NaCl 150 mM, NP-40 1%, DOC 0.5%, SDS 0.1%) durante 30 min a 37° C y las proteínas solubilizadas se cuantificaron utilizando un espectrofotómetro NanoDrop 2000c.
ELISA
Se añadieron proteínas solubilizadas de membranas celulares a los pozos de las placas de ELISA recubiertas con anticuerpos de conejo contra β3, PDI o Hsc70 y se incubaron a 4º C durante la noche, seguido de bloqueo con caseína al 0.6% durante 1 hora 37 ºC. Los pozos de las placas se lavaron tres veces con PBS que contenía Tween 20 al 0.05% (PBS-T) durante 5 min cada lavado. A los pozos se añadieron por separado anticuerpos primarios de cabra (0.2 µg/mL, Santa Cruz Biotechnology Inc.) contra Hsp70, Hsp60 o Hsp40 y se incubaron durante 1 hora 37º C. Después de tres lavados con PBS-T, se añadió anticuerpo secundario de burro anti-cabra conjugado con HRP (0.133 µg/mL, Santa Cruz Biotechnology Inc. SC-2020), seguido de incubación durante 1 hora 37º C. La reacción se reveló usando el sustrato diclorhidrato de o-fenilendiamina (OPD, PIERCE®) en un tampón de sustrato de peróxido estable. La reacción se detuvo con H2SO4 3 N y se midió la absorbancia a 492 nm en un lector de placas de ELISA. Los resultados se expresaron como delta OD obtenido restando los valores de absorbancia de los pozos de control en los que no se añadió proteína solubilizada, pero los tratamientos con anticuerpos fueron idénticos. Los datos se presentaron como media ± desviación estándar (DS) de tres experimentos independientes realizados por duplicado.
En un experimento paralelo, las fracciones de la membrana celular se incubaron por separado con aislamientos de rotavirus WTEW, TRUYO, WWM, Wt1-5 o ECwt-o durante 1 hora 37º C, seguido de solubilización en tampón RIPA (Tris-HCl 50 mM, pH 7,4, NaCl 150 mM, NP-40 al 1%, DOC al 0.5% y SDS al 0.1%) durante 30 min a 37º C. Se añadieron fracciones de membrana celular solubilizadas a placas de ELISA recubiertas con anticuerpos de cobayo contra proteínas estructurales de rotavirus y se incubaron durante 1 hora 37º C. Después de bloquear como se indicó anteriormente y lavar tres veces con PBS-T, las placas se incubaron durante 1 h a 37º C con anticuerpos primarios de cabra (0.2 µg/mL, Santa Cruz Biotechnology Inc.) para Hsp90, Hsp70, Hsp60, Hsp40, PDI e integrina β3. Después de tres lavados con PBS-T, se añadió IgG de burro anti-cabra, conjugado con HRP secundario (0.133 µg/mL, Santa Cruz Biotechnology Inc. SC-2020) y se incubó durante 1 h a 37º C. Después de tres lavados con PBS-T, la reacción se reveló con OPD y se cuantificó como se indicó anteriormente. En los pozos de control se omitió el tratamiento con virus mientras que los otros tratamientos se mantuvieron idénticos.
Ensayo de microdominios lipídicos (rafts)
Las células (1x108) en DMEM sin FBS se trataron o dejaron sin tratar con metil-β-ciclodextrina (10 mM) durante 1 hora 37 °C, se lavaron con PBS y luego se lisaron con tampón de lisis (Tris-HCl 50 mM, pH 7.5, NaCl 150 mM, EDTA 2 mM, DTT 2 mM y Triton X-100 1%) en presencia de PMSF 2 mM a 4º C durante 20 min. Se añadió al lisado un volumen de sacarosa al 80% en tampón de lisis, se colocó en el fondo de un tubo de centrífuga y se cubrió con un gradiente lineal de sacarosa (30 a 5%). Después de centrifugar a 120,000 g durante 4 h a 4° C, se recogieron fracciones (500 µL) de la parte superior del gradiente y las fracciones que contenían las balsas se trataron con Triton X-100 al 1% durante 30 min a 37° C para solubilizar los lípidos.
Las fracciones solubilizadas se aplicaron a placas de ELISA recubiertas con anticuerpos policlonales de conejo contra la integrina β3, se bloquearon con caseína al 0.6% y luego se incubaron durante toda la noche a 4º C. Después de lavar tres veces con PBS-T, los pozos de la placa se trataron con anticuerpos primarios de cabra (0.2 µg/mL, Santa Cruz Biotechnology Inc.) contra Hsp90, Hsp70, Hsp60 o Hsp40 durante 1 h a 37º C. Después de tres lavados con PBS-T, se añadió IgG anti-cabra de burro conjugado con HRP secundario (0.133 µg/mL, Santa Cruz Biotechnology Inc. SC-2020), seguido de incubación durante 1 h a 37º C. Después de lavar tres veces con PBS-T, se usó OPD para visualizar y cuantificar la reacción como se indicó anteriormente. Los pozos sin la adición de fracción solubilizada se utilizaron como pozos de control. Los datos se presentaron como promedio ± desviación estándar (DS) de tres experimentos independientes realizados por duplicado.
Inmunoprecipitación
Las fracciones enriquecidas en la membrana celular de MCF-7 se mezclaron por separado con aislamientos de rotavirus activados con tripsina WTEW, TRUYO, WWM, Wt1-5 o ECwt-o. Como control, las fracciones enriquecidas en la membrana celular MA104 se mezclaron con aislados de rotavirus activados con tripsina Wt1-5. Después de la incubación durante 45 min a 37° C, se añadió tampón RIPA (Tris-HCl 50 mM, pH 8.0, NaCl 150 mM, NP-40 al 1%, DOC al 0.5% y SDS al 0.1%) y la preparación se incubó durante 30 minutos a 37 °C. Las membranas solubilizadas se mezclaron con perlas de agarosa de proteína G que se habían recubierto con anti-rotavirus de conejo y se bloquearon con caseína, seguido de agitación constante durante 30 min a TA. Las perlas lavadas con PBS se sedimentaron y se trataron con tampón de muestra de Laemmli durante 3 min en un baño de agua hirviendo. Las proteínas desnaturalizadas se analizaron mediante SDS-PAGE/Western blotting. Las membranas bloqueadas con caseína se incubaron con una mezcla de anticuerpos de cabra (0.2 μg/mL cada uno, Santa Cruz Biotechnology Inc.) contra Hsp90, Hsp70, Hsp60 y Hsp40 o alternativamente con anticuerpos de conejo contra proteínas estructurales (PE) de rotavirus durante 1 h 37° C. En el caso de las células MA104 con Wt1-5, se utilizaron anticuerpos contra la integrina β3, Hsc70, PDI o HSP (90, 70, 60, 40) en el análisis de SDS-PAGE/Western blotting. Las membranas se lavaron tres veces con PBS-T y se incubaron con anticuerpos de burro anti-IgG de cabra o de cabra anti-conejo conjugados con HRP secundarios (0.2 μg/mL, Santa Cruz Biotechnology Inc.) durante 1 h 37° C. Después del lavado con PBS-T, se reveló la membrana con reactivo de transferencia Western Luminol (Santa Cruz Biotechnology Inc. SC-2048). Como control se utilizaron fracciones de membrana celular solubilizadas tratadas con perlas de agarosa de proteína G sin anticuerpo.
Se probaron las interacciones de HSP (Hsp90, Hsp70, Hsp60 y Hsp40) con aislados de rotavirus usando biotinilación de la superficie celular junto con inmunoprecipitación de receptores para viriones de rotavirus. Las células lavadas con PBS (1x107) se trataron con 0,5 mg/ml de EZ-Link Sulfo-NHS-SS-Biotin (sulfosuccinimidil-2- [biotinamido] etil-1,3-ditiopropionato) durante 30 min a 4° C. Las células se lavaron con PBS que contenía glicina 100 mM durante 30 min a 4° C y luego se inocularon por separado con aislados de rotavirus activados con tripsina WTEW, TRUYO, WWM, Wt1-5 o ECwt-o a MOI 4 durante 30 min a 4° C. Los rotavirus no unidos se eliminaron lavando las células dos veces con PBS antes del tratamiento con tampón RIPA (Tris-HCl 50 mM, pH 8.0, NaCl 150 mM, NP-40 al 1%, DOC al 0.5% y SDS al 0.1%) durante 30 min a 37 ° C. El lisado celular se mezcló con perlas de agarosa de proteína G (Bio-Rad®, 90-180 µm) que se habían recubierto con anticuerpos de conejo contra el rotavirus y se bloquearon con caseína al 0.5% y se incubaron durante 30 minutos a TA con agitación constante. Las perlas se lavaron tres veces con PBS y el sedimento se trató con tampón de muestra de Laemmli durante 3 minutos en un baño de agua hirviendo. Las proteínas solubilizadas se analizaron mediante SDS-PAGE/Western blotting. Las proteínas se transfirieron electroforéticamente a una membrana de PVDF usando tampón CAPS 10 mM, pH 10.5, y la membrana se bloqueó con caseína al 5%. La membrana se hizo reaccionar con estreptavidina-HRP durante 1 hora 37º C y la reacción se visualizó con reactivo de transferencia Western Luminol. Como control se utilizó el lisado celular que contenía rotavirus e incubadas con perlas de agarosa de proteína G sin anticuerpos anti-rotavirus. Los datos se presentaron como media ± desviación estándar de tres experimentos independientes realizados por duplicado.
Análisis FACS
Las células de MCF-7 lavadas con PBS se lavaron adicionalmente con PBS que contenía EDTA 1 mM durante 5 minutos adicionales. Las células se fijaron en paraformaldehído (PFA) al 4% en PBS a 37º C durante 30 min y luego se lavaron dos veces con PBS. La expresión de las proteínas de la superficie celular Hsc70, Hsp90, Hsp70, Hsp60, Hsp40, PDI e integrina β3 se detectó incubando por separado las células con anticuerpos primarios de cabra (0.2 µg/mL, Santa Cruz Biotechnology Inc.) para cada proteína celular durante 45 min a 4° C. Se utilizó un anticuerpo de cabra irrelevante (IgG2a) como control de isotipo de IgG. Después de lavar dos veces con PBS que contenía BSA al 1%, las células se incubaron con IgG anti-cabra de burro acoplado a Alexa Fluor488 (0.4 µg/mL, Santa Cruz Biotechnology Inc. SC-362255) durante 20 minutos a 4º C. Después de lavar con PBS, las células se analizaron usando un citómetro de flujo Dako CyAn (Dako Cytomation). Los datos se presentaron como promedio ± desviación estándar (DS) de tres experimentos independientes realizados por duplicado.
Ensayo de bloqueo de anticuerpos con fragmentos F(ab')2
Las células MCF-7 (1x107) se lavaron con medio RPMI sin FBS y luego se trataron por separado con fragmentos F(ab')2 de conejo generados por digestión con pepsina en nuestro laboratorio contra péptidos sintéticos 20-mer derivados de secuencias de Hsp90, Hsp70, Hsp60 y Hsp40. Las secuencias peptídicas fueron las siguientes: Hsp90 (620-RDNSTMGYMAAKKHLEINPDHS-641); Hsp70 (374) (705-QIQQYMKIISSFKNKEDQYDHLD-727); Hsp70 (375) (646-NSFTLKLEDTENWLYED- 761); Hsp60 (393-RLAKLSDGVAVLKVGGTSDVEVN-415); Hsp40 (251-GSDVIYPARISLREALCGCTVNV-273). Se utilizaron fragmentos F(ab')2 en diluciones 1:40, 1:80, 1: 160, 1: 320; 1: 640 y 1: 1280 y se incubaron durante 1 h a 37° C. Los fragmentos F(ab')2 no unidos se eliminaron resuspendiendo las células en 1 ml de PBS frío antes de centrifugar a 600 g durante 5 min y eliminar el sobrenadante. Se incubaron las células con 100 µL de medio RPMI frío que contiene separadamente aislados de rotavirus WTEW, TRUYO, WWM, Wt1-5 o ECwt-o a MOI 2 y se incubó durante 45 minutos a 4° C. Se retiró el inóculo y se lavaron las células tres veces con PBS y se lisó con tampón RIPA (Tris-HCl 50 mM, pH 8.0, NaCl 150 mM, NP-40 al 1%, DOC al 0.5%, SDS al 0.1% y PMSF 5 mM) durante 1 h a 37º C.
El lisado se aplicó en una placa ELISA de 96 pozos recubierta con anticuerpos de cobayo contra las proteínas estructurales (PE) del rotavirus y que se había bloqueado durante 1 hora 37º C con caseína al 0.6% antes de adicionar el lisado. Después de tres lavados con PBS-T, se agregaron anticuerpos de cabra contra rotavirus PE y luego se incubaron durante 1 h a 37° C. Se añadió anticuerpo secundario de burro anti-cabra conjugado con HRP (0.133 μg/mL, Santa Cruz Biotechnology Inc. SC-2020), seguido de incubación durante 1 h a 37° C. La reacción se visualizó con OPD como se indicó anteriormente. Como control positivo se utilizaron células sin pretratamiento del fragmento F (ab')2 e inoculadas con aislados de rotavirus, mientras que como control negativo se utilizó ovoalbúmina en lugar de rotavirus. Los datos se presentaron como media ± desviación estándar (DS) de tres experimentos independientes realizados por duplicado.
Ensayo de unión viral a proteínas celulares sobre membrana PVDF (VOPBA)
Se lisaron células MCF-7 (1x108) usando buffer de lisis Laemmli (Tris-HCl 0.125 mM pH 6.8, SDS al 2%, glicerol al 10%, 2-ME al 5%, azul de bromofenol al 0.02%) y las proteínas solubilizadas se sometieron a SDS-PAGE (10%) antes de ser transferida electroforéticamente a una membrana de PVDF (Merck Millipore, Billerica, MA, EE. UU.) utilizando tampón CAPS 10 mM, pH 10.5. La membrana bloqueada con caseína al 5% se cortó en dos carriles (1 y 2) y uno de ellos (1) se incubó con TLPs de WTEW activadas con tripsina (10 µg/mL) en medio sin SFB durante toda la noche a 4º C. Este carril de la membrana se lavó inmediatamente con PBS y se incubó con glutaraldehído al 2.5% durante 30 minutos a 37° C antes de lavarse tres veces con PBS y se incubó con anticuerpos de conejo anti-rotavirus. El carril 2 de la membrana se incubó con anticuerpos de cabra contra Hsp90, Hsp70, Hsp60 y Hsp40 (Santa Cruz Biotechnology Inc.). Después ambos carriles de membrana (1 y 2) se incubaron por separado durante 1 h a 37° C y se lavaron dos veces con PBS. Luego, se incubaron durante 1 h a 37º C respectivamente con anticuerpo secundario IgG de cabra anti-conejo conjugado con HRP o IgG de burro anti-cabra, conjugado con HRP. Los carriles de membrana lavados con PBS se revelaron con reactivo de transferencia Western Luminol. Los datos se presentaron como promedio ± desviación estándar (DS) de tres experimentos independientes realizados por duplicado.
Ensayo de bloqueo celular con anticuerpos contra HSPs
Las monocapas de células MCF-7 (80% de confluencia) en placas de cultivo de 96 pozos se incubaron por separado con anticuerpos producidos en conejos contra un péptido sintético de 20 aminoácidos, derivado de Hsp90 (620-641), péptidos sintéticos derivados de tres dominios de Hsp70 (Hsp70 [705-727], Hsp70 [646-668] y Hsp70 [741-761]), Hsp60 [393-415]) o Hsp40 [251-273] a diluciones de 1:40, 1:80 y 1: 160. Después de la incubación durante 1 h a 37° C, las células se lavaron con medio RPMI libre de SFB y se colocaron en hielo durante 15 min. Las células se inocularon con los aislados de rotavirus WTEW, TRUYO, WWM, Wt1-5 o ECwt-o a MOI 0.8 durante 45 min a 4º C, en forma separada cada rotavirus. El virus no unido se eliminó lavando dos veces con PBS y las células se incubaron durante 12 h a 37º C, antes de la detección de células positivas a antígenos de rotavirus usando el ensayo de inmunocitoquímica descrito anteriormente. Los datos se presentaron como media ± desviación estándar de tres experimentos independientes realizados por duplicado.
Tratamiento celular con inhibidores de Hsp90 o Hsp70
Las células al 80% de confluencia se incubaron con inhibidores de Hsp90 17AAG (10, 20, 40 y 80 nM) 33, NVP-AUY922 (2, 4, 8 y 16 nM) 3) o radicicol (7.5, 25, 50 y 100 nM) 3, y los inhibidores de Hsp70 VER-155008 (10, 20, 40 y 80 nM) 3) o quercetina (75, 150, 300 y 600 µM) 3) durante 1 h a 37º C. Las células se inocularon por separado con WWM, TRUYO, Wt1-5, WTEW o ECwt-o a MOI 0.8, durante 45 min a 4º C. Las células lavadas con DMEM se incubaron durante 12 ha 37º C y luego se fijaron con PFA antes de someterlas a análisis inmunocitoquímico. Los datos se presentaron como media ± desviación estándar de tres experimentos independientes realizados por duplicado.
Expresión de HSPs recombinantes en E. coli
Las construcciones de expresión (pRSET-His-HSP90α wt, HSP90α-PGEXgp2, PMS-HSP70 AMP y PET-16b-HSP70) fueron amablemente donadas por el Dr. Y. Miyata (Departamento de Biología Celular y del Desarrollo, Bioestudios de la Escuela de Graduados, Universidad de Kyoto). Se mezclaron alícuotas de células bacterianas competentes (DH5α o BL21 (DE3)) en medio LB (200 µL) que contenía ampicilina (100 µg/mL) con ADN plasmídico (10 ng) en una solución libre de nucleasas (100 µL) (KCl 100 mM , CaCl2 30 mM y MgCl2 50 mM) con agitación suave antes de incubarse en hielo durante 20 min. Se realizó un choque térmico a 42º C de 2 min seguido de una incubación inmediata en hielo durante 2 min. Las células se inocularon en medio LB (5 mL) que contenía ampicilina (100 µg/mL) antes de la incubación a 37º C hasta alcanzar la fase de crecimiento exponencial. Como control se utilizaron células bacterianas competentes sin adición de ADN plasmídico pero sometidas a las mismas etapas de transfección. Se usó el kit Invitrogen ™ Purelink ™ Quick Plasmid Miniprep (Cat # K2100-10) para purificar el ADN de las células bacterianas según las instrucciones del fabricante.
Las células bacterianas (DH5α o BL21 (DE3)) que albergan los plásmidos se sometieron a inducción para expresar proteínas recombinantes cultivándolas en medio LB (25 mL) que contenía ampicilina (100 µg/mL) y etanol al 3% durante 12 h a 37º C con agitación constante (150 rpm) (38. Se usaron como controles células no transfectadas y células transfectadas no inducidas. Se inocularon alícuotas (5 mL) de cultivo bacteriano en medio LB (500 mL) y se incubaron adicionalmente a 37º C con agitación constante (150 rpm) hasta alcanzar la fase de crecimiento exponencial. Para expresar proteínas recombinantes, se añadió 0,5 mM de β-D-1-tiogalactopiranósido (IPTG) durante 4 h a 37º C con agitación constante (150 rpm). La suspensión celular se centrifugó a 12,000 g durante 10 min y las células bacterianas sedimentadas se resuspendieron en PBS que contenía N-lauril sarcosina al 1% y 100 µg/mL de lisozima. Después de la incubación durante 30 minutos a 4 ºC con agitación constante, el lisado celular se sonicó al 45% de amplitud durante 5 ciclos de pulsos de 30 segundos con pausas de 60 segundos. El lisado celular se centrifugó a 12,000 g durante 10 minutos a 4° C y se recogieron las proteínas solubles en el sobrenadante.
Las proteínas presentes en el sobrenadante de 12,000 g se concentraron añadiendo polietilenglicol (PEG) 8,000 (concentración final al 10%), seguido de agitación constante durante 12 horas a TA. Después de centrifugación a 12,000 g durante 30 min a 4° C, se descartó el sobrenadante y se resuspendió una alícuota de proteínas precipitadas en buffer de Laemmli y se calentó en un baño de agua hirviendo durante 3 min. La expresión de proteínas recombinantes se analizó mediante SDS-PAGE/Western blotting, aplicando las mismas cantidades de proteína total en cada pozo del gel. La membrana se trató con anticuerpos primarios contra Hsp90 o Hsp70 y luego con anticuerpo secundario IgG de burro anti-cabra conjugado con HRP (0.4 µg/mL, Santa Cruz Inc. SC-2020). La reacción se visualizó usando reactivo de transferencia Western Luminol. Se usaron como controles lisados de células bacterianas no transfectadas (DH5α o BL21 (DE3)) y células BL21 (DE3) transfectadas no inducidas. La proteína precipitada con PEG restante se resuspendió en PBS que contenía 2-ME 14,2 nM y se midió espectrofotométricamente a 280 nm frente a una curva de calibración de BSA.
Partículas virales pretratadas con HSPs y luego adicionadas al cultivo celular.
Las partículas de virus de los aislamientois de rotavirus TRUYO, WWM, WTEW, WT-15 y ECwt-o se trataron con HSPs durante 24 h a 4º C, mezclando viriones con proteínas recombinantes purificadas (Hsp70 o Hsp90) a concentraciones de 100, 50, 25, 12.5, 6.25 y 3.125 ng/mL. Los viriones tratados con HSPs se inocularon en células MCF-7 seguido de incubación durante 12 h a 37º C. Se utilizaron como control los viriones tratados con BSA (100 ng/mL) o Hsc70 (100 ng/ml) o se dejaron sin tratamiento. Después de la incubación, las células se congelaron y descongelaron dos veces antes del tratamiento con tampón RIPA (NaCl 150 mM, NP-40 al 1%, desoxicolato de sodio al 1%, SDS al 0,1% y Tris-HCl 50 mM, pH 7.5). Después de una incubación de 1 h a 37º C, el lisado se aplicó a una placa de ELISA recubierta con anticuerpos de cobayo contra proteínas estructurales de rotavirus. Se utilizaron anticuerpos primarios de cabra contra partículas de rotavirus y anticuerpos secundarios de conejo anti-cabra conjugados con HRP. La reacción se reveló con sustrato OPD en tampón citrato 100 mM, pH 5.0, y se leyó a 492 nm.
Análisis de transfección y expresión celular de proteínas rHSPs
Los plásmidos Hsp90α-<human>/pFLAG-CMV2, pCDNA3-FLAG Hsp90α, pCDNA3.1 (+) Hsp70 y pCMV-Hsp70 se transfectaron en monocapas de células MCF-7 (85% de confluencia). Plásmidos (0.1 ng/ml) en medio RPMI que contenía 10% de SFB (Gibco®), 20 U/mL de penicilina, 40 U/mL de estreptomicina (Invitrogen®) y 6 μg/mL de bromuro de hexadimetrina (Polybrene®, SIGMA Número CAS 2878- 55-4) se añadieron por separado a las células. Después de 48 horas de cultivo a 37° C en una cámara humidificada con 5% de CO2, las células se fijaron con metanol durante 45 min a TA y se lavaron con PBS. Las células se hicieron reaccionar con anticuerpos producidos en conejos, contra un péptido sintético derivado de Hsp70 de 20 aminoácidos, o mAb (143 µg/mL, Santa Cruz Biotechnology Inc.) al motivo de secuencia de Hsp90 DYKDDD (FLAG). Después de tres lavados con PBS, las células se incubaron con anticuerpos secundarios de bovino anti-conejo conjugado con FITC (0.8 µg/mL, Santa Cruz Biotechnology Inc. SC-2365) o anticuerpo secundario de cabra anti-ratón conjugado con FITC (0.8 µg/mL, Santa Cruz Biotechnology Inc. SC-2010). Las células que expresan rHsp70 o rHsp90 se inocularon por separado con TRUYO, WWM, WTEW, WT-15 o ECwt-o a las 24 h después de la transfección. Las células inoculadas se cultivaron durante 12 horas a 37º C. Las células no transfectadas o transfectadas con vectores vacíos se infectaron con aislados de rotavirus y se usaron como controles. Las células positivas al antígeno del virus se analizaron mediante tinción inmunocitoquímica y microscopía de inmunofluorescencia. Los datos se presentaron como promedio ± desviación estándar de tres experimentos independientes realizados por duplicado.
Células no transfectadas o transfectadas se lavaron con PBS y se trataron con el inhibidor de Hsp90 17AAG (80 nM) durante 1 h a 37º C. Las células se inocularon con WWM, ECwt-o, TRUYO, Wt1-5 o WTEW, a un MOI 0.8 y se incubaron durante 45 min a 4º C, antes de incubar durante 12 a 14 h a 37º C. Las células se fijaron con metanol frío y se lavaron con PBS. El análisis de la infección se realizó con tinción inmunocitoquímica utilizando anticuerpos primarios de cobayo anti-rotavirus e IgG de cabra anti-cobayo conjugado con HRP como anricuerpo secundario (0.133 µg/mL, Santa Cruz Biotechnology Inc. SC -2438), como se indicó anteriormente. Las células se lavaron dos veces con PBS y la reacción se desarrolló con sustrato de 3-amino-9-etilcarbazol (AEC) en tampón de acetato de Na 50 mM, pH 5.0 y H2O2 al 0.03%. Los mismos cubreobjetos desarrollados con AEC se lavaron con PBS y se ensayaron para determinar la expresión de rHsp90 y rHsp70 en las células transfectadas. Las células se hicieron reaccionar con los anticuerpos de conejo generado contra el péptido sintético de Hsp70 o el mAb contra el motivo de la secuencia de Hsp90 DYKDDDDK (FLAG). Después de tres lavados con PBS, las células se incubaron con anticuerpos secundarios IgG de cabra anti-conejo conjugado con FITC o IgG secundario de conejo anti-ratón conjugado con FITC. Se tomaron diez fotografías representativas.
Método estadístico
Los datos obtenidos fueron analizados empleando el programa GraphPad Prism v5.0®. Se aplicó una prueba de ANOVA de doble vía para determinar las diferencias estadísticamente significativas entre los promedios de cada tratamiento. El nivel de significancia se graficó de acuerdo con las siguientes convenciones: *= p <0.1 (Tendencia); ** = p <0.05 (Diferencia significativa); *** = p <0.01 (Diferencia altamente significativa) y ns = diferencias no significativas.
Resultados
Expresión de HSP, PDI e integrina β3 en la superficie celular
El análisis de citometría de flujo mostró que el porcentaje de células positivas para Hsp70, Hsp90, Hsp60, Hsp40, Hsc70, PDI e integrina β3 fue del 83.5%, 51.1%, 58.4%, 67.6%, 75.3%, 35.8% y 47.2% respectivamente. No se detectaron señales fluorescentes para las células de control que consisten en células no tratadas con anticuerpos, solo células tratadas con anticuerpos secundarios o células tratadas con anticuerpos no relacionados (Fig. 1A).
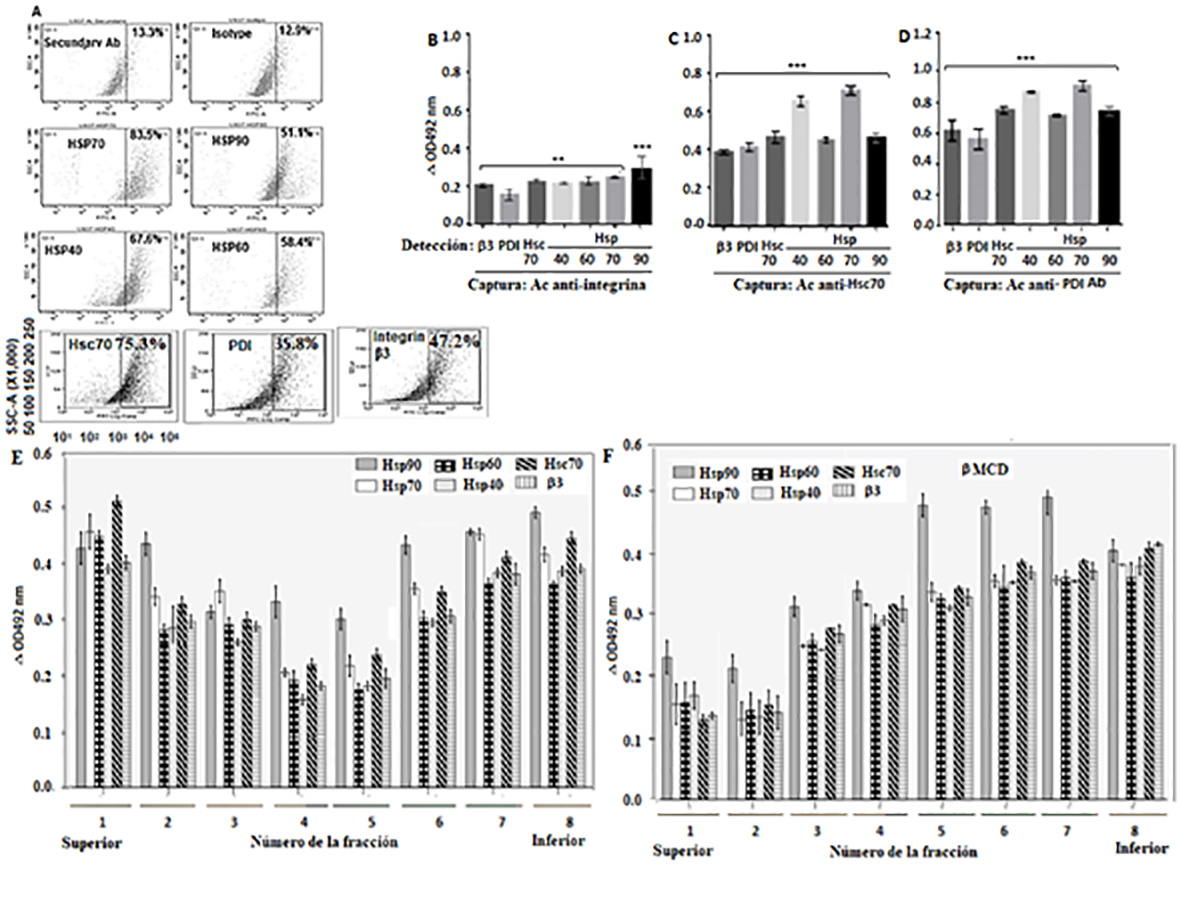
Figura 1 Localización de Hsp90, Hsp70, Hsp60, Hsp40, PDI e integrina β3 en la membrana celular. A. Las células se fijaron en PFD al 4% y se incubaron con anticuerpos primarios de cabra contra las proteínas mencionadas anteriormente. Se usó como anticuerpo secundario IgG de burro anti-cabra conjugado con Alexa Fluor 488. El análisis de inmunofluorescencia se realizó por duplicado utilizando un citómetro de flujo Cyan (Dako) y un software FlowJo. B-D. Se añadieron por separado fracciones enriquecidas en membranas celulares solubilizadas en tampón RIPA a placas de ELISA recubiertas con anticuerpos de captura de conejo contra Hsc70, PDI o integrina β3. Se añadieron anticuerpos primarios de cabra contra Hsp90, Hsp70, Hsp60, Hsp40, Hsc70, PDI o integrina β3 para la detección. Luego se adicionó anticuerpo secundario de burro anti-cabra conjugado con HRP. Las placas se leyeron a 492 nm y los resultados se expresaron como delta OD después de restar los valores del control. E, F. Las células con (E) o sin (F) tratamiento con metil-β-ciclodextrina se lisaron y se sometieron a centrifugación en gradiente de sacarosa. Luego, se recolectaron las fracciones del gradiente que contenían las balsas lipídicas (rafts). Las fracciones se solubilizaron en tampón de RIPA y se añadieron a placas de ELISA recubiertas con anticuerpos de conejo contra la integrina β3. Se añadieron anticuerpos primarios de cabra contra Hsp90, Hsp70, Hsp60, Hsp40 o integrina β3 y luego anticuerpo secundario de burro IgG anti-cabra conjugado con HRP. La reacción se visualizó usando OPD y las placas se leyeron a 492 nm. Los datos se muestran como promedio ± desviación estándar (DS) de tres experimentos independientes realizados por duplicado.
Interacciones de proteínas HSPs con PDI, integrina β3 y Hsc70 en la célula
Después de la detección con anticuerpos secundarios, los valores delta medios de absorbancia del ELISA indicaron que la integrina β3 capturada era capaz de interactuar con PDI, Hsc70, Hsp40, Hsp60, Hsp70 y Hsp90 (Fig. 1B). El Hsc70 capturado interactuó con la integrina β3, PDI, Hsp40, Hsp60, Hsp70 y Hsp90 (Fig. 1C). La PDI capturada interactuó con la integrina β3, Hsc70, Hsp40, Hsp60, Hsp70 y Hsp90 (Fig. 1D). Los resultados de ELISA sugieren que las proteínas celulares probadas están agrupadas probablemente formando un complejo, pero los detalles de las interacciones ya sean directos o indirectos, no están claros en este ensayo.
Proteínas HSP asociadas amicrodominios lipídicos (Rafts)
Mediante la técnica de ELISA, después de la detección con anticuerpos contra Hsp90, Hsp70, Hsp60, Hsp40, Hsc70 e integrina β3, las fracciones de baja densidad (2 a 7) de las células sin tratamiento con metil-β-ciclodextrina mostraron valores de absorbancia delta relativamente más altos para estas proteínas que los mostrados para fracciones de alta densidad (Fig. 1E). En el caso de las células tratadas con metil-β-ciclodextrina, las fracciones 5, 6, 7 y 8 mostraron valores de absorbancia delta relativamente más altos para las proteínas ensayadas que los encontrados para las fracciones de baja densidad (Fig. 1E).
Interacción de rotavirus WTEW, TRUYO, WWM, Wt1-5 y ECwt-o con proteínas celulares Hsp90, Hsp70, Hsp60, Hsp40, integrina β3, PDI y Hsc70
Para los ensayos con fracciones enriquecidas en membrana celular adheridas con virus, el análisis con anticuerpos mostró que todas las proteínas celulares ensayadas fueron capaces de interactuar con todos los aislados de rotavirus estudiados, de acuerdo con los valores de absorbancia delta relativos al control (virus solo en RIPA) (Fig. 2 A-E ). Aunque se encontraron valores diferenciales de absorbancia delta para algunas interacciones particulares de proteína aislada de rotavirus, Hsp70 mostró los valores de absorbancia delta más altos (Fig. 2 A-E ). De manera similar, se observaron interacciones positivas y significativas entre los aislamientos de rotavirus y las proteínas celulares estudiadas cuando se usaron células en lugar de fracciones enriquecidas en la membrana celular (Fig. 2 F-J ).

Figura 2 Unión de aislamientos de rotavirus a Hsp90, Hsp70, Hsp60, Hsp40, Hsc70, PDI e integrina β3. Las células MCF-7 (A-E) o sus fracciones enriquecidas de membrana celular (F-J) se incubaron con los aislamientos de rotavirus indicados. Después del lavado con PBS, las preparaciones se solubilizaron con RIPA y se aplicaron en placas de ELISA recubiertas con anticuerpos de captura de conejo contra proteínas estructurales de rotavirus. Para la detección, se usaron anticuerpos de cabra contra HSP, PDI, Hsc70, integrina β3 o contra rotavirus y se midió la reacción usando anticuerpo secundario de burro anti-IgG de cabra conjugado con HRP y se reveló usando sustrato de OPD.
Inhibición de la unión de rotavirus a la superficie celular con fragmentos F(ab')2 contra HSPs
Se incubó rotavirus a células MCF-7. Después de que se eliminó el exceso de virus, el análisis de los lisados con RIPA mediante ELISA de captura reveló que el tratamiento con el fragmento F(ab')2 disminuyó los antígenos de rotavirus de una manera dependiente de la concentración (Fig. 3 A-E ). Todo el fragmento F(ab')2 utilizado produjo efectos inhibidores comparables en los cinco aislados de rotavirus analizados.
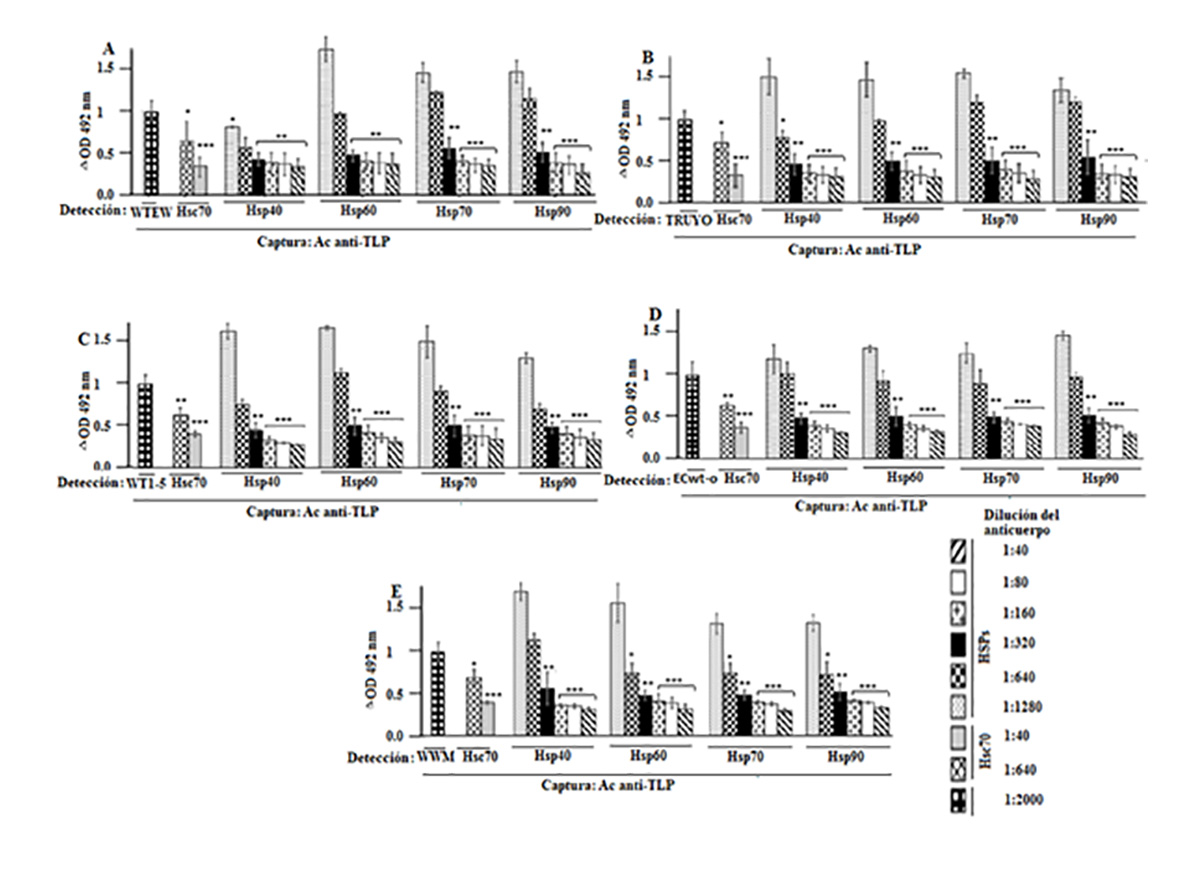
Figura 3 Bloqueo de la unión de rotavirus a la superficie celular con anticuerpos contra Hsp90, Hsp70, Hsp60, Hsp40 y Hsc70. A-F. Las células MCF-7 se incubaron con fragmentos de anticuerpo F(ab')2 contra Hsp90, Hsp70, Hsc70, Hsp60 o Hsp40 en las diluciones indicadas, seguido de incubación con los aislados de rotavirus indicados en los paneles. Después del lavado con PBS, las células se solubilizaron con tampón RIPA antes de la adición a los pozos de ELISA recubiertos con anticuerpos de captura de cobayo contra proteínas estructurales de rotavirus. Para la detección, se usaron anticuerpos de conejo contra partículas de rotavirus y se midió la reacción usando IgG secundario de burro anti-conejo conjugado con HRP y se reveló utilizando sustrato de OPD. Los datos son valores medios ± desviación estándar de tres experimentos independientes realizados por duplicado.
Co-inmunoprecipitación de HSP y antígenos estructurales de aislados de rotavirus
El análisis de SDS-PAGE/Western blotting de las proteínas coinmunoprecipitadas mostró cuatro bandas reactivas correspondientes a Hsp90, Hsp70, Hsp60 y Hsp40 (Fig. 4 A ). Estos resultados sugieren que los aislados de rotavirus estudiados pudieron unirse a estas HSPs. Estos resultados se confirmaron mediante biotinilación de proteínas de la superficie celular antes de SDS-PAGE/análisis de transferencia Western de las HSPs separadas (Anexo 1). El análisis de co-inmunoprecipitación de células MA104 incubadas con Wt1-5 mostró que sólo la integrina β3, Hsc70 y PDI co-inmunoprecipitaban, pero no Hsp90, Hsp70, Hsp60 o Hsp40 (Fig. 4 B ). Estos resultados sugieren que el rotavirus Wt1-5 adaptado a células tumorales ha ganado función ya que puede interactuar con estas HSPs de la superficie celular.
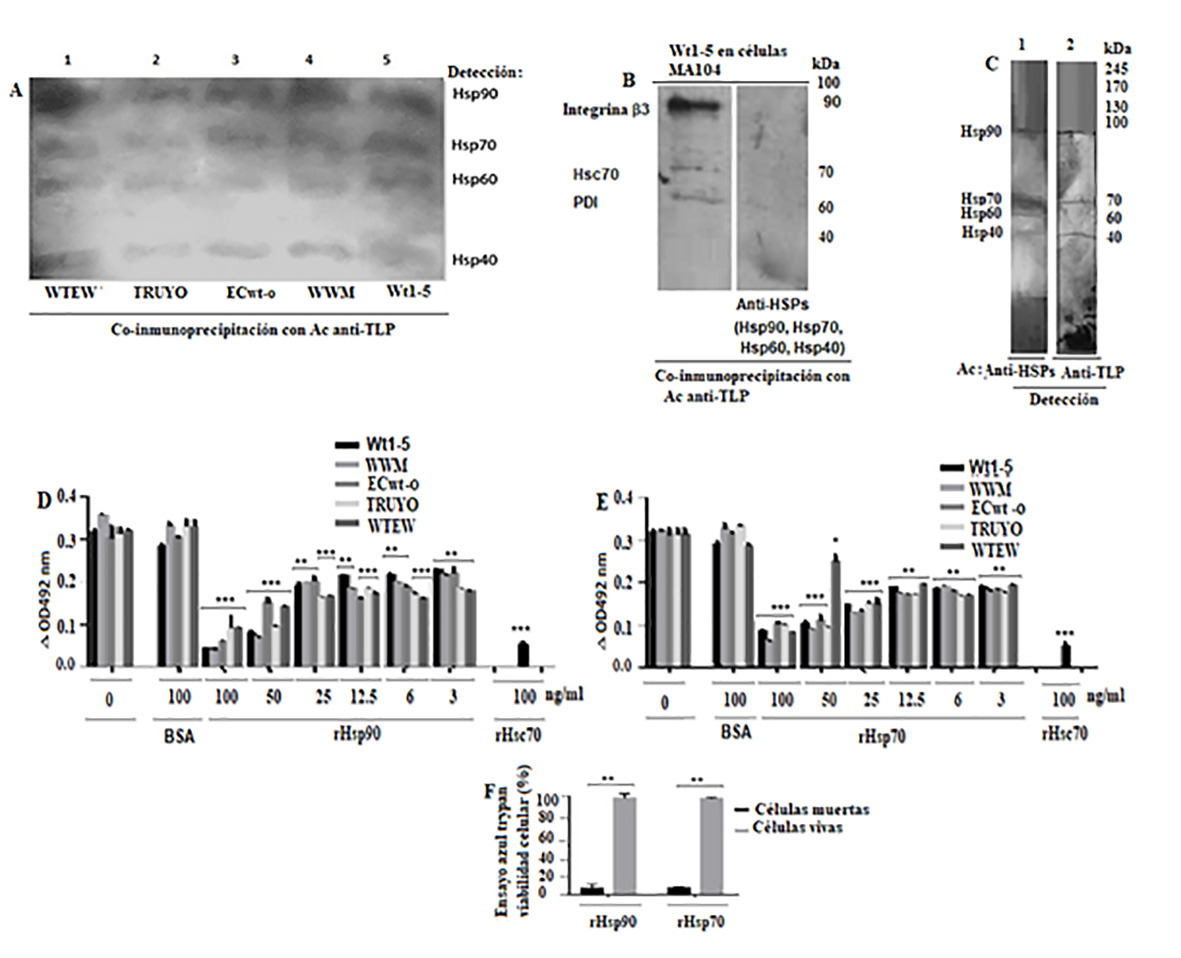
Figura 4 Unión de partículas de rotavirus a Hsp90, Hsp70, Hsp60 y Hsp40, e inhibición de la infección por rotavirus al competir con HSPs de la célula. A. Las fracciones enriquecidas en la membrana celular de MCF-7 se incubaron por separado con los aislados de rotavirus indicados. Las fracciones de membrana celular solubilizadas se inmunoprecipitaron con anticuerpos acoplados a perlas de agarosa contra partículas de rotavirus. Las proteínas inmunoprecipitadas se analizaron mediante SDS-PAGE/Western blotting usando anticuerpos contra las HSPs indicadas y detección basada en luminol. B. Se incubaron fracciones enriquecidas en la membrana celular MA104 con aislado de rotavirus. Las fracciones de membrana celular solubilizadas se inmunoprecipitaron con anticuerpos acoplados a perlas de agarosa contra partículas de rotavirus. Las proteínas inmunoprecipitadas se analizaron mediante SDS-PAGE/Western blotting usando anticuerpos contra la integrina β3, Hsc70, PDI o HSP y detección basada en luminol. C. Se realizó un ensayo de VOBPA lisando células MCF-7 con tampón RIPA seguido de PAGE no desnaturalizante y no reductor y transferencia Western. La membrana se incubó con rotavirus WTEW, se lavó rápidamente y se trató con glutaraldehído al 2,5%. La membrana se trató con una mezcla de anticuerpos contra las HSPs (carril 1) indicadas anteriormente o anticuerpos contra las TLPs de rotavirus (carril 2). Las reacciones se revelaron con reactivo Western Luminol de transferencia. D, E. Los aislamientos de rotavirus (TRUYO, WWM, WTEW, WT-15 y ECwt-o) a un MOI 0.8 se trataron con las concentraciones indicadas de rHsp90 (D) o rHsp70 (E) antes de la incubación con células MCF-7. Los aislamientos de rotavirus se trataron con BSA (100 ng/mL) o Hsc70 (100 ng/ml) o se dejaron sin tratar para utilizarlos como controles. Las células congeladas y descongeladas se trataron con tampón RIPA y el lisado se aplicó sobre placas ELISA recubiertas con anticuerpos de captura de cobayo contra partículas de rotavirus. La detección se realizó con anticuerpos primarios de conejo contra partículas de rotavirus. La reacción se reveló con anticuerpos secundarios IgG de cabra anti-conejo conjugados con HRP y se revelo con sustrato de OPD. F. Se determinó la viabilidad celular de las células tratadas con proteínas recombinantes (rHsp90 o rHsp70) (100 µg/mL) usando una prueba de exclusión con azul de tripano. Los datos se presentan como promedios ± desviación estándar (DS) de tres experimentos independientes realizados por duplicado.
Análisis de “VOPBA” de rotavirus WTEW en proteínas de células MCF-7
La membrana que se incubó con anticuerpos anti-rotavirus de conejo mostró cuatro bandas reactivas que casi coincidían con las bandas de las proteínas Hsp90, Hsp70, Hsp60 y Hsp40 detectadas con los respectivos anticuerpos en un carril paralelo de la membrana de transferencia Western (Fig. 4C). Estos hallazgos confirman que el rotavirus WTEW interactúa con estas HSP.
Inhibición de la infección por rotavirus al incubar viriones con rHsp90 y rHsp70
El análisis ELISA de captura de los lisados celulares después de la incubación durante 12 horas a 37° C mostró que los antígenos virales disminuyeron mediante el pretratamiento con las proteínas recombinantes de una manera dependiente de la concentración. El resultado se comparó respecto a cuando se utilizó el respectivo aislamiento de virus sin el pretratamiento de proteína recombinante como un control (100%) (Fig. 4D). La inhibición más baja del antígeno viral producida por rHsp90 fue de 42.9%, 38.5%, 26.1%, 41.3% y 32.5% para los aislamientos WTEW, TRUYO, ECwt-o, WWM y Wt-15, respectivamente, mientras que la inhibición más alta observada fue 72.0%, 79.0%, 75.5%, 91.2% y 88.0%, respectivamente (Fig. 4D). Se encontraron efectos inhibidores similares cuando las partículas virales se pretrataron con rHsp70. El efecto inhibidor más bajo osciló entre el 22.8% y el 43.5%, mientras que el mayor osciló entre el 60% y el 80% (Fig. 4E). El pretratamiento de partículas virales con Hsc70 (100 ng/mL), utilizado como control positivo, redujo la acumulación de antígeno viral en un 82.2% para WTEW. La viabilidad celular de las células MCF tratadas con rHsp90 y rHsp70 fue del 96% y 94%, respectivamente, de acuerdo con la prueba de exclusión con azul tripán (Fig. 4F). Estos resultados sugieren que las HSPs recombinantes ensayadas se unen de forma estable a partículas virales que les impiden unirse a las contrapartes de la superficie celular. Es menos probable que la unión de rHsp90 o Hsp70 haga inactiva la partícula viral.
Inhibición de la infección de rotavirus por anticuerpos contra HSPs
Como se muestra en la Fig. 5 A-E, el tratamiento con anticuerpos provocó una disminución significativa en la infección por rotavirus según se determinó mediante análisis inmunocitoquímico de los antígenos estructurales de rotavirus acumulados a las 12 h.p.i. La concentración de anticuerpo anti-Hsp90 más alta utilizada fue capaz de reducir la infectividad viral en un 45% (WTEW), 65% (TRUYO), 55% (Wt1-5), 41% (ECwt-o) y 42% (WWM) (Fig.5A). Los anticuerpos contra Hsp70 redujeron la infectividad en un 40% (WTEW), 48% (TRUYO), 55% (WT1-5), 55% (ECwt-o) y 56% (WWM) (Fig. 5 A-E ). La reducción de la infectividad del 50% (WTEW), 50% (TRUYO), 66% (Wt1-5), 69% (ECwt-o) y 40% (WWM) (Fig.5A-E) fue producida por anticuerpos Hsp60, mientras que los anticuerpos contra Hsp40 redujeron la infectividad en un 60% (WTEW), 67% (TRUYO), 51% (Wt1-5), 40% (ECwt-o) y 32% (WWM). Estos resultados sugieren que las HSP probadas están implicadas significativamente en la infección de células MCF-7 con los aislamientos de rotavirus mencionados anteriormente.

Figura 5 Disminución de la infección por rotavirus con anticuerpos contra Hsp90, Hsp70, Hsp60 o Hsp40. A-E. Las células MCF-7 en medio de cultivo se trataron por separado con una dilución en serie de anticuerpos contra las HSPs indicadas, durante 45 minutos a 4 ºC, se lavaron con DMEM y luego se incubaron con los aislados de rotavirus indicados en los paneles. Después de 12 h.p.i., se detectaron antígenos de rotavirus estructurales mediante un ensayo de inmunocitoquímica. Se utilizaron como control células inoculadas con virus sin pretratamiento con anticuerpos. Los datos se expresaron como porcentaje de células infectadas. Los datos se muestran como porcentajes promedios ± desviación estándar (DS) de tres experimentos independientes realizados por duplicado.
Efecto de los inhibidores de Hsp90 y Hsp70 sobre la infección por rotavirus
La preincubación con 17AAG a la concentración más alta utilizada (80 nM) redujo el porcentaje de infección por virus entre un 4% y un 70%, dependiendo del aislado de virus utilizado (Fig. 6 A ). NVP-AUY922 redujo la infección viral entre un 60% y un 66% y el radicicol entre un 80% y un 90%, dependiendo del aislamiento de rotavirus implicado en la concentración más alta de inhibidor utilizado (16 nM y 100 nM, respectivamente) (Fig. 6 B,D). La preincubación con VER-15008 condujo a una inhibición viral del 63% al 80%, mientras que la quercetina inhibió la infección viral del 81% al 90%, dependiendo de los aislados de rotavirus utilizados cuando los inhibidores se utilizaron en sus concentraciones más altas (75 µM y 600 µM, respectivamente) (Fig. 6 C, E ). La preincubación de células con quercetina 300 µM a 43° C durante 30 min antes de la infección con WTEW o Wt1-5, condujo a una inhibición de la infección viral en un 100% y 80%, respectivamente, mientras que la preincubación a 37° C durante 1 hora inhibió la infección por virus en un 70% en ambos casos (Fig. 6 F ). Las concentraciones más altas de los inhibidores ensayados se seleccionaron probando sus efectos sobre la viabilidad celular. La viabilidad celular, determinada por la prueba de exclusión con azul tripán, fue del 99.5%, 98.6%, 99.5%, 94.1% y 90.0% para 17AAG, NVP-AUY922, quercetina, radicicol y VER-155008, respectivamente (Fig. 6 G ). En general, estos resultados son consistentes con la participación de Hsp90 y Hsp70 en el proceso de infección de rotavirus en células MCF-7.
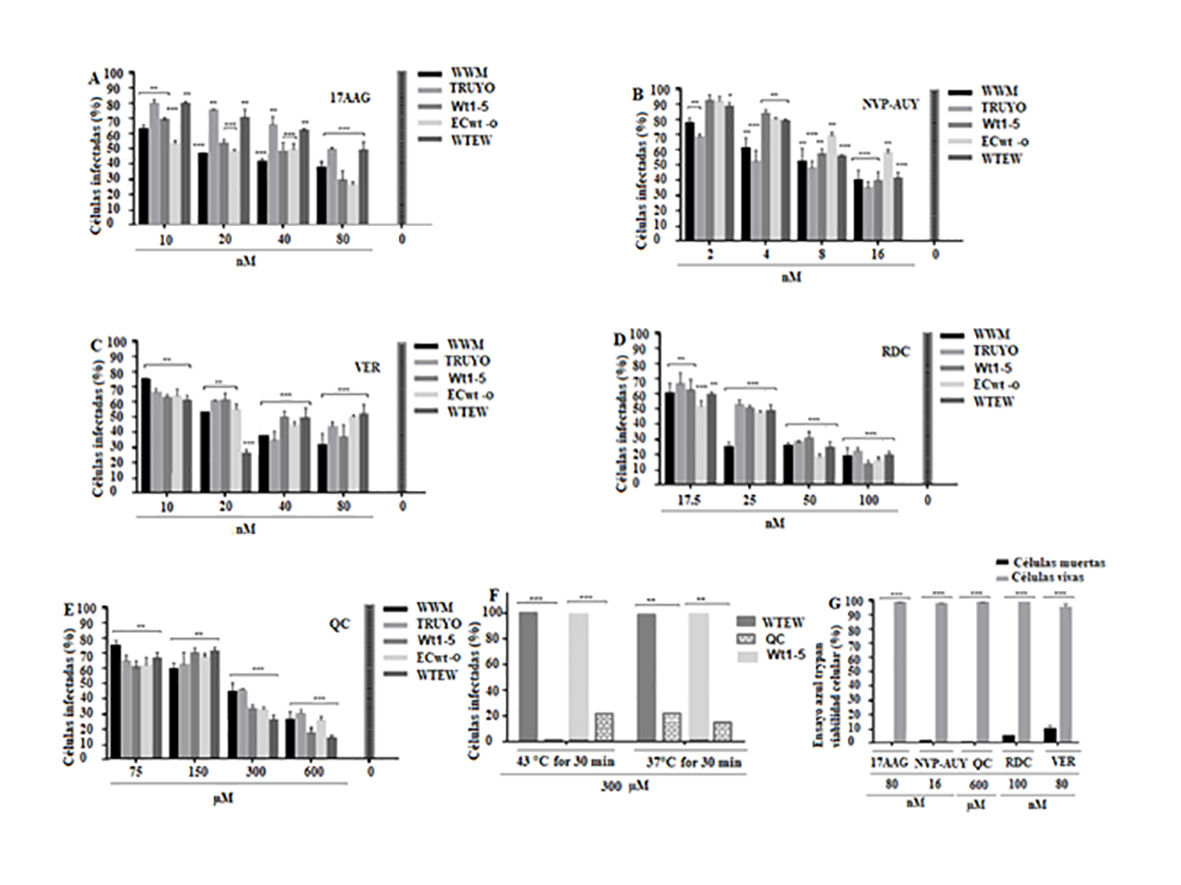
Figura 6 Efecto de la inhibición de Hsp90 y Hsp70 sobre la infección por rotavirus. A-E. Se incubaron células MCF-7 en placas de 96 pozos con diferentes concentraciones de inhibidores de Hsp90 (17AAG, NvP Auy o Radicicol) o inhibidores de Hsp70 (quercetina (QC) o VER155008) durante 1h 37° C. Las células se lavaron con DMEM y se inocularon con los aislados de rotavirus indicados en los paneles durante 45 min a 4° C y luego se incubaron durante 12 ha 37º C. Los antígenos estructurales virales se detectaron mediante un ensayo de inmunocitoquímica y la infección se expresó como porcentaje de células infectadas. F. Las células se incubaron con QC 300 M durante 30 min a 43° C o 37° C y el porcentaje de células infectadas se determinó mediante un ensayo de inmunocitoquímica usando anticuerpos de conejo contra antígenos estructurales de rotavirus. G. Ensayo de viabilidad de células de azul tripán de células tratadas con 600 µM de los inhibidores indicados en la parte inferior del panel. Los datos se muestran como porcentajes medios ± desviación estándar de tres experimentos independientes realizados por duplicado.
Efecto de la sobreexpresión de rHsp90 y rHsp70 sobre la infección por rotavirus
Las células transfectadas mostraron las señales positivas respectivas para cualquiera de las proteínas recombinantes (Fig. 7 A-7D ) mientras que las células no transfectadas mostraron fluorescencia negativa (Fig. 7 E ). Se utilizaron como control los porcentajes de células infectadas sin transfección y sin tratamiento con inhibidor. Estos porcentajes de infección fueron 90%, 82%, 61%, 80% y 70% para WWM, TRUYO, Wt1-5, ECwt-o y WTEW, respectivamente, mientras que estos porcentajes aumentaron a 95%, 95%, 70%, 96% y 83%, respectivamente, en células que expresan rHsp90 (Fig. 7 F ). En el caso de la expresión de rHsp70, todos los aislados virales probados, excepto WWM, mostraron porcentajes de infección significativamente mayores en comparación con los mostrados por las células de control (Fig. 7 F ). El tratamiento de las células con el inhibidor de Hsp90 (17AAG) redujo los porcentajes de infección para todos los aislados de rotavirus analizados entre un 40% y un 71% en relación con los porcentajes observados en las células de control (Fig. 7 F ). Estos resultados confirman que los aislados de rotavirus estudiados utilizan Hsp90 y Hsp70. Se muestran imágenes representativas para cada uno de los tratamientos descritos en la figura 7F (Fig. 7 G )
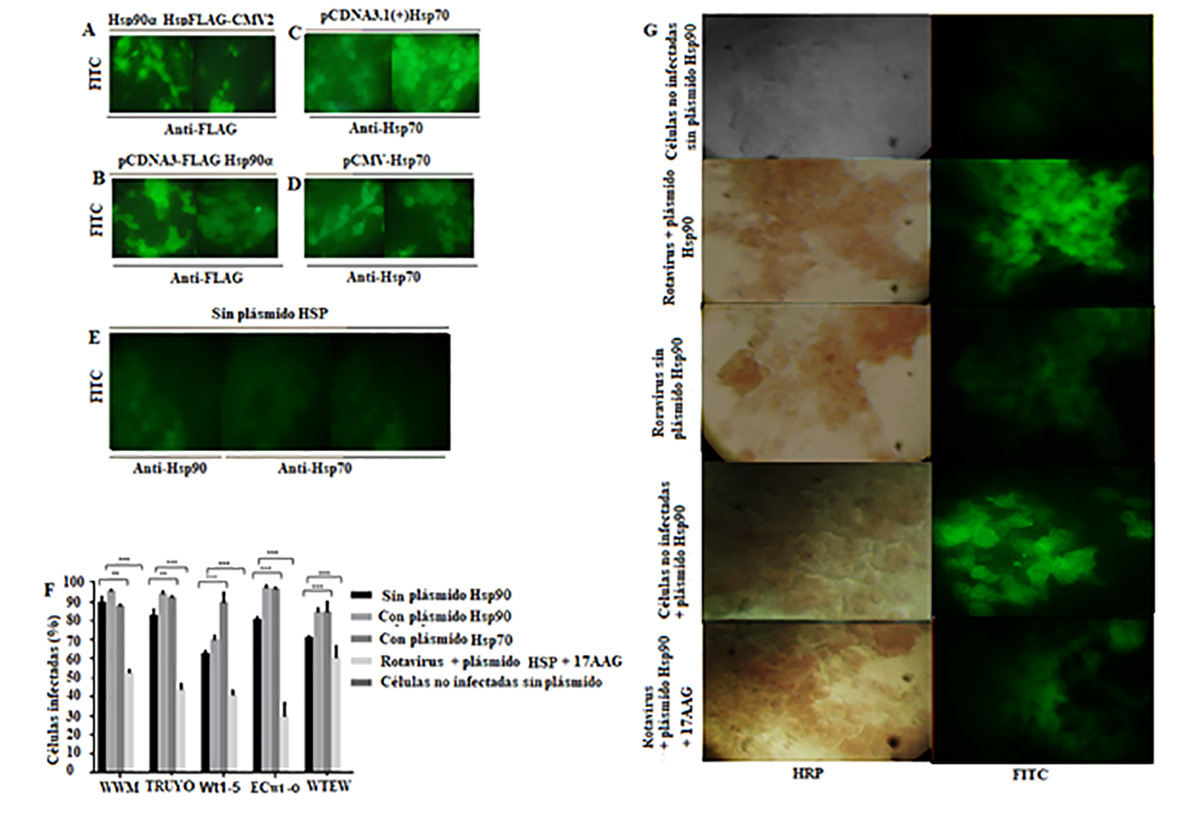
Figura 7 Aumento de la infección por rotavirus por sobreexpresión de rHsp90 y rHsp70. Se transfectaron células MCF-7 con los plásmidos indicados que expresan rHsp90 o rHsp70. Después de 24 h de transfección, las células se infectaron con los aislamientos de rotavirus indicados durante 12 ha 37º C. A-E. La expresión de proteínas recombinantes se detectó mediante inmunofluorescencia usando anticuerpos específicos para ellas. Se muestran fotografías representativas. Se utilizaron células no transfectadas como control. F. Las células transfectadas o no transfectadas con plásmidos que contienen Hsp90 o Hsp70 se infectaron por separado o se dejaron sin infectar con los aislamientos de rotavirus indicados. Se trataron alícuotas de células transfectadas que expresaban rHsp90 con el inhibidor 17AAG antes de la infección con aislados de rotavirus. Las células transfectadas con el plásmido vacío o no transfectadas se usaron como control. La infección se expresó como porcentaje de células positivas a antígenos estructurales de rotavirus. G. Fotografías representativas de células transfectadas infectadas o no infectadas con los aislamientos de rotavirus. Los antígenos de rotavirus se detectaron con anticuerpos secundarios conjugados con HRP (marrón rojizo), y las HSP recombinantes (rHsp90 o rHsp70) se detectaron con anticuerpos secundarios conjugados con FITC (verde). Se utilizó 17AAG como inhibidor de Hsp90. Las células de control consistían de células no infectadas transfectadas con plásmidos de HSPs (ya sea rHsp90 o rHsp70) y células infectadas con rotavirus o no infectadas, pero transfectadas con el plásmido vacío (plásmido -HSP). Los datos se muestran como porcentajes promedios ± desviación estándar (DS) de tres experimentos independientes realizados por duplicado.
Discusión
Las últimas décadas se han caracterizado por la búsqueda de terapias alternativas para el tratamiento del cáncer a partir de los conocidos efectos adversos asociados al uso de la quimioterapia y radioterapia tradicionales. La viroterapia representa una de estas terapias alternativas, ya que los avances recientes han demostrado que el uso de virus oncolíticos es una alternativa segura y eficaz 39. El uso de virus oncolíticos debe cumplir con algunos requisitos, incluida la infección limitada o nula de células distintas de las células tumorales, lo cual depende de su tropismo único por las células tumorales humanas que exhiben receptores celulares que facilitan la entrada viral.
El presente estudio proporciona evidencia de que un porcentaje relativamente alto de células MCF-7 expresa en su superficie celular las HSPs examinadas en este estudio. La presencia de HSPs en las fracciones enriquecidas en membrana celular también está respaldada por su interacción directa o indirecta con Hsc70 y la integrina β3, las cuales se han informado previamente como proteínas marcadoras de la superficie de la membrana celular 40,41. Además, los resultados obtenidos de las fracciones enriquecidas en membranas sugieren que se están produciendo interacciones cuantitativas diferenciales entre los aislamientos de rotavirus y las proteínas celulares. Sin embargo, si estas interacciones son directas o indirectas, no está claro a partir de estos experimentos particulares, ya que no se pueden excluir interacciones mediadas por otras proteínas celulares.
También se proporciona evidencia de que Hsp90, Hsp70, Hsp60 y Hsp40 están implicadas en la infección por rotavirus de las células MCF-7. Los efectos inhibidores sobre la infección viral inducidos por el tratamiento previo de las células con anticuerpos contra estas proteínas celulares sugieren que están involucradas en el proceso de entrada del virus. Esta conclusión se ve fortalecida por el hecho de que estas proteínas celulares se encuentran en microdominios lipídicos (rafts) en donde forman un complejo con PDI, Hsc70 e integrina β3, las cuales se han reportado que forman parte de la maquinaria celular involucrada en la entrada de rotavirus 27. El uso de los fragmentos F(ab')2 en lugar de inmunoglobulinas completas reduce la posibilidad de que los anticuerpos anti-HSPs estén produciendo un impedimento estérico.
Además, el efecto negativo producido sobre la infección de rotavirus por el pretratamiento de células con inhibidores específicos para Hsp90 y Hsp70 también está apoyando la implicación de estas proteínas celulares en el proceso de infección por rotavirus. En esta línea de pensamiento, el aumento de la infección viral provocada por la sobreexpresión de Hsp90 y Hsp70 recombinantes también favorece la participación de estas proteínas celulares en la infección producida por los aislamientos de rotavirus ensayados. La identificación de receptores virales en células no solo es útil para diseñar estrategias antivirales sino también para comprender el tropismo viral en el contexto de la especificidad requerida por la viroterapia oncolítica.
Usamos la línea celular de adenocarcinoma de glándula mamaria humana MCF-7 como un sistema modelo para identificar algunas de las proteínas de la superficie celular involucradas en la unión de rotavirus que median la entrada viral. Encontramos que la unión de las partículas de rotavirus a las HSPs de la superficie celular que funcionan como receptores desencadenó la infección viral que se detectó en términos del porcentaje de células positivas a los antígenos virales o la cantidad total de antígenos virales acumulados en un momento posterior a la infección, lo cual es compatible con al menos un ciclo de infección viral. El uso de Hsp90 y Hsp70 recombinantes para el pretratamiento de viriones de rotavirus antes de inocular este complejo a las células MCF-7, nos permitió concluir que estas proteínas celulares compiten por la unión a rotavirus con su contraparte localizada en la superficie celular, lo que conduce a una disminución de la infección. La interacción entre viriones y Hsp90 y Hsp70 se puso de manifiesto adicionalmente mediante ELISA de captura y ensayo de co-inmunoprecipitación.
Desde el punto de vista del uso de receptores, es muy interesante destacar que los rotavirus parecen utilizar vías de entrada alternativas. En el presente trabajo, los inhibidores de las HSPs y los anticuerpos contra las HSPs no solo fueron incapaces de inhibir completamente la infección por rotavirus, sino que los aislamientos de rotavirus probados respondieron de manera diferente a estos inhibidores y anticuerpos. Cabe destacar que la quercetina inhibió la infección por rotavirus a mayor temperatura. Se ha informado que la quercetina aumenta su efecto inhibidor sobre Hsp70 a temperaturas más altas 42. Aunque el efecto de inhibir Hsc70, PDI e integrina β3 sobre la infección por rotavirus no se probó en el presente estudio, estudios previos han demostrado que estas proteínas celulares se utilizan para la entrada de rotavirus en las células MA104 40,41. Sin embargo, la presencia de estas proteínas en la superficie de células MCF-7 formando complejos con HSPs en microdominios lipídicos y su probable interacción directa con partículas de rotavirus sugieren que podrían ser utilizadas por los aislamientos de rotavirus ensayados en el presente estudio. Estos resultados sugieren que el rotavirus parece haber evolucionado para usar diferentes tipos de moléculas de la superficie celular para ingresar a la célula diana. Las moléculas chaperonas, como las HSPs, además de las moléculas de unión (por ejemplo, ácido acetilsalicílico, integrinas) y moléculas con actividad redox (p. Ej., PDI, Erp57) se han propuesto como receptores candidatos que darían fundamento a vías de entrada alternativas 31. La presencia de varias proteínas de la superficie celular que desempeñan la función de receptores o co-receptores para la entrada de rotavirus nos permite dar más apoyo al modelo de interacción secuencial y alternativo propuesto 43, que incluye la unión y cambios conformacionales de proteínas estructurales de rotavirus antes de la penetración en la célula diana. Sin embargo, nuestros resultados no excluyen que otras moléculas de la superficie celular puedan ayudar a facilitar la entrada de rotavirus en la célula huésped.
Nuestros hallazgos destacan que los aislamientos de rotavirus estudiados utilizan Hsp90, Hsp70, Hsp60 y Hsp40 como receptores para infectar células MCF-7. Las HSPs son una familia de proteínas celulares implicadas en la actividad chaperona dependiente de la hidrólisis de ATP. Se ha informado que Hsc70 induce cambios conformacionales en partículas de rotavirus 26. Concluimos que los aislamientos de rotavirus adaptados a células tumorales estudiados en el presente trabajo pueden infectar con éxito la línea celular de adenocarcinoma de glándula mamaria humana MCF-7 al unirse a Hsp90, Hsp70, Hsp60 y Hsp40 presente en su superficie celular. Este tropismo de estos aislamientos rotavirales dependiente del receptor merece ser destacado, ya que los receptores determinan la entrada del virus en la célula hospedera y las HSPs se sobreexpresan en una variedad de tumores humanos 22. Sin embargo, queda por examinar si las células no tumorales con mayor respuesta al choque térmico son una diana de los aislamientos de rotavirus estudiados aquí. Tomados en conjunto, nuestros hallazgos sugieren que los aislamientos de rotavirus estudiados en el presente trabajo merecen ser investigados más a fondo como un agente oncolítico potencial. Dado que esencialmente toda la población tiene anticuerpos contra el rotavirus, debe buscarse un balance entre la respuesta inmunológica antiviral y la respuesta inmunitaria antitumoral.
REFERENCIAS
1. Dock G. The influence of complicating diseases upon leukaemia. Am J Med Sci. 1904; 127: 1827-1924. [ Links ]
2. Dock G. Rabies virus vaccination in a patient with cervical carcinoma. Am J Med Sci. 1904; 127: 563. [ Links ]
3. Schirrmacher V. From chemotherapy to biological therapy: A review of novel concepts to reduce the side effects of systemic cancer treatment (Review). Int J Oncol. 2019; 54: 407-419. doi: 10.3892/ijo.2018.4661. [ Links ]
4. Pol JG, Lévesque S, Workenhe ST, Gujar S, Le Boeuf F , Clements DR, et al. Trial Watch: Oncolytic viro-immunotherapy of hematologic and solid tumors. Oncoimmunology. 2018; 7(12):e1503032. doi: 10.1080/2162402X.2018.1503032. [ Links ]
5. Meyers DE, Thakur S, Thirukkumaran CM, Morris DG. Oncolytic virotherapy as an immunotherapeutic strategy for multiple myeloma. Blood Cancer J. 2017; 7: 640. doi: 10.1038/s41408-017-0020-0. [ Links ]
6. Marelli G, Howells A, Lemoine NR, Wang Y. Oncolytic viral therapy and the immune system: a double-edged sword against cancer. Front. Immunol. 2018; 9: 866. doi: 10.3389/fimmu.2018.00866. [ Links ]
7. Ekblad M, Hallden G. Adenovirus-based therapy for prostate cancer. Curr Opin Mol Ther. 2010;12(4):421-31. [ Links ]
8. Matthews KS, Alvarez RD, Curiel DT. Advancements in adenoviral based virotherapy for ovarian cancer. Adv Drug Deliv Rev. 2009;61(10):836-41. doi: 10.1016/j.addr.2009.04.012. [ Links ]
9. Zhang Y-F, Zhang B-C, Zhang A-R, Wu T-T, Liu J, YuL-F, et al. Co-transduction of ribosomal protein L23 enhances the therapeutic efficacy of adenoviral-mediated p53 gene transfer in human gastric cancer. Oncol Rep. 2013;30(4):1989-95. doi: 10.3892/or.2013.2663. [ Links ]
10. Fu Y-J, Du J, Yang R-J, Yin L-T, Liang A-H. Potential adenovirus-mediated gene therapy of glioma cancer. Biotechnol. Biotechnol Lett. 2010;32(1):11-8. doi: 10.1007/s10529-009-0132-0. [ Links ]
11. Lundstrom K. New frontiers in oncolytic viruses: optimizing and selecting for virus strains with improved efficacy. Biologics. 2018; 12: 43-60. doi: 10.2147/BTT.S140114. [ Links ]
12. Ranki T, Pesonen S, Hemminki A, Partanen K, Kairemo K, Alanko T, et al. Phase I study with ONCOS-102 for the treatment of solid tumors - an evaluation of clinical response and exploratory analyses of immune markers. J Immunother Cancer. 2016; 4: 17. doi: 10.1186/s40425-016-0121-5. [ Links ]
13. Markert JM, Razdan SN, Kuo H-C, Cantor A, Knoll A, Karrasch M, et al. A phase 1 trial of oncolytic HSV-1, G207, given in combination with radiation for recurrent GBM demonstrates safety and radiographic responses. Mol Ther. 2014; 22(5): 1048-55. doi: 10.1038/mt.2014.22. [ Links ]
14. Filley AC, Dey M. Immune System, Friend or Foe of Oncolytic Virotherapy? Front Oncol. 2017;7:106. doi: 10.3389/fonc.2017.00106. [ Links ]
15. Singh PK, Doley J, Kumar GR, Sahoo AP, Tiwari AK. Oncolytic viruses & their specific targeting to tumour cells. Indian J Med Res. 2012; 136: 571-584. [ Links ]
16. Howells A, Marelli G, Lemoine NR, Wang Y. Oncolytic viruses-interaction of virus and tumor cells in the battle to eliminate cancer. Front Oncol. 2017; 7:195. doi: 10.3389/fonc.2017.00195. [ Links ]
17. Fountzilas C, Patel S, Mahalingam D. Review: Oncolytic virotherapy, updates and future directions. Oncotarget. 2017;8(60):102617-102639. doi: 10.18632/oncotarget.18309. [ Links ]
18. Lawler SE, Speranza M-C, Cho C-F, Chiocca EA. Oncolytic viruses in cancer treatment: a review. JAMA Oncol. 2017;3(6):841-849. doi: 10.1001/jamaoncol.2016.2064. [ Links ]
19. Fukuhara H, Ino Y, Todo T. Oncolytic virus therapy: A new era of cancer treatment at dawn. Cancer Sci. 2016;107(10):1373-1379. doi: 10.1111/cas.13027. [ Links ]
20. Hariharan S, Subramanian S, Pooja C, Renukadevi G, Hashik M. Heat shock proteins-a mini review. Int J. 2014; 1: 65-73. [ Links ]
21. Kim MY, Oglesbee M. Virus-heat shock protein interaction and a novel axis for innate antiviral immunity. Cells. 2012; 1(3): 646-666. doi: 10.3390/cells1030646 [ Links ]
22. Ciocca DR, Calderwood SK. Heat shock proteins in cancer: diagnostic, prognostic, predictive, and treatment implications. Cell Stress Chaperones. 2005; 10(2): 86-103. doi: 10.1379/CSC-99r.1 [ Links ]
23. Calderwood SK, Gong J. Heat Shock proteins promote cancer: it's a protection racket. Trends Biochem Sci. 2016; 41(4): 311-323. doi: 10.1016/j.tibs.2016.01.003 [ Links ]
24. Dutta D, Bagchi P, Chatterjee A, Kant NM, Mukherjee A, Chattopadhyay S, et al. The molecular chaperone heat shock protein-90 positively regulates rotavirus infectionx. Virology. 2009; 391(2): 325-33. doi: 10.1016/j.virol.2009.06.044. [ Links ]
25. Guerrero CA, Bouyssounade D, Zárate S, Isa P, López T, Espinosa R, et al. Heat shock cognate protein 70 is involved in rotavirus cell entry. J Virol. 2002; 76(8): 4096-102. doi: 10.1128/jvi.76.8.4096-4102.2002. [ Links ]
26. Perez-Vargas J, Romero P, Lopez S, Arias CF. The peptide-binding and ATPase domains of recombinant hsc70 are required to interact with rotavirus and reduce its infectivity. J Virol. 2006; 80(7): 3322-31. doi: 10.1128/JVI.80.7.3322-3331.2006. [ Links ]
27. Calderon MN, Guerrero CA, Acosta O, Lopez S, Arias CF. Inhibiting rotavirus infection by membrane-impermeant thiol/disulfide exchange blockers and antibodies against protein disulfide isomerase. Intervirology. 2012;55(6):451-64. doi: 10.1159/000335262. [ Links ]
28. Guerrero CA, Méndez E, Zárate S, Isa P, López S, Arias CF. Integrin alpha(v)beta(3) mediates rotavirus cell entry. Proc Natl Acad Sci U S A. 2000;97(26):14644-9. doi: 10.1073/pnas.250299897. [ Links ]
29. Londrigan SL, Hewish MJ, Thomson MJ, Sanders GM, Mustafa H, Coulson BS. Growth of rotaviruses in continuous human and monkey cell lines that vary in their expression of integrins. J Gen Virol. 2000; 81(Pt 9): 2203-2213. doi: 10.1099/0022-1317-81-9-2203. [ Links ]
30. Abdelhakim AH, Salgado EN, Fu X, Pasham M, Nicastro D, Kirchhausen T, et al. Structural correlates of rotavirus cell entry. PLoS Pathog. 2014; 10(9): e1004355. doi: 10.1371/journal.ppat.1004355. [ Links ]
31. Guerrero CA, Guerrero RA, Silva E, Acosta O, Barreto E. Experimental Adaptation of Rotaviruses to Tumor Cell Lines. PLoS One. 2016; 11(2) :e0147666. doi: 10.1371/journal.pone.0147666. [ Links ]
32. Gualtero DF, Guzmán F, Acosta O, Guerrero CA. Amino acid domains 280-297 of VP6 and 531-554 of VP4 are implicated in heat shock cognate protein hsc70-mediated rotavirus infection. Arch Virol. 2007; 152(12): 2183-96. doi: 10.1007/s00705-007-1055-5. [ Links ]
33. Kao C-Y, Yang P-M, Wu M-H, Huang C-C, Lee Y-C, Lee K-H. Heat shock protein 90 is involved in the regulation of HMGA2-driven growth and epithelial-to-mesenchymal transition of colorectal cancer cells. PeerJ. 2016; 4: e1683. doi: 10.7717/peerj.1683. [ Links ]
34. Yang R, Tang Q, Miao F, An Y, Li M, Han Y, et al. Inhibition of heat-shock protein 90 sensitizes liver cancer stem-like cells to magnetic hyperthermia and enhances anti-tumor effect on hepatocellular carcinoma-burdened nude mice. Int J Nanomedicine. 2015;10:7345-58. doi: 10.2147/IJN.S93758. [ Links ]
35. Gadelle D, Bocs C, Graille M, Forterre P. Inhibition of archaeal growth and DNA topoisomerase VI activities by the Hsp90 inhibitor radicicol. Nucleic Acids Res. 2005;33(7):2310-7. doi: 10.1093/nar/gki526. [ Links ]
36. Schlecht R, Scholz SR, Dahmen H, Wegener A, Sirrenberg C, Musil D, et al. Functional analysis of Hsp70 inhibitors. PLoS One. 2013; 8(11):e78443. doi: 10.1371/journal.pone.0078443. [ Links ]
37. Rong Y, Yang EB, Zhang K, Mack P. Quercetin-induced apoptosis in the monoblastoid cell line U937 in vitro and the regulation of heat shock proteins expression. Anticancer Res. 2000;20(6B):4339-45. [ Links ]
38. Chhetri G, Kalita P, Tripathi T. An efficient protocol to enhance recombinant protein expression using ethanol in Escherichia coli. MethodsX. 2015;2:385-91. doi: 10.1016/j.mex.2015.09.005. [ Links ]
39. Aghi M, Martuza RL. Oncolytic viral therapies - the clinical experience. Oncogene. 2005; 24(52): 7802-16. doi: 10.1038/sj.onc.1209037. [ Links ]
40. Santana AY, Guerrero CA, Acosta O. Implication of Hsc70, PDI and integrin avß3 involvement during entry of the murine rotavirus ECwt into small-intestinal villi of suckling mice. Arch Virol. 2013; 158(6):1323-36. doi: 10.1007/s00705-013-1626-6. [ Links ]
41. Guerrero CA, Moreno LP. Rotavirus receptor proteins Hsc70 and integrin avß3 are located in the lipid microdomains of animal intestinal cells. Acta Virol. 2012; 56(1): 63-70. doi: 10.4149/av_2012_01_63. [ Links ]
42. Elia G, Santoro MG. Regulation of heat shock protein synthesis by quercetin in human erythroleukaemia cells. Biochem J. 1994; 300(Pt 1): 201-9. doi: 10.1042/bj3000201. [ Links ]
43. Guerrero CA, Acosta O. Inflammatory and oxidative stress in rotavirus infection. World J Virol. 2016;5(2):38-62. doi: 10.5501/wjv.v5.i2.38. [ Links ]
Notas:
Financiación: Este estudio fue apoyado por una financiación (No. 110165843297, 658-2014 otorgada por el Departamento Administrativo de Ciencia, Tecnología e Investigación-COLCIENCIAS a C.A. Guerrero.
Anexo
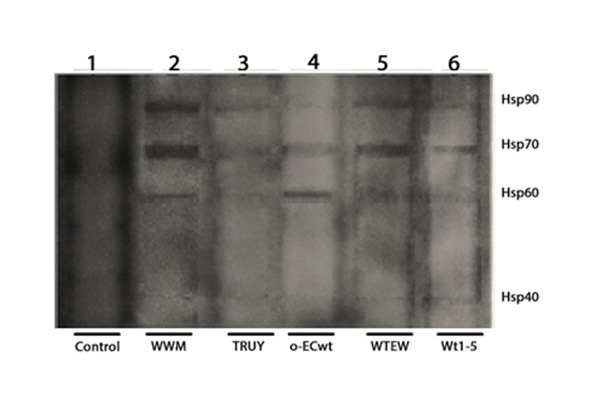
Anexo 1 Biotinilación de proteínas de la superficie celular e inmunoprecipitación. Biotinilación de las proteínas de la superficie celular acoplada a inmuno-precipitación. A células de la línea REH se adicionó 1 mg/mL de NHS-SS-Biotin por 30 minutos a 4° C. Las células se lavaron con PBS que contenía 0.1 M de glicina. Luego, fueron incubadas con los 5 aislamientos de rotavirus. Posteriormente, las células se trataron con glutaraldehído (2.5% concentración final), se lisaron y fueron inmunoprecipitadas usando anticuerpos anti-rotavirus acoplados a perlas de agarosa. Posteriormente, se realizó una electroforesis (SDS-PAGE) y Western blotting y a la membrana se le aplicó estreptavidina-HRP para detectar las HSPs biotiniladas. La membrana fue revelada con el kit Luminol.
Recibido: 10 de Febrero de 2020; Revisado: 31 de Enero de 2021; Aprobado: 02 de Febrero de 2021











 texto en
texto en 



