Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Colombia Médica
On-line version ISSN 1657-9534
Colomb. Med. vol.52 no.3 Cali July/Sept. 2021 Epub Sep 30, 2021
https://doi.org/10.25100/cm.v52i3.4569
Articulo original
Asociación de variantes genéticas en TPMT y NUDT15 con los eventos de toxicidad durante la terapia de mantenimiento en niños colombianos con Leucemia Linfoide Aguda
1 Universidad Nacional de Colombia, Facultad de Medicina, Departamento de Pediatría, Bogotá, Colombia.
2 Universidad Nacional de Colombia, Facultad de Medicina, Instituto de Genética, Departamento de Patología, Bogotá, Colombia.
3 HOMI Fundación Hospital Pediátrico la Misericordia, Division of Pediatric Hematology and Oncology. Bogotá, Colombia.
4 Clínica del Country, Division of Pediatric Hematology and Oncology, Bogota, Colombia.
Objetivo:
la finalidad de este estudio fue evaluar las asociaciones entre los perfiles de los genes NUDT15 y TPMT con los efectos adversos del tratamiento de mantenimiento en pacientes pediátricos con Leucemia Linfoblástica Aguda atendidos en un hospital de referencia durante el 2017.
Métodos:
Este fue un estudio observacional analítico, de corte longitudinal en el que los genotipos de los genes de interés fueron determinados mediante PCR de discriminación alélica con sondas TaqMan® en pacientes que estaban recibiendo quimioterapia de mantenimiento en la Unidad de Oncohematología Pediátrica durante el 2017. Los datos clínicos y sociodemográficos correspondientes a los primeros 6 meses de sus tratamientos de mantenimiento fueron colectados, y se evaluó la correlación entre los genotipos identificados y el desarrollo de efectos secundarios en estos pacientes.
Resultados:
setenta pacientes fueron incluidos en el estudio, de estos, los análisis genéticos para NUDT15 y TPMT (rs1800462 and rs1800460) fueron realizados en 68 pacientes, en tanto que para el polimorfismo rs1142345 se logró la tipificación en 42 pacientes. 4/68 pacientes fueron heterocigotos para NUDT15 y el mismo número de pacientes fueron heterocigotos para rs1800462 and rs1142345, mientras que para rs1800460, 6 pacientes heterocigotos fueron identificados. No se identificaron asociaciones estadísticamente significantes entre las variants genéticas y los resultados clínicos de interés.
Conclusiones:
Estos hallazgos resaltan la importancia de realizar estudios de este tipo con un mayor número de sujetos de estudio, así como plantean la necesidad de evaluar otras variantes genéticas que podrían tener algún impacto en el desarrollo de efectos secundarios durante la quimioterapia de mantenimiento.
Palabras clave: leucemia linfoide aguda; efectos adversos; farmacogenética; niños; tiopurina; pruebas de farmacogenómica
Objective:
This study aimed to correlate the genetic profile of the NUDT15 and TPMT genes with the side effects of the treatment of pediatric patients with acute lymphoid leukemia who were undergoing maintenance therapy at a tertiary care hospital in 2017.
Methods:
This was an analytical, longitudinal, observational study in which the genotypes of the genes of interest were determined by PCR allelic discrimination with TaqMan® probes in patients receiving chemotherapy during the maintenance phase in the Pediatric Hematology and Oncology Unit in 2017. Sociodemographic and clinical data corresponding to the first six months of their maintenance chemotherapy were collected, and the correlation between the genotypes obtained and the development of side effects during the maintenance phase of chemotherapy in these patients was evaluated.
Results:
Seventy pediatric patients were included in the study. Genetic analyses were carried out of these for NUDT15 and TPMT (rs1800462 and rs1800460) on 68 patients, while for the rs1142345 polymorphism, typing was achieved in 42 patients. 4/68 patients were heterozygous for NUDT15, and the same number of patients were heterozygous for rs1800462 and rs1142345, while for rs1800460, 6 heterozygous patients were identified. No statistically significant association was identified between the genetic variants and the outcomes of interest.
Conclusion:
Studies with a larger population size are needed and the evaluation of other genetic variants that may influence the development of side effects during maintenance chemotherapy.
Keywords: Acute lymphoid leukemia; drug-related side effects and adverse reactions; pharmacogenetics; children; mercaptopurine; pharmacogenomic testing
Remark
| 1) ¿Por qué se realizó este estudio? |
| Diversos estudios realizados principalmente en Estados Unidos y Asia han mostrado asociaciones entre las variantes de los genes TPMT y NUDT15 con los resultados de toxicidad en pacientes con leucemia linfoide aguda, la información referente a este tema es escasa en nuestro país, por lo que decidimos evaluar esta asociación en nuestra población la cual representa una muestra con mezcla genética y en la que se han descrito diferencias farmacogenéticas relacionadas con la ancestría. |
| 2) ¿Cuáles fueron los resultados más relevantes del estudio? |
| No se identificaron asociaciones estadísticamente significativas entre las variantes genéticas en TPMT y NUDT15 con los resultados de toxicidad evaluados (mielotoxicidad y toxicidad hepática, así como la reducción o interrupción de la dosis de 6-MP). |
| 3) ¿Qué aportan estos resultados? |
| Nuestros resultados apuntan a la necesidad de realizar estudios nacionales dirigidos a la búsqueda de “biomarcadores autóctonos” que permitan identificar a los pacientes pediátricos oncológicos que presentan mayor riesgo de desarrollar resultados de toxicidad durante sus tratamientos de quimioterapia. |
Introducción
La Leucemia Linfoblástica Aguda (LLA) es el cáncer más frecuente en la edad pediátrica, representando el 25% de todas las neoplasias en niños menores de 15 años 1. Esta se clasifica en dos grupos acorde a su origen celular con 85% de los casos correspondientes a LLA de precursores B y 15% de células T 2,3. La LLA es más frecuente en niños de ancestría hispana y caucásica comparados con los de ancestría africana (4,5. Aunque algunos factores ambientales han sido identificados como factores de riesgo para LLA en niños ( exposición a radiación y a algunos químicos), estas asociaciones solo explican un pequeño grupo de casos (6.
Uno de los grandes hitos en la terapia de niños con LLA fue el desarrollo del régimen intensivo de 8 drogas, 8 semanas de inducción y consolidación, el mismo que fue la base del régimen BFM (Berlín-Frankfurt-Münster) y que es el núcleo de las terapias contemporáneas para LLA, esta estrategia quimioterapéutica incluye además terapias de intensificación y mantenimiento 6. Desde la introducción de este régimen terapéutico, se han venido desarrollando múltiples ensayos clínicos colaborativos que han logrado importantes avances en la supervivencia de los pacientes con LLA, alcanzando tasas de supervivencia total de hasta 90% 6,7.
Pese a las mejoras alcanzadas en supervivencia para la mayoría de pacientes pediátricos, las recaídas ocurren entre el 15-20% y son una causa importante de morbimortalidad en pacientes pediátricos con LLA 8,9. En línea con esto, en la mayoría de los ensayos multicéntricos de tratamiento de primera línea en LLA en niños, hasta el 5% fallecen debido a los efectos tóxicos secundarios del tratamiento 10,11. Las muertes relacionadas al tratamiento ocurren durante episodios recurrentes y prolongados de neutropenia y linfopenia debidas a drogas citotóxicas e inmunosupresoras o porque la leucemia, por sí misma, inhibe la recuperación de la médula ósea durante la terapia de inducción, especialmente en pacientes catalogados como respondedores lentos 10,12.
El avance tecnológico ha permitido la identificación de variaciones genómicas que determinan patrones farmacogenómicos característicos, responsables de las diferencias individuales observadas en la respuesta al tratamiento en términos de efectividad o de toxicidad. Actualmente, el genotipo de TPMT es el principal patrón farmacogenético con implicaciones para los protocolos de tratamiento de los pacientes con LLA con tratamiento basado en tiopurinas (13. La 6-mercaptopurina (6-MP) es esencial en el tratamiento de la LLA principalmente en la fase de mantenimiento, la cual es la fase más larga del tratamiento. Se ha descrito que entre el 85-90% de los pacientes con variantes homocigotas en TPMT se requieren reducciones de dosis de 6-MP 14-17. Otros genes involucrados en el metabolismo de las tiopurinas han sido evaluados con respecto a su comportamiento en LLA 15; dentro de estos, recientemente ha surgido el gen NUDT15 como un candidato importante en la predicción de toxicidad 18.
Yang et al. 19, mediante un estudio de GWAS y su respectiva cohorte de replicación, identificaron dos loci relacionados con la intensidad de dosificación de mercaptopurina: rs1142345 en el gen TPMT y rs116855232 en NUDT15. Esta última variante fue común entre asiáticos e hispanos pero infrecuente entre europeos y no observada en africanos, contribuyendo así a las diferencias en la tolerancia a mercaptopurina relacionada con la ancestría (19. En este mismo estudio, evidenciaron que los pacientes heterocigotos para las variantes de TPMT o NUDT15, requirieron una disminución del 50% de la dosis de mercaptopurina comparados con los de genotipo silvestre (19. El mismo grupo de investigadores, replicó estos resultados en cohortes de Guatemala, Singapur y Japón, adicionando estudios de genómica funcional, con los que evidenciaron que la presencia de los SNPs resulta en una pérdida de la actividad nucleótido difosfatasa, por lo que los pacientes con estos alelos defectivos de NUDT15, presentan niveles elevados de metabolitos activos de tiopurina aumentando así su toxicidad (20. El interés científico en este gen es creciente en los últimos años y ha llevado a que se le asocie con reducciones de dosis de mercaptopurina en relación con la edad (21, mielosupresión 22, desarrollo de toxicidad severa 23, e intolerancia a 6-MP poblaciones específicas 24, convirtiéndolo en un candidato importante a evaluar en estudios de farmacogenética relacionados con tolerancia a 6-MP; aunque con menor soporte en la evidencia que TPMT.
A la fecha, no existen publicaciones sobre enfoques farmacogenómicos relacionados con la respuesta al tratamiento y la toxicidad de la quimioterapia para la LLA en la población pediátrica de Colombia. Esto cobra aún más importancia, cuando se han descrito diferencias farmacogenéticas relacionadas con la ancestría 25-28. Adicionalmente, se ha sugerido que estudios de corte farmacogenético deben ser realizados de manera independiente o colaborativa en distintos países latinoamericanos para un mejor entendimiento de estas relaciones 29. Este estudio tuvo como objetivo evaluar la asociación entre el perfil genético de los genes NUDT15 y TPMT y los efectos secundarios durante la fase de mantenimiento del tratamiento en una muestra de pacientes pediátricos colombianos con leucemia linfoide aguda.
Materiales y Métodos
Diseño
Este fue un estudio observacional analítico, de corte longitudinal, llevado a cabo en un hospital universitario que además es centro de cáncer infantil en Bogotá, que recibe pacientes de áreas rurales y urbanas, siendo un centro nacional de referencia para el cuidado de niños con cáncer.
Pacientes y recolección de información
Se incluyó a pacientes entre 1 y menos de 18 años diagnosticados con LLA que fueron admitidos a la Unidad de Oncohematología y quienes se encontraban recibiendo quimioterapia de mantenimiento del protocolo BFM ALLIC 2009 durante el 2017. Los pacientes menores de un año así como aquellos que presentaban Síndrome de Down como comorbilidad fueron excluidos de este estudio. Para los fines de este estudio, se llevó a cabo un muestreo por conveniencia en el que se incluyó de forma consecutiva a todos los pacientes que cumplían los criterios de selección y aceptaron participar. Las características y resultados clínicos logrados con este protocolo de quimioterapia en este centro, han sido previamente publicados (30. Este estudio se llevó a cabo siguiendo las políticas nacionales para investigación clínica y sujeto a los principios éticos de la Declaración de Helsinki. El protocolo de investigación fue aprobado por los comités de ética de las instituciones involucradas en su ejecución (Acta 015-184-16 del 25/08/16 y CEI-38-16 del 11/08/16). Las muestras de sangre y los datos clínicos fueron obtenidos solo bajo consentimiento de los padres o cuidadores y para cada paciente de 14 años en adelante también se consiguió su asentimiento para participación en el estudio.
El excedente de las muestras de sangre total que son tomadas como parte habitual del control de los pacientes con LLA fue usado para la extracción de ADN. Los pacientes fueron seguidos hasta por seis meses para identificar la aparición de efectos adversos debidos al tratamiento (mielo-supresión, toxicidad hepática, muerte tóxica). Durante este seguimiento, información clínica y paraclínica (hemograma, pruebas de función hepática) así como datos relacionados con ajustes en el tratamiento y el desarrollo de complicaciones fueron colectados. Para este estudio el seguimiento de los pacientes se llevó a cabo de forma paralela con los controles clínicos realizados durante la fase de mantenimiento, que en este centro es llevado a cabo de forma mensual.
Desenlaces
Los desenlaces de interés fueron efectos secundarios como mielotoxicidad y toxicidad hepática, así como la reducción o interrupción de la dosis de 6-MP. Como análisis exploratorio, evaluamos otros resultados como neutropenia febril, recaída y muertes.
Extracción de ADN
Para la extracción de ADN de las muestras de interés se empleó el estuche comercial QIamp DNA Blood Mini Kit™, siguiendo las especificaciones del fabricante y como ha sido previamente descrito (31,32. En breve, 200 μL de sangre total fueron sometidos a la serie de pasos descritos en la guía del fabricante, mediante el empleo de la técnica de filtración en columna mediante microcentrifugación. La cuantificación del ADN se llevó a cabo mediante espectrofotometría en un equipo Nanodrop2000™ y en este mismo equipo se realizaron las mediciones de la razón 260/280 nm para determinar la pureza del material genético. El ADN obtenido se almacenó en tubos eppendorf™ de 1.5 mL a una temperatura de -20° C hasta su empleo en los ensayos de reacción en cadena de la polimerasa (PCR).
PCR de discriminación alélica
La determinación de los genotipos de los SNP (rs1800462, rs1800460, rs1142345 y rs116855232) en los genes TPMT y NUDT15 se llevó a cabo mediante PCR en tiempo real de discriminación alélica por 5’ nucleasa usando los ensayos comercialmente disponibles TaqMan® SNP Genotyping Assays (Identificadores de ensayos: C__12091552_30, C__30634116_20, C_____19567_20 and C_154823200_10, Thermo Fisher Scientific, USA), siguiendo las recomendaciones descritas en “TaqMan® Allelic Discrimination Guide” y protocolos previos. Para esto, se empleó una mezcla de PCR que contenía: 15 ng ADN (3 μL), 1.25 μL de la sonda TaqMan®(20X) correspondiente, 12.5 μL TaqMan® Gene Expression Master Mix y 8.25 μL de agua ultrapura libre de nucleasas para un volumen final por reacción de 25 μL, las muestras se corrieron por duplicado. Los ensayos fueron llevados a cabo empleado el CFX96 Touch™ Real-Time PCR Detection System (Bio-Rad), siguiendo las condiciones de corrido sugeridas para cada sonda por el fabricante.
Análisis de los datos
Los datos son presentados como medianas más rangos interquartiles para las variables cuantitativas, en tanto que para los datos cualitativos se muestras sus frecuencias respectivas.
Las frecuencias alélicas y genotípicas, así como el equilibrio de Hardy-Weinberg fueron evaluados usando el programa snpReady. La tipificación de TPMT se llevó a cabo siguiendo los estándares del Consorcio de Implementación de Farmacogenética Clínica 13. Con base en esto, los haplotipos de TPMT fueron determinados acorde a las combinaciones de los SNPs rs1800462, rs1800460 and rs1142345 siguiendo esta regla: CCT = * 1, GCT = * 2, CTC = * 3A, CTT = * 3B and CCC = * 3C. Para la determinación de la asociación entre las variantes genéticas y los desenlaces de interés, se empleó el estadístico Ji 2. Los análisis estadísticos fueron realizados usando SPSS para Mac, Versión 2.0, se consideró como estadísticamente significante un valor de p <0.05.
Resultados
Características generales de los sujetos de estudio
Un total de 73 pacientes con diagnóstico de LLA se encontraban en mantenimiento durante el 2017 en nuestra institución, de estos, 70 pacientes consintieron y/o asintieron su participación en el proyecto. De los pacientes incluidos, no se logró extraer ADN para los estudios de genotipifcación en 2 pacientes, por lo que los análisis fueron basados en los 68 pacientes en quienes se logró realizar estudios genéticos. La Figura 1 muestra el diagrama de flujo de selección e inclusión de los pacientes en el estudio.
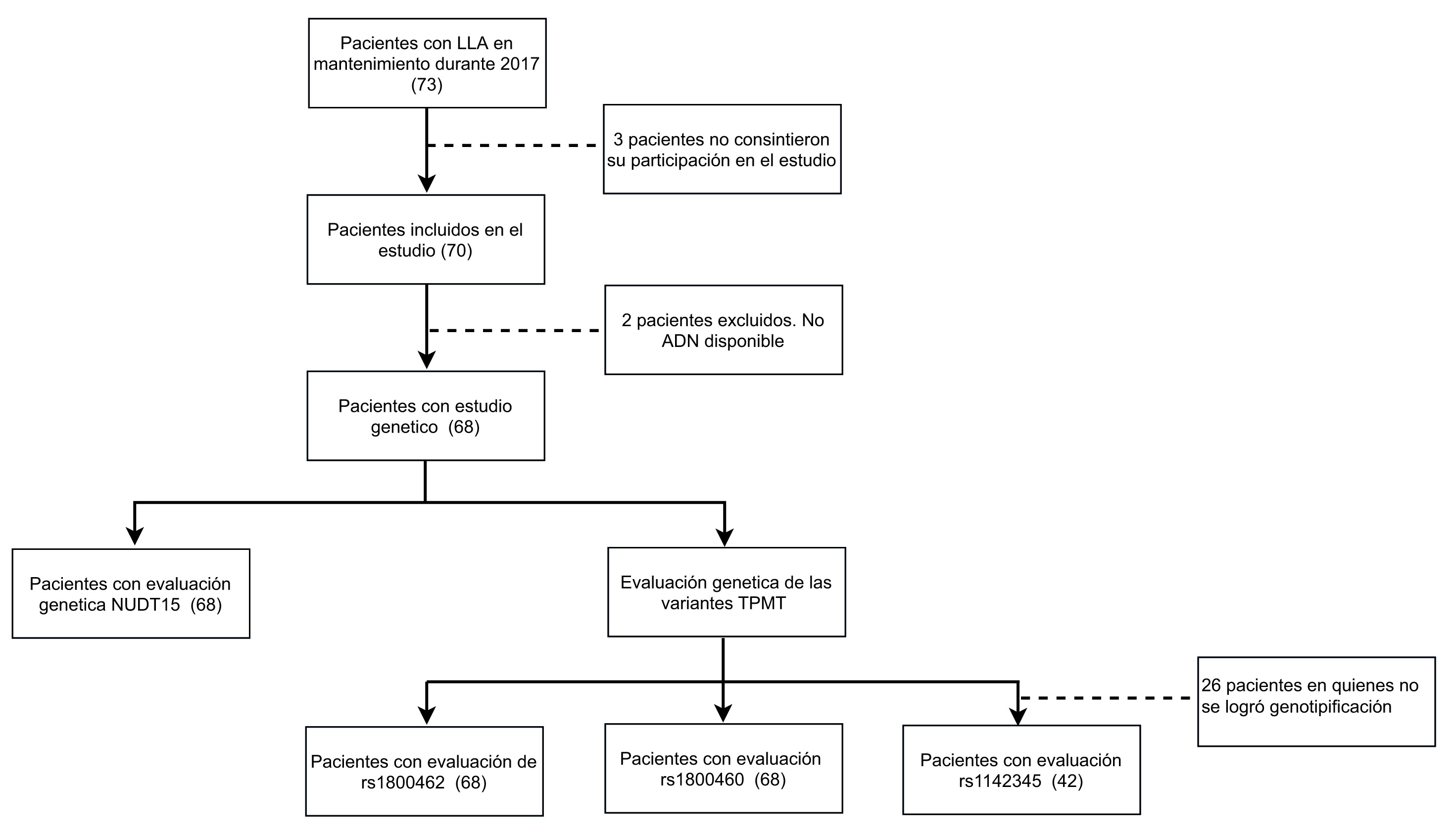
Figura 1 Diagrama de inclusión de pacientes. Este diagrama muestra cómo se llevo a cabo la inclusión de pacientes en el estudio, así cómo los detalles relacionados con la realización de estudios genéticos.
La Tabla 1 resume las características generales de la población de estudio. Un total de 70 pacientes fueron incluidos con una razón hombre a mujer de 1:1. La mediana de edad al inicio de la terapia de mantenimiento fue seis años (RIC: 7 años). Más del 50% de la población de estudio fue de Bogotá y cerca del 42% correspondió a procedentes de zona rural. Con relación a los desenlaces evaluados, se documentó mielotoxicidad en 25%, toxicidad hepática en 27% y 32.9% de los pacientes requirió reducción de su dosis de 6-MP durante el seguimiento. Es importante resaltar que durante el periodo de estudio no se presentó ninguna pérdida de seguimiento.
Tabla 1 Características Generales y seguimiento clínico de la población de estudio.
| Característica | Medición | ||
|---|---|---|---|
| n o mediana | % o RIQ | ||
| Género | Femenino | 35 | 50% |
| Edad (Años cumplidos) | 6 | 7 | |
| Linaje | B | 65 | 92.9% |
| T | 5 | 7.1% | |
| Riesgo | Estándar | 7 | 10% |
| Intermedio | 34 | 48.6% | |
| Alto | 29 | 41.4% | |
| Reducción dosis 6-MP | 23 | 32.9% | |
| Interacción 6-MP | 20 | 28.6% | |
| Neutropenia Febril | 8 | 11.4% | |
| Recaída | 11 | 15.7% | |
| Muerte | 2 | 2.9% | |
| Seguimiento 1 | ALT (IU/L) | 48.7 | 91.25 |
| AST (IU/L) | 37 | 25.3 | |
| Leucocitos/mm3 | 3,315 | 1,370 | |
| Neutrófilos/mm3 | 1,820 | 1,137.5 | |
| Linfocitos/mm3 | 780 | 690 | |
| Bilirubina total (mg/dL) | 0.5 | 0.3 | |
| Seguimiento 2 | ALT (IU/L) | 42.5 | 97.5 |
| AST (IU/L) | 33.55 | 31.125 | |
| Leucocitos/mm3 | 3,060 | 2,160 | |
| Neutrófilos/mm3 | 1,721 | 1,682.5 | |
| Linfocitos/mm3 | 630 | 675 | |
| Bilirubina total (mg/dL) | 0.5 | 0.3 | |
| Seguimiento 3 | ALT (IU/L) | 47 | 98.475 |
| AST (IU/L) | 34 | 37 | |
| Leucocitos/mm3 | 3,095 | 1,487.5 | |
| Neutrófilos/mm3 | 1,615 | 1,196.5 | |
| Linfocitos/mm3 | 742 | 546 | |
| Bilirubina total (mg/dL) | 0.5 | 0.4 | |
| Seguimiento 4 | ALT (IU/L) | 50.35 | 76.425 |
| AST (IU/L) | 32.55 | 23.308 | |
| Leucocitos/mm3 | 3,400 | 1,640 | |
| Neutrófilos/mm3 | 1,940 | 1,276.75 | |
| Linfocitos/mm3 | 760 | 565 | |
| Bilirubina total (mg/dL) | 0.6 | 0.425 | |
| Seguimiento 5 | ALT (IU/L) | 50.8 | 116.9 |
| AST (IU/L) | 37 | 42.1 | |
| Leucocitos/mm3 | 3,360 | 1,700 | |
| Neutrófilos/mm3 | 1,850 | 1,707.5 | |
| Linfocitos/mm3 | 840 | 465 | |
| Bilirubina total (mg/dL) | 0.6 | 0.465 | |
| Seguimiento 6 | ALT (IU/L) | 65.1 | 101.9 |
| AST (IU/L) | 39.8 | 36.2 | |
| Leucocitos/mm3 | 3,370 | 1,920 | |
| Neutrófilos/mm3 | 1,830 | 1,674.5 | |
| Linfocitos/mm3 | 910 | 636 | |
| Bilirubina total (mg/dL) | 0.6 | 0.5 | |
Esta tabla resume las características clínicas generales y de seguimiento de los pacientes incluidos en el estudio. Para cada valor de laboratorio se ha anotado el sistema de medición empleado. Los datos presentada en las columnas de “Medición” corresponden al n o las medianas (subcolumna n o mediana) y la otra subcolumna corresponde a su porcentaje o rango intercuartil
Frecuencias alélicas y genotípicas
Del total de 70 pacientes incluidos en el estudio, se obtuvo material genético con calidad y cantidad adecuadas en 68 casos (concentraciones de 10 a 129 ng/µL y un valor promedio de razón 260/280nm de 1.87; valores óptimos entre 1.8 y 2.0 33,34). Para los otros dos casos, aunque la extracción de ADN se realizó en dos oportunidades adicionales, no se obtuvo material genético con las condiciones de calidad necesarias para realizar los ensayos de reacción en cadena de la polimerasa. Un total de 14 pacientes portaban un alelo de susceptibilidad: 4 para NUDT15, 4 para rs1800462 (TPMT), 6 para rs1800460 (TPMT) y 4 para rs1142345 de TPMT (Figura 2).
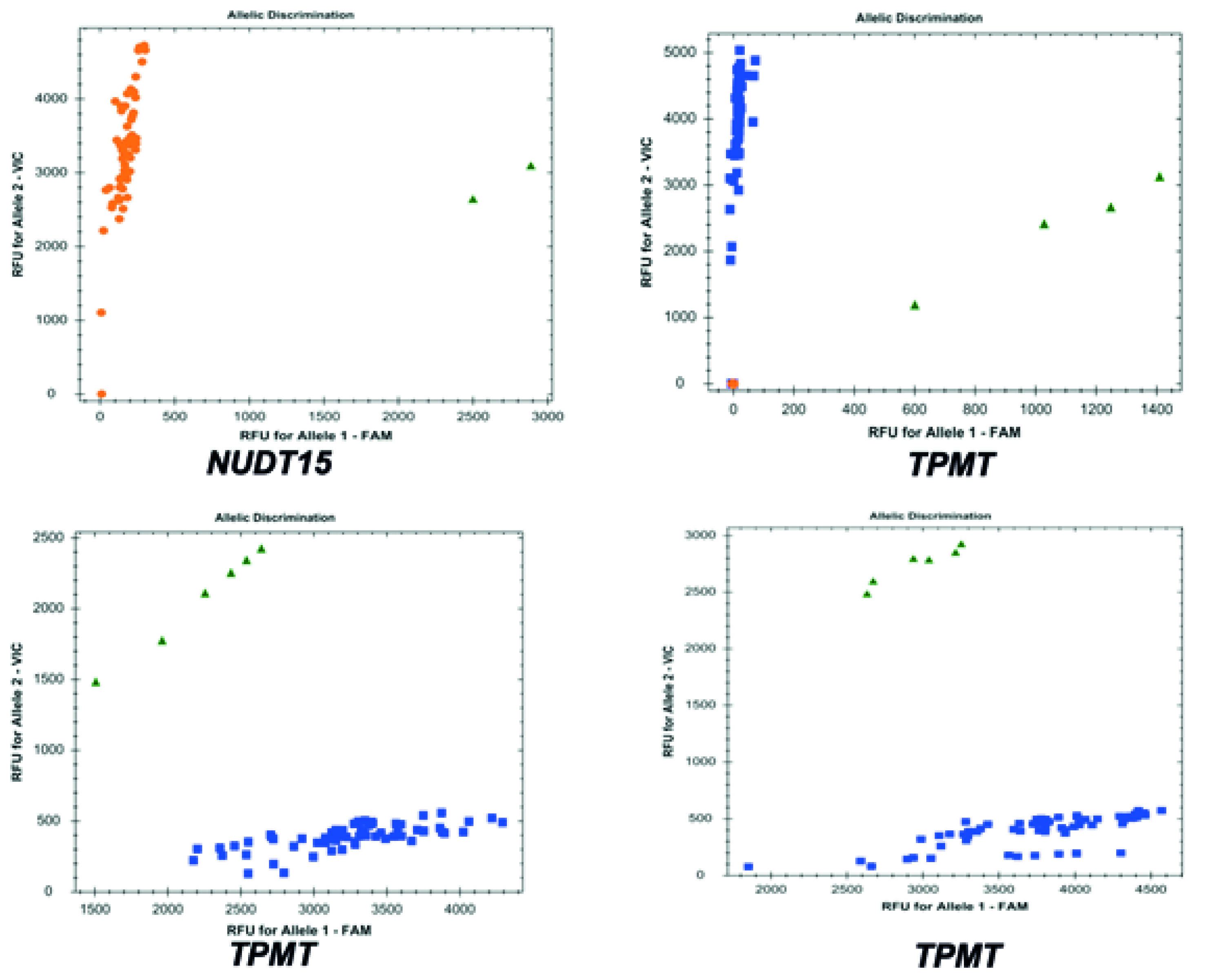
Figura 2 Imagen representativa de las gráficas de discriminación alélica obtenidos por los ensayos de genotipificiación. No hubo pacientes homocigotos para los alelos mutados en ninguno de los polimorfismos estudiados. Los gráficos representan los grupos de muestras con el alelo de tipo salvaje alineado hacia los ejes X o Y según la sonda utilizada para su reconocimiento. Los puntos no alineados corresponden al alelo mutado.
La Tabla 2 muestra el resumen de frecuencias alélicas y genotípicas de cada uno de los SNPs de interés. Se encontró que todas las variantes estaban en equilibrio de Hardy-Weinberg. La Tabla 3 resume la tipificación de TPMT siguiendo los estándares del “Clinical Pharmacogenetic Implementation Consortium” en los 42 pacientes en los que se logró la tipificación de los 3 SNPs 13. Según estos estándares, los haplotipos de TPMT fueron determinados con base a las combinaciones de los SNPs rs1800462, rs1800460 y rs1142345 así: CCT = * 1, GCT = * 2, CTC = * 3ª, CTT = * 3B and CCC = * 3C.
Tabla 2 Frecuencias alélicas y genotípicas de los polimorfismos de interés.
| Gen | Anotación | n | Frecuencia | HW (p Value) | Latinoamérica1 | Latinoamérica2 | |
|---|---|---|---|---|---|---|---|
| NUDT15 | rs116855232 | 68 | 0.802 | n= 804 | n= 974 | ||
| Alelos | C | 132 | 0.97 | 0.994 | 0.953 | ||
| T | 4 | 0.03 | 0.006 | 0.047 | |||
| Genotipos | C/C | 64 | 0.94 | ||||
| C/T | 4 | 0.06 | |||||
| T/T | 0 | 0 | |||||
| TPMT | rs1800462 | 68 | 0.802 | n= 500 | n= 628 | ||
| Alelos | C | 132 | 0.970 | 0.992 | 0.997 | ||
| G | 4 | 0.03 | 0.008 | 0.003 | |||
| Genotipos | C/C | 64 | 0.94 | ||||
| C/G | 4 | 0.06 | |||||
| G/G | 0 | 0 | |||||
| rs1800460 | 68 | 0.703 | n= 1,430 | n= 7,010 | |||
| Alelos | T | 6 | 0.044 | 0.026 | 0.045 | ||
| C | 130 | 0.956 | 0.974 | 0.955 | |||
| Genotipos | T/T | 0 | 0 | ||||
| T/C | 6 | 0.09 | |||||
| C/C | 62 | 0.91 | |||||
| rs1142345 | 42 | 0.746 | n= 1,232 | n= 5,254 | |||
| Alelos | C | 4 | 0.0476 | 0.041 | 0.052 | ||
| T | 80 | 0.9524 | 0.959 | 0.948 | |||
| Genotipos | C/C | 0 | 0 | ||||
| C/T | 4 | 0.10 | |||||
| T/T | 38 | 0.90 |
Esta tabla muestra las frecuencias alélicas y genotípicas de los polimorfismos estudiados. Se encontró que todas las variantes estaban en equilibrio de Hardy-Weinberg (columna HW). Además, las frecuencias alélicas identificadas en nuestro estudio se compararon con en la población latinoamericana anotadas en el proyecto ALFA. No hubo diferencias estadísticamente significantes entre nuestras frecuencias y las reportadas en el proyecto ALFA.
Tabla 3 Tipificación de TPMT.
| Frequencias | ||
|---|---|---|
| Haplotipos | *1 | 78 |
| *2 | 2 | |
| *3A | 4 | |
| Tipos | *1/*1 | 36 |
| *1/*2 | 2 | |
| *1/*3A | 4 | |
Se listan las frecuencias de los tipos de TPMT basadas en los estándares del “Consorcio de implementación de farmacogenética clínica”. Dado que la genotipificación de rs1142345 solo fue posible en 42 sujetos y que para realizar estas clasificaciones es necesario tener genotipificación de los 3 loci de TPMT, estos datos corresponden solo a los 42 pacientes en los que se completó la genotipificación.
Las frecuencias alélicas obtenidas en el presente estudio se compararon con las frecuencias reportadas en las bases de datos dbSNP para poblaciones latinoamericanas descritas en el proyecto ALFA (agregador de frecuencias alélicas) (Tabla 2); La base de datos de frecuencias de genotipos y fenotipos (dbGap) se desarrolló para archivar y distribuir los datos y los resultados de estudios que han investigado la interacción del genotipo y el fenotipo en humanos 35. Los resultados obtenidos para cada SNP analizado por nosotros, no mostraron una diferencia estadísticamente significativa de las frecuencias obtenidas en el proyecto ALFA.
Asociaciones genéticas
La Tabla 4 resume los resultados de los análisis bivariados en los que se muestra la correlación entre las variantes genéticas de los SNPs en NUDT15 y TPMT y los efectos secundarios de interés. No se encontró asociación entre los resultados de interés y la variante genética de NUDT15 o cualquier polimorfismo de TPMT evaluado individualmente. Se llevaron a cabo análisis similares en relación con la tipificación de TPMT, utilizando la variable de homocigosidad para el alelo *1 versus portador *2 o *3ª como heterocigoto, sin evidencia de ninguna asociación estadísticamente significativa (Tabla suplementaria 1S).
Tabla 4 Análisis de asociación entre genotipos y desenlaces.
| Desenlaces | NUDT15 | TPMT | ||||||
|---|---|---|---|---|---|---|---|---|
| rs1800462 | rs1800460 | rs1142345 | ||||||
| C/C | C/T | C/C | C/G | T/C | C/C | C/T | T/T | |
| Reducción 6-MP | ||||||||
| Si | 21 | 2 | 22 | 1 | 3 | 20 | 2 | 8 |
| No | 43 | 2 | 42 | 3 | 3 | 42 | 2 | 30 |
| Interrupción 6-MP | ||||||||
| Si | 19 | 1 | 18 | 2 | 1 | 19 | 0 | 10 |
| No | 45 | 3 | 46 | 2 | 5 | 43 | 4 | 28 |
| Neutropenia | ||||||||
| Si | 7 | 1 | 6 | 2 | 0 | 8 | 0 | 5 |
| No | 57 | 3 | 58 | 2 | 6 | 54 | 4 | 33 |
| Recaída | ||||||||
| Si | 8 | 1 | 8 | 1 | 0 | 9 | 0 | 5 |
| No | 56 | 3 | 56 | 3 | 6 | 53 | 4 | 33 |
| Muerte | ||||||||
| Si | 1 | 1 | 2 | 0 | 0 | 2 | 0 | 2 |
| No | 63 | 3 | 62 | 4 | 6 | 60 | 4 | 36 |
Los datos se presentan como valores de frecuencia absoluta para cada desenlace evaluado. No se encontró asociación estadísticamente significativa entre ninguno de los polimorfismos estudiados y los resultados de interés.
Aunque no se identificaron asociaciones estadísticamente significativas, dos de los cuatro pacientes heterocigotos para NUDT15 requirieron una disminución en 6-MP durante el período de seguimiento. En relación con las variantes genéticas de TPMT, para rs1800462, dos de los cuatro pacientes heterocigotos presentaron neutropenia febril, y estos mismos pacientes requirieron adicionalmente la suspensión de 6-MP durante los primeros seis meses de la terapia de mantenimiento. En el caso de rs1800460, de los seis pacientes que eran heterocigotos, tres requirieron una disminución de la dosis, y de estos, dos también fueron heterocigotos para rs1142345. Además, se evaluaron las diferencias en la mediana de los valores de laboratorio tomados en los primeros 6 meses de tratamiento de mantenimiento: transaminasas, bilirrubina total, leucocitos y recuento celular diferencial; con relación a las variantes genéticas y no se encontraron diferencias estadísticamente significativas (Tabla suplementaria 2S).
Discusión
En nuestro conocimiento, este es el primer estudio en el país, que ha evaluado los polimorfismos de los genes NUDT15 y TPMT y su correlación con los efectos secundarios de la quimioterapia en pacientes pediátricos con leucemia linfoblástica aguda. Aunque nuestra población es considerada como población Latina, diferentes grados de ancestrías Caucásica, Amerindia y Africana han sido descritos en diferentes regiones de Colombia. Nuestros hallazgos varían significativamente de informes anteriores con respecto a la asociación entre variantes y resultados 16,19,36,37; sin embargo, estos estudios han sido realizados principalmente en poblaciones con poca representatividad latina.
La frecuencia alélica de la mutación NUDT15 identificada en nuestro estudio (0.03) fue menor que la informada anteriormente 19,36,38,39, una posible explicación para este hallazgo es que la mayoría de los estudios sobre este polimorfismo han sido realizados en poblaciones de origen asiático, encontrando frecuencias alélicas de hasta 0.27. La muestra evaluada en este estudio, es originada en la recion andina central de Colombia, en la que el componente caucásico (ancestría Europea) representa entre el 50 y 60% de la ancestría genética 40. Otra posible razón puede ser nuestro tamaño de muestra 24,41. Para el caso particular de los polimorfismos en TPMT, previamente solo se ha realizado un estudio en la población colombiana, en el que Isaza y col. (42, describieron frecuencias para el alelo *2 y *3A de 0.004 y 0.035, respectivamente, las cuales son inferiores a las identificadas por nosotros (0.02 y 0.05, respectivamente). En comparación con otros resultados latinoamericanos, encontramos que nuestras frecuencias también son ligeramente diferentes a las descritas por Garrido y col. en pacientes pediátricos con leucemia linfoblástica aguda en Guatemala, identificaron una frecuencia de 0.005 para *2 y 0.0375 para *3A 43. Sin embargo, nuestra frecuencia de *2 es similar a la encontrada en Brasil por Boson y col., Quienes describen una frecuencia de 0.022 pero una frecuencia mucho menor para *3A (0.015) 44. Más recientemente, en pacientes pediátricos uruguayos con LLA se ha descrito una frecuencia de *3A de 0.05, similar a la nuestra, pero con una frecuencia menor de *2 (37.
Cada vez hay más evidencia que destaca el papel de las variantes genéticas de NUDT15 en el desarrollo de efectos secundarios de la toxicidad del 6-MP (19,20,36,37. Sin embargo, al evaluar las posibles asociaciones en nuestra población de estudio, no pudimos identificar ninguna asociación con significación estadística, posiblemente debido al tamaño de la muestra. En relación con TPMT, existe evidencia que respalda su papel tanto en la farmacocinética como en los efectos tóxicos de la mercaptopurina (13,16. En nuestro estudio no encontramos asociación entre las frecuencias alélicas de cada polimorfismo ni en esos 42 casos en los que tuvimos la tipificación completa de TPMT. Entre las posibles razones que explicarían esto podría estar, para el caso de NUDT15, que los estudios originales que describen tales asociaciones se basaron en enfoques GWAS 19, en el que la representación de la población latina fue baja, y esto puede deberse a un efecto relacionado con la ascendencia específica de la población colombiana 45. Por otro lado, en el caso de TPMT, una explicación podría ser que los alelos encontrados en nuestra población (*2 y *3A) corresponden a metabolizadores intermedios, que no suelen requerir muchos ajustes en la terapia 13.
En análisis exploratorios se evaluó si existía asociación entre las variantes genéticas estudiadas y los desenlaces: recaídas o muerte, revisados a octubre de 2018, sin mostrar correlación alguna, lo cual es consistente con lo descrito por Lennard y col.46, quienes, después de genotipificar TPMT en 2,387 pacientes con leucemia linfoblástica aguda, no identificaron asociaciones con la supervivencia libre de eventos. Dentro de las limitaciones del presente estudio, se encuentran el tamaño de la muestra y el sesgo de selección, por tratarse de una muestra cautiva. Sin embargo, los pacientes fueron incluidos de forma prospectiva y consecutiva, lo que permitió un mejor seguimiento de la evolución de los eventos adversos de interés. Por otro lado, una gran fortaleza del presente estudio es el abordaje traslacional, basado en observaciones clínicas previas y la evaluación de aspectos biomédicos básicos que podrían dar respuesta a la búsqueda de factores predictivos para el desarrollo de toxicidad en el tratamiento de pacientes pediátrico con enfermedades oncológicas.
Conclusiones
En resumen, las frecuencias alélicas de las variantes de interés fueron diferentes a las descritas anteriormente, incluso al compararlas con estudios latinoamericanos. No encontramos asociación estadística con eventos de toxicidad para ninguno de los polimorfismos estudiados. Estos hallazgos destacan la necesidad de realizar estudios futuros con una mayor muestra de pacientes; ya sea a través de un estudio multicéntrico con pacientes pediátricos con LLA o incluyendo otras poblaciones pediátricas que reciben manejo con tiopurinas, tales como: pacientes con enfermedad inflamatoria intestinal o artritis idiopática juvenil, que nos permitan tener un adecuado tamaño de muestra a partir del cual identificar mejores inferencias de asociaciones con desenlaces de toxicidad. Además, estos hallazgos plantean la posibilidad de que otros genes puedan estar influyendo en la toxicidad y los efectos secundarios y por otro lado examinar el efecto que la ascendencia podría tener en estas interacciones en poblaciones con una mezcla ancestral diversa como nuestra población.
Agradecimientos
Los autores desean agradecer a los pacientes y sus familias quienes aceptaron participar en este estudio, así como al Instituto de Genética de la Universidad Nacional de Colombia por facilitarnos sus instalaciones para la realización de los estudios de genotipificación. Adicionalmente, agradecemos a los Doctores Jun J. Yang y Takaya Moriyama del St. Jude Children's Research Hospital por su invaluable ayuda en la interpretación de los datos genéticos de TPMT.
REFERENCIAS
1. PDQ Pediatric Treatment Editorial Board. Childhood Acute Lymphoblastic Leukemia Treatment (PDQ(R)): Health Professional Version. In: PDQ Cancer Information Summaries. Bethesda (MD): National Cancer Institute (US); 2002-2021. [ Links ]
2. Cocce MC, Alonso CN, Rossi JG, Bernasconi AR, Rampazzi MA, Felice MS, et al. Cytogenetic and Molecular Findings in Children with Acute Lymphoblastic Leukemia: Experience of a Single Institution in Argentina. Molecular syndromology. 2015;6(4):193-203. doi: 10.1159/000441046 [ Links ]
3. Gowda C, Dovat S. Genetic targets in pediatric acute lymphoblastic leukemia. Advances in experimental medicine and biology. 2013;779:327-40. doi: 10.1007/978-1-4614-6176-0_15 [ Links ]
4. Lim JY, Bhatia S, Robison LL, Yang JJ. Genomics of racial and ethnic disparities in childhood acute lymphoblastic leukemia. Cancer. 2014;120(7):955-62. doi: 10.1002/cncr.28531 [ Links ]
5. Winestone LE, Aplenc R. Disparities in Survival and Health Outcomes in Childhood Leukemia. Curr Hematol Malig Rep. 2019;14(3):179-86. doi: 10.1007/s11899-019-00515-x [ Links ]
6. Hunger SP, Mullighan CG. Acute Lymphoblastic Leukemia in Children. The New England journal of medicine. 2015;373(16):1541-52. doi: 10.1056/NEJMra1400972 [ Links ]
7. Hunger SP, Lu X, Devidas M, Camitta BM, Gaynon PS, Winick NJ, et al. Improved survival for children and adolescents with acute lymphoblastic leukemia between 1990 and 2005: a report from the children's oncology group. Journal of clinical oncology. 2012;30(14):1663-9. doi: 10.1200/JCO.2011.37.8018 [ Links ]
8. Tasian SK, Loh ML, Hunger SP. Childhood acute lymphoblastic leukemia: Integrating genomics into therapy. Cancer. 2015;121(20):3577-90. doi: 10.1038/leu.2008.251 [ Links ]
9. Nguyen K, Devidas M, Cheng SC, La M, Raetz EA, Carroll WL, et al. Factors influencing survival after relapse from acute lymphoblastic leukemia: a Children´s Oncology Group study. Leukemia. 2008;22(12):2142-50. doi: 10.1038/leu.2008.251 [ Links ]
10. Prucker C, Attarbaschi A, Peters C, Dworzak MN, Potschger U, Urban C, et al. Induction death and treatment-related mortality in first remission of children with acute lymphoblastic leukemia: a population-based analysis of the Austrian Berlin-Frankfurt-Munster study group. Leukemia. 2009;23(7):1264-9. doi: 10.1038/leu.2009.12 [ Links ]
11. Christensen MS, Heyman M, Mottonen M, Zeller B, Jonmundsson G, Hasle H, et al. Treatment-related death in childhood acute lymphoblastic leukaemia in the Nordic countries: 1992-2001. British journal of haematology. 2005;131(1):50-8. doi: 10.1111/j.1365-2141.2005.05736.x [ Links ]
12. O´Connor D, Bate J, Wade R, Clack R, Dhir S, Hough R, et al. Infection-related mortality in children with acute lymphoblastic leukemia: an analysis of infectious deaths on UKALL2003. Blood. 2014;124(7):1056-61. doi: 10.1182/blood-2014-03-560847 [ Links ]
13. Relling MV, Schwab M, Whirl-Carrillo M, Suarez-Kurtz G, Pui CH, Stein CM, et al. Clinical Pharmacogenetics Implementation Consortium (CPIC) guideline for thiopurine dosing based on TPMT and NUDT15 genotypes: 2018 update. Clinical pharmacology and therapeutics. 2019;105(5):1095-105. doi: 10.1002/cpt.1304 [ Links ]
14. Mei L, Ontiveros EP, Griffiths EA, Thompson JE, Wang ES, Wetzler M. Pharmacogenetics predictive of response and toxicity in acute lymphoblastic leukemia therapy. Blood reviews. 2015;29(4):243-9. doi: 10.1016/j.blre.2015.01.001 [ Links ]
15. Lopez-Lopez E, Gutierrez-Camino A, Bilbao-Aldaiturriaga N, Pombar-Gomez M, Martin-Guerrero I, Garcia-Orad A. Pharmacogenetics of childhood acute lymphoblastic leukemia. Pharmacogenomics. 2014;15(10):1383-98. doi: 10.2217/pgs.14.106 [ Links ]
16. El-Rashedy FH, Ragab SM, Dawood AA, Temraz SA. Clinical implication of thiopurine methyltransferase polymorphism in children with acute lymphoblastic leukemia: A preliminary Egyptian study. Indian journal of medical and paediatric oncology : official journal of Indian Society of Medical & Paediatric Oncology. 2015;36(4):265-70. doi: 10.4103/0971-5851.171553 [ Links ]
17. Adam de Beaumais T, Fakhoury M, Medard Y, Azougagh S, Zhang D, Yakouben K, et al. Determinants of mercaptopurine toxicity in paediatric acute lymphoblastic leukemia maintenance therapy. British journal of clinical pharmacology. 2011;71(4):575-84. doi: 10.1111/j.1365-2125.2010.03867.x [ Links ]
18. Zgheib NK, Akika R, Mahfouz R, Aridi CA, Ghanem KM, Saab R, et al. NUDT15 and TPMT genetic polymorphisms are related to 6-mercaptopurine intolerance in children treated for acute lymphoblastic leukemia at the Children's Cancer Center of Lebanon. Pediatric blood & cancer. 2017;64(1):146-50. doi: 10.1002/pbc.26189 [ Links ]
19. Yang JJ, Landier W, Yang W, Liu C, Hageman L, Cheng C, et al. Inherited NUDT15 variant is a genetic determinant of mercaptopurine intolerance in children with acute lymphoblastic leukemia. Journal of clinical oncology : official journal of the American Society of Clinical Oncology. 2015;33(11):1235-42. doi: 10.1200/JCO.2014.59.4671 [ Links ]
20. Moriyama T, Nishii R, Perez-Andreu V, Yang W, Klussmann FA, Zhao X, et al. NUDT15 polymorphisms alter thiopurine metabolism and hematopoietic toxicity. Nature genetics. 2016;48(4):367-73. doi: 10.1038/ng.3508 [ Links ]
21. Suzuki H, Fukushima H, Suzuki R, Hosaka S, Yamaki Y, Kobayashi C, et al. Genotyping NUDT15 can predict the dose reduction of 6-MP for children with acute lymphoblastic leukemia especially at a preschool age. Journal of human genetics. 2016;61(9):797-801. doi: 10.1038/jhg.2016.55 [ Links ]
22. Chiengthong K, Ittiwut C, Muensri S, Sophonphan J, Sosothikul D, Seksan P, et al. NUDT15 c.415C´T increases risk of 6-mercaptopurine induced myelosuppression during maintenance therapy in children with acute lymphoblastic leukemia. Haematologica. 2016;101(1):e24-6. doi: 10.3324/haematol.2015.134775 [ Links ]
23. Kimura S, Hasegawa D, Yoshimoto Y, Hirabayashi S, Hosoya Y, Yoshihara H, et al. Severe 6-mercaptopurine-induced hematotoxicity in childhood an ALL patient with homozygous NUDT15 missence variants. Rinsho ketsueki. 2016;57(6):748-53. doi: 10.11406/rinketsu.57.748 [ Links ]
24. Liang DC, Yang CP, Liu HC, Jaing TH, Chen SH, Hung IJ, et al. NUDT15 gene polymorphism related to mercaptopurine intolerance in Taiwan Chinese children with acute lymphoblastic leukemia. The pharmacogenomics journal. 2016;16(6):536-9. doi: 10.1038/tpj.2015.75 [ Links ]
25. Choudhry S, Coyle NE, Tang H, Salari K, Lind D, Clark SL, et al. Population stratification confounds genetic association studies among Latinos. Human genetics. 2006;118(5):652-64. doi: 10.1007/s00439-005-0071-3 [ Links ]
26. Kishi S, Cheng C, French D, Pei D, Das S, Cook EH, et al. Ancestry and pharmacogenetics of antileukemic drug toxicity. Blood. 2007;109(10):4151-7. doi: 10.1182/blood-2006-10-054528 [ Links ]
27. Corvol H, De Giacomo A, Eng C, Seibold M, Ziv E, Chapela R, et al. Genetic ancestry modifies pharmacogenetic gene-gene interaction for asthma. Pharmacogenetics and genomics. 2009;19(7):489-96. doi: 10.1097/FPC.0b013e32832c440e [ Links ]
28. Hoang PT, Ambroise J, Dekairelle AF, Durant JF, Butoescu V, Chi VL, et al. Comparative pharmacogenetic analysis of risk polymorphisms in Caucasian and Vietnamese children with acute lymphoblastic leukemia: prediction of therapeutic outcome? British journal of clinical pharmacology. 2015;79(3):429-40. doi: 10.1111/bcp.12481 [ Links ]
29. Suarez-Kurtz G, Pena SD, Struchiner CJ, Hutz MH. Pharmacogenomic Diversity among Brazilians: Influence of Ancestry, Self-Reported Color, and Geographical Origin. Frontiers in pharmacology. 2012;3:191. doi: 10.3389/fphar.2012.00191 [ Links ]
30. Trujillo AM, Linares-Ballesteros A, Sarmiento IC. Intensive chemotherapy in children with acute lymphoblastic leukemia. Interim analysis in a referral center in Colombia. Rev Fac Med 2016;64(3):417-25. doi: 10.15446/revfacmed.v64n3.53961 [ Links ]
31. Yunis LK, Linares A, Cabrera E, Yunis JJ. Systematic molecular analysis of hemophilia A patients from Colombia. Genet Mol Biol. 2018;41(4):750-7. doi: 10.1590/1678-4685-GMB-2017-0072 [ Links ]
32. Ip SC, Lin SW, Lai KM. An evaluation of the performance of five extraction methods: Chelex(R) 100, QIAamp(R) DNA Blood Mini Kit, QIAamp(R) DNA Investigator Kit, QIAsymphony(R) DNA Investigator(R) Kit and DNA IQ. Sci Justice. 2015;55(3):200-8. doi: 10.1016/j.scijus.2015.01.005 [ Links ]
33. Wilfinger WW, Mackey K, Chomczynski P. Effect of pH and ionic strength on the spectrophotometric assessment of nucleic acid purity. Biotechniques. 1997;22(3):474-6, 8-81. doi: 10.2144/97223st01 [ Links ]
34. Desjardins P, Conklin D. NanoDrop microvolume quantitation of nucleic acids. J Vis Exp. 2010; (45): 2565. doi: 10.3791/2565 [ Links ]
35. NIH. ALFA: Allele Frequency Aggregator. National Center for Biotechnology Information, U.S. 2020. Available from: https://www.ncbi.nlm.nih.gov/snp/docs/gsr/alfa/. [ Links ]
36. Singh M, Bhatia P, Khera S, Trehan A. Emerging role of NUDT15 polymorphisms in 6-mercaptopurine metabolism and dose related toxicity in acute lymphoblastic leukaemia. Leuk Res. 2017;62:17-22. doi: 10.1016/j.leukres.2017.09.012 [ Links ]
37. Soler AM, Olano N, Mendez Y, Lopes A, Silveira A, Dabezies A, et al. TPMT and NUDT15 genes are both related to mercaptopurine intolerance in acute lymphoblastic leukaemia patients from Uruguay. British journal of haematology. 2018;181(2):252-5. doi: 10.1111/bjh.14532 [ Links ]
38. Yang JJ, Whirl-Carrillo M, Scott SA, Turner AJ, Schwab M, Tanaka Y, et al. Pharmacogene Variation Consortium Gene Introduction: NUDT15. Clinical pharmacology and therapeutics. 2019;105(5):1091-4. doi: 10.1002/cpt.1268 [ Links ]
39. Shah SAV, Paradkar MU, Desai DC, Ashavaid TF. Preemptive NUDT15 genotyping: redefining the management of patients with thiopurine-induced toxicity. Drug Metab Pers Ther. 2018;33(1):57-60. doi: 10.1515/dmpt-2017-0038 [ Links ]
40. Ossa H, Aquino J, Pereira R, Ibarra A, Ossa R, Perez LA, et al. Outlining the Ancestry Landscape of Colombian Admixed Populations. PloS one. 2016;11(10):e0164414. doi: 10.1371/journal.pone.0164414 [ Links ]
41. Tanaka Y, Kato M, Hasegawa D, Urayama KY, Nakadate H, Kondoh K, et al. Susceptibility to 6-MP toxicity conferred by a NUDT15 variant in Japanese children with acute lymphoblastic leukaemia. British journal of haematology. 2015;171(1):109-15. doi: 10.1111/bjh.13518 [ Links ]
42. Isaza C, Henao J, Lopez AM, Cacabelos R. Allelic variants of the thiopurine methyltransferase (TPMT) gene in the Colombian population. Methods Find Exp Clin Pharmacol. 2003;25(6):423-9. doi: 10.1358/mf.2003.25.6.769646 [ Links ]
43. Garrido C, Santizo VG, Mullers P, Soriano DR, Avila GB, Dean M, et al. Frequency of thiopurine S-methyltransferase mutant alleles in indigenous and admixed Guatemalan patients with acute lymphoblastic leukemia. Med Oncol. 2013;30(1):474. doi: 10.1007/s12032-013-0474-2 [ Links ]
44. Boson WL, Romano-Silva MA, Correa H, Falcao RP, Teixeira-Vidigal PV, De Marco L. Thiopurine methyltransferase polymorphisms in a Brazilian population. The pharmacogenomics journal. 2003;3(3):178-82. doi: 10.1038/sj.tpj.6500175 [ Links ]
45. Norris ET, Wang L, Conley AB, Rishishwar L, Marino-Ramirez L, Valderrama-Aguirre A, et al. Genetic ancestry, admixture and health determinants in Latin America. BMC Genomics. 2018;19(Suppl 8):861. doi: 10.1186/s12864-018-5195-7 [ Links ]
46. Lennard L, Cartwright CS, Wade R, Vora A. Thiopurine methyltransferase and treatment outcome in the UK acute lymphoblastic leukaemia trial ALL2003. British journal of haematology. 2015;170(4):550-8. doi: 10.1111/bjh.13469 [ Links ]
Notas:
Fuente de financiamiento Este Proyecto fue financiado por la Universidad Nacional de Colombia mediante subvención otorgada a Isabel Sarmiento-Urbina, Código del proyecto 35872.
Material suplementario
Tabla 1S Análisis de asociación entre diplotipos de TPMT y desenlaces.
| Desenlaces | Diplotipos TPMT | Valor p | |
|---|---|---|---|
| *1/*1 | *1/otro | ||
| Reducción 6-MP | |||
| Si | 8 | 2 | 0.616 |
| No | 28 | 4 | |
| Interrupción 6-MP | |||
| Si | 10 | 0 | 0.308 |
| No | 26 | 6 | |
| Neutropenia | |||
| Si | 5 | 0 | 1.000 |
| No | 31 | 6 | |
| Recaída | |||
| Si | 4 | 1 | 0.557 |
| No | 32 | 5 | |
| Muerte | |||
| Si | 2 | 0 | 1.000 |
| No | 34 | 6 | |
Los datos se presentan como frecuencias absolutas de cada uno de los desenlaces evaluados. No se identificaron asociaciones estadísticamente significativas entre los diplotipos y los desenlaces de interés.
Tabla 2S Comparación del seguimiento de laboratorios en relación con los genotipos.
| Seguimiento | NUDT15 | TPMT | |||||||
|---|---|---|---|---|---|---|---|---|---|
| rs1800462 | rs1800460 | rs1142345 | |||||||
| C/C | C/T | C/C | C/G | T/C | C/C | C/T | T/T | ||
| 1 | ALT (IU/L) | 46.20 | 50.50 | 49 | 36 | 45.15 | 48.60 | 49.30 | 53 |
| AST (IU/L) | 34.20 | 41.80 | 36.90 | 30.30 | 42.90 | 35.35 | 42.90 | 38.55 | |
| Leucocitos/mm3 | 3,315 | 3,780 | 3,315 | 3,610 | 2,200 | 3,380 | 2,030 | 3,310 | |
| Neutrófilos/mm3 | 1,820 | 1,995 | 1,820 | 1,737 | 980 | 1,910 | 1,020 | 1,980 | |
| Linfocitos/mm3 | 783 | 409 | 780 | 773 | 630 | 783 | 600 | 725 | |
| Bilirubina total (mg/dL) | 0.50 | 0.60 | 0.51 | 0.40 | 0.60 | 0.50 | 0.45 | 0.52 | |
| 2 | ALT (IU/L) | 45 | 74.50 | 55.40 | 28.80 | 26 | 46 | 53.15 | 45 |
| AST (IU/L) | 33.55 | 33.70 | 33.55 | 33.15 | 37 | 32.70 | 42.80 | 38.25 | |
| Leucocitos/mm3 | 3,135 | 2,170 | 3,020 | 3,980 | 2,160 | 3,225 | 2,305 | 3,245 | |
| Neutrófilos/mm3 | 1,835 | 655 | 1,691 | 2,515.50 | 1,295 | 1,835 | 1,440 | 1,835 | |
| Linfocitos/mm3 | 630 | 1,290 | 640 | 475 | 727 | 630 | 583.50 | 746.50 | |
| Bilirubina total (mg/dL) | 0.50 | 0.23 | 0.50 | 0.45 | 0.40 | 0.50 | 0.40 | 0.59 | |
| 3 | ALT (IU/L) | 40 | 59.10 | 39.50 | 89.45 | 37 | 54 | 80.20 | 54 |
| AST (IU/L) | 34.50 | 33.10 | 33.30 | 38.65 | 42.20 | 33.65 | 42.20 | 35 | |
| Leucocitos/mm3 | 3,070 | 2,865 | 3,100 | 2,085 | 3,100 | 3,050 | 3,130 | 3,055 | |
| Neutrófilos/mm3 | 1,635 | 1,430 | 1,645 | 1,175 | 1,440 | 1,635 | 2,680 | 1,540 | |
| Linfocitos/mm3 | 734 | 1,010 | 753 | 470 | 1,030 | 712 | 875 | 768 | |
| Bilirubina total (mg/dL) | 0.50 | 0.30 | 0.50 | 0.60 | 0.50 | 0.50 | 0.50 | 0.50 | |
| 4 | ALT (IU/L) | 52.40 | 18.10 | 52.40 | 33.70 | 41.60 | 52.40 | 138.30 | 63 |
| AST (IU/L) | 31.10 | 32.10 | 31.20 | 36.30 | 47.21 | 31.20 | 89.90 | 30.40 | |
| Leucocitos/mm3 | 3,400 | 3,135 | 3,400 | 2,980 | 2,570 | 3,510 | 2,615 | 3,635 | |
| Neutrófilos/mm3 | 1,940 | 1,720 | 1,955 | 860 | 1,700 | 1,970 | 1,735 | 1,955 | |
| Linfocitos/mm3 | 760 | 740 | 760 | 1,305 | 535 | 800 | 535 | 800 | |
| Bilirubina total (mg/dL) | 0.70 | 0.40 | 0.60 | 0.70 | 0.45 | 0.70 | 0.40 | 0.70 | |
| 5 | ALT (IU/L) | 54 | 23.40 | 55 | 24 | 29.10 | 54 | 67.10 | 60.75 |
| AST (IU/L) | 40.70 | 35.40 | 42.10 | 25.50 | 42.10 | 38.15 | 55.10 | 47.30 | |
| Leucocitos/mm3 | 3,470 | 3,060 | 3,430 | 3,435 | 2,615 | 3,470 | 2,615 | 3,740 | |
| Neutrófilos/mm3 | 1,930 | 1,410 | 1,905 | 1,764 | 1,360 | 1,930 | 1,360 | 1,800 | |
| Linfocitos/mm3 | 845 | 560 | 830 | 900 | 793 | 850 | 845 | 870 | |
| Bilirubina total (mg/dL) | 0.50 | 0.45 | 0.50 | 0.80 | 0.30 | 0.60 | 0.40 | 0.65 | |
| 6 | ALT (IU/L) | 68.55 | 38 | 70.30 | 35.45 | 71 | 66.05 | 71 | 70.10 |
| AST (IU/L) | 41.50 | 30.90 | 43.95 | 31.35 | 53.80 | 40 | 63 | 44.90 | |
| Leucocitos/mm3 | 3,385 | 2,655 | 3,385 | 3,145 | 3,200 | 3,370 | 2,755 | 3,650 | |
| Neutrófilos/mm3 | 1,820 | 1,415 | 1,790 | 1,790 | 1,515 | 1,800 | 1,185 | 1,750 | |
| Linfocitos/mm3 | 905 | 1,060 | 910 | 810 | 681 | 915.50 | 680 | 921 | |
| Bilirubina total (mg/dL) | 0.60 | 0.50 | 0.60 | 0.71 | 0.56 | 0.60 | 0.38 | 0.60 | |
Los datos son presentados como las medianas de cada laboratorio cuantificado. Al comparar cada mediana acorde a al genotipo de cada SNPs no se logró identificar diferencias estadísticamente significantes para ninguna de las mediciones.
Recibido: 17 de Diciembre de 2020; Revisado: 07 de Abril de 2021; Aprobado: 27 de Septiembre de 2021











 text in
text in 



