Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Colombia Médica
versão On-line ISSN 1657-9534
Colomb. Med. vol.53 no.2 Cali jan./jun. 2022 Epub 14-Jun-2022
https://doi.org/10.25100/cm.v53i2.4874
Original articulo
Análisis de la interacción entre los genes FTO e IRX3 con obesidad y desórdenes metabólicos relacionados en una población mixta de América Latina: un posible aumento del riesgo de exceso de peso
1 Universidad de Cartagena, Facultad de Medicina. Cartagena de Indias, Colombia.
2 Centro de Innovación e Investigación en Metabolismo y Diabetes - Innovatid-. Cartagena de Indias, Colombia.
Introducción:
FTO (Fat Mass and Obesity-related) se ha relacionado de manera consistente con la obesidad. Recientemente, Polimorfismos de Nucleótido Único (SNP) en este gen se han relacionado con el gen IRX3.
Objetivo:
Probar la hipótesis de que: i) SNPs en FTO e IRX3 están asociados con la obesidad y trastornos relacionados; ii) existe desequilibrio de ligamiento (LD) significativo entre ambos genes.
Métodos:
se realizó un estudio transversal en la costa caribe colombiana. Se valoraron variables antropométricas y bioquímicas, la obesidad y trastornos metabólicos. Se genotipificaron 4 SNPs: 3 en FTO (rs17817449, rs8050136, rs9939609) y uno en IRX3 (rs3751723). Se estimó el LD entre estos SNPs. Se aplicó un modelo de regresión logística para estimar asociaciones.
Resultados:
Se incluyeron 792 sujetos. FTO e IRX3 no se encontraron en LD (D' ≤0.03; R2 ≤0.03). El genotipo TT (rs9939609) se encontró asociado con la circunferencia de la cintura (p= 0.04; adj-p= 0.01), y el SNP IRX3 con el Exceso de Peso (EP) (OR= 1.06, adj-p= 0.03). Se encontró un haplotipo FTO-IRX3 asociado con EP (G-A-A-T, rs17817449-rs8050136-rs9939609-rs3751723; OR= 0.67, p= 0.04). Esta asociación persistió después del ajuste para una población mixta (p= 0.03).
Conclusiones:
FTO se encontró asociado con la circunferencia de la cintura e IRX3 con EP en adultos latinoamericanos. Estas asociaciones persistieron tras el ajuste por sexo, edad y ascendencia genética. Aunque estos genes no estaban en LD, los hallazgos de un haplotipo entre FTO-IRX3 sugieren una interacción gen-gen asociada con un mayor riesgo de EP.
Palabras clave: Obesidad; enfermedades metabólicas; polimorfismos de nucleótido único; estudios de asociación genética; américa latina; región del caribe; Colombia
Background:
Fat Mass and Obesity-related (FTO) has been one of the genes consistently related to common obesity. Single nucleotide polymorphisms (SNPs) in FTO have been linked with the IRX3 gene.
Aim:
This study was designed by testing the hypothesis that: i) common SNPs in FTO and IRX3 are associated with obesity and related disorders; ii) there is significant linkage disequilibrium between both genes.
Methods:
A cross-sectional study was carried out on the Colombian Caribbean Coast. Anthropometric and biochemical variables were measured, and obesity and metabolic disorders were diagnosed. Four SNPs were genotyped: 3 at FTO locus (rs17817449, rs8050136, rs9939609) and one at IRX3 locus (rs3751723). LD between these SNPs was estimated. A logistic regression model was applied to estimate associations.
Results:
A total of 792 subjects were included. FTO and IRX3 were not in LD (D’≤ 0.03; R2≤ 0.03). TT genotype (rs9939609) was found to be associated with waist circumference (p= 0.04; adj-p= 0.01), and IRX3 SNP with Body Weight Excess (BWE) (OR= 1.06, adj-p= 0.03). One FTO-IRX3 haplotype was associated with BWE (G-A-A-T, rs17817449-rs8050136-rs9939609-rs3751723; OR= 0.67, p= 0.04). The statistical significance of these relations continued after admixture adjustment for a three-hybrid population (p= 0.03).
Conclusions:
FTO was related to waist circumference, and IRX3 was associated with BWE in Latin American adults. This relation remained statistically significant after an adjustment for sex, age, and genetic ancestry was performed. Despite that these genes were not in LD, findings of a haplotype involving FTO-IRX3 suggest a gene-gene interaction associated with an increased risk of BWE.
Keywords: Obesity; metabolic diseases; single nucleotide polymorphism; genetic association study; genetic predisposition to disease; linkage disequilibrium; haplotypes; waist circumference
Contribución del estudio
| 1) ¿Por qué se realizó este estudio? |
| La población latinoamericana ha estado expuesta a una significativa mezcla étnica. En poblaciones mixtas como ésta, comprender las interacciones genotipo/fenotipo detrás de enfermedades complejas como la obesidad y enfermedades relacionadas con la obesidad sigue siendo una tarea desafiante debido a la estratificación genética, dado que representa una fuente de sesgo de confusión. Sin embargo, la diversidad en este tipo de población también es una oportunidad para descubrir factores genéticos asociados que podrían ser útiles para comprender enfermedades complejas como ésta. El presente estudio aporta evidencia al estudiar la hipótesis de que las variantes comunes en los gene FTO e IRX3 se encuentran asociados con la obesidad y trastornos relacionados con el síndrome metabólico; y que además, existe una interacción significativa entre ambos genes, después de ser puestas a prueba en una población latinoamericana en la que se ha descrito previamente una mezcla genética tri-híbrida. |
| 2) ¿Cuáles fueron los resultados más relevantes del estudio? |
| Las variantes genéticas en el gen FTO no se encontraron relacionadas con la obesidad o variables ocmo el peso corporal, trastornos metabólicos (es decir, la HTA, la hiperglucemia, las dislipidemias, la diabetes mellitus tipo 2) y el síndrome metabólico. Por otro lado, el gen IRX3 se asociado con el exceso de peso corporal incluso después del ajuste por estratificación genética. |
| 3¿Qué aportan estos resultados? |
| Los hallazgos del estudio actual no mostraron evidencia de segregación no aleatoria entre las variantes de los genes FTO e IRX3 en esta muestra de adultos latinoamericanos, posiblemente asociado a la subestructura genética y el reciente proceso de mezcla, lo que podría estar interfiriendo con la medición de las regulaciones intergénicas. No obstante, el diseño de otros estudios destinados a describir el papel del gen IRX3 y su producto en el metabolismo energético parecen ser promisorios en la comprensión de la epidemia actual de exceso de peso corporal en poblaciones con alta diversidad genética |
Introducción
La obesidad consituye una prioridad para la salud pública en casi todas las regiones del mundo (1). Su prevalencia ha aumentado continuamente durante las últimas tres décadas, trayendo consigo a un patrón epidémico que ha impactado negativamente a las comunidades de altos, medianos y bajos ingresos2). A medida que aumenta la prevalencia de la obesidad, también lo hace la frecuencia de trastornos metabólicos (e.g., dislipidemias y hipertensión, etc.), que comparten algunas vías fisiopatológicas 3)..
Se ha identificado ampliamente que los factores genéticos están involucrados en la obesidad y la patogénesis de enfermedades relacionadas con la obesidad, como la Diabetes Mellitus tipo 2, la dislipidemia, la hipertensión y las enfermedades cardiovasculares (4). Entre estos, el gen FTO (Fat Mass and Obesity-related) ha sido uno de los genes más consistentemente relacionados con la obesidad común, según los hallazgos de varios estudios de asociación del genoma completo (GWAS) 5,6). El gen FTO juega un papel importante en la regulación del apetito y la homeostasis energética 7). Además, algunas variantes alélicas se han asociado fuertemente con la acumulación de grasa (7).. A pesar de todo esto, el papel de las variaciones en la región cromosómica FTO y la proteína FTO (una desmetilasa nuclear) en los mecanismos celulares responsables del desarrollo de la obesidad 8,9).
Estudios recientes han mostrado que algunas variantes alélicas en el primer intrón del gen FTO podrían estar influyendo en la regulación de la transcripción de genes ubicados cerca de loci10. En este sentido, Smemo et al. (11) (13, y otros autores han descubierto conexiones funcionales de largo alcance entre las primeras variantes de intrón de FTO y la expresión del gen Iroquois Homeobox 3 (IRX3) (1 2,13) . Además, cada vez se exponen más pruebas de que la epistasis puede desempeñar un papel en el riesgo de enfermedad, por lo que el análisis de la relación entre estos dos genes podría ser particularmente promisorio ya que la actividad de la proteína IRX3 se ha asociado con el control del apetito y el fenotipo de obesidad en animales (11.
La población latinoamericana ha estado expuesta a una mezcla étnica significativa. En poblaciones mixtas como ésta, comprender las interacciones genotipo/fenotipo detrás de enfermedades complejas como la obesidad y las enfermedades relacionadas con la obesidad sigue siendo una tarea desafiante dado que la estratificación genética representa una fuente de sesgo debido a fenómenos de confusión. Sin embargo, la diversidad en este tipo de población también es una oportunidad para descubrir factores genéticos asociados que podrían ser útiles para comprender enfermedades complejas como la obesidad.
Sobre la base de esos hallazgos previos, se han desarrollado algunos estudios para analizar el desequilibrio de ligamiento plausible entre las variantes génicas de los genes FTO e IRX314,15. En conjunto, estos resultados han revelado que esta interacción genómica podría identificarse a nivel poblacional, lo que contribuye a comprender los mecanismos de predisposición genética a la obesidad y cómo los factores genéticos influyen en la epidemia actual. Por lo tanto, el presente estudio fue diseñado para proporcionar más evidencia al probar la hipótesis de que i) los SNP comunes en los genes FTO e IRX3 están asociados con la obesidad y los trastornos relacionados; ii) existe una interacción entre ambos genes. Estas hipótesis fueron probadas en una población latinoamericana en la que se ha descrito previamente una mezcla genética tri-híbrida 16.
Materiales y Métodos
Sujetos
Se realizó un estudio transversal en Cartagena de Indias, ciudad de aproximadamente 1 millón de habitantes ubicada en la Costa Caribe Colombiana (17).. Se empleó una muestra de 792 sujetos para alcanzar el 80% de poder de discriminación de la asociación genética con un odds ratio 2,5/3,5 (heterocigotos/homocigotos), asumiendo un resultado con 25% de prevalencia, 25% de frecuencia de alelos menores, desequilibrio de ligamiento completo (D'= 1) y un coeficiente alfa del 5%, según cálculos descritos por otros autores, y como se ha aplicado en un trabajos anteriores 18).. Para este cálculo se utilizó el software Genetic Power Calculator (http://zzz.bwh.harvard.edu/gpc/cc2.html) 19.
Los criterios de inclusión incluyeron sujetos mayores de 18 años, de ambos sexos, que no fuesen hermanos y que residian en áreas urbanas de Cartagena de Indias (ciudad que cuenta con una población mixta con una ascendencia europea predominante (~ 60%) debido a la colonización española entre los siglos XVI y XIX) (20. Los hermanos fueron identificados inicialmente por comparación de apellidos y luego confirmados por teléfono; por lo tanto, los hermanos de primer y segundo grado fueron excluidos para el análisis.
También se excluyeron aquellos que presentaran antecedentes para enfermedades que afectan su peso corporal como trastornos endocrinos primarios, patologías de origen genético, cáncer o tratamiento quirúrgico para obesidad, así como participantes en estado deembarazo o lactantes. Todos los sujetos dieron su consentimiento informado para participar en el estudio, siguiendo las consideraciones del comité de ética de la Universidad de Cartagena. Se excluyeron los sujetos con datos faltantes.
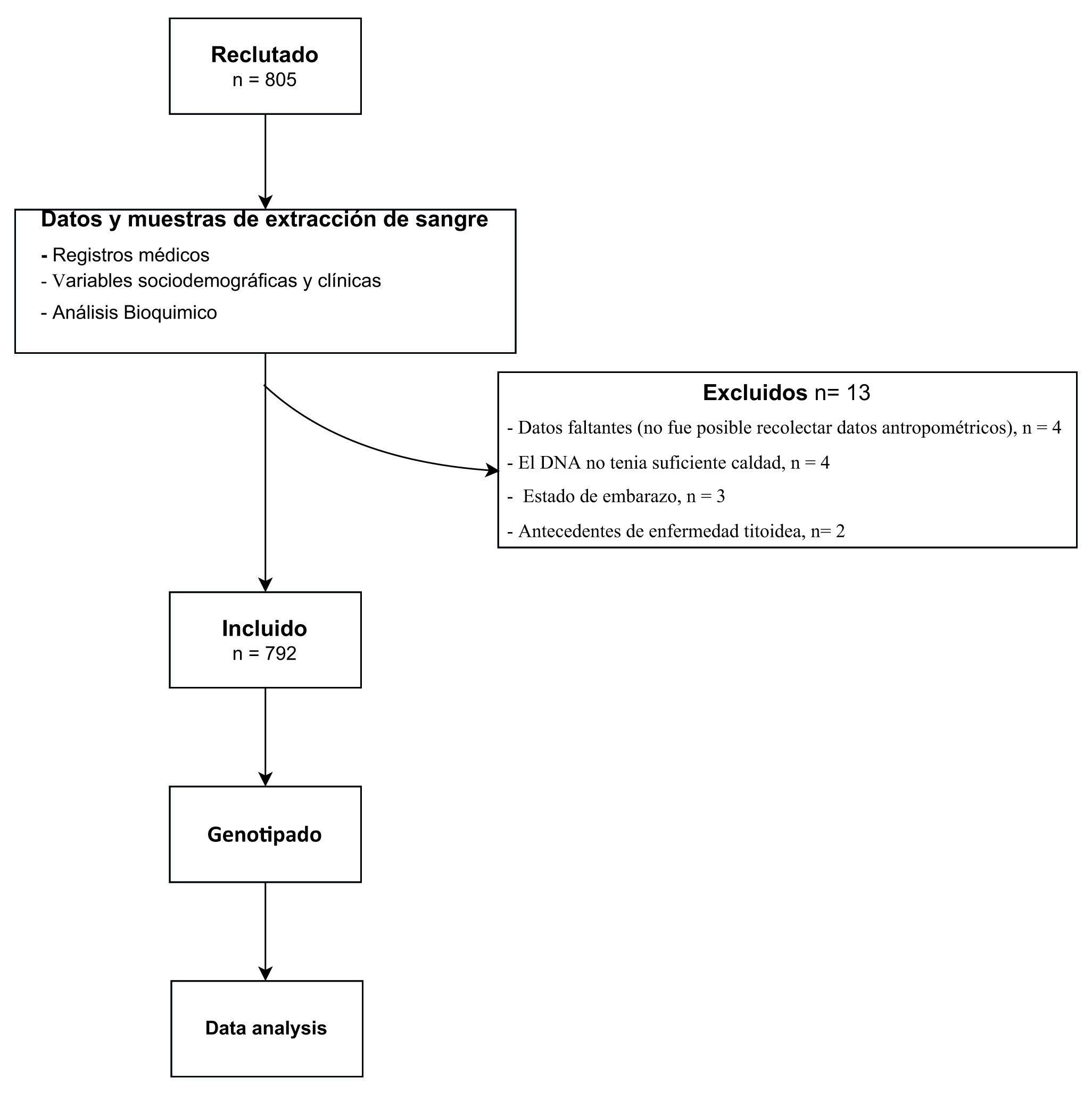
Los participantes fueron valorados por personal médico capacitado encargado de realizar una entrevista y examen físico enfocado en variables sociodemográficas, clínicas y del registro médico de trastornos relacionados con el metabolismo (Figura 1).
Variables antropométricas
Durante el examen físico se obtuvieron los datos antropométricos de cada participante. Se registraron la altura, el peso y la circunferencia Abd. y de la cintura; todas estas mediciones se realizador de acuerdo con las guías internacionales de diabetes para el síndrome metabólico descritas por la Organización Mundial de la Salud (OMS) (21).. La altura se midió en metros (m) utilizando un estadiómetro con el sujeto descalzo y recto. El peso se registró en kilogramos (Kg) a través de una báscula calibrada con el participante usando ropa ligera y sin zapatos. El índice de masa corporal (IMC) se calculó dividiendo el peso en Kg entre el cuadrado de la altura en metros (m2). Se utilizó una cinta métrica inelástica para medir la circunferencia de la cintura y la cadera con el paciente de pie, asegurando que la cinta no comprimiera la piel. La presión arterial se midió mediante un esfigmomanómetro siguiendo el 8º Comité Nacional Conjunto (Joint National Committee , JNC8) 22.
Análisis bioquímico
Se recogió una muestra de sangre total a cada uno de los participantes, que debían contar con un ayuno de al menos 8 horas. Las concentraciones séricas de glucosa, triglicéridos (TG) y colesterol HDL (CHDL) se midieron a través de ensayos colorimétricos en el laboratorio UNIMOL de la Universidad de Cartagena, siguiendo protocolos estándar.
Obesidad y rasgos metabólicos
La obesidad se definió utilizando los puntos de corte para el IMC sugeridos por la OMS 21,23. El exceso de peso corporal (EPC) se definió como sobrepeso u obesidad.
La hipertensión o presión arterial alta (HTA) se determinó de acuerdo con las guías del JNC8 22:
Adultos <60 años: ≥ sistólica 140 mmHg, diastólica ≥90 mmHg
Adultos ≥60 años: sistólica ≥150 mmHg, diastólica ≥90 mmHg
El síndrome metabólico y las condiciones relacionadas se definieron a través de los criterios de la Declaración Provisional Conjunta (JIS), considerando los criterios descritos en otra parte 22,24.
Obesidad Abd.: circunferencia de la cintura en hombres ≥90 cm y mujeres ≥80 cm.
Dislipidemia: Hipertrigliceridemia: TG ≥150 mg/dL o tratamiento por TG sérica altos. CHDL bajo: hombres, CHDL <40 mg/dL; mujeres, CHDL <50 mg/dL.
Hiperglucemia: alteración de la glucosa en ayunas ≥100 mg/dl o tratamiento farmacológico para la glucosa elevada
La diabetes se determinó a través de los criterios de la Asociación Americana de Diabetes (2018) 23,25), considerando la presencia de la enfermedad con valores de glucosa en sangre en ayunas> 126 mg/dL o por valores de HbA1c ≥ 6.5%.
Genotipificación
Las variantes comunes en los genes FTO e IRX3 se identificaron utilizando 1,000 genome project reports. Para ello se utilizó el recurso bioinformático SNiPA26,27. Los SNP con una frecuencia de alelo menor (minor allele frequency, MAF) ≥0.25 y un coeficiente de correlación (R2)= 1.0 se seleccionaron utilizando datos de poblaciones con ascendencia europea. Con este procedimiento, se seleccionaron 3 variantes para FTO: rs9939609 (c.46-23525T>A), rs8050136 (c.46-27777C>A), rs17817449 (c.46-30685T>G) y solo una para IRX3: rs3751723 (c.-235C>A), en base a informes previos donde se analizó el desequilibrio de ligamiento entre ambos genes 14,15,26,28 . Estas variantes son proxies para varias variantes y han mostrado antecedentes de asociación genética con rasgos metabólicos y enfermedades relacionadas con la obesidad 26)..
Los SNP se genotiparon medienta una reacción en cadena de la polimerasa cuantitativa (qPCR) utilizando sondas Taqman (Thermo Fisher Scientific, Inc., Waltham, MA, EE. UU.). Además, la discriminación alélica y los datos fueron analizados por programa informático StepOne Real-Time PCR Software (Thermo Fisher Scientific, Inc., Waltham, MA, USA).
Análisis estadístico
Se describieron variables sociodemográficas, antropométricas, bioquímicas y metabólicas utilizando medidas de tendencia centrales y se describieron las frecuencias según fuera necesario. Se realizó la prueba de Lilliefors para verificar la normalidad. Finalmente, los valores medios se compararon mediante la prueba t y las frecuencias con Ji 2 o la prueba exacta de Fisher.
Las frecuencias alélicas y de genotipo se describieron mediante conteo directo. El equilibrio de Hardy-Weinberg se evaluó a través de los valores del coeficiente de endogamia (Fis) utilizando el software Genetix 4.05. El desequilibrio de ligamiento (LD) entre los SNP seleccionados se estimó con el paquete genetic mediante el software R Program 3.5.0 29; Se reportaron D' (D prima) y R2 (R-cuadrado).
Las asociaciones entre datos continuos y las distribuciones de genotipos se analizaron mediante el análisis de varianza (ANOVA) o la prueba de Kruskal Wallis, según correspondiera. Las relaciones entre variables categóricas (rasgos metabólicos), se determinaron con Ji 2.
Los modelos aditivos se evaluaron mediante modelos de regresión generalizada. También se realizaron modelos de haplotipos para estimar la asociación entre las variante de FTO e IRX3 con la obesidad y las alteraciones metabólicas relacionadas. Estos modelos se ajustaron por edad, sexo e IMC, a excepción de los análisis de exceso de peso corporal (sobrepeso y obesidad), donde esta última variable (IMC) no se empleó debido a la colinealidad. Se empleó la versión 3.5.0 del programa R, con los paquetes genetics y PredictABEL29.
Las asociaciones se ajustaron mediante estratificación genética asumiendo una subestructura de trihíbrida (k= 3) utilizando un enfoque Bayesiano (Markov Chain Monte Carlo, o MCMC) con 100,000 replicaciones utilizando el software STRAT versión 1.1 para DOS / Windows.
El patrón de ancestría tri híbrida aplicada se basó en informes previos de Cartagena de Indias, donde se utilizaron marcadores informativos de ascendencia (AIM) y el cromosoma Y para describir la subestructura genética local y la distribución de la ascendencia 15,16,20,28). En este estudio, repeticiones cortas en tándem del cromosoma 17 Y (Y-STR; AmpFLSTR® Yfiler® PCR Amplification Kit, Thermo Fisher, Inc., EE.UU.) también fueron utilizadas20
Results
Datos generales
Se reclutaron un total de 805 sujetos, 13 fueron excluidos (ver Figura 1). Finalmente, se incluyeron 792 sujetos (55.7% hombres). La mediana de edad fue de 36 RI: (28;52) años. Los rangos de edad de los individuos incluidos se distribuyeron según la pirámide de la población 17,20)..
Variables bioquímicas antropométricas y séricas
Los valores medios de estatura, peso e IMC fueron de 1.66 m RI:(1.60;1.73), 71.3 Kg RI: (60.7;81.5) y 25.6 RI: (22.7;28.6), respectivamente. Para la circunferencia Abd. la mediana fue de 92 cm RI: (84;99). En cuanto a la presión arterial, los valores medios para la presión sistólica y diastólica fueron de 110 mmHg RI: (110-120) y 74 mmHg RI: (70;80) respectivamente.
Respecto a las variables bioquímicas, la mediana del valor glucemia fue de 89.4 RI: (78.6;104.4), la concentración sérica de triglicéridos fue de 151.1 mg/dL RI: (113.9;187.8), de colesterol HDL fue de 46.0 mg/dL RI: (39.2-51.8) y del colesterol total fue de 183.3 mg/dL RI: (160.4;205.4).
Rasgos metabólicos
Alrededor del 56.8% (n= 450/792) de la población general tenía exceso de peso; Entre estos, el 39.7% (n= 315/792) tenía sobrepeso y el 17.1% (n= 135/792) tenía obesidad.
La HTA se encontró en el 13.7% (n= 109/792) de la muestra total. La frecuencia de hiperglucemia fue del 32.0% (n= 254/792), y el 17.1% (n= 136) fue diagnosticada con Diabetes Mellitus tipo 2 (Glucemia ≥126 mg/dL, o tratamiento farmacológico). Más de la mitad de los individuos presentó altas concentraciones séricas de triglicéridos (50.8%, n = 403/792), 43.5% (n = 345/792) bajas concentraciones de colesterol HDL y 38.7% (n = 307/792) tenían altas concentraciones séricas de colesterol total. El síndrome metabólico fue diagnosticado en el 31.9% (n= 253/792) de la población.
Genotipificación
Se realizaron ensayos de genotipificacón en los 792 sujetos. La distribución del genotipo y los valores de Fis para el equilibrio de Hardy-Weinberg se muestran en la Tabla 1.
Tabla 1 Frecuencias alélicas y distribuciones de genotipos para las variantes en los genes FTO e IRX3
| Gen | n | (%) | Fes | p |
|---|---|---|---|---|
| FTO | ||||
| rs9939609 (c.46-23525T>A) | ||||
| Un | 497 | (31.4) | 0.09 | 0.134 |
| T | 1087 | (68.6) | ||
| AA | 89 | (11.2) | ||
| EN | 319 | (40.3) | ||
| TT | 384 | (48.5) | ||
| rs8050136 (c.46-27777C>A) | ||||
| Un | 481 | (30.4) | 0.08 | 0.205 |
| C | 1103 | (69.6) | ||
| AA | 83 | (10.5) | ||
| CA | 315 | (39.8) | ||
| CC | 394 | (49.7) | ||
| rs17817449 (c.46-30685T>G) | ||||
| G | 466 | (29.4) | 0.08 | 0.262 |
| T | 1118 | (70.6) | ||
| GG | 78 | (9.9) | ||
| GT | 310 | (39.1) | ||
| TT | 404 | (51.0) | ||
| IRX3 | ||||
| rs3751723 (c.-235C>A) | ||||
| T | 394 | (24.9) | 0.04 | 0.09 |
| G | 1190 | (75.1) | ||
| TT | 55 | (7.0) | ||
| GG | 453 | (57.2) | ||
| GT | 284 | (35.8) | ||
Fis: coeficiente de endogamia.
Desequilibrio de ligamiento (LD): asociación entre alelos
Los SNP del gen FTO (rs9939609, rs17817449 y rs8050136) no se encontraron en LD con los SNP del gen IRX3 (rs3751723) (D' ≤0.03; R2 ≤0.03).
Asociaciones genéticas
Las variables antropométricas se describieron a partir de medianas y frecuencias, y se compararon según las distribución de los genotipos. En el gen FTO , para el cHDL, los homocigotos TT (rs9939609) mostraron valores más bajos en comparación con el genotipo AT+AA (p= 0.037), los homocigotos CC (rs8050136) a diferencia de los genotipos AC+AA (p= 0.049), y el alelo TT a diferencia del genotipo GG+TT (p= 0.036) (Tabla 2). No se encontró la asociación estadística entre alteraciones metabólicas y genotipos (Tabla 3).
Tabla 2 Mediana de parámetros antropométricos y bioquímicos según el genotipo para SNPs en FTO e IRX3.
| FTO | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| rs9939609 | |||||||||||||
| AA | EN | TT | p | AT+TT | p | AT+AA | p | ||||||
| IMC (Kg/m2) | 25.3 | 22.2-28 | 25.4 | 22.7-28.5 | 25.9 | 22.9-28.9 | 0.2 | 25.7 | 22.8-28.7 | 0.376 | 25.3 | 22.3-28.2 | 0.786 |
| Cir. Cintura (cm) | 88.0 | 80.5-99 | 91.0 | 84.0-99.0 | 93.0 | 85-100 | 0.070 | 92.0 | 85.0-99.0 | 0.079 | 92.0 | 83.0-99.0 | 0.053 |
| Presión Arterial | |||||||||||||
| Sistólica (mmHg) | 110.0 | 100.0-120.0 | 110.0 | 100.0-120.0 | 110.0 | 100.0-120.0 | 0.704 | 110.0 | 100.0-120.0 | 0.403 | 110 | 100.0-120.0 | 0.749 |
| Diastólica (mmHg) | 72.0 | 68.0-80.0 | 72.0 | 68.0-80.0 | 76.0 | 20.0-80.0 | 0.390 | 74.0 | 70.0-80.0 | 0.736 | 74.0 | 68.0-80.0 | 0.258 |
| Concentraciones séricas | |||||||||||||
| Glucosa (mg/dL) | 89.10 | 75.7-99.7 | 89.0 | 78.6-103.6 | 89.5 | 79.6-105.3 | 0.793 | 89.5 | 79.0-104.8 | 0.510 | 89.0 | 78.2-103.0 | 0.697 |
| Triglicéridos | 148.7 | 109.0-175.9 | 152.7 | 114.5-192.0 | 151.9 | 114.0-189.2 | 0.324 | 152.2 | 114.2-191.5 | 0.133 | 151.9 | 113.6-186.6 | 0.581 |
| cHDL | 46.3 | 37.5-51.6 | 46.8 | 40.6-53.7 | 45.1 | 38.9-51.0 | 0.051 | 45.9 | 39.5-51.9 | 0.639 | 46.5 | 39.8-53.5 | 0.037* |
| cTotal | 175.9 | 159.0-197.9 | 184.0 | 162.5-205.5 | 183.9 | 160.1-207.4 | 0.453 | 184.0 | 160.9-206.4 | 0.212 | 182.8 | 161.1-204.0 | 0.786 |
| rs8050136 | |||||||||||||
| AA | AC | CC | p | AC+CC | p | AC+AA | p | ||||||
| IMC (Kg/m2) | 25.3 | 22.2-28 | 25.4 | 22.7-28.2 | 25.9 | 22.9-28.9 | 0.301 | 25.6 | 22.2-28.7 | 0.512 | 25.4 | 22.4-28.2 | 0.123 |
| Cir. Cintura (cm) | 90.0 | 81.5-99.0 | 91.00 | 83.0-99.0 | 92.0 | 85.0-100.0 | 0.310 | 92.0 | 84.0-99.0 | 0.321 | 91.0 | 83.0-99.0 | 0.151 |
| Presión Arterial | |||||||||||||
| Sistólica (mmHg) | 110.0 | 100.0-120.0 | 110.0 | 100.5-120.0 | 110.0 | 100.0-120.0 | 0.846 | 110.0 | 100.0-120.0 | 0.989 | 110.0 | 100.0-120.0 | 0.591 |
| Diastólica (mmHg) | 78.0 | 70.0-81.0 | 72.0 | 68.0-80.0 | 76.0 | 70.0-80.0 | 0.324 | 74.0 | 70.0-80.0 | 0.319 | 74.0 | 68.0-80.0 | 0.475 |
| Concentraciones séricas | |||||||||||||
| Glucosa (mg/dL) | 91.0 | 76.1-103.4 | 88.7 | 78.3-103.2 | 89.6 | 79.8-105.2 | 0.875 | 89.1 | 79.0-104.4 | 0.971 | 88.9 | 78.0-103.2 | 0.620 |
| Triglicéridos | 148.5 | 107.0-175.4 | 151.8 | 114.5-190.3 | 153.4 | 114.1-189.3 | 0.283 | 152.2 | 114.3-189.4 | 0.117 | 151.2 | 113.1-186.4 | 0.427 |
| cHDL | 46.5 | 37.7-52.6 | 46.5 | 40.3-53.7 | 45.1 | 38.9-50.7 | 0.122 | 45.7 | 39.5-51.7 | 0.911 | 46.5 | 39.7-53.5 | 0.049* |
| cTotal | 175.4 | 159.0-198.3 | 184.0 | 161.0-205.4 | 185.0 | 160.9-206.2 | 0.508 | 184.1 | 160.9-206.2 | 0.247 | 182.1 | 160.0-203.8 | 0.605 |
| rs17817449 | |||||||||||||
| GG | GT | TT | p | GT+TT | p | GT+GG | p | ||||||
| IMC (Kg/m2) | 25.3 | 22.2-28.0 | 25.3 | 22.4-28.2 | 25.9 | 22.9-28.9 | 0.232 | 25.6 | 22.8-28.7 | 0.470 | 25.3 | 22.3-28.2 | 0.088 |
| Cir. Cintura (cm) | 89.5 | 81.2-98.7 | 91.0 | 83.0-99.0 | 92.0 | 85.0-100.0 | 0.196 | 92.0 | 84.0-99.0 | 0.304 | 91.0 | 83.0-99.0 | 0.081 |
| Presión Arterial | |||||||||||||
| Sistólica (mmHg) | 100.0 | 100.0-120.0 | 110.0 | 100.0-120.0 | 110.0 | 100.0-120.0 | 0.841 | 110.0 | 100.0-120.0 | 0.613 | 110.0 | 100.0-120.0 | 0.913 |
| Diastólica (mmHg) | 76.0 | 68.5-80.0 | 74.0 | 68.0-80.0 | 76.0 | 70.0-80.0 | 0.499 | 74.0 | 70.0-90.0 | 0.823 | 74.0 | 68.0-80.0 | 0.311 |
| Concentraciones séricas | |||||||||||||
| Glucosa (mg/dL) | 90.6 | 75.2-103.7 | 88.7 | 78.6-102.9 | 89.5 | 79.6-105.3 | 0.924 | 89.2 | 79.0-104.3 | 0.817 | 89.0 | 78.2-103.0 | 0.704 |
| Triglicéridos | 145.2 | 100.5-177.4 | 150.7 | 114.4-187.5 | 154.0 | 114.3-192.6 | 0.135 | 152.2 | 114.4-189.2 | 0.071 | 149.0 | 112.3-185.4 | 0.152 |
| cHDL | 46.7 | 37.5-53.9 | 46.5 | 40.6-53.4 | 45.1 | 38.8-50.8 | 0.100 | 45.7 | 39.5-51.5 | 0.792 | 46.5 | 39.9-53.5 | 0.036* |
| cTotal | 175.2 | 156.0-197.6 | 184.2 | 162.3-206.8 | 184.5 | 160.8-206.0 | 0.336 | 184.3 | 161.3-206.2 | 0.141 | 182.4 | 160.0-204.4 | 0.709 |
| IRX3 | |||||||||||||
| rs3751723 | |||||||||||||
| TT | GT | GG | p | GT+GG | p | GT+TT | p | ||||||
| IMC (Kg/m2) | 25.0 | 21.8-27.8 | 25.4 | 22.7-28.5 | 25.9 | 22.8-28.9 | 0.182 | 25.6 | 22.8-28.8 | 0.132 | 25.3 | 22.7-28.4 | 0.137 |
| Cir. Cintura (cm) | 92.0 | 83.0-100.0 | 92.0 | 84.0-98.7 | 92.0 | 85.0-99.0 | 0.753 | 92.0 | 83.0-99.0 | 0.487 | 92.0 | 83.8-99.0 | 0.621 |
| Presión Arterial | |||||||||||||
| Sistólica (mmHg) | 110.0 | 100.0-120.0 | 110.0 | 102.0-120.0 | 110.0 | 100.0-120.0 | 0.447 | 110.0 | 100.0-120.0 | 0.675 | 110.0 | 102.0-120.0 | 0.204 |
| Diastólica (mmHg) | 76.0 | 70.0-80.0 | 76.0 | 70.0-80.0 | 74.0 | 70.0-80.0 | 0.390 | 74.0 | 70.0-80.0 | 0.266 | 76.0 | 70.0-80.0 | 0.265 |
| Concentraciones séricas | |||||||||||||
| Glucosa (mg/dL) | 90.0 | 81.0-109.2 | 90.0 | 77.0-105.1 | 88.8 | 78.9-103.4 | 0.735 | 89.4 | 78.0-104.2 | 0.435 | 90.0 | 78.0-105.5 | 0.751 |
| Triglicéridos | 151.9 | 119.0-196.9 | 148.4 | 108.9-187.7 | 153.9 | 116.2-186.8 | 0.590 | 152.0 | 113.6-187.3 | 0.729 | 148.6 | 110.0-188.3 | 0.418 |
| cHDL | 45.9 | 39.4-51.0 | 46.1 | 38.9-52.8 | 45.7 | 39.7-51.4 | 0.966 | 46.0 | 39.2-52.2 | 0.874 | 46.1 | 38.9-52.3 | 0.882 |
| cTotal | 177.0 | 153.9-199.1 | 185.9 | 161.7-210.7 | 182.1 | 160.1-203.8 | 0.190 | 183.7 | 160.9-205.7 | 0.296 | 185.5 | 160.4-208.4 | 0.275 |
Los datos son medianas (1er cuartil - 3er cuartil).
Table 3 Frecuencias alélicas y distribuciones de genotipos para los genes FTO e IRX3
| Gen FTO | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| rs9939609 | rs8050136 | rs17817449 | |||||||||||||||||||||||||
| AA | EN | TT | p | AA | CORRIENTE ALTERNA | CC | p | GG | GT | TT | p | ||||||||||||||||
| AA vs AT+TT | TT vs AT+AA | AA vs AC+CC | CC vs AC+AA | GG vs GT+TT | TT vs GT+GG | ||||||||||||||||||||||
| Parámetros antropométricos y bioquímicos | |||||||||||||||||||||||||||
| IMC | |||||||||||||||||||||||||||
| Peso normal | 40 | (44.9) | 148 | (46.4) | 154 | (40.1) | 36 | (43.4) | 145 | (46.0) | 161 | (40.9) | 35 | (44.9) | 144 | (46.5) | 163 | (40.4) | |||||||||
| Sobrepeso | 32 | (36.0) | 123 | (38.6) | 160 | (41.7) | 30 | (36.1) | 124 | (39.4) | 161 | (40.9) | 28 | (35.9) | 119 | (38.4) | 168 | (41.6) | |||||||||
| Obeso | 17 | (19.1) | 48 | (15.0) | 70 | (18.2) | 0.441 | 0.708 | 0.232 | 17 | (20.5) | 46 | (14.6) | 72 | (18.2) | 0,514 | 0.621 | 0.385 | 15 | (19.2) | 47 | (15.1) | 73 | (18.0) | 0,484 | 0.731 | 0.256 |
| Exceso de peso | 49 | (55.1) | 171 | (43.6) | 230 | (59.9) | 47 | (56.6) | 170 | (54.0) | 233 | (59.1) | 43 | (45.1) | 166 | (43.5) | 241 | (59.6) | |||||||||
| CC | |||||||||||||||||||||||||||
| Obesidad Abd. | 59 | (66.3) | 215 | (67.4) | 282 | (73.4) | 57 | (68.7) | 211 | (67.0) | 288 | (73.1) | 54 | (69.2) | 206 | (66.4) | 296 | (73.3) | |||||||||
| No alterado | 30 | (33.7) | 104 | (32.6) | 102 | (26.6) | 0.151 | 0.463 | 0.063 | 26 | (31.3) | 104 | (33.0) | 106 | (26.9) | 0,199 | 0.845 | 0.090 | 24 | (30.8) | 104 | (33.6) | 108 | (26.7) | 0.1399 | 0.946 | 0.064 |
| BP | |||||||||||||||||||||||||||
| HTA | 14 | (15.7) | 46 | (14.4) | 49 | (12.7) | 14 | (16.9) | 47 | (14.9) | 48 | (12.2) | 12 | (15.4) | 44 | (14.2) | 53 | (13.1) | |||||||||
| No alterado | 75 | (84.3) | 273 | (85.6) | 335 | (87.3) | 0.693 | 0.682 | 0.489 | 69 | (83.1) | 268 | (85.1) | 346 | (87.8) | 0,395 | 0.584 | 0.237 | 66 | (84.6) | 266 | (85.8) | 351 | (86.9) | 0.8342 | 0.791 | 0.664 |
| Glucosa sérica | |||||||||||||||||||||||||||
| Hiperglucemia | 24 | (27.0) | 111 | (34.8) | 119 | (30.0) | 26 | (31.3) | 107 | (34.0) | 121 | (30.7) | 24 | (30.8) | 105 | (33.9) | 125 | (30.9) | |||||||||
| No alterado | 65 | (73.0) | 208 | (65.2) | 265 | (70.0) | 0.307 | 0.329 | 0.578 | 57 | (68.7) | 208 | (66.0) | 273 | (69.3) | 0.645 | 0.976 | 0.459 | 54 | (69.2) | 205 | (66.1) | 279 | (69.1) | 0.6843 | 0.895 | 0.535 |
| TG sérica | |||||||||||||||||||||||||||
| HiperTG | 44 | (49.4) | 164 | (51.4) | 195 | (50.8) | 40 | (48.2) | 159 | (50.5) | 204 | (51.8) | 37 | (47.4) | 154 | (49.7) | 212 | (52.5) | |||||||||
| No alterado | 45 | (50.6) | 155 | (48.6) | 189 | (49.2) | 0.945 | 0.859 | 1 | 43 | (51.8) | 156 | (49.5) | 190 | (48.2) | 0.824 | 0.687 | 0.667 | 41 | (52.6) | 157 | (50.3) | 192 | (47.5) | 0.6185 | 0,601 | 0.399 |
| CHDL sérico | |||||||||||||||||||||||||||
| cHDL bajo | 43 | (48.3) | 130 | (40.8) | 172 | (44.8) | 40 | (48.2) | 127 | (40.3) | 178 | (45.2) | 38 | (48.7) | 185 | (40.3) | 182 | (45.0) | |||||||||
| No alterado | 46 | (51.7) | 189 | (59.2) | 212 | (55.2) | 0.353 | 0.397 | 0.544 | 43 | (51.8) | 188 | (59.7) | 216 | (54.8) | 0,288 | 0.433 | 0.400 | 40 | (51.3) | 125 | (56.7) | 222 | (55.0) | 0.2822 | 0.396 | 0.429 |
| TC | |||||||||||||||||||||||||||
| cTotal alto | 59 | (33.7) | 188 | (41.1) | 238 | (38.0) | 29 | (34.9) | 129 | (40.9) | 149 | (37.8) | 25 | (32.1) | 130 | (41.9) | 152 | (37.6) | |||||||||
| No alterado | 30 | (66.3) | 131 | (58.9) | 146 | (62.0) | 0.414 | 0.355 | 0.731 | 54 | (65.1) | 186 | (59.1) | 245 | (62.2) | 0.523 | 0.524 | 0.638 | 53 | (67.9) | 180 | (58.1) | 252 | (62.4) | 0.2214 | 0.246 | 0.549 |
| SMet | |||||||||||||||||||||||||||
| Sí | 27 | (30.3) | 100 | (31.3) | 126 | (32.8) | 27 | (32.5) | 96 | (30.5) | 130 | (33.0) | 25 | (32.0) | 93 | (30.0) | 135 | (33.4) | |||||||||
| No | 62 | (69.7) | 219 | (68.7) | 258 | (67.2) | 0.864 | 0.822 | 0.665 | 56 | (67.5) | 219 | (69.5) | 264 | (67.0) | 0.769 | 1 | 0.579 | 53 | (68.0) | 217 | (70.0) | 269 | (66.6) | 0.624 | 1 | 0.406 |
| Diabetes | |||||||||||||||||||||||||||
| Sí | 13 | (14.6) | 59 | (18.5) | 64 | (16.7) | 14 | (16.9) | 56 | (17.8) | 66 | (16.8) | 13 | (16.7) | 55 | (17.7) | 68 | (16.8) | |||||||||
| No | 76 | (85.4) | 260 | (81.5) | 320 | (83.3) | 0.646 | 0.594 | 0.786 | 69 | (83.1) | 259 | (82.2) | 328 | (83.2) | 0.934 | 1 | 0.827 | 65 | (83.3) | 255 | (82.3) | 336 | (83.2) | 0.942 | 1 | 0.869 |
Los datos son números (%). *p <0.05, Abreviaturas: Obesidad Abd.- obesidad Abdominal., IMC - índice de masa corporal, cHDL - colesterol de lipoproteínas de alta densidad, HiperTG - hipertrigliceridemia, SMet - síndrome metabólico, TG - triglicéridos, CC - circunferencia de la cintura.
En los modelos de regresión donde todos los SNP fueron incluidos como variables, se encontró que el genotipo TT (rs9939609) estaba asociado con la circunferencia de la cintura (p= 0.04; adj-p= 0.01). La variante del gen IRX3 se encotró asociada con EPC (OR= 1.06, adj-p= 0.03) (Tabla 4). Además, un haplotipo FTO-IRX3 se encontró asociado con EPC (G-A-A-T, rs17817449-rs8050136-rs9939609-rs3751723; OR= 0.67, p= 0.04).
Tabla 4 Asociación de variantes de los genes FTO e IRX3 con trastornos metabólicos
| FTO | IRX3 | |||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Rasgos metabólicos | rs9939609 | rs8050136 | rs17817449 | rs3751723 | ||||||||||||||||
| Beta coef. | O | IC del 95% | p* | Ajustado p | Beta coef. | O | IC del 95% | p* | Ajustado p | Beta coef. | O | IC del 95% | p* | Ajustado p | Beta coef. | O | IC del 95% | p* | Ajustado p | |
| Exceso de peso | -0.036 | 0.96 | 0.91-1.01 | 0.156 | 0.225 | -0.026 | 0.97 | 0.92-1.02 | 0.319 | 0.363 | -0.036 | 0.96 | 0.91-1.01 | 0.167 | 0.194 | 0.057 | 1.06 | 1.00-1.12 | 0.039* | 0.024 |
| Abd. Obesidad | -0.043 | 0.95 | 0.91-1.00 | 0.068 | 0.346 | -0.035 | 0.96 | 0.92-1.01 | 0.141 | 0.152 | -0.037 | 0.96 | 0.91-1.01 | 0.123 | 0.131 | -0.007 | 0.99 | 0.94-1.04 | 0.764 | 0.057 |
| HTA | 0.015 | 1.01 | 0.98-1.05 | 0.394 | 0.124 | 0.024 | 1.02 | 0.98-1.06 | 0.175 | 0.076 | 0.011 | 1.01 | 0.97-1.04 | 0.547 | 0.323 | -0.002 | 0.99 | 0.95-1.03 | 0.898 | 0.948 |
| Hiperglucemia | -0.001 | 0.99 | 0.95-1.04 | 0.965 | 0.618 | 0.013 | 1.01 | 0.96-1.06 | 0.590 | 0.396 | 0.010 | 1.01 | 0.96-1.06 | 0.683 | 0.476 | -0.011 | 0.98 | 0.93-1.04 | 0.657 | 0.722 |
| Hiper TG | -0.002 | 0.99 | 0.94-1.05 | 0.925 | 0.879 | -0.016 | 0.98 | 0.93-1.03 | 0.542 | 0.501 | -0.026 | 0.97 | 0.92-1.02 | 0.328 | 0.304 | 0.043 | 1.04 | 0.98-1.10 | 0.124 | 0.254 |
| CHDL bajo | -0.001 | 0.99 | 0.94-1.05 | 0.958 | 0.709 | -0.007 | 0.99 | 0.94-1.04 | 0.787 | 0.650 | -0.005 | 0.99 | 0.94-1.04 | 0.829 | 0.664 | -0.007 | 0.99 | 0.93-1.04 | 0.785 | 0.348 |
| Alto Col | -0.004 | 0.99 | 0.94-1.04 | 0.859 | 0.798 | 0.001 | 1.00 | 0.95-1.05 | 0.952 | 0.824 | -0.001 | 0.99 | 0.94-1.05 | 0.944 | 0.950 | -0.007 | 0.99 | 0.94-1.04 | 0.790 | 0.940 |
| SMet | -0.013 | 0.98 | 0.94-1.03 | 0.592 | 0.758 | -0.010 | 0.98 | 0.94-1.03 | 0.678 | 0.760 | -0.016 | 0.98 | 0.93-1.03 | 0.500 | 0.552 | -0.003 | 0.99 | 0.94-1.04 | 0.889 | 0.499 |
| Diabetes | -0.0009 | 0.99 | 0.96-1.03 | 0.961 | 0.663 | 0.003 | 1.00 | 0.96-1.04 | 0.843 | 0.646 | 0.002 | 1.00 | 0.96-1.04 | 0.889 | 0.705 | -0.004 | 0.99 | 0.95-1.03 | 0.839 | 0.976 |
*p <0.017 se consideraron estadísticamente significativos (corrección de Bonferroni). † ajuste de mezcla con la cadena de Monte-Carlo-Markov (k= 3). Se evaluó un modelo aditivo a través de una regresión logística donde los alelos de riesgo se interpretaron como la unidad. Las variaciones genéticas en rs9939609 (AA= 2, AT= 1 y TT= 0), rs8050136 (AA= 2, AC= 1 y CC= 0), rs17817449 (GG= 2, GT= 1 y TT= 2) y rs3751723 (TT= 0, GT= 1 y GG= 2), se incluyeron como variables independientes. La edad, el sexo y el índice de masa corporal se incluyeron como variables de confusión (excepto para el análisis del exceso de peso corporal en el que solo se utilizaron la edad y el sexo). Se realizó un ajuste de mezcla a través de una cadena Monte-Carlo-Markov donde se asumió una estratificación genética de tres híbridos. Abreviaturas: Abd. obesidad - obesidad Abd., cHDL - colesterol de lipoproteínas de alta densidad, HiperTG - hipertrigliceridemia, SMet - síndrome metabólico.
El modelo aditivo mostró una asociación significativa entre el exceso de peso y el SNP rs3751723 en la generación IRX3 (OR= 1.06, p= 0.024), tomando "G" como alelo de riesgo. La significación estadística de estas relaciones continuó después del ajuste por mezcla para una población de tres híbridos (p= 0.03) (Tabla 4).. Sin embargo, se realizó un análisis adicional y no se encontró significación estadística (Tabla 5).
Tabla 5 Análisis de interacciones entre FTO rs9939609, rs8050136, rs17817449 e IRX3 rs3751723 a obesidad y alteraciones metabólicas
| Rasgos metabólicos | Mejor modelo de interacción | Precisión del equilibrio de entrenamiento | Comprobación de la precisión de la balanza | CVC | O | IC del 95% | p* |
|---|---|---|---|---|---|---|---|
| Exceso de peso | IRX3 rs3751723 | 0.5376 | 0.4937 | 7/10 | 1.05 | 0.28-3.85 | 0.8281 |
| IRX3 rs3751723, FTO rs17817449 | 0.5541 | 0.5198 | 9/10 | 1.22 | 0.32-4.70 | 0.0547 | |
| FTO rs9939609, FTO rs17817449, IRX3 rs3751723 | 0.5642 | 0.5173 | 7/10 | 1.20 | 0.32-4.48 | 0.1719 | |
| >Obesidad Abd. | FTO rs9939609 | 0.5388 | 0.5187 | 6/10 | 1.31 | 0.32-5.39 | 0.3770 |
| FTO rs17817449, IRX3 rs3751723 | 0.5434 | 0.5057 | 4/10 | 1.13 | 0.27-4.65 | 0.3770 | |
| FTO rs9939609, FTO rs8050136, IRX3 rs3751723 | 0.5497 | 0.4902 | 5/10 | 0.98 | 0.23-4.12 | 0.6230 | |
| HTA | FTO rs8050136, | 0.5320 | 0.4940 | 9/10 | 1.32 | 0.16-10.3 | 0.8281 |
| FTO rs8050136, IRX3 rs3751723 | 0.5437 | 0.4315 | 6/10 | 1.93 | 0.88-4.23 | 0.9893 | |
| FTO rs9939609, FTO rs8050136, IRX3 rs3751723 | 0.5524 | 0.4462 | 7/10 | 1.57 | 0.11-21.3 | 0.9893 | |
| Hiperglucemia | FTO rs9939609 | 0.5282 | 0.4902 | 8/10 | 0.98 | 0.24-3.95 | 0.8281 |
| FTO rs9939609, IRX3 rs3751723 | 0.5363 | 0.4818 | 5/10 | 0.89 | 0.21-3.66 | 0.8281 | |
| FTO rs9939609, FTO rs8050136, IRX3 rs3751723 | 0.5430 | 0.4780 | 8/10 | 0.83 | 0.19-3.63 | 0.9893 | |
| Hiper TG | IRX3 rs3751723 | 0.5323 | 0.5164 | 10/10 | 1.27 | 0.34-4.70 | 0.0547 |
| FTO rs9939609, IRX3 rs3751723 | 0.5362 | 0.4992 | 5/10 | 1.11 | 0.29-4.12 | 0.1719 | |
| FTO rs9939609, FTO rs17817449, IRX3 rs3751723 | 0.5426 | 0.5014 | 4/10 | 1.12 | 0.30-4.14 | 0.1719 | |
| cHDL bajo | FTO rs8050136 | 0.5266 | 0.4987 | 5/10 | 1.09 | 0.27-4.39 | 0.6230 |
| FTO rs8050136, IRX3 rs3751723 | 0.5322 | 0.4741 | 5/10 | 0.86 | 0.22-3.29 | 0.8281 | |
| FTO rs9939609, FTO rs8050136, IRX3 rs3751723 | 0.5372 | 0.4691 | 6/10 | 0.89 | 0.22-3.48 | 0.9453 | |
| cTotal alto | IRX3 rs3751723 | 0.5320 | 0.4972 | 7/10 | 1.06 | 0.27-4.09 | 0.6230 |
| FTO rs17817449, IRX3 rs3751723 | 0.5385 | 0.4921 | 8/10 | 1.04 | 0.26-4.21 | 0.8281 | |
| FTO rs9939609, FTO rs17817449, IRX3 rs3751723 | 0.5456 | 0.4778 | 9/10 | 0.85 | 0.19-3.73 | 0.9893 | |
| Diabetes | IRX3 rs3751723 | 0.5291 | 0.4653 | 8/10 | 0.68 | 0.08-5.73 | 0.9990 |
| FTO rs9939609, IRX3 rs3751723 | 0.5468 | 0.4419 | 9/10 | 0.68 | 0.11-4.27 | 0.9990 | |
| FTO rs9939609, FTO rs8050136, IRX3 rs3751723 | 0.5524 | 0.4514 | 10/10 | 0.65 | 0.10-4.20 | 1.00 | |
| SMet | FTO rs17817449 | 0.5195 | 0.4858 | 8/10 | 1.00 | 0.22-4.40 | 0.8281 |
| FTO rs17817449, IRX3 rs3751723 | 0.5306 | 0.4683 | 9/10 | 0.85 | 0.21-3.45 | 0.8281 | |
| FTO rs9939609, FTO rs17817449, IRX3 rs3751723 | 0.5355 | 0.4548 | 6/10 | 0.72 | 0.17-3.02 | 0.9990 |
Abd. obesidad - obesidad Abdominal, cHDL - colesterol de lipoproteínas de alta densidad, HiperTG - hipertrigliceridemia, SMet - síndrome metabólico.
Discusión
En el presente estudio, se evaluaron las variantes genéticas de los genes FTO e IRX3 y su asociación genética con la obesidad y los trastornos relacionados. Además, se analizó el desequilibrio de ligamiento entre ambos genes. De acuerdo con nuestros resultados, se las variantes genéticas en el gen FTO no se encontraron relacionadas con la obesidad o el peso corporal, los trastornos metabólicos (e.g., la HTA, la hiperglucemia, las dislipidemias, la diabetes mellitus tipo 2 y el síndrome metabólico). Por otro lado, IRX3 se encontró asociado con el exceso de peso corporal incluso después del ajuste por estratificación genética. Además, a pesar de informes previos, se encontró que ambos genes estaban en equilibrio de ligamiento en esta población latinoamericana heterogenea (28)..
La variante en FTO se ha asociado repetidamente con la obesidad en poblaciones europeas y asiáticas. En América Latina, Saldaña-Alvárez et al. (30, publicó un estudio de asociación genética en población mexicana en donde cinco SNP en FTO (rs1121980, rs17817449, rs3751812, rs9930506 y rs17817449) se encontraron relacionados con la obesidad clase II/III, mientras que el SNP rs9930506 se asoció con todos los grados de obesidad (I-III) (29,30. Del mismo modo, el SNP rs17817449 en la región FTO se asoció con rasgos glucémicos en adultos de una ciudad andina colombiana 31).. Por otro lado, otros estudios han coincidido con el trabajo actual. Por ejemplo, en un estudio realizado en adultos y niños mexicanos no encontraron una asociaciones significativa entre FTO e IMC (β = 0.187, p = 0.143), y se informó un hallazgo similar para la circunferencia de cintura (β = 0.409, p = 0.208) 32. Tales discrepancias entre los estudios latinoamericanos muestran la complejidad de los análisis de asociación genética en poblaciones mezcladas. Podría pensarse que algunos de los resultados no significativos se deben a la rigurosidad en la aplicación de los procedimientos estadísticos (33).
Es importante tener en cuenta que la mayoría de las comunidades latinoamericanas están experimentando una intensa transición nutricional. Bajo este fenómeno, una gran proporción de la variabilidad interpersonal del IMC estaría influenciada por factores sociales y económicos, como la urbanización y los ingresos, entre otros. Es plausible que la influencia de las variables socioeconómicas podría haber atenuado el impacto de las variaciones genéticas en el gen FTO en la susceptibilidad a la obesidad. Se han observado interacciones similares en otros estudios donde el tamaño del efecto y la significación estadística se modifican por factores ambientales (e.g, comportamientos dietarios) (34,35).
La prevalencia de exceso de peso corporal se asoció con IRX3. Se encontró un aumento del 10% en la frecuencia de sobrepeso u obesidad cuando el genotipo rs3751723 cambió de GG a TT. Este resultado coincide con estudios recientes de múltiples poblaciones en los que la región genómica IRX3 se ha relacionado con rasgos antropométricos y obesidad 14,28,36). Teniendo en cuenta que este gen ha sido identificado como un efector plausible para la influencia de FTO en el peso corporal (10-13), las asociaciones genéticas actuales podrían ser evidencia que apoya el papel biológico de IRX3 en el control de peso, como se ha sugerido en modelos animales 12,13 . De acuerdo con esto, se justifican más estudios centrados en los mecanismos celulares y fisiológicos detrás del papel de este gen en el desarrollo de esta enfermedad.
Además de la sospecha de regulación central del apetito, algunos autores han descrito una asociación de la expresión de IRX3 en el tejido de adipocitos con la disipación de energía (a través de la termogénesis mitocondrial) y el almacenamiento de grasa (12).. Este hallazgo sugiere que los alelos de riesgo en IRX3 podrían estar involucrados en la patogénesis de la obesidad a través de varios mecanismos y que rs3751723 podría estar relacionado con la expresión génica y el metabolismo de los adipocitos. Si los sujetos con esta variación genética muestran un perfil de expresión diferente y la fisiología del tejido adiposo podría ser un enfoque interesante en futuras investigaciones.
Srivastrava et al. (28, reportaron desequilibrio de ligamiento entre ambos genes en una población del norte de la India. Aunque se cree que un fenómeno similar está ocurriendo en otros grupos, los hallazgos del estudio actual no mostraron evidencia de segregación no aleatoria entre los SNPs de FTO e IRX3 en esta muestra de adultos latinoamericanos. A este respecto, la subestructura genética y el reciente proceso de mezcla siguen siendo cuestiones que podrían estar interfiriendo con la medición de las regulaciones intergénicas.
En conclusión, se encontró una asociación genética entre IRX3 y el peso corporal en adultos latinoamericanos. Además, esta relación persistió como estadísticamente significativa después de que se realizó un ajuste por sexo, edad y ascendencia genética. De acuerdo con estos resultados, es plausible que la expresión del gen IRX3 pueda estar involucrada en la prevalencia de la obesidad en esta población; por lo tanto, otros estudios destinados a describir el papel de este gen y su producto en el metabolismo energético parecen ser promisorios en la comprensión de la epidemia actual de exceso de peso corporal.
Reconocimiento:
Este trabajo se llevó a cabo con el apoyo financiero de la Vicerrectoría de Investigaciones, Octava convocatoria para la financiación de proyectos de investigación (Res 02565 de 2016), Colombia. La autor fue apoyado por el Ministerio de Ciencia, Tecnología e Innovación de Colombia (MINCIENCIAS) mediante la resolución no. 2286, 2015.
REFERENCIAS
1. World Health Organization. Global status report on noncommunicable diseases 2014. Geneva: World Health Organization; 2014 2014. https://apps.who.int/iris/handle/10665/148114 [ Links ]
2. GBD 2015 Mortality and Causes of Death Collaborators. Global, regional, and national life expectancy, all-cause mortality, and cause-specific mortality for 249 causes of death, 1980-2015: a systematic analysis for the Global Burden of Disease Study 2015. Lancet. 2016;388(10053):1459-544. DOI: 10.1016/S0140-6736(16)31012-1 [ Links ]
3. Tchernof A, Despres JP. Pathophysiology of human visceral obesity: an update. Physiol Rev. 2013;93(1):359-404. DOI: 10.1152/physrev.00033.2011 [ Links ]
4. Singh RK, Kumar P, Mahalingam K. Molecular genetics of human obesity: A comprehensive review. C R Biol. 2017;340(2):87-108. DOI: 10.1016/j.crvi.2016.11.007 [ Links ]
5. Lindstrom S, Loomis S, Turman C, Huang H, Huang J, Aschard H, et al. A comprehensive survey of genetic variation in 20,691 subjects from four large cohorts. PLoS One. 2017;12(3):e0173997. DOI:https://doi.org/10.1371/journal.pone.0173997 [ Links ]
6. Wang HJ, Hinney A, Song JY, Scherag A, Meng XR, Grallert H, et al. Association of common variants identified by recent genome-wide association studies with obesity in Chinese children: a case-control study. BMC Med Genet. 2016;17:7. DOI: 10.1186/s12881-016-0268-4 [ Links ]
7. Zhao NN, Dong GP, Wu W, Wang JL, Ullah R, Fu JF. FTO gene polymorphisms and obesity risk in Chinese population: a meta-analysis. World J Pediatr. 2019;15(4):382-9. DOI: 10.1007/s12519-019-00254-2 [ Links ]
8. Mauer J, Jaffrey SR. FTO, m(6) Am , and the hypothesis of reversible epitranscriptomic mRNA modifications. FEBS Lett. 2018;592(12):2012-22. DOI: 10.1002/1873-3468.13092 [ Links ]
9. Akbari ME, Gholamalizadeh M, Doaei S, Mirsafa F. FTO Gene Affects Obesity and Breast Cancer Through Similar Mechanisms: A New Insight into the Molecular Therapeutic Targets. Nutr Cancer. 2018;70(1):30-6. DOI: 10.1080/01635581.2018.1397709 [ Links ]
10. Ragvin A, Moro E, Fredman D, Navratilova P, Drivenes O, Engstrom PG, et al. Long-range gene regulation links genomic type 2 diabetes and obesity risk regions to HHEX, SOX4, and IRX3. Proc Natl Acad Sci U S A. 2010;107(2):775-80. DOI: 10.1073/pnas.0911591107 [ Links ]
11. Smemo S, Tena JJ, Kim KH, Gamazon ER, Sakabe NJ, Gomez-Marin C, et al. Obesity-associated variants within FTO form long-range functional connections with IRX3. Nature. 2014;507(7492):371-5. DOI: 10.1038/nature13138 [ Links ]
12. Nowacka-Woszuk J, Pruszynska-Oszmalek E, Szydlowski M, Szczerbal I. Nutrition modulates Fto and Irx3 gene transcript levels, but does not alter their DNA methylation profiles in rat white adipose tissues. Gene. 2017;610:44-8. DOI: 10.1016/j.gene.2017.02.002 [ Links ]
13. Landgraf K, Scholz M, Kovacs P, Kiess W, Korner A. FTO Obesity Risk Variants Are Linked to Adipocyte IRX3 Expression and BMI of Children - Relevance of FTO Variants to Defend Body Weight in Lean Children? PLoS One. 2016;11(8):e0161739. DOI: 10.1371/journal.pone.0161739 [ Links ]
14. Sobalska-Kwapis M, Suchanecka A, Slomka M, Siewierska-Gorska A, Kepka E, Strapagiel D. Genetic association of FTO/IRX region with obesity and overweight in the Polish population. PLoS One. 2017;12(6):e0180295. DOI: 10.1371/journal.pone.0180295 [ Links ]
15. Srivastava A, Mittal B, Prakash J, Srivastava P, Srivastava N, Srivastava N. A multianalytical approach to evaluate the association of 55 SNPs in 28 genes with obesity risk in North Indian adults. Am J Hum Biol. 2017;29(2). DOI: 10.1002/ajhb.22923 [ Links ]
16. Noguera MC, Schwegler A, Gomes V, Briceno I, Alvarez L, Uricoechea D, et al. Colombia's racial crucible: Y chromosome evidence from six admixed communities in the Department of Bolivar. Ann Hum Biol. 2014;41(5):453-9. DOI: 10.3109/03014460.2013.852244 [ Links ]
17. FAO, OMS. Panorama de la Inseguridad Alimentaria y Nutricional en América Latina y el Caribe 2016. Santiago de Chile. 2017. DOI: 10.37774/9789275319727 [ Links ]
18. Mora-Garcia G, Ruiz-Diaz MS, Espitia-Almeida F, Gomez-Camargo D. Variations in ADIPOR1 But Not ADIPOR2 are Associated With Hypertriglyceridemia and Diabetes in an Admixed Latin American Population. Rev Diabet Stud. 2017;14(2-3):311-28. DOI: 10.1900/RDS.2017.14.311 [ Links ]
19. Hong EP, Park JW. Sample size and statistical power calculation in genetic association studies. Genomics Inform. 2012;10(2):117-22. DOI: 10.5808/GI.2012.10.2.117 [ Links ]
20. Gómez D. Genetic Structure of Cartagena de Indias Population Using Hypervariable Markers of Y Chromosome. Open Journal of Genetics. 2015; 5(1):15. DOI: 10.4236/ojgen.2015.51003 [ Links ]
21. Alberti KG, Zimmet P, Shaw J, Group IDFETFC. The metabolic syndrome--a new worldwide definition. Lancet. 2005;366(9491):1059-62. DOI: 10.1016/S0140-6736(05)67402-8 [ Links ]
22. James PA, Oparil S, Carter BL, Cushman WC, Dennison-Himmelfarb C, Handler J, et al. 2014 evidence-based guideline for the management of high blood pressure in adults: report from the panel members appointed to the Eighth Joint National Committee (JNC 8). JAMA. 2014;311(5):507-20. DOI: 10.1001/jama.2013.284427 [ Links ]
23. Obesity WHOCo, World Health Organization. Division of Noncommunicable D, World Health Organization. Programme of Nutrition F, Reproductive H. Obesity: preventing and managing the global epidemic: report of a WHO Consultation on Obesity, Geneva, 3-5 June 1997. Geneva: World Health Organization; 1998. https://apps.who.int/iris/handle/10665/63854 [ Links ]
24. Mora GG, Salguedo MG, Ruiz DM, Ramos CE, Alario BA, Fortich A, et al. Concordancia entre cinco definiciones de síndrome metabólico. Cartagena, Colombia. Rev Esp Salud Publica. 2012;86(3):301-11. DOI: 10.1590/S1135-57272012000300009 [ Links ]
25. American Diabetes Association. 2 Classification and Diagnosis of Diabetes: Standards of Medical Care in Diabetes-2018. Diabetes Care. 2018;41(Suppl 1):S13-S27. DOI: 10.2337/dc18-S002 [ Links ]
26. Arnold M, Raffler J, Pfeufer A, Suhre K, Kastenmuller G. SNiPA: an interactive, genetic variant-centered annotation browser. Bioinformatics. 2015;31(8):1334-6. [ Links ]
27. Genomes Project C, Abecasis GR, Auton A, Brooks LD, DePristo MA, Durbin RM, et al. An integrated map of genetic variation from 1,092 human genomes. Nature. 2012;491(7422):56-65. DOI: 10.1093/bioinformatics/btu779 [ Links ]
28. Srivastava A, Mittal B, Prakash J, Srivastava P, Srivastava N, Srivastava N. Association of FTO and IRX3 genetic variants to obesity risk in north India. Ann Hum Biol. 2016;43(5):451-6. DOI: 10.3109/03014460.2015.1103902 [ Links ]
29. R Core Team. R: A Language and Environment for Statistical Computing. Vienna, Austria: R Foundation for Statistical Computing; 2018. [ Links ]
30. Saldana-Alvarez Y, Salas-Martinez MG, Garcia-Ortiz H, Luckie-Duque A, Garcia-Cardenas G, Vicenteno-Ayala H, et al. Gender-Dependent Association of FTO Polymorphisms with Body Mass Index in Mexicans. PLoS One. 2016;11(1):e0145984. DOI: 10.1371/journal.pone.0145984 [ Links ]
31. Caro-Gomez MA, Naranjo-Gonzalez CA, Gallego-Lopera N, Parra-Marin MV, Valencia DM, Arcos EG, et al. Association of Native American ancestry and common variants in ACE, ADIPOR2, MTNR1B, GCK, TCF7L2 and FTO genes with glycemic traits in Colombian population. Gene. 2018;677:198-210. DOI: 10.1016/j.gene.2018.07.066 [ Links ]
32. Villalobos-Comparan M, Antuna-Puente B, Villarreal-Molina MT, Canizales-Quinteros S, Velazquez-Cruz R, Leon-Mimila P, et al. Interaction between FTO rs9939609 and the Native American-origin ABCA1 rs9282541 affects BMI in the admixed Mexican population. BMC Med Genet. 2017;18(1):46. DOI: 10.1186/s12881-017-0410-y [ Links ]
33. Little J, Higgins JP, Ioannidis JP, Moher D, Gagnon F, von Elm E, et al. STrengthening the REporting of Genetic Association Studies (STREGA): an extension of the STROBE statement. PLoS Med. 2009;6(2):e22. DOI: 10.1371/journal.pmed.1000022 [ Links ]
34. Wang T, Heianza Y, Sun D, Huang T, Ma W, Rimm EB, et al. Improving adherence to healthy dietary patterns, genetic risk, and long term weight gain: gene-diet interaction analysis in two prospective cohort studies. BMJ. 2018;360:j5644. DOI: 10.1136/bmj.j5644 [ Links ]
35. Ma J, Hennein R, Liu C, Long MT, Hoffmann U, Jacques PF, et al. Improved Diet Quality Associates With Reduction in Liver Fat, Particularly in Individuals With High Genetic Risk Scores for Nonalcoholic Fatty Liver Disease. Gastroenterology. 2018;155(1):107-17. DOI: 10.1053/j.gastro.2018.03.038 [ Links ]
36. Liu C, Chu C, Zhang J, Wu D, Xu D, Li P, et al. IRX3 is a genetic modifier for birth weight, adolescent obesity and transaminase metabolism. Pediatr Obes. 2018;13(3):141-8. DOI: 10.1111/ijpo.12214 [ Links ]
Notas:
Financiación: Este trabajo fue apoyado por vicerrectoría de Investigaciones, Octava convocatoria para la financiación de proyectos de investigación (Res 02565 de 2016) Colombia. M.S. Ruiz-Díaz fue apoyado por el Ministerio de Ciencia, Tecnología e Innovación de Colombia (MINCIENCIAS) mediante la resolución no. 2286, 2015.
Recibido: 21 de Mayo de 2021; Revisado: 08 de Marzo de 2022; Aprobado: 10 de Mayo de 2022











 texto em
texto em