Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Colombia Médica
versão On-line ISSN 1657-9534
Colomb. Med. vol.54 no.1 Cali jan./mar. 2023 Epub 30-Mar-2023
https://doi.org/10.25100/cm.v54i1.5300
Articulo original
Extracción automatizada de información en español de texto libre de informes de patología oncológica
1 Universidad Nacional de Colombia, Facultad de Medicina, Departamento de Patología, Bogotá, Colombia.
2 Quantil SAS. Bogotá, Colombia
3 Centro de Analítica para Políticas Públicas. Bogotá, Colombia
4 Fundación Valle del Lili; Departamento de Patología, Cali, Colombia
5 Universidad de los Andes, Facultad de Economía. Bogotá, Colombia
6 Fundación Valle del Lili; Departamento de Hemato-Oncología, Cali, Colombia
7 Fundación Valle del Lili, Centro de Investigaciones Clínicas, Cali, Colombia
8 Universidad Icesi, Centro PROESA, Cali, Colombia
Introducción:
Los reportes de patología están almacenados como texto libre sin estructura, gramática, fragmentados o abreviados, con variabilidad lingüística entre patólogos. Por esta razón, la extracción de información de tumores requiere un esfuerzo humano significativo. Almacenar información en un formato eficiente y de alta calidad es esencial para implementar y establecer un registro hospitalario de cáncer.
Objetivo:
Este estudio busca describir la implementación de un algoritmo de Procesamiento de Lenguaje Natural para reportes de patología oncológica.
Métodos:
Desarrollamos un algoritmo para procesar reportes de patología oncológica en Español, con el objetivo de extraer 20 descriptores médicos. El abordaje se basa en la coincidencia sucesiva de expresiones regulares.
Resultados:
La validación se hizo con 140 reportes de patología. La identificación topográfica se realizó por humanos y por el algoritmo en todos los reportes. La morfología fue identificada por humanos en 138 reportes y por el algoritmo en 137. El valor de coincidencias parciales (fuzzy matches) promedio fue de 68.3 para Topografía y 89.5 para Morfología.
Conclusiones:
Se hizo una validación preliminar del algoritmo contra extracción humana sobre un pequeño grupo de reportes, con resultados satisfactorios. Esto muestra que múltiples atributos del espécimen pueden ser extraídos de manera precisa de texto libre de reportes de patología en Español, usando un abordaje de expresiones regulares. Adicionalmente, desarrollamos una página web para facilitar la validación colaborativa a gran escala, lo que puede ser beneficioso para futuras investigaciones en el tema.
Palabras clave: Registro del programa nacional de cancer; inteligencia artificial; aprendizaje de ontologia; ciencia de los datos; reportes em patologia del cancer; expresiones regulares; algoritmo
Background:
Pathology reports are stored as unstructured, ungrammatical, fragmented, and abbreviated free text with linguistic variability among pathologists. For this reason, tumor information extraction requires a significant human effort. Recording data in an efficient and high-quality format is essential in implementing and establishing a hospital-based-cancer registry
Objective:
This study aimed to describe implementing a natural language processing algorithm for oncology pathology reports.
Methods:
An algorithm was developed to process oncology pathology reports in Spanish to extract 20 medical descriptors. The approach is based on the successive coincidence of regular expressions.
Results:
The validation was performed with 140 pathological reports. The topography identification was performed manually by humans and the algorithm in all reports. The human identified morphology in 138 reports and by the algorithm in 137. The average fuzzy matching score was 68.3 for Topography and 89.5 for Morphology.
Conclusions:
A preliminary algorithm validation against human extraction was performed over a small set of reports with satisfactory results. This shows that a regular-expression approach can accurately and precisely extract multiple specimen attributes from free-text Spanish pathology reports. Additionally, we developed a website to facilitate collaborative validation at a larger scale which may be helpful for future research on the subject.
Key words: National Program of Cancer Registries; artificial intelligence; ontology learning; data science; cancer pathology reports; regular expressions; algorithm
Contribución del estudio
| 1) ¿Por qué se realizó este estudio? |
| Nuestro Registro de Cáncer de base Hospitalario es implementado en Enero de 2014. Este estudio fue realizado por la necesidad de extracción efectiva y análisis de características tumorales de reportes de oncología almacenados en el egistro. |
| 2) ¿Cuáles fueron los resultados más relevantes del estudio? |
| Se desarrolló un algoritmo usando inteligencia artificial para procesar lenguaje natural. Se consiguió concordancia adecuada con respecto a la evaluación humana en relación a los parámetros críticos para determinar frecuencias, topografía y morfología de los tumores. |
| 3¿Qué aportan estos resultados? |
| Este estudio presenta una herramienta para clasificar enfermedades oncológicas y un sistema de notificación que facilita la implementación de un registro de cáncer. |
Introducción
Los registros de cáncer recolectan, almacenan, analizan y acceden a información de cáncer de una determinada población 1. Estos guardan datos demográficos, características del cáncer, información de tratamiento y desenlaces del paciente para monitorizar e identificar prevenciones de cáncer y métodos de control. La información viene de bases de datos de cuidados de la salud, incluyendo registros electrónicos, imágenes diagnósticas, exámenes de laboratorio y reportes de patología, los cuales resultaron en variables estructuradas y datos no estructurados 2. Usualmente la información más relevante para casos de cáncer está incluida en el reporte de patología. Esos reportes siguen un formato preestablecido en un texto no estructurado que no tiene gramática, es fragmentado, abreviado y con variabilidad lingüística entre patólogo 3) . En este escenario, la tarea de extracción requiere de un esfuerzo tedioso que los humanos realizan manualmente.
El Procesamiento de Lenguaje Natural es un campo de la inteligencia artificial que combina técnicas lingüísticas, estadísticas y computacionales para analizar y representar el lenguaje humano en un formato legible por máquinas 4. El Procesamiento de Lenguaje Natural ha demostrado potencial para automatizar procesos de extracción de información del sector de la salud 5,6. Se han publicado estudios que usan aplicaciones de Procesamiento de Lenguaje Natural para extraer información de reportes de patología de cáncer en Inglés, Holandés, Francés, Alemán 7, e Italiano 8 y están principalmente enfocados en extraer características sencillas 8 o sólo unas cuantas 9. Se ha realizado un esfuerzo similar referente a la extracción de datos de reportes radiológicos 10 y de salud pública en Español 11. Usando técnicas adicionales como el “deep learning”, otro subcampo de la inteligencia artificial, los investigadores han extraído características de registros clínicos de texto libre de cáncer de pulmón, en Español 12. Para hacer esto, siguen un proceso de tres pasos que incluye el uso de Procesamiento de Lenguaje Natural para reconocimiento de la entidad. Sin embargo, su modelo usa técnicas de aprendizaje supervisadas (e.j. deep learning), lo que requiere anotar manualmente siete características (entidad del cáncer, estadio, fechas, eventos, miembros familiares, tratamiento y fármacos) en 14,759 frases. Otros autores buscan posteriores afinamientos 13, usando técnicas de “deep learning”, para extraer once características similares.
En este proyecto apuntamos a implementar un algoritmo que automáticamente extrajera 20 características claves del cáncer en reportes de patología oncológica, escritos en Español, de un registro de cáncer hospitalario.
Materiales y Métodos
Base de datos
La Fundación Valle del Lilí es un Hospital Universitario de alta complejidad, sin ánimo de lucro, ubicado en Cali, Colombia; su registro de cancer de base hospitalario incluye pacientes diagnosticados con cáncer desde el 1ro de Enero del 2014 14. Los datos están almacenados en una plataforma digital propiedad de la institución, que se ajusta a las recomendaciones del Facility Oncology Registry Data Standards (FORDS) 2016 15.
Obtuvimos un corpus de texto de reportes de patología oncológica del registro de cáncer intrahospitalario. El corpus consistía de texto no estructurado de 22,322 reportes de patología oncológica, anonimizados, diagnosticados desde el 1 de enero del 2014 hasta el 13 de noviembre del 2019. Cada reporte incluía tres secciones de texto libre: diagnóstico patológico, descripción macroscópica y microscópica (Tabla 1).
Tabla 1 Ejemplos de texto libre de reportes de patología en Español
| Descripción macroscópica | Descripción microscópica | Diagnóstico |
|---|---|---|
| Tres fragmentos de mucosa gástrica. Se procesa todo en 1 canastilla | Mucosa gástrica antral infiltrada por glándulas malignas | Mucosa gástrica antral. Biopsia Adenocarcinoma bien diferenciado |
| Se recibe en un tubo con EDTA, aproximadamente 4 ml de médula ósea | Población patológica: 48% de blastos mieloides CD34+, CD117+, CD33+, CD13+, cMPOdim, CD56 parcial, HLA-DR+. | Proliferación de blastos mieloides del 48% compatibles con leucemia mieloide aguda con cambios relacionados a mielodisplasia |
| Se recibe rotulado como “dorso lumbar izquierda”, fragmento de piel de 5.5x4.5x3.0 cm | Melanoma nodular fase de crecimiento vertical Nivel de Clark IV Espesor de Breslow 1.5 cm | Dorso lumbar izquierdo. Lesión. Biopsia: Los hallazgos histológicos observados muestran melanoma nodular |
| “mama derecha” se reciben 11 fragmentos de tejido, el mayor de 1.6x0.2cm. Se procesa todo en 3 canastillas. | 5. Patrón morfológico y tipo histológico: Carcinoma invasivo, tipo indeterminado | Mama derecha. Biopsia Trucut: Carcinoma invasivo, tipo indeterminado score de Nottingham 3 (9/9) |
| “Tumor colon derecho”: siete fragmentos de tejido blanquecino y blando, el mayor de 0.2x0.2 cm. Se procesa todo en una canastilla. | La totalidad de la muestra corresponde a una lesión neoplásica maligna de origen epitelial | Mucosa de colon. Colonoscopia. Lesión. Biopsia: Adenocarcinoma |
Descriptores a extraer de reportes de patología
Se extrajeron veinte características claves del cáncer descrito en reportes de patología oncológicos almacenados en el registro de cáncer hospitalario. Se incluyeron estos descriptores de interés en el módulo de “Identificación del Cáncer”. Se adaptaron las recomendaciones del FORDS 2016 al Registro de Notificación Obligatoria establecidos por el Instituto Nacional de Salud de Colombia 16 en la Resolución 247 del 2014.
Dividimos cada descriptor extraído en cuatro grupos de acuerdo con su relevancia clínica y el tipo de valor que podían tomar.
Descriptores principales
Las variables topografía (identifica la localización anatómica donde se encontró la malignidad) y morfología (determina el tipo microscópico de las células tumorales) contienen la información más relevante en el reporte patológico ya que constituyen la base de la clasificación del caso. Ambos descriptores toman valores en la forma de texto libre
Descriptores complementarios
Estos descriptores contienen información valiosa relacionada al tumor primario (identificado con los descriptores primarios). Pueden ser clasificados en diversas categorías como se muestra en la Tabla 2.
Tabla 2 Descriptores extraídos de cada reporte de patología oncológica. La primera columna muestra el nombre del descriptor y su definición, la segunda el tipo de valor que puede tomar, la tercera la descripción de esos valores.
| Nombre del descriptor y definición | Valor | Significado | |
|---|---|---|---|
| Descriptores principales | Topografía. Identifica el sitio anatómico donde se encontró la malignidad | Texto libre | Como escribió el patólogo |
| Morfología. Identifica el tipo de células tumorales microscópicamente | Texto libre | Como escribió el patólogo | |
| Descriptores complementarios | Lateralidad Identifica el lado de un órgano par o el lado del cuerpo de donde se originó el tumor | 0 | Órgano impar |
| 1 | Lado derecho | ||
| 2 | Lado izquierdo | ||
| 9 | Órgano par, lateralidad desconocida | ||
| Comportamiento Describe el comportamiento clínico tumoral | 0 | Benigno | |
| 1 | Limítrofe | ||
| 2 | In situ | ||
| 3 | Invasivo | ||
| Grado Describe la similitud tumoral con el tejido normal | 1 | Bien diferenciado | |
| 2 | Moderadamente diferenciado | ||
| 3 | Pobremente diferenciado | ||
| 4 | No diferenciado | ||
| 5 | Células T | ||
| 6 | Células B | ||
| 8 | Células NK | ||
| 9 | Desconocido | ||
| Método de Evaluación del Tumor Sólido Registra el método diagnóstico usado para diagnosticar el cáncer sólido | 0 | No tumor sólido | |
| 1 | Histología positiva | ||
| 2 | Citología positiva | ||
| 9 | Desconocido | ||
| Método de Evaluación para Tumores Hematológicos Registra el método diagnóstico usado para diagnosticar el cáncer hematológico | 0 | No tumor hematológico | |
| 3 | Histología positiva | ||
| Procedimiento diagnóstico Registra el procedimiento diagnóstico realizado para confirmar el cáncer | 1 | La biopsia no es el sitio primario | |
| 2 | La biopsia es el sitio primario | ||
| 3 | Exploración | ||
| 5 | Cirugía | ||
| 9 | Desconocido | ||
| Invasión linfovascular Indica la presencia o ausencia de células tumorales en canales linfáticos o vasos sanguíneos | 0 | Ausente | |
| 1 | Presente | ||
| 8 | No aplica | ||
| 9 | Desconocido | ||
| Márgenes quirúrgicas Registra si las márgenes del tumor presentaron el compromiso macroscópico o microscópico | 0 | Sin compromiso residual | |
| 1 | Con tumor residual; NOS | ||
| 2 | Tumor microscópico residual | ||
| 3 | Tumor macroscópico residual | ||
| 9 | Desconocido | ||
| Metástasis hepáticas Identifica si el hígado es un sitio con compromiso metastásico | 0 | Ausente | |
| 1 | Presente | ||
| 8 | No aplica | ||
| 9 | Desconocido | ||
| Metástasis pulmonar Identifica si el pulmón es un sitio con compromiso metastásico | 0 | Ausente | |
| 1 | Presente | ||
| 8 | No aplica | ||
| 9 | Desconocido | ||
| Metástasis cerebral Identifica si el cerebro es un sitio con compromiso metastásico | 0 | Ausente | |
| 1 | Presente | ||
| 8 | No aplica | ||
| 9 | Desconocido | ||
| Metástasis ósea Identifica si el hueso es un sitio con compromiso metastásico | 0 | Ausente | |
| 1 | Presente | ||
| 8 | No aplica | ||
| 9 | Desconocido | ||
| Metástasis de nódulos linfáticos distales Identifica si los nódulos linfáticos distales son un sitio con compromiso metastásico | 0 | Ausente | |
| 1 | Presente | ||
| 8 | No aplica | ||
| 9 | Desconocido | ||
| Otras metástasis Identifica si hay compromiso metastásico diferente al hígado, pulmón, cerebro y nódulos linfáticos distantes | 0 | Ausente | |
| 1 | Presente | ||
| 8 | No aplica | ||
| 9 | Desconocido | ||
| Descriptores especiales | TNM Registra la estadificación TNM registrada por el Patólogo | Texto libre | Como escribió el patólogo |
| Tamaño tumoral Registra la medida más precisa de un tumor sólido primario | Numérico | Dos o tres dimensiones | |
| Nódulos linfáticos examinados Registra el número exacto de nódulos linfáticos examinados por el patólogo | Numérico | Numérico | |
| Nódulos linfáticos positivos Registra el número exacto de nódulos linfáticos regionales examinados por el Patólogo, en los que se encontrara cáncer | Numérico | Numérico |
Descriptores relacionados a la metástasis
Estos descriptores identifican si el órgano mencionado es un sitio metastásico y evalúan el compromiso pulmonar, óseo, hepático, cerebral y de nódulos linfáticos distales, así como otras metástasis. La puntuación de los descriptores fue: 0: NO sitio metastásico; 1: sitio metastásico; 8: no aplica; 9: desconocido.
Descriptores especiales
Este grupo de descriptores tienen diferentes posibles valores y proveen información complementaria que puede no estar presentes o incluso no ser aplicables en una cantidad considerable de reportes patológicos. Estos descriptores son: Número de nódulos linfáticos examinados, número de nódulos linfáticos seccionados cerca al tumor positivos, el tamaño del tumor y la clasificación según Tumor, nódulos Linfáticos y Metástasis (TNM).
Cada descriptor podía tomar hasta dos valores diferentes: No aplica (NA) y desconocido o no reportado (NR). El No aplica se usaba cuando el descriptor no aplicaba al procedimiento o tipo de cáncer reportado; por ejemplo, no tiene sentido evaluar el tumor residual y márgenes en caso de una biopsia. No reportado se usaba cuando el descriptor aplicaba pero no lo mencionaban en el reporte.
Construcción del algoritmo
Los descriptores del texto de reporte de patología fueron extraídos usando técnicas de Procesamiento de Lenguaje Natural, particularmente el procesamiento de expresiones regulares y la coincidencia aproximada de cadenas.
Este proyecto fue desarrollado en Python, y se implementó un módulo que contenía un algoritmo para extraer cada descriptor. Cada algoritmo seguía en cierta medida los siguientes pasos (Figura 1):
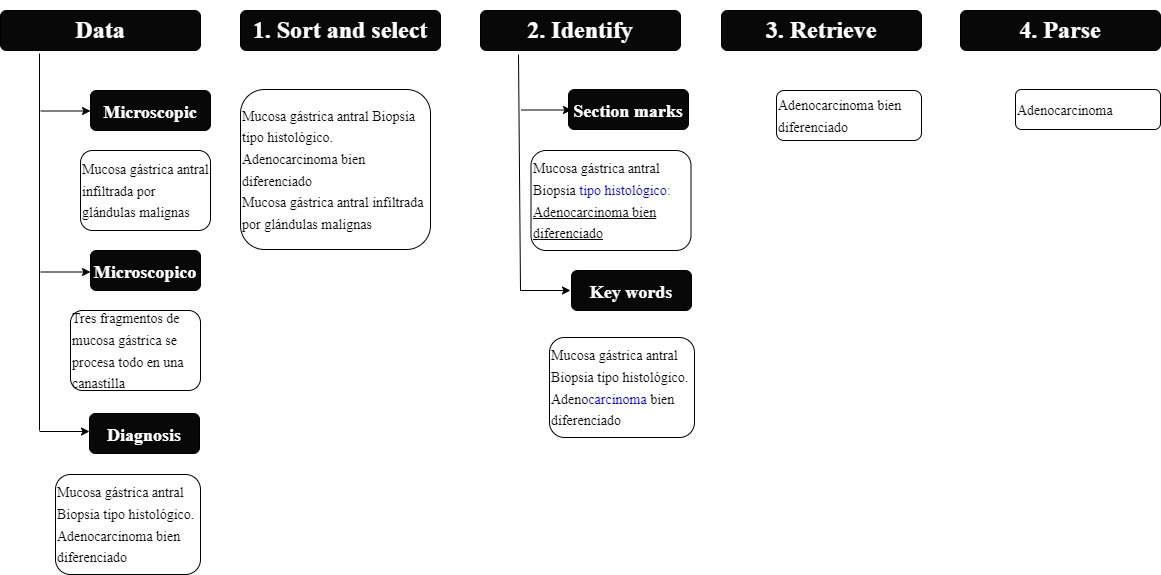
Figura 1 Algoritmo: la figura muestra el proceso aplicado para identificar y recuperar las características relevantes del reporte de patología oncológico. El algoritmo se alimenta de tres tipos de datos: microscópico, macroscópico y datos de diagnóstico. Luego, sigue un proceso de cuatro pasos en el que los datos se sortean (paso 1), luego se identifican las características en el texto (paso 2) para finalmente ser traídos (paso 3) y analizados o “monetizados” en partes gramaticales (paso 4).
1. Elección de las secciones de patología y su orden para cada búsqueda de la descripción.
2. Identificar el marcador que introdujo el valor del descriptor (en caso de ser explícito). Por ejemplo, al tamaño tumoral usualmente lo precedía la frase “Tamaño del tumor”.
3. Identificar palabras clave directamente relacionadas al descriptor en caso de que el valor estuviera tácitamente mencionado en el texto.
4. Extracción de texto relevante.
5. Análisis del valor de dicho texto.
Los siguientes párrafos describen los algoritmos para cada tipo de descriptor en mayor detalle.
Descriptores principales
Topografía y morfología
Para cada variable se armó un diccionario basado en la sección correspondiente de la Clasificación Internacional de Enfermedades para Oncología (CIE-O) 17. Estos diccionarios identificaron las palabras clave en todas las categorías de topografía y morfología. Se buscaron esas palabras clave (e.j. “carcinoma”) primero en la sección de diagnóstico del texto de reporte de patología, continuando con otras secciones. Una vez se encontraba el igual, se hacía una búsqueda secundaria de modificadores cerca a la palabra, relevantes para dicha palabra clave (e.j. “ductal”, “papilar”, etc.).
Descriptores complementarios
Este grupo de descriptores ofrecía información complementaria a la búsqueda realizada y los resultados encontrados. Todos fueron calculados posterior a que se determinaran la topografía y morfología. Cada descriptor tenía unos cuantos posibles valores, dependiendo de si el cáncer se había establecido como un tumor sólido o malignidad hematológica (cuya distinción se puede hacer en base a la topografía y morfología).
La lateralidad se implementó como computación tomográfica lateral, primero verificando si el órgano era par y después su lado con respecto a los modificadores encontrados. El comportamiento se encontró en la sección de diagnóstico, usualmente cerca a la morfología, en algunos casos implícito en esta. Dada la naturaleza de los datos, el valor predeterminado era malignidad si no estaba descrito explícitamente.
El grado se determinaba de tres posibles fuentes: 1. Una palabra clave para la diferenciación escrita explícitamente o cercano a la declaración de morfología, por ejemplo: bien diferenciado. 2. Un número de grado global o un valor numérico para un grupo especificado de topografías. Por ejemplo, la puntuación de Nottingham en cáncer de mama. 3. Para malignidades hematológicas, el tipo de linfocito implicado era explícito o se determinaba por un marcador biológico.
El método de evaluación y el procedimiento diagnóstico dependían sustancialmente de la distinción entre sólido y hematológico. El tipo de evaluación complementa esta información, y la búsqueda de palabras clave entre descripciones microscópicas o macroscópicas.
La evaluación de tumor residual y márgenes quirúrgicas sólo procedía cuando se realizaba un procedimiento quirúrgico y se especificaba como micro o macro dependiendo del tamaño tumoral residual. La presencia o ausencia de invasión linfovascular usualmente era explícito en la evaluación de la descripción microscópica.
Descriptores relacionados a metástasis
Seis descriptores estudian la propagación del cáncer de acuerdo con los órganos comprometidos. Estos fueron calculados simultáneamente siguiendo un procedimiento de dos pasos: primero, identificar cada metástasis mencionada en el reporte y extraer de textos aledaños. Luego, se buscaba una mención por cada órgano especificado en los textos; si no se encontraba un órgano pero se mencionan metástasis en una forma NO negativa, se clasificaban como “otras metástasis”.
Se tenían en cuenta dos condiciones especiales en este algoritmo: primero, la exclusión de cáncer en el órgano primario como un posible sitio metastásico, y segundo, la diferenciación entre nódulos linfáticos regionales y distantes.
Descriptores especiales
Estos se determinaron basándose en la aplicabilidad de reglas del descriptor y algo de manipulación de los números reportados. Finalmente, se extraía la clasificación TNM con una búsqueda global basada en expresiones regulares, considerando repetición y declaraciones de código.
Por ejemplo, el código TNM puede estar distribuido en un párrafo primero indicando el valor T y unas frases después declarar el valor de N y M.
El tamaño tumoral sólo se buscaba cuando se hacía resección. Para extraerlo, se inspeccionaba el contexto de cada número que parecía una medida (e.j. 1.2 cm) para establecer si se mencionaba el tumor. El número de nódulos linfáticos evaluados y nódulos positivos se calculaba desde un contexto de inspección de los números presentes en el diagnóstico o en la descripción microscópica de la patología.
Evaluación del algoritmo
Durante el desarrollo del algoritmo, un grupo de expertos en nuestra institución seleccionaron un subgrupo de reportes de patología y ejecutaron una extracción manual de los descriptores de dichos reportes. Este equipo humano incluía un médico general, un patólogo y un hemato-oncólogo. Los reportes para extracción manual fueron cuidadosamente elegidos para asegurar la inclusión de una gran gama de reportes de patología. Se prestó atención especial a incluir representantes de cada base de datos, la mayoría de cánceres y estadios comunes y cada tipo de procedimiento.
Para evaluar y mejorar el algoritmo, se comparó la extracción manual y algorítmica en tres ciclos progresivos (primero 20 reportes, luego 42 y por último 140). Después de cada ciclo, se identificaban posibles errores en el algoritmo y se hacían e implementaban muchas sugerencias para su mejora.
La métrica usada para medir el desempeño del algoritmo dependía del tipo de valores que tomara cada descriptor:
Los valores se consideraban texto libre para el descriptor primario, y se calculaba un puntaje de coincidencia parcial. Este puntaje se basa en la distancia de Levenshtein entre el texto extraído por el algoritmo y el equipo humano; esta distancia mide el número de ediciones (adiciones, sustracciones o reemplazos de caracteres) necesarios para transformar una palabra en otra. La distancia se escala para obtener un puntaje que varía entre 0 a 100. Donde un puntaje de 100 significa que la palabra en ambos textos es idéntica, y 0 significa que los textos no tienen caracteres en común.
Para los otros descriptores, se separaban los valores en un número pequeño de clases. Así, usamos cuatro métricas comunes para un problema de clasificación multiclase: la precisión general y la precisión macro promedio, capacidad de recordar (referido como recordar), y puntaje f.
La precisión general mide la fracción de reportes correctamente clasificados entre todos los reportes, donde “correctamente” significa que la extracción humana y algorítmica coinciden.
Para cada valor posible del descriptor, computamos la precisión, recordar y puntaje f en una estrategia de uno contra el resto según la siguiente fórmula:
La precisión mide qué tan bueno es el algoritmo diferenciando una clase de las demás, y el recordar mide qué tan bueno es el algoritmo capturando todas las instancias de la misma clase. Ya que ambos objetivos son complementarios, el puntaje f es un resultado de ambos.
Finalmente, el promedio aritmético de cada métrica se toma sobre todos los valores posibles de un descriptor. Esto se conoce como promedio macro.
Adicionalmente, para los descriptores especiales donde los valores no aplicables o no reportados representan una proporción significativa, se realizó un análisis categórico entre clases reportadas, no reportadas y no aplicables antes de proceder al análisis de los valores reportados.
Para realizar una validación a gran escala del algoritmo, se desarrolló una página web para el mismo (disponible en una de nuestras plataformas institucionales), de acceso abierto para todo usuario externo interesado, que desee participar voluntariamente en su evaluación y mejora (https://oncologia-web-app-dev.uc.r.appspot.com/polls/).
Resultados
Esta sección resume la comparación entre la extracción de descriptores humanos y algorítmicos para los reportes de patología elegidos para validación. Se realizó la evaluación como fue descrito en la sección previa.
Descriptores primarios
Se realizó la validación en 140 reportes patológicos. La topografía se identificó por humano y algoritmo en todos los reportes. El humano identificó la morfología en 138 reportes y el algoritmo en 137.
Un puntaje de coincidencias parciales se calculó entre los valores en los reportes donde tanto humano como algoritmo extrajeron el descriptor. La Tabla 3 resume la distribución de dicho puntaje calculado para cada descriptor. Nótese que el puntaje de procesamiento fue superior a 90,0 en tres cuartos de los casos para el texto de Morfología.
Tabla 3 Resumen estadístico para el puntaje de coincidencias parciales entre la extracción humana y algorítmica de descriptores en texto libre. La tabla muestra el número de reportes validados y la media, desviación estándar y cuartiles del puntaje.
| Descriptor | Conteo | Media | DE | Mín | 25% | 50% | 75% | Máx |
| Topografía | 140 | 68.27 | 25.22 | 0.0 | 45.0 | 77.0 | 90.0 | 100 |
| Morfología | 137 | 89.45 | 10.64 | 31.0 | 90.0 | 90.0 | 95.0 | 100 |
Descriptores complementarios
Se calculó la precisión, recordar y puntaje f para cada valor posible del descriptor y luego fueron promediados. La precisión global corresponde a la fracción de reportes donde la extracción manual y algorítmica del descriptor concuerda. La Tabla 4 resume la precisión, recordar y exactitud para cada descriptor categórico en el subgrupo de validación de 42 reportes.
Tabla 4 Medidas del desempeño de extracción algorítmica cuando se aplica a características categóricas. La precisión mide el número de reportes correctamente clasificados entre el número total de reportes asignados a la clase por el algoritmo. “Recordar”, mide el número de reportes correctamente clasificados entre el número de reportes verdaderos (e.j. clasificados por humanos) en esa misma clase. El puntaje f es la media armoniosa de precisión y recordar. Para características multiclase, precisión, recordar y el puntaje f se promediaban sobre las clases (promedio macro). La exactitud global es el número de reportes correctamente clasificados entre el número total de reportes evaluados.
| Descriptor | Precisión macro (%) | Recordar macro (%) | Puntaje-f macro (%) | Exactitud global. % (n/N) | |
|---|---|---|---|---|---|
| Descriptores complementarios | Lateralidad | 66.2 | 50.0 | 52.9 | 64.3 (27/42) |
| Comportamiento | 57.1 | 92.7 | 58.6 | 85.7 (36/42) | |
| Grado | 70.3 | 64.8 | 79.6 | 76.2 (32/42) | |
| Método de Evaluación de Tumores Sólidos | 78.6 | 94.8 | 78.4 | 85.7 (36/42) | |
| Método de Evaluación de Tumores Hematológicos | 100 | 100 | 100 | 100 (42/42) | |
| Procedimiento diagnóstico | 95.0 | 83.7 | 87.2 | 90.5 (38/42) | |
| Invasión linfovascular | 82.5 | 91.2 | 83.9 | 85.7 (36/42) | |
| Márgenes quirúrgicas | 94.4 | 77.2 | 82.8 | 90.5 (38/42) | |
| Metástasis pulmonares | 100 | 100 | 100 | 100 (42/42) | |
| Metástasis óseas | 92.9 | 50.0 | 96.3 | 92.9 (39/42) | |
| Metástasis hepáticas | 75.0 | 66.7 | 83.3 | 97.6 (41/42) | |
| Metástasis cerebrales | 50.0 | 50.0 | 100 | 97.6 (41/42) | |
| Metástasis a nódulos linfáticos distales | 50.0 | 97.6 | 98.8 | 97.6 (41/42) | |
| Otras metástasis | 98.8 | 75.0 | 82.7 | 97.6 (41/42) | |
| Descriptores especiales | Nódulos regionales examinados | 92.3 | 100 | 96.0 | 41.7 (5/12) |
| Nódulos regionales positivos | 92.3 | 100 | 96.0 | 58.3 (7/12) | |
| Tamaño tumoral | 85.7 | 75.0 | 80.0 | 50.0 (6/12) | |
| Estadificación TNM | 100 | 75.0 | 85.7 | 100 (3/3) |
Descriptores especiales
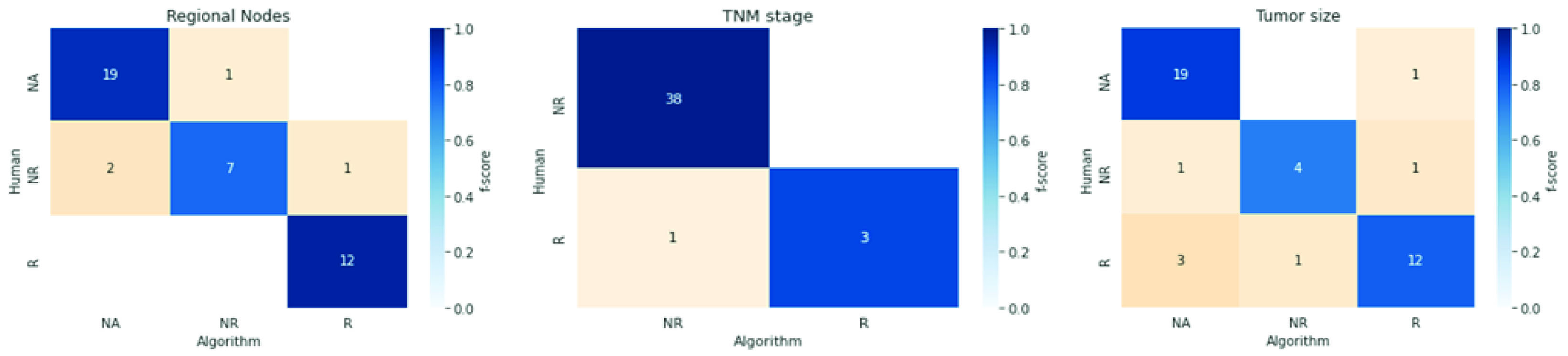
Para estos descriptores, se ejecutó el análisis en dos pasos. Primero, se midió el desempeño del algoritmo para diferenciar valores reportados de los no reportados o no aplicables. A continuación, medimos la precisión de los valores reportados. La Figura 2 muestra matrices de confusión por puntaje f para cada descriptor.
Discusión
Aquí presentamos un esfuerzo para combiner el desarrollo del Procesamiento de Lenguaje Natural con el esfuerzo humano para optimizar los resultados de extracción de información para el módulo “tumor” de nuestro registro hospitalario de cáncer.
La extracción de datos usando el abordaje de expresiones regulares puede extraer múltiples atributos del espécimen de reportes de patología en texto libre en español, con exactitud y precisión aceptables. En los casos seleccionados para validación, el puntaje promedio de coincidencias parciales fue 68.3 para topografía y 89.5 para morfología. Los descriptores complementarios mostraron precisión y tasa de verdaderos positivos entre 50% y 100% y un puntaje f entre 52.9% y 100%. Entre los casos reportados, la precisión varió entre 92.3% y 100%, la tasa de verdaderos positivos entre 75% a 100% y el puntaje f entre 80% a 96%.
Estos desarrollos podrían asistir en extraer información veraz de registros de cáncer hospitalarios en los que se impone el desafío de manejar volúmenes enormes de información. Basado en la extracción algorítmica de descriptores, ahora es factible el análisis estadístico de estos tipos de reportes patológicos.
Aunque la precisión del modelo es alta, otras métricas como exhaustividad muestran que hay oportunidad de mejora. La exhaustividad muestra que las reglas creadas mediante las expresiones regulares no fueron suficientes para capturar un número significativo de características tumorales. Esto puede estar causado por la existencia de patrones de lenguaje subyacentes que los doctores no tienen presente porque (1) son infrecuentes o (2) pueden ser muy complejos para identificar. Estos dos obstáculos parecen insuperables usando expresiones regulares y coincidencias parciales de cadenas ya que todos los casos potenciales necesitarían ser incluidos, muchos de los cuales son desconocidos para los patólogos. Esta es la limitación más crítica de las expresiones regulares. Sin embargo, otras metodologías del Procesamiento de Lenguaje Natural podrían demostrar ser más exactas en estos casos; esta herramienta es un acercamiento que podría incrementar significativamente la exhaustividad de esta aplicación por medio de modelos de aprendizaje de máquinas.
Los modelos de aprendizaje de máquinas en datos de texto pueden identificar patrones subyacentes que los humanos no, superando la limitación de brechas en el conocimiento. Las metodologías de deep learning como redes neuronales recurrentes, word2vec y transformers pueden capturar el significado de palabras/términos en su contexto; entendiendo el contexto como el lenguaje a su alrededor. Con suficientes datos, estos modelos pueden aprovechar información en el texto, como longevidad, ubicación en el texto y orden de ocurrencia, para deducir correlaciones complejas y extraer las características de manera precisa. Creemos que la investigación en un futuro estará encaminada a esto, donde los modelos de aprendizaje serán entrenados para sobreponerse a las limitaciones generadas por patrones lingüísticos complejos o infrecuentes.
Aplicar el algoritmo en reportes de patología con textos estructurados o no estructurados podría ayudar a las instituciones a implementar los registros de cáncer hospitalarios. Los datos extraídos clasifican un tumor (CIE-O-M) según ubicación, tamaño, compromiso linfovascular, compromiso de nódulos linfáticos, metástasis y estadificar con el TNM.
Las limitaciones del algoritmo incluyen que requiere supervisión humana para la extracción de información. El algoritmo mejora cuando se registra información esencial de la malignidad en reportes patológicos con plantillas de protocolos de cáncer. Se necesitan estudios para demostrar el alcance del algoritmo en un corpus extenso de información.
References
1. Ruiz A, Facio Á. Hospital-based cancer registry: A tool for patient care, management and quality. A focus on its use for quality assessment. Rev Oncol. 2004; 6(2): 104-13. Doi: 10.1007/BF02710038 [ Links ]
2. Kreimeyer K, Foster M, Pandey A, Arya N, Halford G, Jones SF, et al. Natural language processing systems for capturing and standardizing unstructured clinical information: A systematic review. J Biomed Inform. 2017; 73: 14-29. doi: 10.1016/j.jbi.2017.07.012. [ Links ]
3. Alawad M, Gao S, Qiu JX, Yoon HJ, Blair Christian J, Penberthy L, et al. Automatic extraction of cancer registry reportable information from free-text pathology reports using multitask convolutional neural networks. J Am Med Informatics Assoc. 2020; 27(1): 89-98. Doi: 10.1093/jamia/ocz153 [ Links ]
4. Nadkarni PM, Ohno-Machado L, Chapman WW. Natural language processing: an introduction. J Am Med Inform Assoc. 2011; 18(5): 544-51. doi: 10.1136/amiajnl-2011-000464 [ Links ]
5. Meystre S, Savova G, Kipper-Schuler KC, Hurdle JF. Extracting information from textual documents in the electronic health record: a review of recent research. Yearb Med Inf. 2007; 128-44. [ Links ]
6. Velupillai S, Suominen H, Liakata M, Roberts A, Shah AD, Morley K, et al. Using clinical Natural Language Processing for health outcomes research: Overview and actionable suggestions for future advances. J Biomed Inform. 2018; 88: 11-9. Doi: 10.1016/j.jbi.2018.10.005 [ Links ]
7. Burger G, Abu-Hanna A, de Keizer N, Cornet R. Natural language processing in pathology: A scoping review. J Clin Pathol. 2016; 69: jclinpath-2016. doi: 10.1136/jclinpath-2016-203872. [ Links ]
8. Hammami L, Paglialonga A, Pruneri G, Torresani M, Sant M, Bono C, et al. Automated classification of cancer morphology from Italian pathology reports using Natural Language Processing techniques: A rule-based approach. J Biomed Inform. 2021; 116: 103712. Doi: 10.1016/j.jbi.2021.103712 [ Links ]
9. Aalabdulsalam A, Garvin J, Redd A, Carter M, Sweeny C, Meystre S. Automated Extraction and Classification of Cancer Stage Mentions fromUnstructured Text Fields in a Central Cancer Registry. AMIA Jt Summits Transl Sci Proc. 2018; 2017: 16-25. [ Links ]
10. Koza W, Filippo D, Cotik V, Stricker V, Muñoz M, Godoy N, et al. Automatic Detection of Negated Findings in Radiological Reports for Spanish Language: Methodology Based on Lexicon-Grammatical Information Processing. J Digit Imaging. 2019; 32(1):19-29. doi: 10.1007/s10278-018-0113-8. [ Links ]
11. Villena F, Dunstan J. Obtención automática de palabras clave en textos clínicos: una aplicación de procesamiento del lenguaje natural a datos masivos de sospecha diagnóstica en Chile. Rev Med Chil. 2019; 147(10): 1229-38. Doi: 10.4067/s0034-98872019001001229 [ Links ]
12. Solarte-Pabón O, Blazquez-Herranz A, Torrente M, Rodríguez-Gonzalez A, Provencio M, Menasalvas E. Extracting Cancer treatments from clinical text written in spanish: a deep learning approach. IEEE 8th Int Conf Data Sci Adv Anal DSAA 2021; 2021 [ Links ]
13. Solarte-Pabón O, Torrente M, Provencio M, Rodríguez-Gonzalez A, Menasalvas E. Integrating speculation detection and deep learning to extract lung cancer diagnosis from clinical notes. Appl Sci. 2021; 11(2): 865. doi: 10.3390/app11020865 [ Links ]
14. Parra-Lara LG, Mendoza-Urbano D, Zambrano Á, Valencia-Orozco A, Bravo-Ocaña JC, Bravo-Ocaña LE, et al. Methods and Implementation of a Hospital-Based Cancer Registry in a Major City in a Low-to Middle-Income Country: The Case of Cali, Colombia. Cancer Causes Control. 2022; 33(3): 381-392. doi: 10.1007/s10552-021-01532-z.. [ Links ]
15. American College of Surgeons. Facility oncology registry data standards (FORDS): Revised for 2016; 2017. Available from: https://www.facs.org/quality-programs/cancer-programs/national-cancer-database/ncdb-call-for-data/fordsmanual/ [ Links ]
16. Instituto Nacional de Salud. Fichas y Protocolos; 2022. Available from: https://www.ins.gov.co/buscador-eventos/Paginas/Fichas-y-Protocolos.aspx [ Links ]
17. Fritz A, Percy C, Jack A, Shan K. Clasificación internacional de enfermedades para oncología (CIE-O). Rev Esp Salud Publica. 2003;77(5):659-659. [ Links ]
Recibido: 10 de Junio de 2022; Revisado: 02 de Agosto de 2022; Aprobado: 20 de Septiembre de 2022











 texto em
texto em