Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biosalud
Print version ISSN 1657-9550
Biosalud vol.11 no.2 Manizales July/Dec. 2012
EVALUATION OF SENSITIVITY AN SPECIFICITY OF MOLECULAR DIAGNOSIS FOR Staphylococcus aureus IN MILK OF COWS AFFECTED BY MASTITIS
Juan Restrepo1, 2
Luisa Ortiz1
Ximena Cardona2
Martha Olivera3
1 Escuela de Ciencias Agrarias, Grupo Vericel, Universidad de Antioquía juanrb@colanta.com.co, cel.310 407 11 40. Teléfono (4) 445 3000. Dirección Carrera 64c # 72 157 luifernanda@gmail.com cel 317 817 95 02
2 Colanta Ltda. Departamento de Asistencia Técnica ximenacl@colanta.com.co cel 321 799 40 41
3 Biogénesis, Universidad de Antioquia. syngamia@gmail.com cel 317 538 39 21.
Entre los agentes contagiosos que inducen mastitis severas en la vaca, se encuentra el Staphylococcus aureus (S. aureus), de difícil cura bacteriológica y alta resistencia antimicrobiana. Debido a que el cultivo microbiológico de las muestras clínicas, solo ofrece resultado en un 50% de los casos (1) el diagnóstico por PCR es una alternativa. El objetivo de este estudio fue probar si los cebadores descritos por Cremonesi et al. (2) para el diagnóstico de S. aureus, como de buena sensibilidad y especificidad, sirven para ser usados en muestras clínicas. Los resultados demostraron que las siguientes secuencias de nucleótidos como cebadores: F 5' AGC TGT GGA TTG TCC TTT GG 3' y R 5' TCG CTC GCT CAC CTT AGA A 3', para obtener un amplificado de 499 pb no sirven en muestras clínicas por su baja especificidad (62,95%). Se requiere buscar nuevos cebadores que amplifiquen regiones del S. aureus que no se compartan con otras bacterias, en especial aquellas que producen mastitis en vacas productoras de leche.
Palabras clave: Staphylococcus aureus, PCR, mastitis.
Staphylococcus aureus (S. aureus) is one of the infectious agents that induce severe mastitis in cows with a difficult bacteriological cure and high antimicrobial resistance. Because the microbiological culture of clinical samples only shows results in 50% of the cases (Koskinen et al., 2009), diagnostic through PCR is an alternative. The aim of this study was to prove if the primers described by Cremonesi et al. (2006) for the S. aureus diagnosis, with good sensitivity and specificity, could be used in clinical samples too. The results showed that the following nucleotide sequences can be used as primers: F 5' AGC TGT GGA TTG TCC TTT GG 3' and R 5' TCG CTC GCT CAC CTT AGA A 3' in order to obtain a 499 pb enlargemenr are not useful in clinical samples due their low specificity (62.95%). It is required to search new primers to amplify S. aureus regions not shared with other bacteria, especially those cauding mastitis in dairy cows.
Key words: Staphylococcus aureus, PCR, mastitis.
La mastitis bovina, definida como la inflamación de la glándula mamaria, es la enfermedad más costosa y común del ganado lechero; y se presenta en forma endémica en gran parte del mundo (3, 4). Las mastitis contagiosas son producidas por los patógenos Streptococcus agalactiae (S. agalactiae) y Staphylococcus aureus (S. aureus) (5, 6). Cuando el patógeno es S. agalactiae son mastitis controlables que se pueden erradicar del hato (7), mientras que las producidas por S. aureus presentan baja cura bacteriológica por su resistencia a los antibacterianos (8, 9) y por la capacidad que tiene de producir biopelícula (10).
La patogenicidad del S. aureus se debe a su adhesión intercelular (10), su capacidad de colonizar las células epiteliales y macrófagos de la glándula mamaria (10, 11), así como a su habilidad de producir fibrosis y micro abscesos que dificultan el acceso de agentes antimicrobianos (12). Produce mastitis crónicas recurrentes y cuando son agudas, generalmente se producen recidivas después del tratamiento (13).
El diagnóstico de S. aureus es importante para implementar la estrategia de manejo del hato frente a la enfermedad previniendo la diseminación de este patógeno en el resto del hato, revisando las prácticas de ordeño, mejorando el uso de preselladores y selladores, propiciando que los operarios usen guantes de látex (7), organizando el orden de ordeño para que las vacas con un diagnóstico positivo a S. aureus, entren de últimas, para posteriormente ser eliminadas lo más pronto del hato (3).
La mastitis en la vaca es una de las enfermedades más costosas en la industria lechera. En EE.UU., se estima que el costo por año a los productores de leche oscila entre 1,2 a 1,7 billones de dólares (1), que es aproximadamente el 6% del valor total de la producción del país (14). Se estiman las pérdidas anuales por vaca en 184 dólares; este valor incluye la disminución en la producción de leche, la leche que se descarta, el gasto en medicamentos, los honorarios del médico veterinario y animales con un descarte prematuro (1). El volumen de leche dejado de producir de acuerdo a la severidad de la mastitis subclínica oscila entre el 2,8 y el 45% de la producción por cuarto/día (3). Las pérdidas económicas en Colombia se estiman en diferentes estudios a partir de los hallazgos en mastitis subclínica. En diez fincas de la Sabana de Bogotá se calculó que lo dejado de producir en leche costaba en pesos colombianos $41.580 diarios (15), en Boyacá se calcularon las pérdidas en $1'130.226 al día en 34 empresas ganaderas (16), y en Antioquia se reportaron pérdidas estimadas de $4'608.000 mensuales en siete hatos (17).
Prevalencia de la enfermedad
La prevalencia de la enfermedad por el patógeno S. aureus ha sido objeto de estudio en diferentes trabajos de investigación. En Colombia se tienen reportes de Corpoica en el Valle del Cauca, donde de 235 muestras recolectadas se aisló Staphylococcus spp. en el 37,45%, Streptococcus sp. en 17,87% y E. coli en 8,94% (18). Los autores sugieren que la mayor prevalencia del género Staphylococcus spp. puede ser atribuida a la persistencia del agente en la glándula mamaria (18, 19). Ramírez et al. en investigaciones realizadas en San Pedro de los Milagros (Antioquia), encontraron una prevalencia de S. aureus del 13%, de un total de 1.679 cultivos (19). Calderón y Rodríguez realizaron la prueba de California Mastitis Test (CMT) en 2.854 vacas en 40 hatos de la sabana cundiboyacense, tomaron muestras de leche de las vacas CMT positivo y de allí aislaron S. aureus en el 29,09% de los casos (4). Calvinho y Tirante revisaron la prevalencia de S. aureus en los últimos 25 años, en diferentes regiones de Argentina y encontraron rangos entre el 17% y el 54% en las diferentes cuencas lecheras (20). En la región del sur de Chile reportan que la prevalencia de mastitis clínica causada por S. aureus era del 27% (21). Ferraro, Scaramelli y Troya, en trece estados de Venezuela realizaron CMT en 60 fincas lecheras, a 24.599 cuartos, cultivaron los positivos y encontraron S. aureus en el 28,4% (22). Sabour et al. en Ontario (Canadá) hallaron una prevalencia entre el 7 y el 44% (23), y Sumathi, Veeregowda y Amitha en Bangalore (Canadá) encontraron el 24% de S. aureus (24).
Estudios de diagnóstico molecular del S. aureus
Según la Organización Internacional de Epizootias –OIE– (2008), la experiencia de las últimas dos décadas indica que la técnica de reacción de la polimerasa en cadena (PCR) con el tiempo remplazará a muchos de los métodos clásicos de detección directa de agentes infecciosos, especialmente sustituyendo el aislamiento de virus o bacterias así como en la detección de agentes que son difíciles o imposibles de cultivar (25). Según varios autores, la sensibilidad y especificidad de la PCR es generalmente mayor que el aislamiento (2, 26). La aplicación de esta técnica, con un formato sencillo y rápido, es adecuada a la rutina diaria de un laboratorio de microbiología y contribuye a realizar un diagnóstico precoz. Si se detecta más temprano el microorganismo podría prevenirse la aparición de brotes y permitiría implementar controles previos a la ocurrencia de la enfermedad. Sin embargo, la especificidad y la sensibilidad de los cebadores para el diagnóstico del patógeno se deben validar (1). Hasta la fecha se han publicado diferentes tipos de cebadores para la amplificación de una región del genoma del S. aureus, los cuales se ilustran en la Tabla 1.
El diagnóstico convencional de la leche proveniente de glándulas mamarias afectadas con mastitis para las infecciones por S. aureus, de acuerdo con las directrices del National Mastitis Council Wisconsin (1998), es la siembra en agar sangre y tipificación con pruebas bioquímicas: oxidasa negativa, catalasa positiva, coagulasa positiva, no descarboxila ornitina y con lectura tres a cinco días después de la siembra (14). En la búsqueda de realizar diagnósticos más rápidos con alta sensibilidad y especificidad, Cremonesi et al. así como otros investigadores (2, 26-31), han propuesto el uso de la reacción de la polimerasa en cadena (PCR) para identificación de la bacteria en cuestión de horas (2, 24, 30). Esta rutina aún no se ha implementado debido a que lo que reportan estos autores es el uso de cebadores aplicados a cultivos puros y no en muestras clínicas. El objetivo de la presente investigación fue evaluar la sensibilidad y especificidad en el diagnóstico molecular del Staphylococcus aureus en leche de vacas afectadas por mastitis clínica, utilizando los cebadores reportados por Cremonesi et al. (2).
Análisis in silico
Se realizó una simulación de PCR empleando el programa in silico –simulation of molecular biology experiment– (http://insilico.ehu.es/) (32), para probar diferentes cebadores reportados en la literatura (Tabla 1). Con el programa Blast (http://blast.ncbi.nlm.nih.gov/Blast.cgi) se determinó la especificidad de los mismos. Se seleccionaron los cebadores descritos por Cremonesi et al. (2) porque al realizar el in silico amplificaron todas las cepas de S. aureus reportadas.
Estandarización de la prueba
Cepas controles
Se usaron además de una cepa de S. aureus ATCC 29213, diez cepas de S. aureus aisladas de muestras de leche, cuatro cepas de referencia ATCC Streptococcus equisimilis ATCC 35666, Enterococcus spp. ATCC 151299, Klebsella pneumoniae ATCC 70063, Streptococcus uberis ATCC 9927C, y seis cepas de patógenos cultivados e identificados como causantes de mastitis clínica: Streptococcus agalactiae, Streptococcus pyogenes, Streptococcus dysgalactiae, Pseudomonas spp., Escherichia coli, Streptococcus intermedius. A todas estas cepas se les realizó extracción de DNA.
Extracción de DNAPara la extracción del DNA de las muestras de leche, se empleó el kit de DNeasy® Tissue de Qiagen, empleando la metodología de columnas a partir de tejidos celulares, siguiendo el instructivo descrito por el fabricante.
Sensibilidad y especificidad
Para determinar la sensibilidad y la especificidad de los cebadores a utilizar, se trabajó con la cepa de S. aureus ATCC 29213 con una concentración de 1,2 x 10-8 UFC según la escala de McFarland. La extracción de DNA se realizó y se obtuvo una concentración de 815,6 ng/ul medida en un Nanodrop 2000. Partiendo de esta concentración se realizaron diluciones seriadas hasta obtener una concentración de 16,31 atogramos (10-18) y a cada una de estas se le realizó una prueba de PCR con el fin de determinar el límite de detección de la prueba.
Muestras clínicas
Para calcular la sensibilidad y la especificidad de la PCR para el S. aureus en muestras de leche provenientes de vacas con mastitis clínica, se tomaron 43 muestras por duplicado. La primera, para cultivo y aislamiento, en agar sangre y tipificación con pruebas bioquímicas: oxidasa negativa, catalasa positiva, coagulasa positiva y prueba de decarboxilación de la ornitina negativa. La segunda muestra se usó para la prueba molecular. El protocolo de extracción fue descrito anteriormente.
PCR
Para la reacción de PCR se siguió el protocolo descrito por Cremonesi et al. (2), que amplifica una región específica del genoma del DNA que corresponde al gen 16S rRNA. Se utilizaron las siguientes secuencias de nucleótidos como cebadores: F 5' AGC TGT GGA TTG TCC TTT GG 3' y R 5' TCG CTC GCT CAC CTT AGA A 3' para obtener un amplificado de 499 pb. El volumen final de la PCR fue de 25 uL que contenían 200 ng de DNA extraído, buffer 1X, MgCl2 1,5 mM, 1U de Taq polimerasa Fermentas®, dNTPS 0,2 mM, cebadores 0,25 uM. El proceso se realizó en un termociclador DNA Engine PTC 200. Las condiciones para la PCR fueron 5 minutos de precalentamiento a 94ºC, seguido de 32 ciclos compuestos de un minuto de desnaturalización a 94ºC, un minuto de anillamiento de los cebadores a 56ºC, un minuto de extensión a 72ºC y una extensión final de diez minutos a 72ºC.
Electroforesis
Los productos de la PCR fueron visualizados en gel de agarosa al 2%, teñidos con 0,3 ug/ml de bromuro de etidio, corrido a 80 voltios durante 45 minutos y visualizado bajo luz ultravioleta. Se utilizó un marcador de peso molecular de 50 pb de laboratorio Molecular Bio Laboratory®.
Análisis estadístico
El diseño estadístico que usó fue un doble ciego, utilizando una tabla de contingencia de 2 x 2, comparando los resultados de la prueba de cultivo microbiológico, como prueba de oro, con los resultados de la PCR. Para el análisis de sensibilidad y especificidad se empleó el programa SPSS versión 18 (Tabla 3) (5, 33).
De las 43 muestras de leche provenientes de vacas afectadas con mastitis y cultivadas, se obtuvo crecimiento en 36 de ellas con diferentes tipos de bacterias de las cuales se obtuvo amplificación en 24 (Tabla 2). El límite de detección de la prueba cuandó se usó el DNA de cepas puras de S. aureus fue inferior a 16,31 atogramos (10-18).
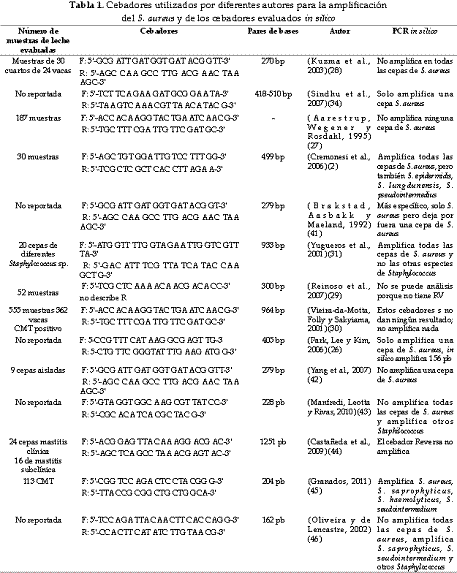
En la Figura 1 se presentan los productos del amplificado de las cepas de S. aureus que se usaron como controles y de la cepa de referencia; todas amplificaron el fragmento de 499 pb con el cebador F 5' AGC TGT GGA TTG TCC TTT GG 3' y R 5' TCG CTC GCT CAC CTT AGA A 3'.
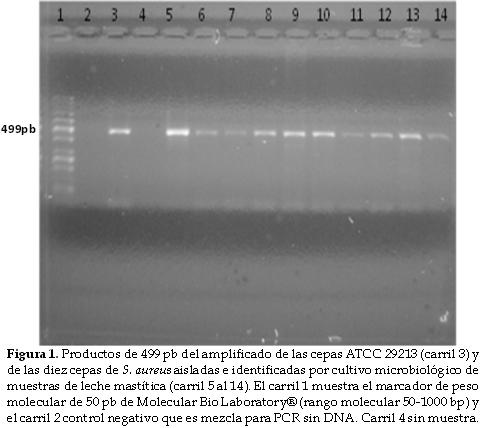
Una foto de un gel con 12 muestras de DNA extraído de las leches de casos clínicos se presenta en la Figura 2; 19 muestras no amplificaron (Tabla 3). Ninguna de las muestras que dio negativo para el S. aureus en el cultivo produjo amplificado en la PCR.
Los resultados de sensibilidad y especificidad de la prueba de PCR cuando se comparan con la prueba de oro, se calcularon con base en la Tabla 3. Para la tabulación se tomaron en cuenta los datos de aislamiento bacteriológico tanto puro, como mixto, para un total de 15 muestras con aislamiento de S. aureus.
La sensibilidad para la prueba de PCR con estos cebadores fue del 93,8% y la especificidad del 66,7%.
Cuando se probó con todas las cepas de referencia diferentes al S. aureus, si el cebador estudiado amplificaba otras bacterias, se encontró que amplifica también Enterococcus spp. ATCC 151299.
Según los resultados que se muestran en la Tabla 2, cuando en el cultivo microbiológico crece S. aureus puro, la prueba de PCR identifica el 92,3% de los casos. Sin embargo, cuando crece en un cultivo mixto se puede confundir con Streptococcus pyogenes o Streptococcus agalactiae. Además, el cebador usado también fue capaz de amplificar muchas otras bacterias (Tabla 2), entre ellas una de las contagiosas importantes como es el Streptococcus agalactiae. Cuando el Veterinario requiere un diagnóstico rápido y confiable para la toma de decisión sobre el manejo de la vaca con mastitis con un patógeno contagioso, el resultado de la prueba de PCR con este cebador no es adecuado.

Es claro que cuando Cremonesi et al. usan estos cebadores en cepas de cultivo puro tiene un 100% de certeza, como también lo obtuvimos en este trabajo con la cepa ATCC 29213, las cepas control y los aislamientos de S. aureus de campo, pero tratándose de pruebas clínicas que se compararon con la prueba de oro (aislamiento), el resultado indica que el DNA de muestras clínicas en presencia de otras cinco bacterias también se amplificó el mismo segmento: Streptococcus spp., Streptococcus agalactiae, Streptococcus pyogenes y el Staphylococcus haemoliticus.
Sin embargo, cuando se aplicó el cebador a las cepas de referencia puras de Streptococcus agalactiae y Streptococcus pyogenes, no amplificaron la secuencia esperada como se hubiese esperado según lo obtenido de algunas cepas de campo.
Según lo demuestran algunos autores los cultivos de muestras clínicas aunque tienen una alta especificidad, tienen baja sensibilidad, ya que solamente hay crecimiento bacteriano entre un 50 y 76% de los casos cultivados de mastitis (1, 4, 25, 34, 37).
Aunque Cremonesi et al., Sindhu et al., Granda et al. y Abu y Radstrom (2, 34-36), habían demostrado que la PCR es un método eficiente para la detección de microorganismos y puede ser útil en el diagnóstico partiendo de cultivos puros, podemos deducir a partir de nuestros resultados que dicho cebador es compartido con otras bacterias (1, 36, 38).
Los resultados anteriores soportan la baja especificidad encontrada, cuando se comparan con las obtenidas con otros cebadores como los descritos con Koskinen et al. (1) y por Kim et al. (5), que fueron del 99% y del 100%, respectivamente.
Es posible que la inespecificidad de la prueba, como lo reportan Ramesh et al. (39), se deba a los sebadores de unen inespecíficamente a fragmentos de DNA diferentes al del S. aureus, generando dando falsos positivos.
En este trabajo se encontró que al utilizar una concentración de 16,31 atogramos (10-18 g) de DNA bacteriano, los cebadores son capaces de amplificar las diferentes cepas, mostrando una alta sensibilidad en el límite de detección. Si los cebadores fuesen específicos para el S. aureus, el límite de detección hubiese sido muy apropiado ya que puede detectar fragmentos de DNA de al menos una bacteria. Para Abu y Radstrom (36) las concentraciones mínimas de detección (0,1 ng) fueron mayores que las encontradas en este trabajo.
En cuanto a la técnica de extracción del DNA de muestras de leche, usando el kit de DNeasy® Tissue de Qiagen, se concluye que produce DNA de buena calidad que permite amplificar la banda esperada y no produce interferencia en el proceso. Ercolini et al. (38) proponen, antes de realizar la metodología de la extracción de DNA, enriquecer la muestra de leche con una preincubación para aumentar las UFC (38). Otros métodos para extracción de DNA a partir de leche, han reportado una extracción eficiente del DNA, como los es el método de fenol-cloroformo (2, 5, 33), el de kit Chelex-100 modificado por Cremonesi et al. (2) al adicionar guanidina tiocianato para aumentar la calidad del DNA; la ebullición a la cual Khan et al. (40) le adicionaron cuatro lavados con PBS obteniendo resultados satisfactorios.
Se requiere buscar nuevos cebadores que amplifiquen regiones del S. aureus que no se compartan con otras bacterias, en especial aquellas que producen mastitis en vacas productoras de leche.
1. Koskinen M, Holopainen J, Pyörälä S, Bredbacka P, Pitkälä A, Barkema H, et al. Analytical specificity and sensitivity of a real-time polymerase chain reaction assay for identification of bovine mastitis pathogens. Journal of Dairy Science 2009; 92(3):952-959. [ Links ]
2. Cremonesi P, Castiglioni B, Malferrari G, Biunno I, Vimercati C, Moroni P, et al. Technical Note: Improved Method for Rapid DNA Extraction of Mastitis Pathogens Directly from Milk. Journal of Dairy Science 2006; 89(1):163-169. [ Links ]
3. Philpot WN. Relación entre el manejo del hato y la mastitis. http://colanta.comunidadpmb.org/opac_css/index.php?lvl=author_see&id=8039. 2001, 3, 2003. [ Links ]
4. Calderón A, Rodríguez VC. Prevalencia de mastitis bovina y su etiología infecciosa en sistemas especializados en producción de leche en el altiplano cundiboyacence (Colombia). Revista Colombiana de Ciencias Pecuarias 2008; 21(4):582-589. [ Links ]
5. Kim CH, Khan M, Morin D, Hurley W, Tripathy D, Kehrli Jr M, et al. Optimization of the PCR for detection of Staphylococcus aureus nuc gene in bovine milk. Journal of Dairy Science 2001; 84:74-83. [ Links ]
6. Philpot WN, Nickerson SC. Ganando la lucha contra la mastitis. Naperville, USA; 2001. [ Links ]
7. Keefe TM, Ceballos A, Jaramillo M, Londoño M, Chaffer M, Montoya M. Prevalencia del Streptococcus agalactiae en tanques de enfiamiento de la Cooperativa COLANTA. En: VII Seminario Internacional Competitividad Carne y Leche. COLANTA ed. Medellín Octubre 21, 22, 2010. Vol. 1, p. 53. [ Links ]
8. Russi NB, Bantar C, Calvinho LF. Antimicrobial susceptibility of Staphylococcus aureus causing bovine mastitis in Argentine dairy herds. Revista Argentina de Microbiología 2008; 40(2):116-119. [ Links ]
9. San Martín B, Kruze J, Morales M, Agüero H, León B, Esppinoza S, et al. Resistencia bacteriana en cepas patógenas aisladas de mastitis en vacas lecheras de la V Región, Región Metropolitana y X Región, Chile. Archivos de Medicina Veterinaria 2002; 34:221-234. [ Links ]
10. Cucarella C, Solano C, Valle J, Amorena B, Lasa I, Penadés JR. Bap, a Staphylococcus aureus surface protein involved in biofilm formation. Journal of Bacteriology 2001; 183(9):2888-2896. [ Links ]
11. Melchior M, Vaarkamp H, Fink-Gremmels J. Biofilms: a role in recurrent mastitis infections? The Veterinary Journal 2006; 171(3):398-407. [ Links ]
12. Loeza-Angeles H., López-Meza JE, Ochoa A. Efecto del medio condicionado de células endoteliales que expresan el péptido antimicrobiano tionina Thi2.1 de Arabidopsis thaliana sobre aislamientos de Staphylococcus aureus. Revista Mexicana de Ciencias Farmacéuticas 2010; 41(2):36-41. [ Links ]
13. Sharma N, Pandey V, Sudhan, N. Comparison of some indirect screening tests for detection of subclinical mastitis in dairy cows. Bulgarian Journal of Veterinary Medicine 2010; 13(2):98-103. [ Links ]
14. National-Mastitis-Council. Current Concepts of Bovine Mastitis In: National Mastitis Council, Madison, WI 1998; Vol. 4a ed. [ Links ]
15. Rodríguez G. Comportamiento de la Mastitis Bovina y su impacto económico en algunos hatos de la Sabana de Bogotá, Colombia. Revista de Medicina Veterinaria 2006; 12:35-55. [ Links ]
16. Pinzón A, Moreno FC, Rodríguez, G. Efectos de la mastitis subclínica en algunos hatos de la cuenca lechera del Alto Chicamocha (departamento de Boyacá). Revista de Medicina Veterinaria 2009; 17:23-36. [ Links ]
17. Trujillo CM, Gallego AF, Ramírez N, Palacio Baena L.G. Prevalence of mastitis in dairy herds in Eastern Antioquia. Revista Colombiana de Ciencias Pecuarias 2011; 24:11-18. [ Links ]
18. Corpoica. Prevalencia de los principales microorganismos causantes de mastitis en vacas lecheras del Centro del Valle del Cauca en los años 2003 a 2006. 2007. Disponible en: http://www.monografias.com/trabajos59/microorganismos-mastitis-vacas-lecheras/microorganismos-mastitis-vacas-lecheras3.shtml [ Links ]
19. Ramírez N, Gaviria G, Arroyave O, Sierra B, Benjumea J. Prevalencia de mastitis en vacas lecheras lactantes en el municipio de San Pedro de los Milagros, Antioquia. Revista Colombiana de Ciencias Pecuarias 2009; 14:76-87. [ Links ]
20. Calvinho LF, Tirante L. Prevalencia de microorganismos patógenos de mastitis bovina y evolución del estado de salud de la glándula mamaria en Argentina en los últimos 25 años. Rev. FAVE Sección Cs. Vet. 2005; 4:29-40. [ Links ]
21. Shim EH, Shanks RD, Morin DE. Milk loss and treatment costs associated with two treatment protocols for clinical mastitis in dairy cows. J Dairy Sci 2004; 87(8):2702-8. [ Links ]
22. Ferraro L, Scaramelli A, Troya H. Prevalencia de la mastitis subclínica bovina en Venezuela y evaluación de la prueba de mastitis de California (CMT) como prueba diagnóstica. Revista Científica FCV-LUZ 1999; IX(2):81-90. [ Links ]
23. Sabour P, Gill J, Lepp D, Pacan J, Ahmed R, Dingwell R, Leslie K. Molecular typing and distribution of Staphylococcus aureus isolates in Eastern Canadian dairy herds. Journal of Clinical Microbiology 2004; 42:3449-3455. [ Links ]
24. Sumathi B, Veeregowda B, Amitha RG. Prevalence and antibiogram profile of bacterial isolates from clinical bovine mastitis. Veterinary World 2008; 1(8):237-238. [ Links ]
25. OIE. Manual of Diagnostic Tests and Vaccines for Terrestrial Animals : Validation and quality control of Polymerase Chain Reaction methods used for the diagnosis of infectious diseases. OIE: Ginebra, Suiza. 2008 vol 1., chapter 1.1.5. 46- 55 [ Links ]
26. Park YS, Lee SR, Kim YG. Detection of Escherichia coli O157:H7, Salmonella spp., Staphylococcus aureus and Listeria monocytogenes in kimchi by multiplex polymerase chain reaction (mPCR). Journal Microbiol 2006; 44(1):92-97. [ Links ]
27. Aarestrup FM, Wegener H, Rosdahl V. Evaluation of phenotypic and genotypic methods for epidemiological typing of Staphylococcus aureus isolates from bovine mastitis in Denmark. Veterinary Microbiology 1995; 45(2-3):139-50. [ Links ]
28. Kuzma K, Malinowski E, Lassa H, Klossowska A. Detection of genes for enterotoxins and toxic shock syndrome toxin-1 in Staphylococcus aureus isolated from bovine mastitis. Bull. Vet. Inst. Pulawy 2003; 47:419-426. [ Links ]
29. Reinoso E, Betterra S, Odierno L, Bogni C. rep-PCR of Staphylococcus aureus strains isolated from bovine mastitis in Argentina. Vet. Res. Anim. Sci. 2007; 40:115-121. [ Links ]
30. Vieira-da-Motta O, Folly MM, Sakyiama CCH. Detection of different Staphylococcus aureus strains in bovine milk from subclinical mastitis using PCR and routine techniques. Brazilian Journal of Microbiology 2001; 32(1):27-31. [ Links ]
31. Yugueros J, Temprano A, Sánchez M, Luengo JM, Naharro G. Identification of Staphylococcus spp. by PCR-Restriction fragment length polymorphism of gap gene. Journal of Clinical Microbiology 2001; 39(10):3693-3695. [ Links ]
32. San Milln R, Garaizar J, Bikandi J. In silico simulation of fingerprinting techniques based on double endonuclease digestion of genomic DNA. In silico biology 2005; 5(3):341-6. [ Links ]
33. Poutou R, Burbano M, Sierra S, Torres K, Carrascal A, Mercado, M. Estandarización de la extracción de ADN y validación de la PCR múltiple para detectar Listeria monocytogenes en queso, leche, carne de res y pollo. Pontificia Universidad Javeriana: Revista de la Facultad de Ciencias 2005; 10(2):61-78. [ Links ]
34. Sindhu N, Sharma A, Kumar S, Jain V. Polymerase chain reaction assay for detection of Staphylococcus aureus in buffalo milk. Italian Journal of Animal Science 2007; 6(2):862-864. [ Links ]
35. Gandra Á, Silva A, Macedo R, Ribeiro M, Mata M, Silva W. Differentiation between Staphylococcus aureus, S. intermedius and S. hyicus using phenotypical tests and PCR. Alimentos e Nutrição Araraquara 2005; 16(2):99-103. [ Links ]
36. Abu AS, Radstrom P. Capacity of nine thermostable DNA polymerases to mediate DNA amplification in the presence of PCR-inhibiting samples. Applied and Environmental Microbiology 1998; 64:3748-3753. [ Links ]
37. Rainard P, Fromageau A, Cunha P, Gilbert FB. Staphylococcus aureus lipoteichoic acid triggers inflammation in the lactating bovine mammary gland. Veterinary Research 2008; 39(5):52. [ Links ]
38. Ercolini D, Blaiotta G, Fusco V, Coppola S. PCR-based detection of enterotoxigenic Staphylococcus aureus in the early stages of raw milk cheese making. Journal of Applied Microbiology 2004; 96(5):1090-1096. [ Links ]
39. Ramesh A, Padmapriya B, Chrashekar A, Varadaraj M. Application of a convenient DNA extraction method and multiplex PCR for the direct detection of Staphylococcus aureus and Yersinia enterocolitica in milk samples. Molecular and Cellular Probes 2002; 16:307-314. [ Links ]
40. Khan M, Kim C, Kakoma I, Morin E, Hansen R, Hurley W, Tripathy D, Baek, B. Detection of Staphylococcus aureus in milk by use of polymerase-chain-reaction analysis. American Journal of Veterinary Research 1998; 59(7):807-813. [ Links ]
41. Brakstad O, Aasbakk K, Maeland JA. Detection of Staphylococcus aureus by polymerase chain reaction amplification of the nuc gene. Journal of Clinical Microbiology 1992; 30(7):1654-1660. [ Links ]
42. Yang Y, Su X, Yuan Y, Kang C, Li Y, Zhang W, Zhong X. Detection of Staphylococcus aureus in Dairy Products by Polymerase Chain Reaction Assay. Agricultural Sciences in China 2007; 6(7):857-862. [ Links ]
43. Manfredi E, Leotta G, Rivas M. PCR múltiple para la detección de los genes sea, seb, sec, sed y see de Staphylococcus aureus. Caracterización de aislamientos de origen alimentario. Revista Argentina de Microbiología 2010; 42(3):212-215. [ Links ]
44. Castañeda Vázquez H, El Sayed A, Jager S, Joachim A, Lammler C, Woter W. Estudio comparativo de las características genotípicas de cepas de Staphylococcus aureus aisladas de casos de mastitis clínica y subclínica en México. Veterinaria México 2006; 37(2):165-179. [ Links ]
45. Granados Beltrán E. Caracterizacion de aislamientos de Staphylococcus aureus asociados a mastitis bovina en las localidades de Cotzio y Tajaro, mediante secuenciacion del gen de arn robosomal 16s. Universidad Michoacán de San Nicolás de Hidalgo, 2011. http://bibliotecavirtual.dgb.umich.mx.8083/jspui/bitstream/123456789/422/1/CARACT~2.PDF [ Links ]
46. Oliveira DC, de Lencastre H. Multiplex PCR strategy for rapid identification of structural types and variants of the mec element in methicillin-resistant Staphylococcus aureus. Antimicrobial Agents and Chemotherapy 2002; 46:2155-2161. [ Links ]













