Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biosalud
Print version ISSN 1657-9550
Biosalud vol.14 no.2 Manizales jJuly/Dec. 2015
https://doi.org/10.17151/biosa.2015.14.2.4
DOI: http://dx.doi.org/10.17151/biosa.2015.14.2.4
CHARACTERIZATION OF THE SECONDARY STRUCTURE OF HISTONE H1 SUBTYPES BY CIRCULAR DICHROISM (CD)
Mary Orrego Cardozo2
Inma Ponte3
Pedro Suau4
1 Trabajo realizado en el grupo de investigación Expresión Génica del Departamento de Bioquímica y Biología Molecular de la Facultad de Biociencias de la Universidad Autónoma de Barcelona, España.
2 Ph.D. en Bioquímica y Biología Molecular. Docente Investigadora Universidad Autónoma de Manizales - Docente Catedrática Universidad Nacional de Colombia, sede Manizales. Departamento de Bioquímica y Biología Molecular, Facultad de Biociencias, Universidad Autónoma de Barcelona. Barcelona, España. Autor para correspondencia. Correo electrónico: morregoc@unal.edu.co - maryorrego@autonoma.edu.co
3 Ph.D. en Biología. Departamento de Bioquímica y Biología Molecular, Facultad de Biociencias, Universidad Autónoma de Barcelona. Barcelona, España. Correo electrónico: inma.ponte@uab.es
4 Ph.D. en Ciencias Biológicas. Departamento de Bioquímica y Biología Molecular, Facultad de Biociencias, Universidad Autónoma de Barcelona. Barcelona, España. Correo electrónico: pere.suau@uab.es
Introducción: Las histonas H1 modulan la estructura y la función de la cromatina. Las células somáticas de mamífero contienen los subtipos H1º, H1a, H1b, H1c, H1d y H1e; en células germinales de testículo y en ovocito, se encuentran respectivamente H1t y H1oo. Su estructura está conformada por un dominio central globular flanqueado por los dominios N-Terminal (DNT) y C-Terminal (DCT). Objetivo: Caracterizar la estructura secundaria de subtipos de la histona H1 mediante dicroísmo circular (DC). Materiales y Métodos: La histona H1 total se extrajo de núcleos de cerebro de rata por cromatografía de intercambio catiónico; la H1º se purificó por filtración en gel y las H1a, H1b, H1c y H1e por cromatografía líquida de alta resolución de fase reversa (RF-HPLC). Los espectros de DC se realizaron en tampón fosfato 10 mM; tampón fosfato 10 mM, 20% TFE (trifluoroetanol); tampón fosfato 10 mM, 40% TFE; tampón fosfato 10 mM, 60% TFE; tampón fosfato 10 mM, 150 mM NaCl y tampón fosfato 10 mM, 1 M NaCl. El análisis de los espectros se realizó con el programa Standard Analysis. Resultados: El porcentaje de hélice-alfa se calculó por diferentes métodos matemáticos teniendo en cuenta elipticidad molar a 193 nm y a 222 nm; con programa de deconvolución K2D y con relaciones cualitativas R1 y R2. El TFE induce la estructura en hélice-alfa en cada uno de los subtipos, mientras que NaCl no induce ningún cambio importante. Conclusión: Los subtipos con mayor contenido de hélice-alfa son H1a y H1c. Las diferencias observadas en el porcentaje de hélice-alfa entre los diferentes subtipos puede ser importante para su diferenciación funcional.
Palabras clave: dicroísmo circular, histonas H1, estructura secundaria, hélice-alfa, estructura-beta, ADN, trifluoroetanol.
H1 histones modulate the structure and function of chromatin. Mammalian somatic cells contain H1º, H1a, H1b, H1c, H1d and H1e subtypes; H1t and H1oo are found in testicular germ cells and oocyte, respectively. Its structure consists of a globular core domain flanked by N-terminal (DNT) and C-terminal (DCT) domains. Objective: To characterize the secondary structure of histone H1 subtypes through circular dichroism (CD). Materials and Methods: Total histone H1 was extracted for rat brain nuclei by cation exchange chromatography; histone H1º was purified by gel filtration and the histones H1a, H1b, H1c and H1e were purified by reversed phase high performance liquid chromatography (RP-HPLC). CD spectra were performed in 10 mM phosphate buffer; 10 mM, 20% TFE phosphate buffer (trifluoroethanol); 10 mM, 40% TFE; phosphate buffer 10 mM, 60% TFE; phosphate buffer 10 mM, 150 mM NaCl and phosphate buffer 10 mm, 1 M NaCl. The analysis of the spectra was performed with JASCO Standard Analysis. Results: The percentage of alpha-helix was calculated using different mathematical methods, taking into account the molar ellipticity at 193 nm, and 222 nm, with K2D deconvolution program and the R1 and R2 qualitative relationships. The results indicate that TFE induced the alpha-helix structure in each of the subtypes, whereas NaCl did not induce any significant change. Conclusion: H1a and H1c are subtypes with highest content of alpha-helix. The observed differences in the percentage of alpha-helix between different subtypes may be important for their functional differentiation.
Key words: circular dichroism, histone H1, secondary structure, alpha-helix, beta-structure, DNA, trifluoroethanol.
En las células eucariotas la cromatina está formada por un complejo de ADN y proteínas que compactan eficazmente grandes cantidades de ADN dentro del pequeño volumen nuclear. Las histonas modulan la estructura y la función de la cromatina, las cuales, a su vez, afectan la utilización del ADN genómico. La estructura de la cromatina está sujeta a procesos de dinámica estructural que hacen posibles la replicación, la transcripción y la regulación de la expresión génica (1, 2).
Las histonas H1 son proteínas básicas que interaccionan con el ADN internucleosomal (3-5). Se han descrito numerosos subtipos de la histona H1 en vertebrados, invertebrados y plantas, pero los mejor caracterizados son los siete subtipos de los mamíferos, cinco somáticos, H1a, b, c, d, e, H1º, y dos específicos de células germinales, H1t y H1oo (6), la H5 de eritrocito de ave (7) y la B4 de Xenopus (8).
Los diferentes subtipos de histonas sufren modificaciones postraduccionales como: fosforilación, acetilación, metilación, poli-ADP-ribosilación y ubiquitinización. Recientemente, se han identificado nuevas modificaciones postraduccionales en histonas H1 de eritrocitos de pollo: cuatro acetilaciones, tres formilaciones y dos fosforilaciones (9). Las histonas H1 difieren en estabilidad evolutiva (10) y en las tasas relativas de síntesis y degradación, en células en división y en células quiescentes, propiedades que permiten cambios en sus proporciones relativas durante el desarrollo y la maduración de los tejidos. Las histonas H1 presentan diferencias de afinidad relativa por el ADN y por la cromatina; la H1a es el subtipo con más baja afinidad, las H1b y H1c tienen afinidades intermedias y las H1d, H1e y H1º tienen las afinidades más altas (11).
La estructura de las histonas H1 está conformada por un dominio central globular flanqueado por los extremos amino-terminal (N-terminal) y carboxilo-terminal (C-terminal); su dominio globular interacciona con el ADN de enlace a la entrada y salida del nucleosoma (12) y está constituido por un haz de 3 hélices-alfa y una lámina-beta antiparalela (beta-hairpain) en el extremo carboxílico. El dominio N-terminal (DNT) presenta dos regiones claramente diferenciadas: la región más cercana al N-terminal tiene carácter apolar, por la presencia de residuos de alanina y prolina y la otra región, adyacente al dominio globular, es similar al dominio C-terminal en composición aminoacídica, contiene residuos básicos, especialmente lisina. Por su parte, el dominio C-terminal (DCT), rico en lisina, serina, prolina y alanina, interacciona con el ADN entre los nucleosomas, neutraliza su carga y facilita la compactación de la cromatina (13). El primer nivel de compactación de la cromatina, el nucleosomal, puede deberse, en parte, a interacciones electrostáticas entre las cargas positivas de la histona H1 y los fosfatos del ADN internucleosomal (14). Es probable que los dominios terminales de la H1 se plieguen en hélice-alfa y lámina-beta al interaccionar con el ADN (15).
Por otra parte, los extremos N-terminal y C-terminal son altamente básicos. En disolución, estos dominios están desestructurados, probablemente como consecuencia de la distribución regular de residuos de prolina y por las repulsiones entre las cargas positivas de las cadenas laterales de los numerosos residuos de lisina y arginina, estos residuos constituyen casi el 30% del total (16).
La variación en secuencia y estructura de los subtipos de la histona H1 ha sido ampliamente reconocida como un importante factor en la determinación de la estabilidad local de la cromatina. La variación en las secuencias de aminoácidos entre los subtipos de una misma especie radica en la extensión de los dominios policatiónicos (N-terminal y C-terminal), mientras que el dominio globular es conservado en su secuencia de aminoácidos. Dentro de un mismo organismo, el peso molecular de dos subtipos de la histona H1 puede diferir en aproximadamente 1 kDa (Tabla 1) (17, 18); esta diferencia se debe al dominio C-terminal de la molécula. La distinta composición y conformación de los dominios de los subtipos pueden jugar múltiples papeles funcionales en la estructura de la cromatina y en el proceso de regulación génica.
En presencia de inductores de hélice-alfa_ el DCT adopta una estructura en cortos segmentos de hélice, interrumpidos por los residuos de prolina (19, 20), que podrían interaccionar con uno de los surcos del ADN (21). Se ha propuesto que los motivos (S/T)P(K/R)(K/R), presentes en el DCT, adoptan una estructura de giro _eta (b ) (22). A su vez, estudios con péptidos, correspondientes a los extremos N-terminal y C-terminal de las histonas H1º y H1e, mediante dicroísmo circular (DC) y resonancia magnética nuclear en dos dimensiones demostraron que los péptidos se estructuran en hélice-alfa y codos b,s o s,b (23). Igualmente, se determinó que los elementos de estructura secundaria, presentes en el DCT unido a ADN, incluyen hélice-alfa, estructura-beta, giros y bucles abiertos (15).
En presencia de agentes aglomerantes, el DCT de las histonas H1t y H1º se estructura en hélice-alfa, banda-beta y bucles abiertos; además, el DCT no unido se encuentra en un estado de glóbulo fundido, como en la estructura secundaria nativa, pero carece de la estructura del dominio cooperativamente unida al ADN (24).
El efecto de la fosforilación sobre la estructura secundaria del complejo DCT-ADN es específico de sitio y depende del número de grupos fosfato. La fosforilación completa aumenta la proporción de estructura-beta y disminuye la de hélice-alfa, mientras que la fosforilación parcial aumenta la cantidad de estructura indefinida y causa disminución de la hélice-alfa, sin un aumento significativo de la estructura-beta. El grado de fosforilación también tiene efecto sobre la afinidad del DCT unido al ADN; la fosforilación parcial reduce drásticamente la agregación de fragmentos de ADN por el DCT, pero la fosforilación completa restaura, en gran medida, la capacidad de agregación del DCT no fosforilado (25).
La unión del dominio C-terminal al ADN es básicamente electrostática; esto implica compensación de la carga recíproca de los fosfatos del ADN y los grupos amino de la lisina. La eliminación de la carga de la lisina a pH alcalino induce el plegamiento del DCT con proporciones de motivos de estructura secundaria similares a los observados en complejos con ADN; estas observaciones condujeron a sugerir que el aumento de hidrofobicidad de las cadenas laterales anfipáticas de la lisina, que genera la neutralización de la carga en la interacción con el ADN, es el responsable del plegamiento del dominio C-terminal (26).
Se ha determinado el efecto de varios detergentes sobre la estructura secundaria del DCT, utilizando espectroscopia de infrarrojo (27). Los detergentes neutros, Brij 35 y Triton X-100, y el detergente iónico, SDS, inducen el plegamiento del DCT no fosforilado, con proporciones de estructura secundaria similares a los encontrados en complejos ADN-DCT. La fosforilación del DCT se comporta como un interruptor estructural; en la presencia de detergentes, aumenta la cantidad de estructura- beta y disminuye la hélice-alfa. El mayor efecto se observa en el DCT completamente fosforilado, en presencia de SDS, cuando se convierte en una proteína todo beta y se forman fibras amiloideas. En condiciones similares, cuando la H1º está completamente fosforilada forma fibras amiloideas (27).
Por otra parte, el DC es una técnica estándar para medir la actividad óptica de moléculas asimétricas en disolución, como las proteínas y los ácidos nucleicos, mediante la absorción desigual de la luz polarizada circularmente a la derecha y a la izquierda por moléculas activas ópticamente. El espectro de DC se puede observar en dos regiones espectrales: en el ultravioleta (UV) lejano, o región amida (170 nm - 250 nm), debido a los enlaces peptídicos, y en el UV cercano (250 nm - 330 nm), debido a los aminoácidos aromáticos. La región amida da información sobre la organización estructural de una proteína y es usada para caracterizar la estructura secundaria y sus cambios; por ejemplo, la estructura en hélice-alfa muestra un fuerte y característico espectro en la región del UV lejano.
Espectros de referencia observados en varias proteínas, con conformación en hélice-alfa, muestran dos picos negativos: uno con un valor de elipticidad molar de -35700 a 222 nanómetros de longitud de onda ([Ø]222 - 35700) y otro con un valor de elipticidad molar de -32600 a 208 nm ([Ø]208 - 32600); además, aparece un pico positivo de +76900 a una longitud de onda de 191 nm ([Ø]191 + 76900). El espectro característico de la banda beta muestra una banda negativa de -18400 de elipticidad molar ([Ø]217 - 18400) y una banda positiva con valor de elipticidad molar de + 31900 ([Ø]195 + 31900). La conformación desestructurada (random coil) presenta un pequeño pico a 217 nm de 4600 y uno mayor a 197 nm (-41900) (28).
Las conformaciones básicas de las proteínas son fácilmente distinguibles por su espectro de DC. Las histonas disueltas en agua, o en disolución a muy baja fuerza iónica, se encuentran desestructuradas. Las histonas H1 tienen un espectro de DC característico de hélice-alfa en presencia de sal o de inductores como TFE (trifluoroetanol) o NaClO4 (perclorato de sodio). Por tanto, a partir de los espectros de DC en la región amida, se pueden utilizar varios modelos matemáticos para determinar la estructura secundaria de las proteínas (29-32).
Como se ha descrito en párrafos anteriores, la mayoría de estudios sobre la estructura de las histonas H1 incluye determinación de estructura secundaria de péptidos correspondientes a los dominios terminales y determinación de estructura de los dominios amino y carboxi-terminales de algunos subtipos como H1º, H1e y H1t y su interacción con moléculas como ADN, agentes aglomerantes y trifluoroetanol, a través de diversas técnicas, pero, poco hay reportado sobre la determinación de la estructura secundaria de las histonas H1 completas; por ejemplo, se ha determinado la estructura secundaria en histonas H1 parcialmente purificadas como en la mezcla de H1bcde, en H1a, y H1t (33); en este contexto, por lo tanto, este trabajo de investigación tuvo como objetivo caracterizar la estructura secundaria de subtipos de la histona H1 (H1º, H1a, H1b, H1c, H1e) mediante DC.
Purificación de núcleos
Los núcleos se purificaron a partir de cerebro de ratas, de la cepa OFA Sprague Dawley, de dos grupos de edad, de 15 días, para la posterior purificación de H1a y H1b, y de 30 días o más, para purificar H1º (34, 35). El tejido se homogeneizó con sacarosa 1 M en cacodilato sódico 4 mM, KCl 100 mM, espermina 0,60 mM, espermidina 200 mM, EDTA 4 mM, tiodiglicol 4%, PMSF 0,1 mM, 0,1 mg de aprotinina, 0,1 mg de leupeptina y 0,1 mg de pepstatina. Los núcleos se precipitaron por centrifugación del homogeneizado sobre una disolución de sacarosa 2M, a 28.000 rpm, en un rotor Beckman SW 28. El precipitado nuclear se resuspendió en cacodilato sódico, KCl, espermina, espermidina, CaCl2 y tiodiglicol y se comprobó el estado de los núcleos al microscopio óptico.
Extracción de la histona H1 totalPara purificar la H1 total de cerebro de rata se modificó el método de extracción salina (36). Teniendo en cuenta la concentración de ADN en la disolución de núcleos, se realizó digestión con 5 ml de nucleasa micrococal (0,5 unidades/ ml) durante 10 min a 37ºC. Los núcleos se recuperaron en el precipitado después de centrifugar durante 10 min a 12.000 rpm; se resuspendieron en 2 ml de EDTA 1 mM, Tris 10 mM, inhibidores a pH 7,4 y se dializaron toda la noche a 4ºC frente a EDTA 1 mM, Tris 10 mM a pH 7,4. Después de este proceso de lisis nuclear, la cromatina quedó libre en la disolución.
La cromatina solubilizada se ajustó a una concentración 0,35 M de NaCl con gotas de NaCl 4 M a pH 8,8. Después, se añadió Carboximetil-Sephadex C-25 hidratado (CM-S) (24 mg/ml) y se agitó en hielo durante 2 horas; en este período, las histonas H1 cargadas positivamente fueron selectivamente extraídas de la cromatina y unidas a la resina de CM-S –operación por tandas– (36, 37).
Después del proceso de intercambio catiónico, la suspensión se centrifugó a 10.000 rpm durante 10 min para recuperar, en el precipitado, las histonas unidas a la resina de CM-S y, para eliminar, en el sobrenadante, los restos de cromatina, ADN e histonas no unidas. La suspensión de CM-S, con histonas unidas, se lavó cuatro veces más con NaCl 0,35 M, Tris 10 mM, PMSF 0,1 mM a pH 8,8 para retirar todos los restos.
Posteriormente, el CM-S, con histonas unidas y equilibrado con NaCl 0,35 M, Tris 10 mM, PMSF 0,1 mM a pH 8,8, se cargó en una columna que contenía 3 ml de Sephadex G-25, también, previamente equilibrada con NaCl 0,35 M, Tris 10 mM y pH 8,8.
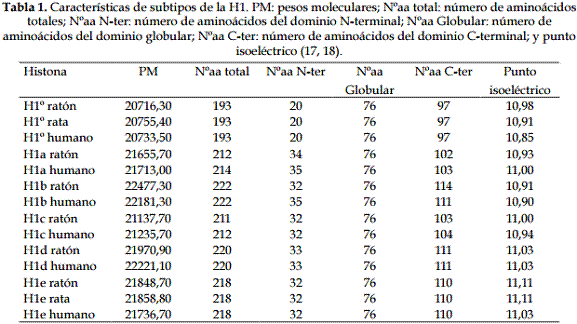
Las histonas H1, unidas a la resina de CM-Sephadex, se eluyeron con 60 ml de un gradiente lineal de NaCl 0,35 M a 1,2 M, Tris 10 mM/pH 8,8. El perfil de elución se registró a 226 nm con un detector Unicord SII de Pharmacia-Biotech y se recogieron fracciones de 1 ml. La composición de las fracciones se comprobó en geles de poliacrilamida con SDS (38, 39); el gel de resolución se preparó con poliacrilamida al 15 %, porque se obtiene mejor resolución de la H1 de rata (38); el gel de concentración se preparó al 5%. Las fracciones correspondientes al pico en el cromatograma (Figura 1) se dializaron frente a ácido acético al 1%, se liofilizaron y se utilizaron como material de partida para separar cada uno de los subtipos: H1º, H1a, H1b, H1c y H1e.
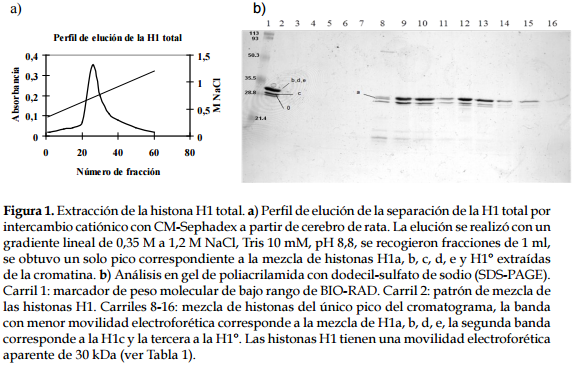
Separación de la histona H1º
La separación de la H1º se realizó a partir de H1 total purificada de cerebro de rata de 30 días o más; a esta edad, esta proteína se encuentra en mayor proporción (40). Se separó por filtración en gel (41) con Biogel P100 en una columna de 160 cm de longitud y un diámetro de 2 cm. Entre 1,25 y 3 mg de H1 total liofilizada, obtenidos por intercambio catiónico con CM-S, se resuspendieron en 2 ml de urea 4 M, Tris 10 mM, se cargaron en la superficie del gel y se realizó elución con 10 mM HCl, 0,02 azida sódica (NaN3), a temperatura ambiente, a una velocidad de 10 ml/hora y se recogieron fracciones de 3,5 ml. El perfil de elución se registró con un detector Unicord SII de Pharmacia-Biotech. Las fracciones de los picos se analizaron por electroforesis de poliacrilamida con SDS (38, 39).
Purificación de las histonas H1a, H1b, H1c, H1eA partir de la histona H1 total, extraída por intercambio catiónico, las histonas H1a, b, c y e se separaron por Cromatografía Líquida de Alta Resolución de Fase Reversa (RP-HPLC) (42). La cromatografía se realizó en un sistema modular Waters, que consiste en dos bombas (modelo 150), un inyector de líquido (UGK) y un detector de absorbancia modelo 490E. El sistema fue controlado por el programa Máxima 820 (Dynamic Solution, Division of Millipore Corp.). Se usó una columna Vydac C18 de 28 cm de longitud. Se preparó como disolución A, ácido trifluoracético al 0,1%, y como disolución B, ácido trifluoracético al 0,1% y acetonitrilo (CH3CN) al 95%. La histona H1 total liofilizada (obtenida por intercambio catiónico) se resuspendió en 1 ml de disolución A y se centrifugó a 13.000 rpm durante 5 min, para eliminar cualquier material particulado antes de inyectar la muestra en el sistema de HPLC; posteriormente, se realizó la inyección manual de la muestra. La elución se realizó durante 140 min a un flujo de 0,7 ml/min con el siguiente gradiente: de 0 a 10 min 100% de A, de 10 a 15 min 80% de A y 20% de B, de 15 a 115 min 60% de A y 40% de B, de 115 a 120 min 10% de A y 90% de B, y finalmente de 120 a 140 min 100% de A. El perfil de elución se registró a 214 y a 280 nm de longitud de onda.
Las fracciones se analizaron por PAGE-SDS y PAGE-AU, geles de poliacrilamida con urea-acético de alta resolución (38, 40, 43). El gel de resolución con 15% de acrilamida, urea 2,5 M, ácido acético 0,9 M, persulfato de amonio al 0,5% y 400 ml TEMED (tetra-metil-etilen-diamina). se dejó polimerizar toda la noche5. El gel de concentración se preparó con acrilamida al 7%, urea 2,5 M, ácido acético 0,9 M, persulfato de amonio al 0,5% y TEMED.
Se realizó pre-electroforesis, a 200 voltios (v) durante 12 horas a 4ºC, para remover radicales libres del gel (38); se usó ácido acético 0,9 M como tampón de recorrido. Después de la pre-electroforesis, el tampón se cambió por ácido acético 0,9 M fresco y se cargaron las muestras. Previamente, un volumen de 50 ml de cada muestra fue evaporado y disuelto en 10 ml de tampón de muestras (urea 8 M, tiodiglicol 5%, ditiotreitol 0,1 mM, pironina 0,01% y ácido acético 0,9M) (38). La electroforesis se corrió a 320 v durante 29 horas. El gel se sumergió en una disolución de metanol/agua/acético (5:5:1) durante 30 min, con el fin de fijar las proteínas, y luego se tiñó durante toda la noche con negro amido. El gel se destiñó con una disolución de metanol/agua/acético (45:45:10).
Desnaturalización-renaturalización de las proteínasAntes de medir los espectros de DC, todos los subtipos de la H1 se sometieron a un proceso de desnaturalización-renaturalización. La desnaturalización se realizó con urea 6 M y la renaturalización se realizó por diálisis frente a concentraciones decrecientes de urea 6 M, 3 M, 1,5 M, 0,75 M, 0,3 M en NaCl 0,2 M y tampón fosfato 10 mM a pH 7; finalmente, las proteínas se dializaron frente a NaCl 0,14 M y tampón fosfato 10 mM a pH 7.
Dicroísmo circularLos espectros de DC se registraron con un espectropolarímetro JASCO J-715. El análisis de los espectros se realizó con el programa Análisis Estándar de JASCO, que permite realizar los espectros de diferencia, el cambio de unidades a elipticidad molar _q__ (deg.cm²/dmol de residuo) y la reducción de ruido de fondo, si es necesario. Los espectros se realizaron con una cubeta de cuarzo rectangular de 0,1 cm de paso de luz a 20ºC; los parámetros seleccionados fueron: rango 260-190 nm, amplitud de la banda 2,0 nm, resolución 0,5 nm, acumulación 4, sensibilidad 50 mdeg, respuesta 2 s y velocidad 50 nm/min. Para cada uno de los subtipos H1º, H1a, b, c y e, se realizó una serie de espectros en: tampón fosfato 10 mM; tampón fosfato 10 mM, 20% TFE; tampón fosfato 10 mM, 40% TFE; tampón fosfato 10 mM, 60% TFE; tampón fosfato 10 mM, 150 mM NaCl y tampón fosfato 10 mM, 1M NaCl. Las concentraciones de las proteínas se midieron espectrofotométricamente usando e2101% = 205 (44, 45).
Los porcentajes de hélice-alfa se calcularon por diferentes modelos matemáticos, a partir de los espectros de DC. El primer modelo se basa en la ecuación empírica que expresa que: % hélice a = 100 [q]222 / (-39500 (1- 2,57 / n))], donde n es el número de enlaces peptídicos y [q]222] es la elipticidad molar de las proteínas a 222 nm de longitud de onda (29). El segundo modelo, utiliza la elipticidad molar a 193 nm, usando la ecuación: % hélice- a = 14,769 + 0,0010261 [q]193], en la cual [q] 93] es la elipticidad molar de la proteína a 193 nm de longitud de onda (30). La ecuación fue obtenida por correlación de los cambios de elipticidad molar a 193 nm con el contenido de hélice obtenido del método de Chen et al. (29); con esta ecuación, es posible obtener el contenido de hélice-alfa corregido por la contribución de aminoácidos aromáticos en proteínas o péptidos con fenilalanina.
Puesto que la elipticidad molar a 222 nm y a 193 nm depende de la exactitud en la determinación de la concentración de las proteínas, con el propósito de eliminar posibles errores en su determinación, se usaron dos parámetros independientes de la concentración: R1 y R2, que son relaciones de intensidades en los espectros de DC (31). R1 es definido como la relación entre [q] máxima en el rango de longitud de onda de 190 nm a 195 nm y la q mínima en el rango 195 nm a 210 nm, y R2 se define como la proporción entre [q] 222 nm y q mínima.
Por último, se usó el programa de deconvolución K2D, que proporciona una estimación de los porcentajes de estructura secundaria de las proteínas, a partir de los espectros de DC, teniendo en cuenta el intervalo entre 200 nm a 240 nm (32), que tampoco depende de la concentración.
Purificación de los subtipos de la H1
La histona H1 se extrajo por cromatografía de intercambio catiónico como se describe en Materiales y Métodos. En el perfil de elución, se obtuvo un único pico (Figura 1a) porque el proceso de intercambio catiónico, en este caso, es un proceso preparativo de la mezcla de histonas H1 (H1º, H1a, b, c, d, e), en el cual estas proteínas se separan de la cromatina y se unen al CM-Sephadex a una concentración 0,35 M de NaCl. El único pico eluyó a partir de 0,6 M y el máximo se registró a 0,7 M.
Cuando la histona H1 total se separa en geles de poliacrilamida con SDS da origen a un patrón de tres bandas electroforéticas. La banda con menor movilidad corresponde a la mezcla de histonas H1a, b, d, e; por debajo de esta, corre la H1c y la tercera banda con mayor movilidad corresponde a la H1º, carril 2 (Figura 1b) (38, 43).
En el análisis electroforético de la H1 total, extraída por intercambio catiónico (carriles 8-16, Figura 1b), las bandas más intensas corresponden al máximo del pico observado (carriles 9, 10, Figura 1b). En todos los carriles, se observa la banda con menor movilidad electroforética formada por H1b, d, e; en el carril 8, por debajo de la anterior, corre con una movilidad ligeramente superior, la H1a; la siguiente banda corresponde a la H1c que se ve más intensa en los carriles 9-13, y por último aparece la H1º a partir del carril 12.
La H1a eluyó a una concentración 0,6 M, mientras que la H1º eluyó al final del pico, a una concentración 0,8M, (a partir del carril 12). La elución más rápida de la H1a indica una menor afinidad de este subtipo por el CM-Sephadex; esto significa que existe un mecanismo de separación electrostático, con la mayor carga positiva en los subtipos que eluyeron al final.
La histona H1º se separó por filtración en gel. En el cromatograma (Figura 2a) se observaron dos picos, el primero corresponde a la mezcla de histonas H1a, b, c, d y e, que empezó a eluir a los 166 ml y se recogió en un volumen de 17 ml (fracciones 50-54) y el segundo corresponde a la H1º, que empezó a eluir a los 190 ml y se recogió en un volumen de 14 ml (fracciones 57-60). En el análisis electroforético (Figura 2b), en los carriles 4, 5, 6, 7 y 8, se observa la mezcla de histonas del primer pico que se separa en las dos bandas electroforéticas típicas, y en los carriles 11, 12, 13 y 14, se observa la H1º que tiene la mayor movilidad en geles de poliacrilamida con SDS.
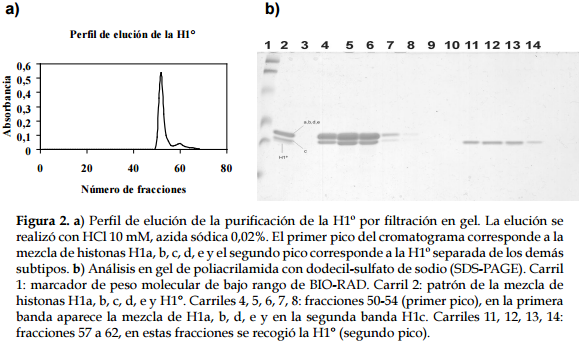
La separación de las histonas H1a, b, c y e, por RP-HPLC, se realizó a partir de H1 total purificada de cerebro de rata de 15 días, cuando la H1a y la H1b se encuentran en mayor proporción (40). Con el gradiente utilizado, como se describe en Materiales y Métodos, las histonas se separaron en 5 picos (cromatograma, Figura 3a). El análisis de las fracciones correspondientes a cada uno de los picos se realizó en geles de poliacrilamida de urea-acético de alta resolución (Figura 3b); se usó como marcador de peso molecular una mezcla de histonas (carriles 1, 15, 16 y 27); en el carril 27 del gel, se observa que el subtipo con menor movilidad es H1a, luego aparecen H1b y H1d; luego se observa la banda con la mezcla H1c-H1e, que tiene la misma movilidad, y por último, H1º, que se separa en sus dos componentes H1ºa y H1ºb. Estas bandas electroforéticas corresponden con las descritas por otros investigadores (38, 40, 43).
El análisis en gel de urea-acético muestra que el primer pico del cromatograma corresponde a una mezcla de H1b y H1º, el segundo pico corresponde a H1b, el tercer pico a H1a y el cuarto a H1e; estos cuatro picos eluyeron de la columna, respectivamente, a los 90, 104 y 107 min. En el quinto pico, que eluyó a los 110 min, se solaparon en gran porcentaje H1c y H1d. La histona H1c se recogió manualmente al final del quinto pico.
Como en el gel de urea-acético la H1e y la H1c tienen la misma movilidad electroforética (38, 40, 43), las fracciones correspondientes a los 5 picos de la RP-HPLC se separaron en gel de poliacrilamida con SDS para comprobar su identidad (Figura 3c). En este gel, la H1e está ubicada en la primera banda, de acuerdo con su movilidad electroforética característica en SDS-PAGE (carril 4), y la H1c en la segunda banda (carril 7). También, se observa que las proteínas no están mezcladas con otros subtipos y su movilidad electroforética es la característica en cada uno de los subtipos (38, 43).
Predicción de estructura secundaria a partir de los espectros de dicroísmo circularEn los espectros de DC de las histonas realizados con 150 mM de NaCl y con 1 M de NaCl en tampón fosfato 10 mM, no se aprecian cambios importantes, si se comparan con los espectros a baja fuerza iónica con 10 mM TP (Figura 4).

En los espectros de las histonas H1º, H1a, H1b, H1c y H1e con TFE (Figura 5), se observa que, al añadir TFE, va disminuyendo el valor de la elipticidad molar a 222 nm y van aumentando los valores con longitudes de onda menores a 200 nm; es decir, el perfil de elipticidad va adquiriendo la forma típica de hélice-alfa. En las histonas H1º, H1a, H1c y H1e el cambio más importante se produjo al pasar de un 20% a un 40% de TFE. En el caso de la H1b, en contraste con los demás subtipos, el aumento de hélice-alfa se produjo de manera gradual al añadir cantidades crecientes de TFE.

Purificación de los subtipos
Para extraer la histona H1, la edad se tuvo en cuenta porque los subtipos H1a y H1b se encuentran en mayor proporción en el cerebro de ratas jóvenes y la H1º se acumula en neuronas en proceso de diferenciación, es decir, a partir de 10 días de edad. Los subtipos H1c, d y e son los más abundantes a cualquier edad, aunque también se encuentran en diferente proporción según el grado de desarrollo del tejido (35, 40).
Teniendo en cuenta que la histona H1 es una proteína policatiónica, se realizó una extracción preparativa usando como intercambiador catiónico carboxi-metil-Sephadex C-25, en la cual, en un primer paso, las histonas H1 se unieron a una suspensión de CM-S, operación por tandas (36, 37) y, en un segundo paso, la suspensión de resina-histonas unidas se cargó en una columna de 28 cm sobre 3 ml de Sephadex G-25; el paso por Sephadex G-25 evitó que las histonas separadas del CM-S en el proceso de elución con el gradiente lineal de NaCl se volvieran a unir a esta resina. Esta modificación se realizó al método de extracción salina (36), porque, en los primeros ensayos, las histonas quedaban retenidas en la resina de CM-S y no se recuperaban en el tampón de elución; además, si se aplicaba un gradiente con mayor concentración de NaCl se unían las histonas internas (H2A, H2B, H3 y H4). Las histonas H1 se separan a una fuerza iónica de 0,35 M porque se encuentran unidas a la entrada y a la salida de los nucleosomas; por el contrario, las histonas internas forman el centro del nucleosoma y es necesario aplicar mayor fuerza iónica para su separación de la cromatina (3-5).
En el análisis electroforético de la purificación de la H1, se tuvo en cuenta que aunque la electroforesis en geles que contienen SDS separa proteínas de acuerdo con su peso molecular, las histonas realizan contribuciones sustanciales a la carga de sus complejos con SDS y tienen un comportamiento anómalo; estas histonas con peso molecular entre 20 y 22 kDa (Tabla 1) (17, 18) migran como un grupo de proteínas de peso molecular aparente entre 30 y 35 kDa (38), como se observa en el gel respecto al marcador de peso molecular (Figura 1b).
A pH ácido y baja fuerza iónica la precipitación de histonas es mínima; sin embargo, bajo estas condiciones, las histonas existen como cadenas extendidas, se comportan de manera anómala en cromatografía de filtración en gel y exhiben pesos moleculares que pueden estar en un factor de 2 a 6 veces más altos que sus pesos moleculares normales (41, 46); esta característica de mostrar pesos moleculares anómalos permite separar la H1º de la mezcla de histonas H1a, b, c, d, e y H1º por filtración en gel con Biogel P100, producto utilizado por los investigadores para separar histonas (41, 46, 47) y que en este trabajo permitió la separación de la H1º de los demás subtipos.
Por RP-HPLC se logró la separación de los subtipos H1a, H1b, H1c y H1e. Los resultados obtenidos se contrastaron con distintos protocolos de separación realizados por varios investigadores (42, 48-50). En uno de los trabajos, el proceso de separación de las histonas H1 de hígado de rata se realizó durante 45 min a un flujo de 0,5 ml/min, con gradiente lineal desde 60% de disolvente A y 40% de disolvente B hasta 42% de A y 58% de B (disolvente A: 0,1% de ácido trifluoroacético en agua; disolvente B: 0,1% de ácido trifluoroacético en 70% de acetonitrilo) y obtuvieron 6 picos, correspondientes a las histonas H1º, H1b, H1a, H1d, H1e+c y H1c (48). Estos autores, también, separaron H1 de testículo de rata, entre ellas, la H1t, específica de células germinales masculinas, que eluyó a los 38 min en el pico 7. El gradiente y el tiempo utilizado por estos autores es diferente al utilizado en esta investigación, en la cual se obtuvieron 5 picos que corresponden, consecutivamente, a las histonas H1º+b, H1b, H1a, H1e, H1d+c y al final del quinto pico, se recogió manualmente la H1c.
Otros investigadores separaron histonas H1 fosforiladas, utilizando RP-HPLC con una columna C4 de Nucleosil 300-5; en este caso, la cromatografía se realizó durante 35 min, a un flujo de 1,5 mil/min, con un gradiente lineal de acetonitrilo. En este gradiente, se utilizó como solvente A 0,1% de TFA y como solvente B 70% de acetonitrilo y 0,1% TFA; el gradiente empezó con 53% de A y 47% de B; la concentración de B aumentó linealmente de 47 a 55% en 35 min. Con esta cromatografía, las histonas se separaron en dos picos: en el primero, se separó la H1b, y el segundo correspondió a una mezcla de los otros subtipos. Para separar los subtipos de la mezcla, se utilizó una cromatografía líquida de interacción hidrofílica (49).
Otra contrastación se realizó con el trabajo de investigadores que utilizaron células 3T3 de ratón para separar las histonas H1º, H1b, H1c y H1e; además, separaron las histonas totales con un gradiente lineal creciente de tampón A (0,1% TFA y 95% de acetonitrilo). Los pasos del gradiente fueron: 0,1-25% durante 10 min, 25-30% durante 15 min, 30-55% durante 100 min y 55-90% durante 5 min (42).
En ninguno de los casos descritos se logró la purificación completa de los 5 subtipos somáticos de histona H1. En cada uno de estos trabajos, el material de partida, los gradientes y los tiempos de elución fueron diferentes; estas variables pueden influir en la separación de cada uno de los subtipos. Como consecuencia, los protocolos de purificación por HPLC varían; esta situación hace necesaria su optimización en cada caso.
En resumen, se sabe que las técnicas cromatográficas no permiten la purificación simultánea de todos los subtipos de la histona H1 y sus formas modificadas (49). Generalmente, se requieren sistemas cromatográficos combinados. Por este motivo, en esta investigación se realizaron diferentes tipos de cromatografía para la purificación de los subtipos H1a, b, c, e y H1º. Primero, se usó la cromatografía de intercambio catiónico con CM-S, para purificar la H1 total, a partir de cerebro de rata. La purificación por este método, que utilizó el gradiente lineal de NaCl, evita el tratamiento de las proteínas con ácido perclórico o ácido clorhídrico, sistemas más usados para su extracción, protegiendo, de esta manera, la desestructuración del dominio globular (51).
En segundo lugar, se utilizó la cromatografía de filtración en gel, la cual permitió la separación de la histona H1º de las demás histonas de manera completamente pura. La histona H1 se obtuvo a partir de ratas con más de 30 días de edad, debido a que este es el período del desarrollo durante el cual se acumula mayor cantidad de H1º en neuronas en proceso de diferenciación (40).
Por último, se usó RP-HPLC para la purificación de las histonas H1a, b, c, e. Algunos autores (43, 49) han descrito que este método permite la separación de formas modificadas y no modificadas de las histonas y también la separación parcial de varios subtipos de histona H1 y sus formas fosforiladas. En comparación con el proceso de filtración en gel para purificar la H1º, la RP-HPLC tiene importantes ventajas como el tiempo de duración de la cromatografía y los componentes de la fase móvil, que protegen las proteínas de procesos de desnaturalización.
Predicción de estructura secundaria a partir de los espectros de dicroísmo circularDe los porcentajes de hélice-alfa para cada uno de los subtipos estudiados (Tabla 2), se deduce que los espectros de DC de cada uno de los subtipos en tampón fosfato 10 mM presentan un espectro característico de hélice-alfa, aunque en muy bajo porcentaje, debido posiblemente a que los iones fosfato en la disolución estabilizan parcialmente las regiones helicoidales del dominio globular de las proteínas (33). Si se compara el valor de elipticidad molar de los diferentes subtipos, se observa que la H1b es el subtipo que menos se estructura en tampón fosfato, mientras que el subtipo que más se estructura es H1c.
Al incrementar progresivamente el porcentaje de TFE, los valores de elipticidad molar, a menos de 200 nm, aumentan progresivamente hasta hacerse claramente positivos, mientras que los valores entre 208 y 230 nm disminuyen, formándose dos mínimos, uno entre 205 y 208 nm y el otro a 222 nm. Estos cambios indican que el TFE induce cada vez más un espectro típico de estructura en hélice-alfa e incrementa el contenido de hélice-alfa, posiblemente porque estabiliza los puentes de hidrógeno intramoleculares entre los aminoácidos (20).
Al comparar los porcentajes de hélice-alfa calculados (Tabla 2) se observa variabilidad en los valores obtenidos a partir de la elipticidad molar a 193 nm, la elipticidad molar a 222 nm y el programa de deconvolución K2D. Esta variabilidad en el porcentaje de hélice-alfa según el modelo matemático utilizado, ha sido ampliamente reflejada en la literatura, al no existir un método claramente establecido (20, 30, 31, 52, 53). Así, para Pintar et al. (52), los porcentajes de hélice-alfa obtenidos a partir de la elipticidad a 222 nm son afectados por la presencia de otras conformaciones (distintas a las de hélice) y por la contribución de residuos aromáticos. Algunos autores han establecido un método para el cálculo del porcentaje de hélice-alfa en péptidos, a partir del valor de elipticidad molar a 193 nm, con el fin de corregir el error producido por la presencia de anillos aromáticos (30). En el caso de los programas de deconvolución, el problema radica en saber hasta qué punto las proteínas de la base de datos del programa son adecuadas para las proteínas en estudio. En la determinación de contenido de hélice-alfa a partir de espectros de DC realizados con distintos métodos, se obtienen diferencias de más del 50% (52).
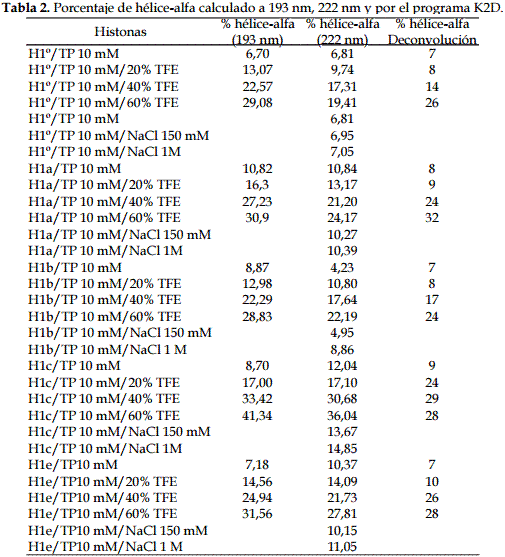
La variabilidad de los porcentajes, debido a los distintos modelos utilizados, es del mismo orden que las diferencias encontradas en los porcentajes de hélice-alfa entre los diferentes subtipos. En consecuencia, estas diferencias de porcentajes entre los distintos subtipos no se pueden considerar significativas. No obstante, podemos observar que la H1c presentó siempre un mayor contenido de hélice-alfa en tampón fosfato, en los distintos porcentajes de TFE y en presencia de NaCl.
En estudios anteriores de DC, realizados con subtipos H1 parcialmente purificados (H1bcde, H1a y H1t, en 1 M NaCl y 60% de TFE), se obtuvieron valores de elipticidad molar a 220 nm, similares a los del presente trabajo y se propuso que la hélice-alfa observada a 1 M de NaCl se debe tan solo a la estructuración del dominio globular, mientras que el porcentaje de hélice-alfa en presencia de TFE se debe a la inducción de hélices-alfa en los dominios terminales (33).
Por otro lado, se ha observado que el TFE incrementa el porcentaje de hélice-alfa de la histona H1 y de la histona H5 de pollo más eficazmente que el NaClO4 y el NaCl (19). Estos autores proponen que la unión de los ligandos al ADN estabiliza fragmentos de hélice-alfa en los dominios C-terminales de la H1 y de la H5. En este sentido, resultaría interesante estudiar los cambios que se producen en el espectro de DC del ADN, al añadir cada uno de los subtipos de la histona H1 (54).
Por espectroscopia de infrarrojo (IR) se determinó la estructura secundaria de los dominios C-terminal de los subtipos H1º y H1t unidos a ADN; el dominio C-terminal tiene poca estructura en solución acuosa, pero llega a ser extensivamente plegado en interacción con el ADN, encontrándose elementos de estructura secundaria como hélice-alfa, estructura-beta, y giros. La estructura del dominio C-terminal de la H1º unido a ADN muestra dependencia de la concentración de sal; en concentración fisiológica de sal, 140 mM, llega a ser altamente estructurado con 24% de hélice-alfa, 25% de estructura-beta, 17% de loops abiertos y 33% de giros (15).
Se ha observado que la fosforilación del DCT de la H1º genera un reordenamiento estructural caracterizado por una disminución en la proporción de hélice-alfa y un aumento en la proporción de banda-beta. A una relación, proteína/ADN (r) (peso/peso) de 0,25 o menos, la hélice-alfa disminuye del 24 al 8% y la banda-beta aumenta del 24 al 36% en el DCT fosforilado; a r = 0,5, la hélice disminuye hasta una proporción de 6% y la banda-beta aumenta hasta 46% y cuando, r = 0,7, aproximadamente la proporción de saturación, el CTD finalmente se convierte en una proteína todo-beta, sin hélice-alfa, 16% de regiones flexibles, 20% de giros y 74% estructura-beta. El DCT de H1e de ratón (CH1e) no fosforilado presentó una proporción significativamente mayor de hélice-alfa (34%) y menor proporción de estructura-beta (18%). El CH1e fosforilado presentó menor cantidad de hélice-alfa, que de estructura-beta; pero a diferencia del DCT de H1º, el CH1e completamente fosforilado conserva una proporción significativa de hélice-alfa (17%) y el aumento de estructura fue menos pronunciado (55%) (25).
Se ha estudiado el efecto de detergentes neutros, Brij 35 y Triton X-100, y detergente aniónico, SDS, sobre la estructura secundaria del DCT de histona H1, por espectroscopia de IR a concentración de sal fisiológica (140 mM NaCl). En presencia de TritonX-100, las proporciones de motivos de estructura secundaria encontrados son 34% de giros, 27% de hélice-alfa, 19% plegamientos al azar y regiones flexibles y 20% de estructura-beta. En presencia de SDS, las proporciones son similares con 38% de giros, 26% de hélice-alfa, 16% de plegamientos al azar y flexibles y 20% estructura-beta. Debido a su carácter aniónico, el SDS es capaz de realizar interacciones electrostáticas e hidrófobas (27).
El porcentaje de hélice-alfa en TFE al 60% encontrado en este trabajo, desde 19,41% para H1º hasta 30,68% para H1c a 222 nm (Tabla 2), es semejante al porcentaje de hélice-alfa encontrado en el C-terminal de la H1 unido al ADN (15, 25) y unido a detergentes (27). Este hallazgo aporta a la hipótesis de que en presencia de TFE la estructuración en hélice-alfa de las histonas H1 se debe a los dominios C-terminales.
Ahora bien, las relaciones R1 y R2 son pruebas útiles para determinar la cantidad relativa de hélice-alfa presente en dos o más proteínas similares, y son característicos de la forma del espectro. Además, proporcionan bases precisas para la comparación de la cantidad de hélice-alfa presente, independiente de la concentración de las proteínas (31). Teniendo en cuenta estos criterios, se calcularon los valores R1 y R2 para todos los subtipos en las diferentes condiciones. Los subtipos H1º, H1a, H1b y H1e presentaron un valor positivo de R1 en TP 10 mM, mientras que para la H1c es negativo (Figura 6). En TFE, al 20%, R1 es negativo para todos los subtipos, y al incrementar el TFE al 40% y al 60% los valores se hacen cada vez más negativos. Además, se observa que el cambio más significativo sucede cuando la concentración de TFE pasa del 20% al 40% (Figura 6).
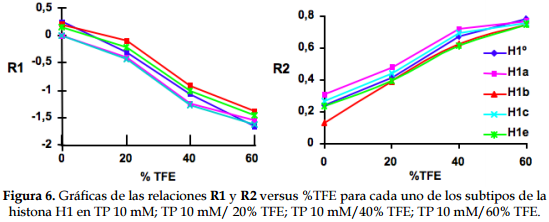
R1 es positivo cuando la [q] máxima y la [q] mínima son negativas e indica bajos niveles de formación de hélice-alfa. Cuando la [q] máxima es positiva y la [q] mínima es negativa, R1 disminuye, lo cual indica que el contenido de hélice-alfa aumenta; estos valores confirman que, a medida que se incrementa el porcentaje de TFE en cada uno de los subtipos, aumenta el porcentaje de hélice-alfa; a medida que los valores R2 se acercan a 1 aumenta el contenido de hélice-alfa. Teniendo en cuenta los valores R1 y R2, los subtipos con mayor contenido de hélice-alfa son la H1a y la H1c.
Se han realizado muchos estudios sobre la estructura secundaria de péptidos correspondientes a los dominios N y C-terminales (23), sobre la estructura del DCT desfosforilado, del DCT fosforilado, sobre complejos ADN-DCT, DCT-aglomerantes, DCT-detergentes (15, 25, 27), pero muy pocos estudios con los subtipos completos, como el realizado por infrarrojo en presencia de detergentes (27). En este sentido, los resultados de esta investigación son importantes porque contribuyen a la comprensión de la estructura secundaria de subtipos de la histona H1 completos.
Por último, los resultados indican que los porcentajes de hélice-alfa son similares en los métodos estudiados; sin embargo, se observaron pequeñas diferencias que podrían ser importantes en la diferenciación funcional de los subtipos, junto a otras variables como la longitud de los dominios, el número y la distribución de cargas y las diferentes afinidades por el ADN y la cromatina.
Los autores expresan su agradecimiento a todas las personas que colaboraron con el desarrollo de este trabajo.
Este trabajo fue patrocinado por el Ministerio de Educación y Ciencia (España), Grant BFU2005-02143.
Los autores declaran que no existe ningún conflicto de interés.
Notas al Pie
5 Una característica del ácido acético es que inhibe el proceso de polimerización, razón por la cual es necesario utilizar mayor cantidad de TEMED que en PAGE-SDS.
1. Van Holde K, Zlatanova J. Chromatin higher order structure: chasing a mirage? Journal of Biological Chemistry 1995; 270(15):8373-6. [ Links ]
2. Wolffe A. Chromatin: structure and function. Academic Press; 1998. [ Links ]
3. Thoma F, Koller T, Klug A. Involvement of histone H1 in the organization of the nucleosome and of the salt-dependent superstructures of chromatin. The Journal of Cell Biology 1979; 83(2 Pt 1):403-27. [ Links ]
4. Clark DJ, Thomas JO. Salt-dependent co-operative interaction of histone H1 with linear DNA. Journal of Molecular Biology 1986; 187(4):569-80. [ Links ]
5. Thomas JO, Rees C, Finch JT. Cooperative binding of the globular domains of histones H1 and H5 to DNA. Nucleic Acids Research 1992; 20(2):187-94. [ Links ]
6. Cole RD. A minireview of microheterogeneity in H1 histone and its possible significance. Analytical Biochemistry 1984; 136(1):24-30. [ Links ]
7. Coles LS, Robins AJ, Madley LK, Wells JR. Characterization of the chicken histone H1 gene complement. Generation of a complete set of vertebrate H1 protein sequences. The Journal of Biological Chemistry 1987; 262(20):9656-63. [ Links ]
8. Smith RC, Dworkin-Rastl E, Dworkin MB. Expression of a histone H1-like protein is restricted to early Xenopus development. Genes & Development 1988; 2(10):1284-95. [ Links ]
9. Sarg B, López R, Lindner H, Ponte I, Suau P, Roque A. Identification of novel post-translational modifications in linker histones from chicken erythrocytes. Journal of Proteomics 2015; 113:162-77. [ Links ]
10. Ponte I, Vidal-Taboada JM, Suau P. Evolution of the vertebrate H1 histone class: evidence for the functional differentiation of the subtypes. Molecular Biology and Evolution 1998; 15(6):702-8. [ Links ]
11. Orrego M, Ponte I, Roque A, Buschati N, Mora X, Suau P. Differential affinity of mammalian histone H1 somatic subtypes for DNA and chromatin. BMC Biology 2007; 5:22. [ Links ]
12. Wolffe AP, Khochbin S, Dimitrov S. What do linker histones do in chromatin? Bioessays 1997; 19(3):249-55. [ Links ]
13. Allan J, Mitchell T, Harborne N, Bohm L, Crane-Robinson C. Roles of H1 domains in determining higher order chromatin structure and H1 location. Journal of Molecular Biology 1986; 187(4):591-601. [ Links ]
14. Clark DJ, Kimura T. Electrostatic mechanism of chromatin folding. Journal of Molecular Biology 1990; 211(4):883-96. [ Links ]
15. Roque A, Iloro I, Ponte I, Arrondo JL, Suau P. DNA-induced secondary structure of the carboxyl-terminal domain of histone H1. The Journal of Biological Chemistry 2005; 280(37):32141-7. [ Links ]
16. Wolffe AP. Histone H1. The International Journal of Biochemistry & Cell Biology 1997; 29(12):1463-6. [ Links ]
17. Drabent B, Franke K, Bode C, Kosciessa U, Bouterfa H, Hameister H, et al. Isolation of two murine H1 histone genes and chromosomal mapping of the H1 gene complement. Mammalian Genome 1995; 6(8):505-11. [ Links ]
18. Albig W, Meergans T, Doenecke D. Characterization of the H1.5 gene completes the set of human H1 subtype genes. Gene 1997; 184(2):141-8. [ Links ]
19. Clark DJ, Hill CS, Martin SR, Thomas JO. Alpha-helix in the carboxy-terminal domains of histones H1 and H5. The EMBO Journal 1988; 7(1):69-75. [ Links ]
20. Luo P, Baldwin RL. Mechanism of helix induction by trifluoroethanol: a framework for extrapolating the helix-forming properties of peptides from trifluoroethanol/water mixtures back to water. Biochemistry 1997; 36(27):8413-21. [ Links ]
21. Ramakrishnan V, Finch JT, Graziano V, Lee PL, Sweet RM. Crystal structure of globular domain of histone H5 and its implications for nucleosome binding. Nature 1993; 362(6417):219-23. [ Links ]
22. Suzuki M, Gerstein M, Johnson T. An NMR study on the DNA-binding SPKK motif and a model for its interaction with DNA. Protein Engineering 1993; 6(6):565-74. [ Links ]
23. Vila R, Ponte I, Jiménez MA, Rico M, Suau P. A helix-turn motif in the C-terminal domain of histone H1. Protein Science: a publication of the Protein Society 2000; 9(4):627-36. [ Links ]
24. Roque A, Ponte I, Suau P. Macromolecular crowding induces a molten globule state in the C-terminal domain of histone H1. Biophysical Journal 2007; 93(6):2170-7. [ Links ]
25. Roque A, Ponte I, Arrondo JL, Suau P. Phosphorylation of the carboxy-terminal domain of histone H1: effects on secondary structure and DNA condensation. Nucleic Acids Research 2008; 36(14):4719-26. [ Links ]
26. Roque A, Ponte I, Suau P. Role of charge neutralization in the folding of the carboxy-terminal domain of histone H1. The Journal of Physical Chemistry B 2009; 113(35):12061-6. [ Links ]
27. Roque A, Teruel N, López R, Ponte I, Suau P. Contribution of hydrophobic interactions to the folding and fibrillation of histone H1 and its carboxy-terminal domain. Journal of Structural Biology 2012; 180(1):101-9. [ Links ]
28. Nicolini CA (Ed.). Chromatin structure and function. New York: Plenum Press; 1979. [ Links ]
29. Chen YH, Yang JT, Chau KH. Determination of the helix and beta form of proteins in aqueous solution by circular dichroism. Biochemistry 1974; 13(16):3350-9. [ Links ]
30. Viguera AR, Serrano L. Side-chain interactions between sulfur-containing amino acids and phenylalanine in alpha-helices. Biochemistry 1995; 34(27):8771-9. [ Links ]
31. Bruch MD, Dhingra MM, Gierasch LM. Side chain-backbone hydrogen bonding contributes to helix stability in peptides derived from an alpha-helical region of carboxypeptidase A. Proteins 1991; 10(2):130-9. [ Links ]
32. Andrade MA, Chacón P, Merelo JJ, Morán F. Evaluation of secondary structure of proteins from UV circular dichroism spectra using an unsupervised learning neural network. Protein Engineering 1993; 6(4):383-90. [ Links ]
33. Khadake JR, Rao MR. Condensation of DNA and chromatin by an SPKK-containing octapeptide repeat motif present in the C-terminus of histone H1. Biochemistry 1997; 36(5):1041-51. [ Links ]
34. Thompson RJ. Studies on RNA synthesis in two populations of nuclei from the mammalian cerebral cortex. Journal of Neurochemistry 1973; 21(1):19-40. [ Links ]
35. Pina B, Martínez P, Simón L, Suau P. Differential kinetics of histone H1(0) accumulation in neuronal and glial cells from rat cerebral cortex during postnatal development. Biochemical and Biophysical Research Communications 1984; 123(2):697-702. [ Links ]
36. García-Ramírez M, Leuba SH, Ausio J. One-step fractionation method for isolating H1 histones from chromatin under nondenaturing conditions. Protein Expression and Purification 1990; 1(1):40-4. [ Links ]
37. Segura JMG, et al. Técnicas instrumentales de análisis en Bioquímica. Síntesis; 2002. [ Links ]
38. Lennox RW, Cohen LH. Analysis of histone subtypes and their modified forms by polyacrylamide gel electrophoresis. Methods in Enzymology 1989; 170:532-49. [ Links ]
39. Sambrook J, Fritsch EF, Maniatis T. Molecular cloning. New York: Cold Spring Harbor Laboratory Press; 1989. [ Links ]
40. Pina B, Martínez P, Suau P. Changes in H1 complement in differentiating rat-brain cortical neurons. European Journal of Biochemistry / FEBS 1987; 164(1):71-6. [ Links ]
41. Bohm EL, Strickland WN, Strickland M, Thwaits BH, van der Westhuizen DR, von Holt C. Purification of the five main calf thymus histone fractions by gel exclusion chromatography. FEBS Letters 1973; 34(2):217-21. [ Links ]
42. Brown DT, Alexander BT, Sittman DB. Differential effect of H1 variant overexpression on cell cycle progression and gene expression. Nucleic Acids Research 1996; 24(3):486-93. [ Links ]
43. Lennox RW, Oshima RG, Cohen LH. The H1 histones and their interphase phosphorylated states in differentiated and undifferentiated cell lines derived from murine teratocarcinomas. The Journal of Biological Chemistry 1982; 257(9):5183-9. [ Links ]
44. Scopes RK. Protein purification: principles and practice. Springer; 1994. [ Links ]
45. Wellman SE, Sittman DB, Chaires JB. Preferential binding of H1e histone to GC-rich DNA. Biochemistry 1994; 33(1):384-8. [ Links ]
46. Phillips D, Clarke M. Behaviour of histones in exclusion chromatography and gel electrophoresis in relation to their molecular weights. Journal of Chromatography A 1970; 46:320-3. [ Links ]
47. Gorovsky MA, Pleger GL, Keevert JB, Johmann CA. Studies on histone fraction F2A1 in macro- and micronuclei of Tetrahymena pyriformis. The Journal of Cell Biology 1973; 57(3):773-81. [ Links ]
48. Lindner H, Helliger W, Puschendorf B. Separation of rat tissue histone H1 subtypes by reverse-phase h.p.l.c. Identification and assignment to a standard H1 nomenclature. The Biochemical Journal 1990; 269(2):359-63. [ Links ]
49. Lindner H, Sarg B, Helliger W. Application of hydrophilic-interaction liquid chromatography to the separation of phosphorylated H1 histones. Journal of Chromatography A 1997; 782(1):55-62. [ Links ]
50. Talasz H, Helliger W, Puschendorf B, Lindner H. In vivo phosphorylation of histone H1 variants during the cell cycle. Biochemistry 1996; 35(6):1761-7. [ Links ]
51. Khadake JR, Rao MR. DNA- and chromatin-condensing properties of rat testes H1a and H1t compared to those of rat liver H1bdec; H1t is a poor condenser of chromatin. Biochemistry 1995; 34(48):15792-801. [ Links ]
52. Pintar A, Chollet A, Bradshaw C, Chaffotte A, Cadieux C, Rooman MJ, et al. Conformational properties of four peptides corresponding to alpha-helical regions of Rhodospirillum cytochrome c2 and bovine calcium binding protein. Biochemistry 1994; 33(37):11158-73. [ Links ]
53. Prieto J, Serrano L. C-capping and helix stability: the Pro C-capping motif. Journal of Molecular Biology 1997; 274(2):276-88. [ Links ]
54. Morán F, Montero F, Azorín F, Suau P. Condensation of DNA by the C-terminal domain of histone H1. A circular dichroism study. Biophysical Chemistry 1985; 22(1-2):125-9.5 [ Links ]














