Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biosalud
Print version ISSN 1657-9550
Biosalud vol.15 no.1 Manizales Jan./June 2016
https://doi.org/10.17151/biosa.2016.15.1.5
DOI: 10.17151/biosa.2016.15.1.5
DETECTION OF PATHOGENIC Leptospira spp IN HOUSE MOUSE (Mus musculus) KIDNEY TISSUE IN SINCELEJO, COLOMBIA
Anaís Castellar M.1
Plácido Arrieta B.2
Libardo Caraballo B.3
María Paulina Torres P.4
Linda Ríos M.5
1 Magíster en Microbiología Tropical. Universidad de Córdoba. Docente Investigador, Universidad de Sucre - Grupo Investigaciones Biomédicas. Sincelejo, Colombia. Correo: anaiscastellar@gmail.com ORCID: 0000-0001-6383-8562
2 Biólogo. Estudiante de Maestría en Ciencias Ambientales, SUE-CARIBE - Grupo Investigaciones Biomédicas, Universidad de Sucre. Sincelejo, Colombia. Correo electrónico: arrietabo84@gmail.com ORCID: 0000-0003-2148-3024
3 Biólogo. Estudiante de Maestría en Biotecnología, Universidad de Córdoba. Montería, Colombia - Grupo Investigaciones Biomédicas, Universidad de Sucre. Correo electrónico: blanenr@gmail.com ORCID: 0000-0002-5850-8910
4 Bióloga. Estudiante de Maestría en Ciencias Ambientales, SUE-CARIBE - Grupo de investigación CRHIA, Universidad de Sucre. Sincelejo, Colombia. Correo electrónico: mria-paulina17@hotmail.com ORCID: 0000-0002-0043-5544
5 Bióloga. Estudiante de Maestría en Ciencias Ambientales, SUE-CARIBE - Grupo investigación Biología de los Microorganismos, Universidad de Sucre. Sincelejo, Colombia. Correo electrónico: lindaestefania22@hotmail.com ORCID: 0000-0001-9829-9992
La leptospirosis es una enfermedad zoonótica de distribución mundial, con mayor frecuencia en los países tropicales. Algunos roedores, y entre ellos Mus musculus o ratón casero, son portadores crónicos asintomáticos de Leptospira spp. patógenas, debido a que alojan la bacteria en sus riñones y la diseminan al ambiente a través de la orina. El presente trabajo tuvo como objetivo detectar molecularmente Leptospira spp. patógenas en riñones extraídos de M. musculus en el municipio de Sincelejo (Sucre, Colombia). La captura de los roedores se realizó en el área urbana del municipio; se instalaron trampas tipo Sherman® intra y peridomicilio en el segundo periodo de 2010 y el primero de 2011 por un lapso de 14 y 15 horas. Durante la necropsia se retiró los riñones de los individuos para la extracción de ADN y análisis de PCR para la amplificación del segmento génico de 423pb entre las posiciones 270 y 692 de la región codificante LipL32. Se capturaron 154 individuos, de los cuales 5 fueron positivos para Leptospira spp. patógenas. El análisis de correspondencias mostró que los ratones positivos se encuentran mayoritariamente en la comuna 2, seguido por la comuna 4 del municipio. El estudio sugiere que el saneamiento básico establece ambientes propicios para que se presente el proceso epidemiológico de la bacteria, y se demuestra la circulación de Leptospira spp. patógenas en M. musculus en el municipio de Sincelejo. Por lo que se infiere que la infestación de roedores en la zona de estudio es fuente potencial de transmisión de Leptospiras patógenas para el ser humano, que es susceptible a todas las especies patógenas del género.
Palabras clave: leptospirosis, Leptospira, Mus musculus, PCR, LipL32.
Leptospirosis is a zoonotic disease of worldwide distribution with more frequency in tropical countries. Some rodents and among them the Mus musculus or house mouse are asymptomatic chronic carriers of pathogenic Leptospira spp because they have them in their kidneys and spread it to the environment through the urine. This study aimed to detect Leptospira spp molecularly in kidneys removed from M. musculus in the municipality of Sincelejo (Sucre, Colombia). The capture of rodents was conducted in the urban area of the city: intra and peridomiciliar Sherman® traps were installed in the second quarter of 2010 and the first quarter of 2011 for 14 to 15 hours. During the necropsy the kidneys of individuals were removed for DNA extraction and PCR analysis for the amplification of the 423 pb gene segment between positions 270 and 692 of the coding region LipL32. A total of 154 individuals were captured, of which five were positive for pathogenic Leptospira spp. The correlation analysis showed that mice are mostly positive in commune 2, followed by commune 4 in the municipality. The study suggests that basic sanitation establishes favorable environments for the epidemiological process of the bacteria and the circulation of pathogenic Leptospira spp. in M. musculus is demonstrated in the municipality of Sincelejo. It is inferred that rodent infestation in the studied area is a potential source for the transmission of pathogenic Leptospira spp. to human beings, which is susceptible to all pathogenic species of the genus.
Key words: leptospirosis, Leptospira, Mus musculus, PCR, LipL32.
La leptospirosis es la enfermedad infecciosa con mayor distribución en el mundo (1, 2), con una mayor ocurrencia en las regiones tropicales (3). Esta es causada por espiroquetas patógenas del género Leptospira. La bacteria infecta tanto animales domésticos como silvestres, aunque también se ha aislado de otros vertebrados como aves y reptiles. Usualmente, es transmitida a humanos por contacto directo con orina de animales infectados a través de abrasiones en la piel y por contacto indirecto con agua y suelos contaminados (4). Dentro de los animales sinantrópicos, los roedores son animales significativos en la epidemiologia de la enfermedad (5, 6), y entre ellos M. musculus es considerado huésped o reservorio de mantenimiento de la bacteria, debido a que puede albergar en la superficie de las células epiteliales de los túbulos proximales de los riñones bacterias patógenas del género Leptospira sin presentar sintomatología alguna y, de esta manera, diseminar la bacteria a través de la orina durante todo el ciclo de vida del animal (3). En Colombia, Agudelo-Flórez et al. (6) reportaron la circulación de Leptospira spp., en Rattus norvegicus naturalmente expuesta en una zona urbana de Colombia, encontrando que el 25,2% de los roedores capturados no solo tienen seropositividad para la serovariedad Icterohaemorrhagiae, sino que también lo están otras serovariedades como Grippotyphosa y Canicola. Asimismo, Agudelo et al (7) encontraron que el 23% (12/52) de las Rattus norvegicus capturadas fueron positivas por cultivo y el 20% (52/254) fueron positivas para Leptospira patógenas por PCR en la ciudad de Medellín. En el departamento de Sucre hay pocos estudios relacionados con la leptospirosis, solo se tiene un estudio realizado por Ríos et al (8) en el que reportaron una alta seroprevalencia de Leptospira spp. en trabajadores rurales. Además, el Sistema Nacional de Vigilancia en Salud Pública –SIVIGILA– indica que entre los años 2007-2015 Sucre reportó 274 casos, de los cuales 37 fueron notificados por el municipio de Sincelejo, lo que demuestra la circulación de Leptospiras patógenas en esta parte del país. Por lo anterior, el objetivo de este estudio fue detectar molecularmente Leptospiras patógenas en riñones extraídos del ratón casero (Mus musculus) en el municipio de Sincelejo (Sucre, Colombia).
Área de estudio y muestreo
Las capturas de los roedores se realizaron en el área urbana del municipio de Sincelejo, ubicado al noroeste de Colombia (9º18" N, 75º23" W), conformado por nueve comunas, entre el mes de julio de 2010 y febrero de 2011, teniendo en cuenta como criterios de inclusión los barrios de cada comuna con saneamiento básico precario (cercanías a arroyos, zonas enmontadas y botaderos improvisados de basura) favorables para la proliferación de Mus musculus y transmisión de las Leptospira spp. (9), y de exclusión aquellos barrios con saneamiento básico satisfactorio. Se utilizaron 50 trampas de captura viva tipo Sherman® por comuna (1-9), cebadas con maíz molido y atún e instaladas intra y peridomicilio en las horas de la tarde y recogidas en las horas de la mañana del día siguiente por un periodo de 14 y 16 horas con un éxito de trampeo de 26, 38, 20, 38, 44, 38, 32, 38, 34% por cada comuna. Durante el desarrollo de este estudio se tuvieron en cuenta los criterios propuestos por Romero et al. (10) Los procedimientos de manejo y disección de los animales fueron aprobados por el Comité Institucional de Ética de la Universidad de Sucre.
Captura y necropsia de los ratones capturadosSe capturaron 154 M. musculus que fueron trasladados en bolsas plásticas al Laboratorio de Investigaciones Biomédicas de la Universidad de Sucre, donde se llevó a cabo su disección de acuerdo a los protocolos de bioseguridad sugeridos por la CDC (11). Se determinaron datos morfométricos como largo total, largo de la cola, largo del cuerpo, largo del miembro posterior derecho y largo de la oreja de cada individuo, posteriormente fueron anestesiados con cloroformo y sacrificados por dislocación cervical (10, 12). Durante la necropsia se desinfectó la zona ventral con alcohol al 70% y se procedió a la extracción de los riñones (10, 13), y se determinó la especie con las claves genéricas para roedores del Nuevo Mundo (14, 15).
Extracción de ADNA partir de la muestra de tejido renal se extrajo ADN total, aplicando el método de altas concentraciones de sales propuesto por Watts (16, 17).
Amplificación por PCRLa PCR se llevó a cabo mediante cebadores previamente descritos por Levett et al. (18), los cuales amplifican una región de 423 pb entre las posiciones 270 y 692 del gen que codifica para la lipoproteína de membrana externa LipL32, altamente conservada en especies patógenas del genero Leptospira. Para el proceso de PCR se usó un volumen final de reacción de 25 ul, 3 mM de MgCl2, 0,25 mM de dNTP's, 0,1 uM de ambos cebadores, 1X de buffer de PCR, 1U/ul de taq polimerasa y entre 10-150 ng/ul de ADN. Un control positivo y uno negativo fue usado en cada reacción de PCR; para el control positivo se utilizó ADN de Leptospira interrogans serovar Copenhageni serogrupo Icterohaemorrhagiae cebado con ADN de riñón de roedor negativo por MAT, y para el control negativo agua estéril. El perfil de amplificación fue el propuesto por Levett et al (18); Moreno y Agudelo Flórez (19) con modificaciones. Desnaturalización inicial a 95°C durante cinco min, seguido de una fase de 35 ciclos a 94°C durante un min, alineamiento a 55°C durante un min y extensión a 72°C durante dos min, posteriormente se llevó a cabo una extensión final a 72°C durante cinco min.
ElectroforesisLos productos de PCR se corrieron en gel de agarosa al 2% usando buffer TBE (Tris-base, Ácido Bórico, EDTA) 5X con un marcador de peso molecular (100 bp DNA Ladder; Invitrogen™). El revelado se hizo con GelStar. Posteriormente se efectuó el corrido electroforético a 90 V por 45 min y los productos se visualizaron en un fotodocumentador Quantum ST4 (Vilber Lourmat).
Secuenciación y análisis genéticoLos productos amplificados se purificaron con un Kit comercial (PureLinkTM Quick gel Extraction), atendiendo las recomendaciones del fabricante. Las muestras fueron enviadas a secuenciar a la empresa Macrogen (Seoul, South Korea). Los electroferogramas resultantes fueron editados y alineados con el programa MEGA 5 (19) para obtener una secuencia consenso por muestra. Estas, se alinearon con secuencias consignadas en GenBank usando la herramienta BlastN, para determinar mediante los criterios de máxima identidad, cobertura y puntuación total en el alineamiento múltiple, la identidad de las secuencias obtenidas a partir del ADN amplificado de tejido renal de roedores.
Análisis estadísticoLos datos de los roedores capturados fueron analizados mediante estadística descriptiva, obteniendo tasas y proporciones de lugar, sexo y PCR (+). Las variables: lugar, sexo y PCR (+), se sometieron a un análisis de correspondencia para determinar el grado de relación entre ellas con ayuda del software estadístico InfoStat versión estudiantil (20).
Captura y necropsia de los ratones capturados
Durante el estudio fueron capturados 154 roedores, que de acuerdo a la determinación taxonómica corresponden a la especie M. musculus, en las nueve comunas que conforman al municipio de Sincelejo, de los cuales el 55% fueron hembras y el 45% machos.
LipL32 – PCRSe logró detectar ADN de Leptospiras patógenas en 5 muestras de riñón de roedores de M. musculus, el producto de la PCR mostró un tamaño de 423 pb aproximadamente. Tras su separación en la electroforesis, se revelaron y se representaron para todas las bandas la movilidad (distancia avanzada o, mejor, cociente entre esta y el avance del frente de electroforesis) frente al logaritmo del tamaño (longitud en pb). Sobre la recta ajustada se interpolaron las distancias de avance de las muestras problema, calculando así su tamaño en pb (Figura 1).
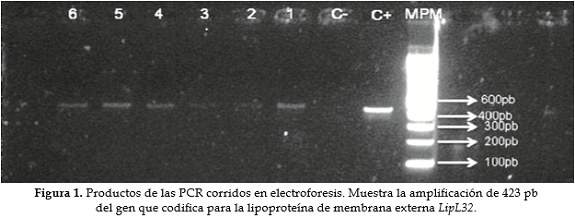
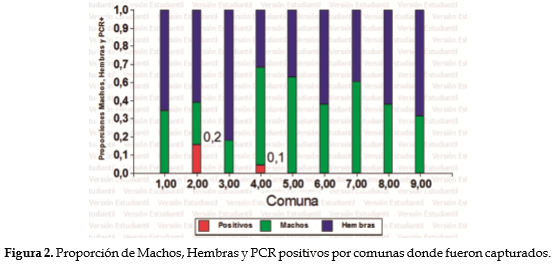
Después de identificar las muestras positivas por PCR, se realizó la determinación de proporciones de PCR positivos por comunas (Figura 2).
Secuenciación y alineamiento de los segmentos génicos obtenidos a partir de la PCR
Se obtuvieron en total 5 secuencias consenso, provenientes de muestras de riñón de M. musculus urbanos de Sincelejo. El alineamiento de estas secuencias mostró que 5 de estas presentaban mayor homología con Leptospira interrogans y que el primer nucleótido es homólogo al nucleótido ubicado en la posición 270 del gen LipL32 de L. interrogans consignado en GenBank con el número de acceso JQ013520. El análisis de alineamiento local con Blast, confirmó la identidad de las secuencias, mostrando una identidad de 100% con la especie Leptospira interrogans.
Análisis estadísticoDe los 5 roedores positivos para L. interrogans por PCR, la proporción de 0,2 correspondió a la comuna 2 y de 0,1 a la comuna 4 (Figura 2). El análisis de correspondencias (AC) (Figura 3) en su primer eje (con una inercia de 13,89%) mostró que los ratones (Mus musculus) positivos para Leptospira interrogans se encuentran mayoritariamente en la comuna 2 del municipio de Sincelejo, seguido de la comuna 4, y habitan predominantemente en las comunas 2 y 4.
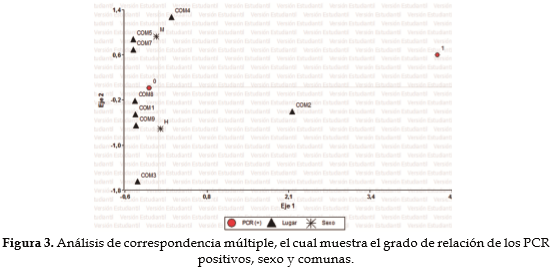
El porcentaje de detección de ADN de Leptospira interrogans fue de 3,25% (5/154). Los roedores que mostraron positividad por PCR se encontraron distribuidos en las comunas 2 y 4 con una proporción de infección de 0,2 y 0,1 (Figura 2) respectivamente. Posiblemente, porque estas comunas tienen las condiciones apropiadas como zonas enmontadas, residuos sólidos, casas en madera, condiciones de alcantarillados precarios, presencia o cercanía con arroyos de aguas negras (Figura 4) que podrían facilitar no solo el crecimiento poblacional de roedores, sino también la diseminación y transmisión de la bacteria (21-23). Por lo que, el saneamiento ambiental tiene un rol significativo en el proceso epidemiológico de Leptospira spp. patógenas y saprofitas, este proceso puede explicarse de acuerdo al modelo número dos propuesto por Fainé et al. (25) y Céspedes (26), donde señala que la transmisión ocurre en áreas tropicales donde hay serovares que infectan a humanos y animales, además hay un gran número de reservorios como los roedores y la exposición humana no está limitada a la ocupación sino a la contaminación ambiental y de acuerdo a las características ambientales de las comunas donde se detectó la presencia de L. interrogans en M. musculus presentan las tipologías dichas por los autores (Figura 4). Además, la relación entre los PCR (+) y las comunas 2 y 4 (inercia de 13,89%) podría deberse a las condiciones favorables (Figura 4) tanto para la bacteria causante de la enfermedad como para la proliferación de los ratones. Y como M. musculus, o ratón casero, hace parte de las tres especies de roedores con amplia distribución mundial, es asociado comúnmente con la infección y transmisión de Leptospira spp. patógena (24). Por lo que, este puede estar cumpliendo con la función de transmisión y diseminación de la bacteria causante de la leptospirosis en el municipio de Sincelejo.

La detección de ADN de Leptospira interrogans en tejido renal de estos ratones a través de la técnica PCR con el uso de los cebadores LipL32 ha demostrado ser eficiente, dado el grado de homología e identidad que presentó hacía Leptospira interrogans en el análisis de alineamiento, lo cual coincide con los estudios realizados por Moreno y Agudelo-Flórez (25), quienes afirman que la detección de ADN de Leptospira spp. patógenas por PCR con los cebadores LipL32 resulta ser bastante específica, debido a que no se puede detectar ADN de especies saprofitas por que estas no tienen gen ortólogo de LipL32. De igual forma, en otro estudio realizado por Agudelo et al (7) lograron detectar ADN de Leptospira spp. en Rattus norvegicus, indicando la especificidad de la PCR LipL32 en la detección de Leptospiras patógenas. Asimismo, el estudio realizado por Rosario et al. (28), quienes caracterizaron cepas patógenas del género Leptospira con el uso de estos cebadores. Estos antecedentes demuestran que los cebadores LipL32 son importantes a la hora de detectar y diferenciar entre Leptospiras patógenas y saprofitas (26-28), pero no es posible llegar al serovar debido a que se necesitan de otros estudios como los realizados por Jung et al. (32) y Romero-Vivas et al. (29, 30).
Los hallazgos en este trabajo son relevantes, debido a que se encontraron Leptospira spp. patógenas en tejido renal de M. musculus en el municipio de Sincelejo. Asimismo, el uso de métodos moleculares para detectar el marcador de virulencia LipL32 de Leptospira spp. patógenas en tejido renal de M. musculus. Además, es el primer reporte que se tiene para el departamento y municipio de la presencia de L. interrogans en el ratón casero M. musculus y estaría participando en la transmisión de la bacteria.
Agradecimientos a Virginia Rodríguez Rodríguez y a Germán Arrieta Bernate del Instituto de Investigaciones Biológicas del Trópico de la Universidad de Córdoba, por su colaboración en el suministro de las cepas utilizadas como controles positivos.
Parte de este trabajo fue financiado con recursos del Departamento de Investigación de la Universidad de Sucre (DIUS). Resolución número 30 de 2012.
1. Bharti AR, Nally JE, Ricaldi JN, Matthias MA, Diaz MM, Lovett MA, et al. Leptospirosis: a zoonotic disease of global importance. The Lancet infectious diseases. 2003;3(12):757-71. [ Links ]
2. Fornazari F, da Silva RC, Richini-Pereira VB, Beserra HEO, Luvizotto MCR, Langoni H. Comparison of conventional PCR, quantitative PCR, bacteriological culture and the Warthin Starry technique to detect Leptospira spp. in kidney and liver samples from naturally infected sheep from Brazil. Journal of microbiological methods. 2012;90(3):321-6. [ Links ]
3. Sacsaquispe R, Glenny M, Céspedes M. Estudio preliminar de leptospirosis en roedores y canes en salitral, Piura-1999. Revista Peruana de Medicina Experimental y Salud Pública. 2003;20(1):39-40. [ Links ]
4. Lomar AV, Diament D, Torres JR. Leptospirosis in Latin America. Infectious disease clinics of North America. 2000;14(1):23-39. [ Links ]
5. Collares-Pereira M, Mathias M, Santos-Reis M, Ramalhinho M, Duarte-Rodrigues P. Rodents and Leptospira transmission risk in Terceira island (Azores). European journal of epidemiology. 2000;16(12):1151-7. [ Links ]
6. Agudelo-Flórez P, Arango JC, Merizalde E, Londoño AF, Quiroz VH, Rodas JD. Evidencia serológica de circulación de Leptospira spp en Rattus norvegicus naturalmente expuestos en una zona urbana colombiana. Revista de Salud Pública. 2010;12(6):990-9. [ Links ]
7. Agudelo-Flórez P, Londoño AF, Quiroz VH, Ángel JC, Moreno N, Loaiza ET, et al. Prevalence of Leptospira spp. in urban rodents from a groceries trade center of Medellin, Colombia. The American journal of tropical medicine and hygiene. 2009;81(5):906-10. [ Links ]
8. Ríos R, Franco S, Mattar S, Urrea M, Tique V. Seroprevalence of Leptospira sp., Rickettsia sp. and Ehrlichia sp. in rural workers of Sucre, Colombia. Infectio. 2008;12(2):90-5. [ Links ]
9. Ferro BE, Rodriguez AL, Perez M, Travi BL. [Seroprevalence of Leptospira infection in habitants of peripheral neighborhoods in Cali, Colombia]. Biomedica: revista del Instituto Nacional de Salud. 2006;26(2):250-7. [ Links ]
10. Romero Almaraz MdL, Sánchez Hernández C, García Estrada C, Owen RD. Mamíferos pequeños. Manual de técnicas de captura, preparación, preservación y estudio: Las Prensas de Ciencias. Coordinación de Servicios Editoriales, Facultad de Ciencias, UNAM; 2007. [ Links ]
11. Mills JN, Childs JE, Ksiazek TG, Peters C, Velleca WM. Methods for trapping and sampling small mammals for virologic testing: US Department of Health and Human Services, Public Health Service, Centres for Disease Control and Prevention; 1995. [ Links ]
12. Natarajaseenivasan K, Vijayachari P, Sharma S, Sugunan AP, Vedhagiri K, Selvin J, et al. FlaB PCRbased identification of pathogenic Leptospiral isolates. Journal of Microbiology, Immunology and Infection. 2010;43(1):62-9. [ Links ]
13. Marder G, Ruiz R, LM-Zorzo L, Merino D. Detección de Leptospiras en riñon de roedores de la ciudad de Corrientes: estudio preliminar. [ Links ]
14. Emmons L, Feer F. Neotropical rainforest mammals: a field guide. 1997. [ Links ]
15. Nowak RM. Walker's Mammals of the World: JHU Press; 1999. [ Links ]
16. Watts P. Extraction of DNA from tissue: High salt method. Liverpool. School of Biological Sciences. 2001; Version 1.0: 5. [ Links ]
17. Lopera-Barrero NM, Povh JA, Ribeiro RP, Gomes PC, Jacometo CB, Silva Lopes Td. Comparación de protocolos de extracción de ADN con muestras de aleta y larva de peces: extracción modificada con cloruro de sodio. Ciencia e investigación agraria. 2008;35(1):77-86. [ Links ]
18. Levett PN, Morey RE, Galloway RL, Turner DE, Steigerwalt AG, Mayer LW. Detection of pathogenic leptospires by real-time quantitative PCR. Journal of Medical Microbiology. 2005;54(1):45-9. [ Links ]
19. Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: Molecular Evolutionary Genetics Analysis Using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Molecular biology and evolution. 2011;28(10):2731-9. [ Links ]
20. Di Rienzo JAC, F. Balzarini, M. G. Gonzalez, L. Tablada, M. Robledo, C. W. InfoStat. 2011. [ Links ]
21. Johnson MA, Smith H, Joseph P, Gilman RH, Bautista CT, Campos KJ, et al. Environmental exposure and leptospirosis, Peru. Emerging infectious diseases. 2004;10(6). [ Links ]
22. Donaires LF, Céspedes MJ, Sihuincha MG, Pachas PE. Determinantes ambientales y sociales para la reemergencia de la leptospirosis en la región amazónica del Perú, 2012. Revista Peruana de Medicina Experimental y Salud Pública. 2012;29(2):280-4. [ Links ]
23. Nardone A, Capek I, Baranton G, Campese C, Postic D, Vaillant V, et al. Risk factors for leptospirosis in metropolitan France: results of a national case-control study, 1999-2000. Clinical Infectious Diseases. 2004;39(5):751-3. [ Links ]
24. Houemenou G, Ahmed A, Libois R, Hartskeerl R. Leptospira spp. prevalence in small mammal populations in Cotonou, Benin. ISRN Epidemiology. 2013;2013. [ Links ]
25. Moreno N, Agudelo-Flórez P. Aplicación de las pruebas de PCR convencional simple y múltiple para la identificación de aislamientos de Leptospira spp. en Colombia. Revista Peruana de Medicina Experimental y Salud Pública. 2010;27(4):548-56. [ Links ]
26. Guerreiro H, Croda J, Flannery B, Mazel M, Matsunaga J, Reis MG, et al. Leptospiral proteins recognized during the humoral immune response to leptospirosis in humans. Infection and immunity. 2001;69(8):4958-68. [ Links ]
27. Haake DA, Matsunaga J. Characterization of the Leptospiral outer membrane and description of three novel Leptospiral membrane proteins. Infection and immunity. 2002;70(9):4936-45. [ Links ]
28. Zhang X-Y, Yu Y, He P, Zhang Y-X, Hu B-Y, Yang Y, et al. Expression and comparative analysis of genes encoding outer membrane proteins LipL21, LipL32 and OmpL1 in epidemic leptospires. Acta biochimica et biophysica Sinica. 2005;37(10):649-56. [ Links ]
29. Jung LRC, Bomfim MRQ, Kroon EG, Nunes ÁC. Identification of Leptospira serovars by RFLP of the RNA polymerase beta subunit gene (rpoB). Brazilian Journal of Microbiology. 2015;46(2):465-76. [ Links ]
30. Romero-Vivas CM, Thiry D, Rodríguez V, Calderón A, Arrieta G, Máttar S, et al. Molecular serovar characterization of Leptospira isolates from animals and water in Colombia. Biomédica. 2013;33:179-84. [ Links ]
Como citar este artículo: Castellar A, Arrieta P, Caraballo L, Torres MP, Ríos L. Detección de Leptospiras patógenas en tejido renal de ratón casero (Mus musculus) en Sincelejo, Colombia. Revista Biosalud 2016; 15(1):41-49. DOI: 10.17151/biosa.2016.15.1.5




![9-[(E)-2-phenylethenyl]anthracene AND 9-[(E)-2-(naphthalen-2-yl)ethenyl] anthracene AS TRAPS FOR SINGLET OXYGEN: PHOTOSENSITIZED OXIDATION AND PHOTODYNAMIC EFFECT ON Leishmania tarentolae PARASITES](/img/en/prev.gif)









