INTRODUCCIÓN
El género Mycobacterium está conformado por más de 170 especies. Las micobacterias no tuberculosas ―MNT― incluyen aquellas micobacterias que no son miembros del complejo Mycobacterium tuberculosis. En 1950, los investigadores Timpe y Runyon propusieron una clasificación útil para el género Mycobacterium. De acuerdo a su velocidad de crecimiento y producción de pigmento en la oscuridad o por exposición a la luz se clasifican en: fotocromógenos, crecimiento abundante de los cultivos que producen pigmentos (cristales amarillos de caroteno por exposición a la luz, aunque no lo generan en la oscuridad); escotocromógenos, los organismos desarrollan un pigmento amarillo brillante en la luz y en la oscuridad; no fotocromógenos, que incluyen un número de especies que producen pequeñas cantidades de pigmento amarillo pálido (sin embargo si son expuestas a la luz brillante no intensifican su coloración); finalmente las micobacterias de crecimiento rápido, según los grupos de Runyon.
Las especies de micobacterias incluyen especies patógenas y no patógenas que pueden ser divididas en tres grupos con base en su significancia clínica 1.
El primer grupo incluye “patógenos obligados” en humanos y animales, por ejemplo: Mycobacterium tuberculosis complex (M. africanum, M. bovis, M. canetti, M. caprae, M. microti, M. pinnipedii y M. mungi); M. leprae; M. lepraemurium y avium paratuberculosis; generalmente estos son patógenos estrictos cuya gran mayoría no suelen ser hallados en el medio ambiente.
El segundo grupo de micobacterias consiste en “especies potencialmente patógenas a humanos y animales”, estas son: M. ulcerans; M. leprae y M. lepraemurium; las cuales generalmente son halladas en el ambiente. Recientes investigaciones han demostrado la presencia de M. leprae en el suelo y en alguna asociación con protozoos 2,3. De igual manera pueden llegar a ser patógenas bajo determinadas condiciones; por esta razón son llamadas “micobacterias oportunistas” o “patógenos ocasionales” para distinguirlas de los “patógenos obligados”, algunas de ellas son excepcionales (como M. ulcerans y M. malmoense) puesto que podrían estar en ambos grupos.
El tercer grupo son especies saprófitas, no patógenas o solo “excepcionalmente patógenas”, a estas pertenecen las micobacterias ambientales o también llamadas MNT en las que se incluyen: las micobacterias de crecimiento lento y rápido; las micobacterias de crecimiento lento son formadoras de colonias con más de 7 días de incubación y micobacterias de crecimiento rápido en menos de 7 días. Muchas especies exhiben gran variedad en su tiempo de generación entre 2 y 48 horas 4,5.
La identificación de bacterias convencionales está bien establecida en los laboratorios clínicos, sin embargo para la tipificación de MNT el personal del laboratorio clínico debe estar familiarizado con esa especie de bacterias. El reconocimiento tradicional de las MNT se basa en el análisis de ciertas características fenotípicas (biológicas y bioquímicas) tales como la velocidad de crecimiento, la producción de pigmento y la actividad de ciertas enzimas. Muchas de esas técnicas requieren de algunas semanas, por lo que no es posible en determinados casos la diferenciación entre los miembros pertenecientes a un mismo complejo de micobacterias. Las pruebas bioquímicas para MNT son muy amplias y muchas veces tediosas, pero algunas de ellas son relativamente sencillas y permiten la identificación de especies. A mediados de los años 90 se identificaron nuevos requerimientos para la identificación de micobacterias de crecimiento lento; dentro de estos requisitos se encontró un número limitado de pruebas bioquímicas que, además de la aplicación de técnicas moleculares, permitían obtener la identificación de micobacterias. Las pruebas como niacina positivas, reducción de nitritos y la prueba de catalasa termolábil, son útiles para el complejo tuberculosis. Las pruebas bioquímicas son de utilidad para la identificación de algunas especies, sin embargo suelen ser incompletas debido a la falta de disponibilidad de un número suficiente de ensayos discriminatorios para identificar todas las especies; siendo las herramientas moleculares las más apropiadas y rápidas. Dentro del genoma de las micobacterias existen regiones bien conservadas de ADN, específicas de género que flanquean regiones hipervariables determinadas de especie. Las regiones más empleadas y amplificadas en tuberculosis han sido las regiones de la subunidad ribosómica IS6110 y 16S rRNA. Existen otras regiones empleadas para la tipificación como son: rpoB que codifica la subunidad β de RNA polimerasa; el recAgen y el gen hsp65 que codifica una proteína en las micobacterias de 65KDa. Esta es una proteína de choque térmico: 65kDa contiene epítopes que son comunes a diversas especies de micobacterias. Por tanto, es viable el uso de la secuenciación del gen hsp65 para identificar micobacterias y analizar su variabilidad genética. La secuenciación hsp65 tiene el potencial de ser preciso, confiable y eficaz para la identificación de Mycobacterium de crecimiento rápido aisladas de muestras clínicas y del medio ambiente 6.
Con base en ello se emplearon algunas de estas herramientas moleculares para establecer la identidad de las especies de micobacterias aisladas. En Perú hay pocos estudios sobre las micobacterias ambientales, por lo que rara vez son reportadas. El objetivo de este estudio fue aislar y caracterizar molecularmente MNT del sistema de distribución de agua en un hospital de Lima.
MATERIALES Y MÉTODOS
El estudio se llevó a cabo en un hospital oncológico de Lima entre 2003-2004. Sin embargo los análisis de secuenciación, filogenia y el manuscrito se realizaron en 2014.
Recolección de agua del hospital
Las muestras de agua se recolectaron en diferentes áreas del hospital, el cual se ubica en Lima Este; en general se tomaron 37 muestras de agua divididas en 5 ensayos. Se obtuvieron las muestras en el séptimo piso en el área de pediatría (neutropenia, zona de infectados y procedimientos oncológicos especiales). Se prosiguió en el sexto y quinto piso: clínica. En el cuarto piso, en el área de la unidad de cuidados intensivos ―UCI―. En el tercer piso servicio de protección al inmuno neutropénico ―SEPIN―. Igualmente en el comedor que se encuentra en el sótano y finalmente en el primer piso en el pozo de distribución de agua que abastece todo el hospital.
La recolección de la muestra se realizó en frascos de vidrio estériles de tapa rosca, los cuales fueron lavados y autoclavados a una presión de 15 mmHg a 121 °C por 15 minutos. Se procedió a tomar las muestras de agua de cada piso del hospital, el volumen de agua fue entre 800 ml y 900 ml tanto para agua fría (-23 °C) como para agua caliente (>50 °C); inmediatamente obtenidas las muestras fueron procesadas. En el caso de la muestra de agua caliente, esta se dejó enfriar para ser filtrada después.
Filtrado de muestras de agua y detección de micobacterias
Se empleó el método del filtrado al vacío, el cual permitió retener los microorganismos que se deseaban aislar. Entre 800-900 ml de agua fueron tratados a través de un filtro Millipore 0,45 µm de diámetro; se procedió con una cuidadosa limpieza del equipo de filtrado al vacío con agua destilada estéril y alcohol (de 70°) antes y después de filtrar cada muestra de agua. Luego de pasar el agua por la membrana, esta se colocó en placas de Petri conteniendo medio de cultivo Ogawa; estas fueron incubadas a 37 °C y 42 °C por 10 días para las muestras de agua fría y de 15 a 20 días mínimo para las muestras de agua caliente.
Cultivo de las muestras
El medio de cultivo empleado fue el Ogawa, a base de huevo, usado para el aislamiento primario. El medio se coaguló en placas de Petri, lo que permitió colocar la membrana en el centro de la placa. Se realizaron 5 ensayos.
Primer ensayo: el aislamiento de micobacterias del agua fría se incubó a 37 °C con adición de antibióticos (dicloxacilina 40µg/ml, cefalexina 40µg/ml) en condiciones de aerobiosis.
Segundo ensayo: el aislamiento de micobacterias del agua caliente se incubó a 37 °C y 42 °C con adición de antibióticos (dicloxacilina 1mg/ml, cefalexina 1mg/ml, fluconazol 1 mg/ml) en condiciones de microaerofília; se incubaron por un mes.
Tercer ensayo: el aislamiento de micobacterias del agua caliente se incubó a 37 °C y 42 °C con adición de antibióticos (dicloxacilina 1mg/ml, cefalexina 1mg/ml, fluconazol 1mg/ml) en condiciones de aerobiosis.
Cuarto ensayo: sembrado y aislamiento de micobacterias del agua caliente incubadas a 37 °C y 42 °C en condiciones de aerobiosis, sin antibióticos.
Quinto ensayo: repetición del primer ensayo con el agua fría en el que se incubó a 37 °C en condiciones aeróbicas, sin antibióticos.
Las membranas se colocaron en la placa de Petri luego de realizar una impronta a un lado de la misma placa, esto para observar las características de las colonias que pudieran crecer.
Extracción de ADN
Se tomó una asada de la cepa y se suspendió en 400 µL de TE 1x, se incubó a 80 °C por 20-30 minutos para inactivarla. Después de su enfriamiento se agregó 50 µL de lisozima (50mg/ml), que fue agitado en vórtex, luego incubado a 37 °C por al menos 1 hora en movimiento o 12 horas en reposo. Se agregó 70 µL de SDS 10 % y 5 µL de proteinasa K (20mg/ml), se pasó por el vórtex y se incubó a 65 °C por 10 minutos, se agregó 100 µL de NaCl 5M y 100 µL de CTAB/NaCl solución. Se pasó por el vórtex hasta que el líquido estuvo lechoso y se incubó a 65 °C por 10 minutos. Se agregó 750 µL de cloroformo/alcohol isoamílico 24:1. Se pasó por el vórtex por 10 s y se centrifugó a temperatura ambiente por 5 minutos a 14,000 g, transfiriéndose el sobrenadante a un nuevo tubo con 0,6 de volumen (450 µL de isopropanol), se incubó a -20 °C por 30 minutos y se centrifugó 15 minutos a 14,000 g; el sedimento se volvió a suspender en 1 ml de etanol al 70 % y se centrifugó a 14,000 g por 5 minutos, se decantó el sobrenadante y el sedimento se dejó secar 20 minutos y luego se añadió 30-40 µL TE y se refrigeró 4 °C o -20 °C.
Análisis bioquímico
Los análisis bioquímicos fueron realizados por el Instituto Nacional de Salud ―INS― de Lima (Perú) y algunas cepas por el Instituto de Medicina Tropical ―IMT― de Amberes (Bélgica). Las pruebas bioquímicas fueron básicamente: coloración de Ziehl-Neelsen; temperatura de crecimiento (37-42 °C); velocidad de crecimiento (días); morfología de las colonias y la producción de pigmento; así como métodos bioquímicos tales como niacina, nitrato, fosfatos, urea y algunas enzimáticas como la catalasa; basados en el manual de Kent y Kubica de 1985 7.
Análisis del genotipado molecular
El genotipado de cepas se llevó a cabo en el Instituto de Medicina Tropical Alexander von Humboldt ― IMT-AvH― (Perú) y el IMT de Amberes Bélgica por el laboratorio Unidad de Tuberculosis. Las cepas fueron analizadas amplificando el gen 16S rRNA, en este caso el cebador empleado fue el directo (forward) pA: (5’-AGAGTTTGATCCTGGCTCAG-3’) 8,9. También realizamos el análisis de secuenciación del gen hsp65 para la diferenciación de especies de micobacterias, en donde se amplificó una secuencia parcial de 441 aproximadamente. Se empleó el cebador directo (forward) Tb11 (5’-ACCAACGATGTGTGTCCAT-3’) y el cebador reverse Tb12 (TTGTCGAACCGCATACCCT) (6, 7). Para la amplificación de los productos de PCR se empleó el siguiente protocolo: 1 ciclo de 95 °C durante 5 minutos; 30 ciclos de 94 °C durante 1 minuto, 65 °C durante 1 minuto, 72 °C durante 1 minuto y 1 ciclo de 72 °C durante 7 minutos; como describe el manual de Leao et al. 10.
Los estudios de secuenciación fueron realizados con un analizador automatizado (Applied Biosystems 3130 Series Genetic Analyzers).
Análisis filogenético
Todas las secuencias obtenidas fueron analizadas empleando el software MEGA versión 6.0. El análisis de alineación fue con CLUSTALW empleando el software MEGA 6. Para el análisis del árbol filogenético fue empleado el método neighbor-joining con el modelo de 2-parámetros de Kimura y con 1,000 Bootstrap de replicaciones.
RESULTADOS
En total se analizaron 37 muestras de agua potable del hospital. Fueron divididas en 5 ensayos individuales. Solo se detectaron 16/37 bacilos ácido alcohol resistentes, es decir el 43,2 %. Las 16 (100 %) cepas fueron aisladas en agua fría, ninguna en agua caliente. La temperatura del agua caliente del hospital alcanzó un promedio de 73 °C y un pH entre 7,2-7,4. (Tabla 1).
Tabla 1 Características fisicoquímicas de las muestras de agua en el hospital.
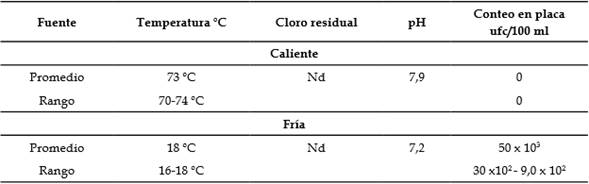
Nd: no detectado; ufc: unidades formadoras de colonia.
Fuente: elaboración propia.
Las muestras de agua contenían más de dos especies de micobacterias. La identificación molecular fue llevada a cabo por el IMT-AvH y por el Laboratorio Nacional de Referencia e Investigaciones en Tuberculosis (Bélgica) (Tabla 2). Todas las colonias se desarrollaron dentro de 5 a 7 días.
Tabla 2 Identificación de las cepas de micobacterias no tuberculosas aisladas en agua potable del hospital.
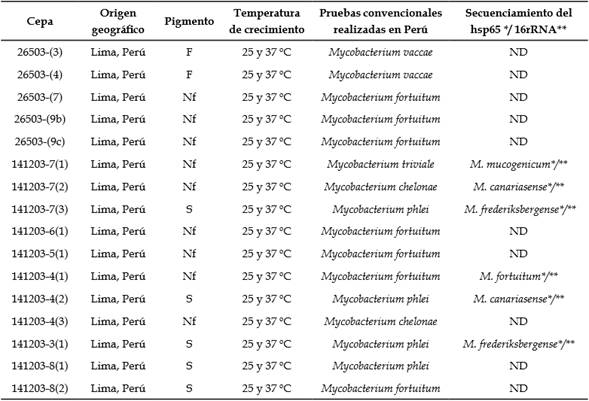
Hsp65= proteína de shock térmico; ND= no determinado; F= fotocromógeno; S= escotocromógeno; Nf = no fotocromógeno. Todas las cepas crecieron a 37 °C en incubación, pero también a temperatura ambiente 25 °C.
Fuente: elaboración propia.
La identificación bioquímica fue llevada a cabo por el INS y el IMT (Bélgica). Todas las colonias aisladas crecieron en medio Ogawa y a temperatura de 37 °C. El rango de acidez fue registrado en agua fría con pH 7,2 y para el agua caliente de 7,4 (Tabla 1).
En el ensayo 1 el volumen de agua filtrada fue de 840 ml y 990 ml, recolectado a las 11:00 a. m. En estas muestras se identificó bacilos ácido alcohol resistentes (9) 100 %. Las cepas crecieron a los 5 días de incubación a 37 °C (Figuras 1, 2).
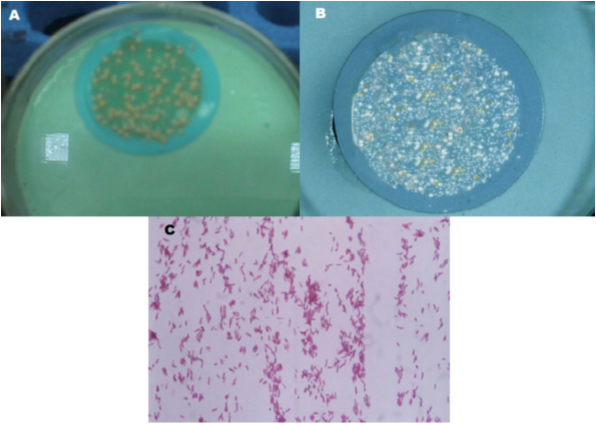
Figura 1 A y B) colonias de micobacterias aisladas del agua potable del grifo del hospital y crecidas en medio Ogawa empelando una membrana de 0.22 µ de poro. C) Coloración de Ziehl- Neelsen.
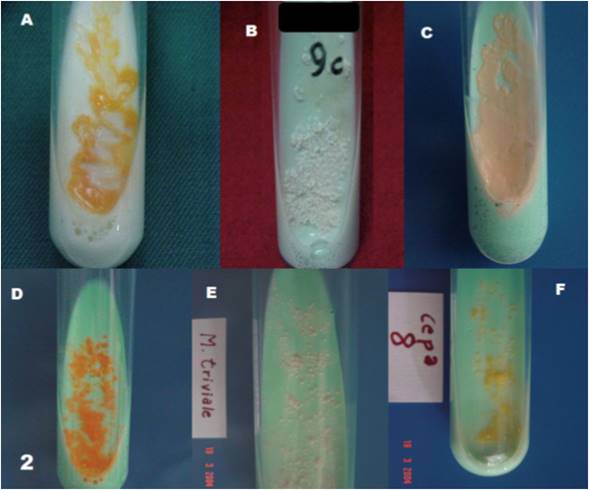
Figura 2 Cepas de Micobacterias aisladas en medio Ogawa, A) Mycobacterium frederickbergense 4to piso B) M fortuitum C) Mycobacterium chelonae complejo aislado de UCI. D) Mycobacterium frederickbergense aislado 7mo piso. E) Mycobacterium fortuitum. y F) Mycobacterium frederickbergense aislada del comedor.
En la muestra 5 se observó que la morfología de los bacilos era más ancha y larga, bajo la tinción de Ziehl-Neelsen, respecto a las demás muestras estudiadas. Luego de 11 días de crecida la colonia se observó que esta se tornó de color anaranjado. En la muestra 9 se identificaron 3 colonias: 1 colonia amarilla, 1 colonia amarilla pálida y 1 colonia blanca y rugosa. En general, solo se tomó una colonia en representación de cada placa de Ogawa. Con la excepción de la muestra 9, en donde se tomaron las 3 colonias diferentes (9a, 9b, 9c).
En los ensayos 2, 3 y 4, no se aisló ningún microorganismo en estos experimentos.
En el ensayo 5, por su parte, se identificó bajo la tinción de Ziehl-Neelsen la presencia de bacilos ácido alcohol resistentes en (9) 100 % de las muestras de agua. Las cepas aisladas de las muestras de agua crecieron dentro de los 5 días de incubación a 37 °C (Figura 3).
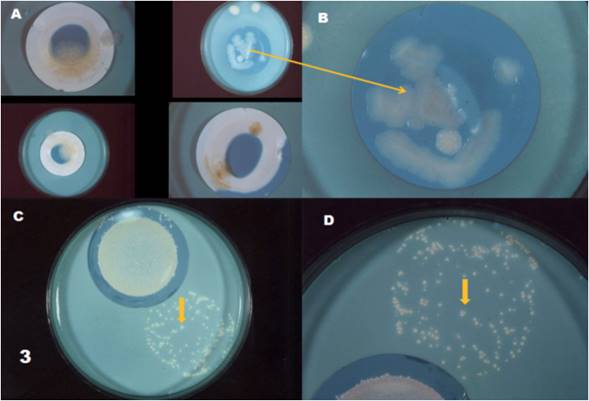
Figura 3 A, B) Muestras de agua incubadas a 42 °C en las que se observa deshidratación después de un mes de incubación y en una placa crecimiento de Bacillus sp. B) Ampliación de la imagen donde se indica con la flecha amarilla. C y D) Colonias de Micobacteria aislada de agua del pozo del hospital (fuente de ingreso del agua al hospital).
Las especies identificadas en el ensayo 1, por pruebas fenotípicas, fueron: 3 cepas de M. fortuitum y 2 cepas de M. vaccae (Tablas 2, 3). En el ensayo 5, fueron: M. chelonae 2/16 (12,5 %); M. triviale 1/16 (6,25 %); M. phlei 4/16 (25 %); M. vaccae 2/16 (12,5 %) y M. fortuitum 7/16 (43,75 %) (Tablas 3, 4).
Tabla 3 Identificación bioquímica de micobacterias aisladas de agua del hospital de Lima.
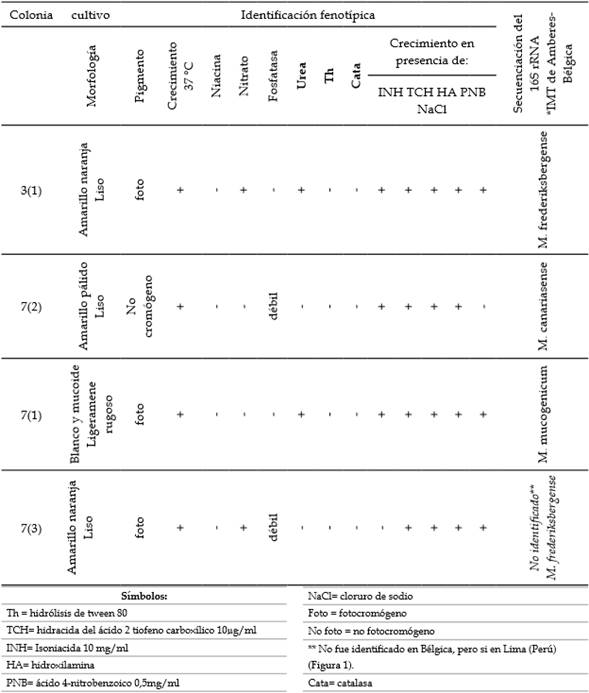
Fuente: elaboración propia.
Tabla 4 Hallazgos microbiológicos del 1 y 5 ensayo.
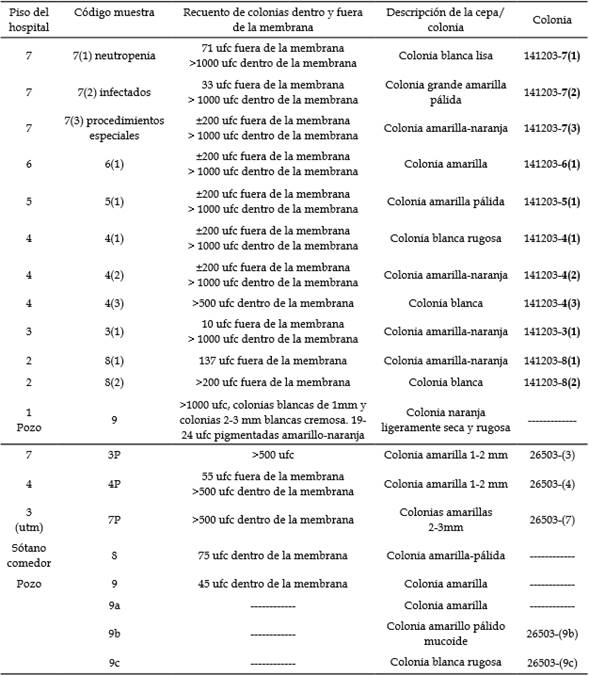
ufc= unidad formadora de colonia; utm= unidad de trasplante de médula.
Fuente: elaboración propia.
Identificación por biología molecular
El 7/16, es decir el 38 % de las cepas, fueron analizadas para la secuenciación del gen hsp65 y por secuenciación parcial del 16S rRNA. Una de las 7 cepas fue duplicada y por ello no se contabilizó. Las 6 cepas restantes se estudiaron con un cebador universal con lo que se obtuvo entre 477-640 pb y con el gen hsp65 conseguimos un amplicón de 441pb empleando los cebadores Tb11 y Tb12.
Identificación de micobacterias por análisis del gen del 16S rRNA
La identificación de las cepas fue así: 3(1) y 7(3) se encuentran relacionadas con Mycobacterium frederiksbergense y muestran una similitud del 99 %, así como a M. neoaurum en un 93 %. Las cepas 4(2) y 7(2) están relacionadas a M. canariasense en un 97 % y M. diemhoferi. La cepa 7(1) está relacionada a M. chelonae y 4(1) relativamente cercana a M. fortuitum (Tabla 2, Figura 4).
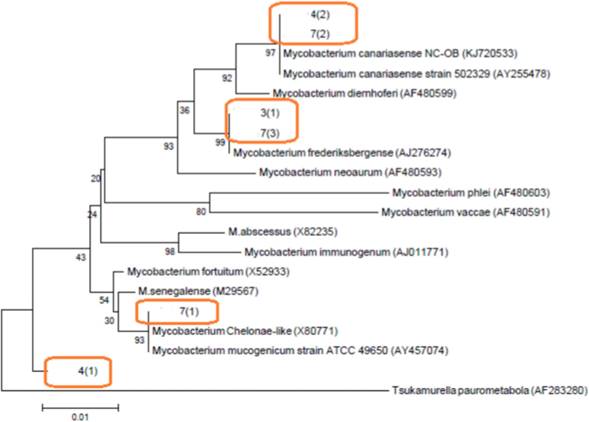
Figura 4 Árbol filogenético de micobacterias de crecimiento rápido, indicando la relación de las cepas de Micobacterias aisladas del Hospital. Árbol basado en las secuencias parciales obtenidas del PCR se han comparado con las secuencias disponibles en el GenBank para el gen ARNr 16S. El algoritmo utilizado para construir el árbol fue Neighbor-joining con el modelo de 2-parámetros de Kimura. La cepa NC-OB (KJ720 533) 27. El grupo externo fue Tsukamurella paurometabola.
Identificación de micobacterias por análisis del gen hsp65
Las 6 cepas fueron analizadas secuenciando el gen hsp65 (Tabla 2). Los fragmentos amplificados de las cepas fueron de 430 a 448 pb aproximadamente. Las cepas 4(2) y 7(2) están relacionadas filogenéticamente a Mycobacterium canariasense, así como a Mycobacterium neoaurum, lo que confirma el análisis del 16S. En el caso de la cepa 7(1) muestra una similitud (del 100 %) con Mycobacterium mucogenicum. Las cepas 3(1) y 7(3) son cepas idénticas a M. frederiksbergense y cercanas a M. neoaurum. La cepa 4(1) está relacionada a Mycobacterium fortuitum (Figura 5).
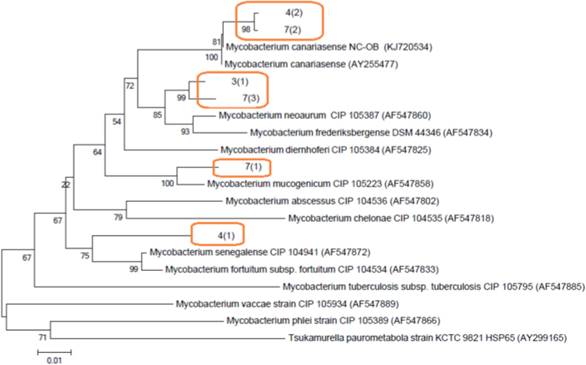
Figura 5 Árbol filogenético de micobacterias de crecimiento rápido, indicando la relación de las cepas de Micobacterias aisladas del Hospital. Las secuencias obtenidas del PCR se han comparado con las secuencias disponibles en el GenBank para el gen hsp65. El algoritmo utilizado para construir el árbol fue Neighbor-joining con el modelo de 2-parámetros de Kimura. La cepa NC-OB (KJ720533) 27. El grupo externo fue Tsukamurella paurometabola.
DISCUSIÓN
Las MNT han sido implicadas en enfermedades crónicas como son las infecciones respiratorias y cutáneas 1,5.
Las MNT tales como M. fortuitum, M. chelonae, M. peregrinum, M. szulgai, M. gordonae han sido involucradas en infecciones nosocomiales y pseudobrotes; por esta razón deben de ser estudiadas con mayor profundidad para establecer claramente la causa de un brote o pseudobrote nosocomial.
Kirschner et al. 11 demostraron que la población de M. avium está relacionada directamente con los niveles de zinc. La presencia de micobacterias en los sistemas de distribución de agua de los hospitales se da en parte porque se usan cañerías galvanizadas, las cuales son hechas con aleaciones de zinc. Debe considerarse que el zinc es raro en aguas naturales, pero bastante común en agua potable y en los desagües por el uso común de material de hierro (metal galvanizado). La remoción de zinc se da por corrosión de las aleaciones de bronce y zinc-cobre, siendo conocida como descincificación. La descincificación es agravada por la cloración del agua y puede ocurrir tanto en agua caliente como en agua fría 12. Otro factor que contribuye a la presencia de estas micobacterias en los sistemas de distribución de agua en los hospitales es su resistencia a la desinfección por cloro (Cl-), por ejemplo: Mycobacterium intracellulare, M. xenopi, M. avium, M. fortuitum, M. chelonae, son conocidos por su resistencia a los metales pesados y oxianiones; Mycobacterium scrofulaceum, M. avium y M. intracellulare, son anormalmente resistentes al cadmio (Cd), mercurio (Hg), plata (Ag) y telurio (Te). Queda demostrado así que en la fisiología de las micobacterias es un claro determinante en su ecología y en su distribución geográfica 11,12, lo que impide erradicarlas completamente.
Las infecciones por micobacterias no son exclusivamente pulmonares, Mycobacterium scrofulaceum fue encontrada como agente causante de linfoadenitis en niños 13.
Las investigaciones de du Moulin y Stottmeier 14 determinaron las potenciales rutas de infección para los pacientes hospitalizados. Estas se podrían identificar como: el caño en el baño del paciente; el agua de la jarra en la mesa de noche del paciente; el equipo de nebulización y el urinario entre otros. En el mismo estudio 14 aislaron 43 cepas de micobacterias de agua potable en agua caliente y fría del hospital de una comunidad costera en EEUU. En esa investigación se encontró que 39 cepas (90,7 %) fueron Mycobacterium avium complex, M. gordonae (7,0 %) y M. scrofulaceum (2,3 %); la concentración de M. avium fue de 10 a 500 ufc/100ml. En hospitales de otros países se han encontrado complicaciones post cirugía de trasplante renal y en los pacientes con leucemia infectados por varias especies de micobacterias 15,16.
Es importante señalar que el nicho ecológico y la fisiología de las MNT son significativamente diferentes a Mycobacterium tuberculosis. Muchas micobacterias son de vida libre y han sido detectadas en un amplio rango de ecosistemas incluyendo agua, suelos y aerosoles 16,17.
Las MNT se hallan en el medio ambiente o reservorios de agua como lo es el sistema de distribución de agua potable, no hay una contaminación de otro origen; estas son residentes capaces de crecer, persistir y sobrevivir 11,16,17 en su medio acuoso. Los factores que permiten a las micobacterias persistir y mantenerse viables serían: la temperatura; los nutrientes gaseosos y la interrupción de flujo de agua. Las micobacterias más comúnmente halladas son: Mycobacterium avium complex; Mycobacterium gordonae; M. flavescens; M. fortuitum y M. chelonae.
La capacidad de las micobacterias para sobrevivir en fuentes de agua de los hospitales ha sido reportada hace más de 30 años 18. Las micobacterias patógenas han sido aisladas de agua de hospital en varias ocasiones, von Reyn 18 aisló Mycobacterium avium 10 de 33 (30 %) muestras ambientales en Boston y New Hampshire entre 1990-1992; otros investigadores han reportado resultados similares 19,20,21. Por otro lado en este estudio no se ha detectado micobacterias en agua caliente, la temperatura del agua del hospital alcanzó en promedio los 73 °C. Las MNT crecen en un rango de 52-57 °C 18,21, esto podría explicar porque no se han aislado micobacterias del sistema de distribución de agua caliente (Tabla 1).
Hace algunos años se han venido reportando nuevas especies de micobacterias en fuentes de agua de hospitales; estos estudios han demostrado una fuerte asociación entre las especies de micobacterias en el agua y brotes nosocomiales en pacientes inmunosuprimidos 13-16,19,21.
En 1988 21 34 áreas de un hospital fueron analizadas en búsqueda de micobacterias, 14 (41 %) demostraron la presencia de M. avium por métodos fenotípicos y genotípicos. Once áreas correspondieron a muestras de agua caliente con una temperatura promedio de 55 °C, sugiriendo que el agua caliente no es factor limitante para algunas micobacterias. Otros investigadores 22 detectaron MNT en el 54 % de las muestras de hielo, donde el 35 % fueron de agua potable, en lugares públicos de 21 Estados de EEUU. Hace algún tiempo se publicó una extensa revisión bibliográfica por Vaerewijck et al. 23 en la que se recomienda la evaluación de los sistemas de distribución de agua y las biopelículas como método de control de micobacterias ambientales.
Consideramos que debe realizarse una limpieza minuciosa de las máquinas de uso clínico, de igual modo de toda agua potable del hospital y toda fuente de agua que pueda ser reservorio de patógenos. Los estudios preventivos o de mantenimiento como de limpieza o desinfección deben ser hechos por personal especializado y respaldado por un laboratorio clínico, dejando informes para futuras comparaciones de resultados. La presencia de micobacterias en el sistema de distribución de agua de los hospitales de Lima debería ser notificada a los órganos de control y considerada seriamente como una fuente importante de patógenos agresivos, sobre todo para los pacientes inmunosuprimidos por quimioterapia o cirugías de trasplante.
Para finales de los años 90, Hillebrand-Haverkort et al. 24 demostraron la presencia de M. genavense en agua de hospital ≥10 bacterias/ml por técnicas moleculares como reacción en cadena de la polimerasa ―RCP―; esta micobacteria es de difícil cultivo e identificación.
Las especies de micobacterias aisladas en este hospital han sido reportadas anteriormente como responsables de infecciones nosocomiales en humanos en otras sedes hospitalarias de Lima. Es importante que el personal del laboratorio esté familiarizado con cepas tipo Mycobacterium fortuitum y Mycobacterium chelonae. En este estudio también hemos identificado a Mycobacterium frederiksbergense, una cepa aislada por primera vez en Dinamarca en 2001; la cual recientemente ha sido implicada en infecciones en humanos, en especial en un paciente pediátrico con leucemia linfoblástica aguda 25. Así Mycobacterium canariasense fue aislada más de una vez del agua del hospital. La primera descripción de Mycobacterium canariasense fue por Campos-Herrero et al. y Jiménez et al. 15,26, ellos identificaron la bacteria en 12 pacientes con leucemia y linfomas. Los pacientes tuvieron cuadros de bacteriemia; nunca identificaron la fuente de la infección, pero lo más probable es que fuera el agua. La presencia de M. canariasense y M. frederiksbergense en agua hospitalaria podría representar una amenaza potencial, ya que pueden considerarse como patógenos oportunistas en pacientes con cáncer 27. Hoy no son consideradas las MNT un problema de salud pública en Perú. Durante este estudio no se ha hecho la búsqueda de casos clínicos que se correlacionen con infecciones por estas micobacterias en este hospital. Sin embargo los hallazgos ponen de manifiesto la presencia y potencial peligro en pacientes inmunosuprimidos por cáncer, trasplantes o cirugías oncológicas de emergencia.
CONCLUSIONES
Este es el primer estudio en Perú que revela la presencia de micobacterias potencialmente patógenas en el sistema de distribución de agua de un hospital de Lima y que podría comprometer a pacientes inmunocomprometidos. Este estudio pretende servir como punto de partida para profundizar en investigaciones sobre MNT en el Perú. En algunos de los hospitales del país existe la necesidad de investigar potenciales reservorios de patógenos de difícil identificación o aislamiento. Las pruebas bioquímicas son útiles para algunas especies de micobacterias, pero para discriminar especies muy cercanas filogenéticamente es necesario el uso de herramientas moleculares. La presencia de MNT en fuentes de agua dentro del hospital podría conllevar a infecciones intrahospitalarias. La sospecha de casos debe partir del personal clínico y el laboratorio, por ello se cree que serían recomendables las evaluaciones periódicas de las fuentes de agua en los hospitales para determinar la carga microbiana; y poderlas relacionar así con los posibles brotes o pseudobrotes que pueden darse en un determinado período al interior de los hospitales.














