INTRODUCCIÓN
Las bacterias del género Staphylococcus tienen una gran capacidad para adaptarse al medio en el que habitan; en el ámbito hospitalario, la aparición de técnicas invasivas favorece la producción de infecciones por estos microorganismos 1. Dentro de este género destaca el S. aureus como la especie más patógena que causa un amplio rango de infecciones nosocomiales desde infecciones en piel, mucosas, huesos hasta infecciones sistémicas como bacteriemias y endocarditis 2,3,4. Esta especie es uno de los patógenos más comunes en el ambiente hospitalario, ocasionando muchos problemas por la capacidad de adquirir genes de resistencia a los antibióticos.
Algo similar ocurre con los estafilococos coagulasa negativos ―ECN―, considerados patógenos nosocomiales oportunistas que tienen un impacto en la salud humana debido al uso de terapias inmunosupresoras y métodos invasivos como implantes y prótesis. Estas bacterias se encuentran como parte de la microbiota de piel y mucosas, siendo la principal fuente de infección endógena. A este grupo pertenece S. epidermidis, uno de los estafilococos más frecuentes en el ambiente nosocomial 5,6,7.
La resistencia a la meticilina en estas dos especies de estafilococos se considera un problema creciente en el mundo. Los estafilococos resistentes a meticilina son también resistentes a β-lactámicos y a otros grupos de antibióticos; recientemente se ha reportado resistencia a glicopéptidos. In vitro la susceptibilidad a clindamicina, trimetoprimsulfametoxazol, eritromicina, quinolonas y tetraciclinas ha sido frecuentemente reportada. Sin embargo se han presentado cepas resistentes al grupo de antibióticos MLSB ―macrólidos, lincosamidas y estreptograminas B― comúnmente usados para tratar infecciones por Staphylococcus spp.; razón por la cual monitorear el perfil de susceptibilidad antimicrobiana es determinante para la evaluación de la terapia empírica en pro de generar alertas sobre la presencia de mecanismos de resistencia emergentes o inusuales 8,9.
La aparición de cepas resistentes de estafilococos conlleva a un riesgo de mortalidad, incremento de la estancia hospitalaria y costos comparados con las infecciones por cepas susceptibles; por ello es importante vigilar los cambios en los patrones de resistencia de estos patógenos circulantes en hospitales para así contribuir con el estudio epidemiológico de estas infecciones y con el manejo de la mejor opción de tratamiento antibacteriano para el paciente.
Por todo lo anterior, está investigación tiene como objetivo evaluar la tendencia de los perfiles de resistencia antimicrobiana de S. aureus y S. epidermidis aislados en un hospital de tercer nivel de la ciudad de Cartagena.
MATERIALES Y MÉTODOS
Estudio transversal, realizado con 520 aislamientos de S. aureus y 230 aislamientos de S. epidermidis obtenidos en un hospital de alta complejidad entre 2010 y 2016. La información analizada proviene de la base de datos en soporte digital del laboratorio de microbiología del hospital, que fue positiva para Staphylococcus spp.
Las muestras clínicas de las que se obtuvieron los aislamientos fueron: sangre; orina; abscesos; secreciones; fluidos corporales (líquido pleural, líquido peritoneal) y otras (punta de catéter, escara, retrocultivo, piel, lavado bronquial). En esta investigación se consideraron las secreciones como sustancias que fueron liberadas de una herida y fluidos corporales como los líquidos biológicos que se producen en el organismo.
Susceptibilidad antimicrobiana
Se utilizó el sistema automatizado MicroScan® para la determinación de la concentración inhibitoria mínima ―CIM―, teniendo en cuenta los protocolos recomendados por el Clinical & Laboratory Standards Institute ―CLSI― durante los años 2010-2016 para los siguientes antibióticos con sus correspondientes siglas de acuerdo al WHONET: ciprofloxacina ―CIP―; eritromicina ―ERI―; gentamicina ―GEN―; levofloxacina ―LVX―; rifampicina ―RIF―; trimetoprim-sulfametoxazol ―SXT―; vancomicina ―VAN―; linezolid ―LZD―; clindamicina ―CLI―; daptomicina ―DAP― y moxifloxacina ―MFX―. Los aislamientos fueron clasificados como sensible (S) o resistente (R). Se calculó el índice de resistencia múltiple a antibióticos ―MRA―, definido como a/b donde ‘a’ es el número de antibióticos a los que una cepa fue resistente y ‘b’ es el número de antibióticos a los que la cepa estuvo expuesta. Un índice mayor a 0,2 indicó que una bacteria tiene resistencia múltiple 10. Los aislamientos con perfiles de resistencia antimicrobiana a tres o más familias de antibióticos fueron interpretados como bacterias multirresistentes 11.
Análisis estadístico
La información fue procesada y analizada con el programa estadístico SPSS v23. Para la descripción de las variables se utilizaron frecuencias absolutas y porcentajes con su respectivo intervalo de confianza al 95 %. Se estimó la frecuencia de Staphylococcus en la UCI y otros servicios, porcentaje de especies de Staphylococcus por muestra clínica y los perfiles de resistencia a antibióticos de los aislamientos. Además, se realizaron comparaciones de los perfiles de resistencia teniendo en cuenta el aislamiento de interés usando la prueba de chi-cuadrado; de igual manera se realizó el análisis multivariado para estimar la fuerza de la asociación. Se utilizó la prueba de chicuadrado de tendencia lineal para analizar el comportamiento de la resistencia a lo largo del período de estudio. La diferencia estadísticamente significativa fue establecida a través de la interpretación de los intervalos de confianza de la diferencia (IC 95 %dif) de proporciones, en conjunto con la interpretación de los p-valores según los criterios de Sterne y Smith 12.
RESULTADOS
Entre 2010 y 2016 fueron aislados 1218 cocos grampositivos de los cuales el 42,7 % (520/1218; IC 95 %, 39,9 % a 45,5 %) fueron Staphylococcus aureus; el 18,9 % (230/1218; IC 95 %, 16,8 % a 21,2 %) pertenecieron a la especie S. epidermidis y el 38,4 % (468/1218; IC 95 %, 3,8 % a 41,2 %) fueron otros ECN (S. hominis, S. haemolyticus, S xylosus, S. simulans y S. capitis).
Con relación al origen de los aislamientos, la mayoría de las cepas de interés fueron obtenidas de muestras como sangre (58,4 %; 711/1218; IC 95 %, 55,6 % a 61,1 %) y secreciones (24,0 %; 292/1218; IC 95 %, 21,7 % a 26,5 %) (Tabla 1).
De las cepas de S. aureus, el 47,5 % (247/520; IC 95 %, 43,2 % a 51,8 %) fueron meticilino resistentes (SARM); además, se encontró una alta frecuencia de SARM en secreciones (43,3 %; 107/247; IC 95 %, 37,3 % a 49,6 %). En el caso de S. epidermidis, el 68,7 % (158/230; IC 95 %, 62,4 % a 74,3 %) fueron meticilino resistentes y fueron aislados principalmente en sangre (76,9 %; 120/156; IC 95 %, 69,7 % a 82,8 %). Otros resultados se presentan en la Tabla 1.
Tabla 1 Frecuencia de aislamientos de Staphylococcus en muestras clínicas.
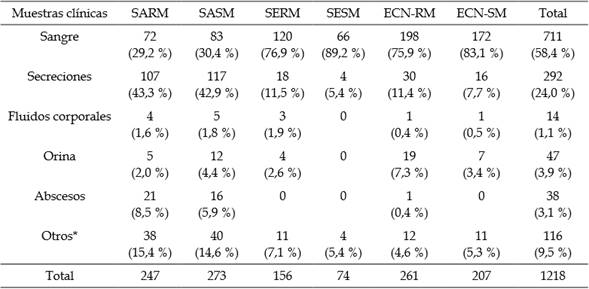
SARM: Staphylococcus aureus, resistente a meticilina; SASM: S. aureus, sensible a meticilina; SERM: Staphylococcus epidermidis, resistente a meticilina; SESM: S. epidermidis, sensible a meticilina; ECNRM: estafilococo coagulasa negativo resistente a meticilina; ECN-SM: estafilococo coagulasa negativo sensible a meticilina. Otros: punta de catéter, escara, retrocultivo, piel, lavado bronquial, heridas.
Fuente: elaboración propia.
Analizando el tipo de servicio, el 80,2 % (198/247; IC 95 %, 74,7 % a 84,7 %) de SARM se encontraron en servicios diferentes a la UCI; al igual que los aislamientos de S. aureus sensibles a la meticilina (SASM) (76,6 %, 209/273; IC 95 %, 71,2 % a 81,2 %). En cuanto al S. epidermidis resistente a la meticilina (SERM), el mayor número de aislamientos se encontró en la UCI (50,6 %; 79/76; IC 95 %, 42,9 % a 58,4 %); contrario a los aislamientos de S. epidermidis sensibles a la meticilina (SESM) que se hallaron principalmente en otros servicios (56,8 %, 42/74; IC 95 %, 45,4 % a 67,4 %). No se pudo detectar asociación entre el origen de los aislamientos y la resistencia a la meticilina (SARM: UCI frente a no-UCI, p=0,415; SERM: UCI frente a no-UCI, p=0,682).
Entre 2015 y 2016 se observó una reducción significativa de la resistencia a meticilina en la UCI; mientras la resistencia a meticilina en otros servicios fue constante. En cambio, tanto en la UCI como en otros servicios, la resistencia en los aislamientos de SERM mostró tendencia decreciente en estos dos años (Figura 1). En general, se encontró que la tendencia de los fenotipos de resistencia fueron similares durante los siete años de estudio (prueba de chi-cuadrado de tendencia lineal: SARM-UCI p=0,362; SARM no-UCI p=0,575; SERM-UCI p=0,627; SERM-noUCI p=0,701).
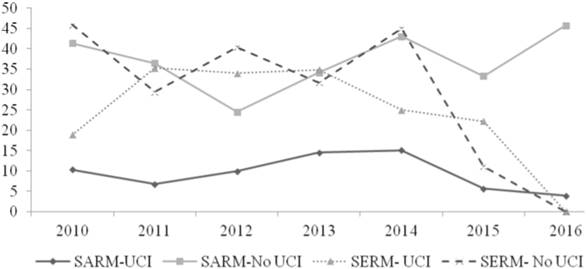
Figura 1 Frecuencia de SARM y SERM de acuerdo al tipo de servicio, 2010-2016. SARM-UCI: S. aureus, resistente a meticilina de UCI; SARM-no-UCI: S. aureus, resistente a meticilina de otros servicios; SERM-UCI: S. epidermidis, resistente a meticilina de UCI; SERMno-UCI: S. epidermidis, resistente a meticilina de otros servicios.
Teniendo en cuenta los perfiles de resistencia de los aislamientos de SARM y SERM, las cepas SARM fueron principalmente resistentes a: ciprofloxacina (25,8 %; IC 95 %, 20,5 % a 31,9 %); eritromicina (42,0 %; IC 95 %, 35,9 % a 48,3 %) y clindamicina (24,3 %; IC 95 %, 19,0 % a 30,5 %) (Tabla 2).
Tabla 2 Comparación de los fenotipos resistentes a antibióticos entre aislamientos de SARM y SASM.
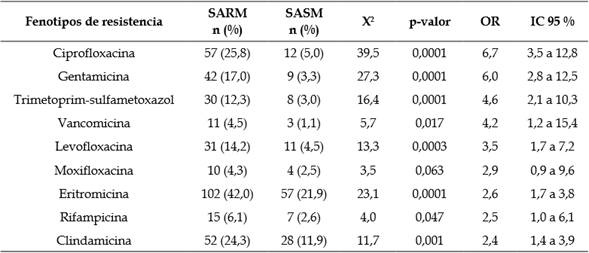
SARM: S. aureus, resistente a meticilina; SASM: S. aureus, sensible a meticilina.
Fuente: elaboración propia.
Continuando con los aislamientos SERM se observó una mayor resistencia a: eritromicina (70,9 %; 105/148; IC 95 %, 63,2 % a 77,7 %); vancomicina (1,3 %; 2/154; IC 95 %, 0,4 % a 4,6 %) y gentamicina (53,9 %; 82/152; IC 95 %, 46,0 % a 61,7 %) (Tabla 3).
Tabla 3 Comparación de fenotipos de resistencia entre aislamientos de SERM y SESM.
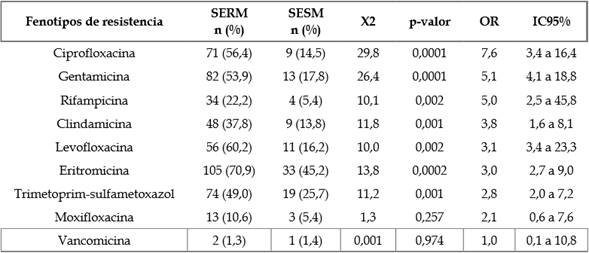
SERM: S. epidermidis, resistente a meticilina; SESM: S. epidermidis, sensible a meticilina.
Fuente: elaboración propia.
Se encontró asociación estadísticamente significativa entre la frecuencia de aislamientos de S. aureus eritromicina-resistentes y meticilinaresistentes; se detectó que cuando un aislamiento de S. aureus es resistente a meticilina tiene aproximadamente tres veces más riesgo de ser resistente a eritromicina. El resto de asociaciones y sus respectivos tamaños del efecto (OR) se presentan en la Tabla 2.
De igual forma se encontró asociación significativa entre la resistencia a gentamicina y eritromicina en S. epidermidis. Para este caso, se estimó que está bacteria tiene un riesgo aproximadamente seis veces mayor de ser resistente a gentamicina cuando es SERM (Tabla 3).
Con respecto al índice MRA se observaron valores entre 0 y 1 en SARM y valores de 0 a 0,8 en SERM, sin considerar el tipo de servicio. Aproximadamente el 46,6 % (115/247; IC 95 %, 40,4 % a 52,8 %) de las cepas SARM y el 74,4 % (116/156; IC 95 %, 67,0 % a 80,6 %) de las cepas SERM pueden ser consideradas multirresistentes (índice MRA>0,2).
Se detectaron 28 antibiotipos, siendo 10 de ellos multirresistentes en cepas SARM procedentes de la UCI, la mayoría Ant-3 (36,8 %; IC 95 %, 19,2 % a 59,0 %); mientras que en las cepas SARM de otros servicios se presentaron 9 antibiotipos multirresistentes, siendo los más frecuentes: Ant-3 y Ant-6 (23,7 %; IC 95 %, 13,0 % a 39,2 %).
Los aislamientos de SERM presentaron un menor número de antibiotipos en comparación con las cepas SARM dado que se encontraron 22 antibiotipos de los cuales siete se consideraron multirresistentes dentro del grupo de aislamientos SERM procedentes de la UCI, siendo el Ant-C (26,9 %; IC 95 %, 16,8 % a 40,3 %) el más abundante. Algo similar fue encontrado en el caso de SERM de otros servicios (Ant-C: 21,0 %; IC 95 %, 11,1 % a 36,3 %) (Tablas 4, 5). No fue posible realizar comparaciones entre los antibiotipos debido a la baja cantidad detectada.
Tabla 4 Antibiotipos de multirresistencia de aislamientos SARM.
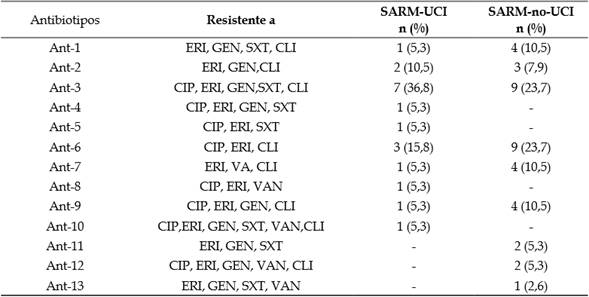
SARM-UCI: S. aureus, resistente a meticilina de UCI; SARM-no-UCI: S. aureus de otros servicios; CIP: ciprofloxacina; ERI: eritromicina; GEN: gentamicina; SXT: trimetoprim-sulfametoxazol; VAN: vancomicina; CLI: clindamicina.
Fuente: elaboración propia.
Tabla 5 Antibiotipos de multirresistencia de aislamientos SERM.
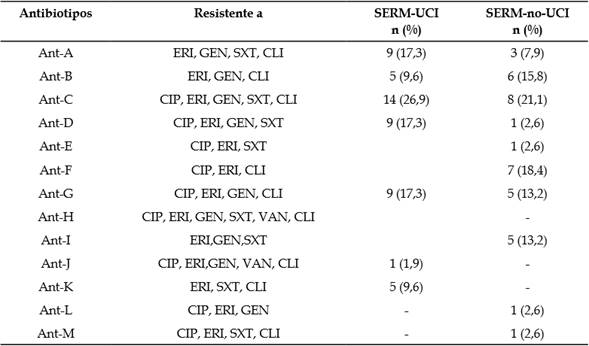
SERM-UCI: S. epidermidis, resistente a meticilina de UCI; SERM-no-UCI: S. epidermidis resistente a Meticilina de otros servicios. CIP: ciprofloxacina; ERI: eritromicina; GEN: gentamicina; SXT: trimetoprim-sulfametoxazol; VAN: vancomicina; CLI: clindamicina.
Fuente: elaboración propia.
DISCUSIÓN
Las infecciones asociadas con el cuidado de la salud ―y producidas por bacterias grampositivas como los estafilococos― están implicadas en: hospitalización prolongada; UCI; altos costos y pronóstico desfavorable 9.
Durante los siete años de estudio se identificaron 1218 cocos grampositivos de los cuales el 42,7 % fueron S. aureus y 18,9 % correspondió a S. epidermidis; estas frecuencias fueron mayores que las obtenidas por Martínez et al. en clínicas y hospitales de Cali (Colombia), aunque el período de estudio en esa investigación fue de 3 años 9. Otros autores también han reportado frecuencias menores 13,14.
La presencia de bacterias en sangre es consecuencia de una amplia variedad de infecciones y su tratamiento es urgente. En estas situaciones se debe conocer el agente etiológico y perfiles de resistencia para seleccionar un tratamiento eficaz. Es conocido que en la etiología de las bacteriemias nosocomiales los cocos grampositivos tienen los tres primeros lugares 15.
En el caso de esta investigación se reportó una alta frecuencia de cocos grampositivos en muestras de sangre de pacientes con bacteriemia: el 89,2 % de los aislamientos SESM y el 76,9 % de SERM fueron obtenidos de sangre; con respecto a los aislamientos de SASM y SARM se obtuvieron frecuencias de 30,4 % y 29,2 %, respectivamente.
Estos porcentajes son altos al compararse con el 31,0 % de Staphylococcus aislados en un hospital de Valledupar (Colombia) 16 y el 36,8 % de cocos grampositivos encontrados en un estudio sobre bacteriemias realizado en Monterrey (México) (6,7 % a 27,6 %) 17. En el caso del S. aureus es importante mencionar que en otras investigaciones esta bacteria fue aislada de abscesos, sangre y secreciones de herida quirúrgica 18; tal como en este estudio en el que una considerable cantidad de esta bacteria fue obtenida de estas muestras clínicas.
Las infecciones asociadas al cuidado de la salud se presentan en cualquier tipo de servicio de hospital, aunque los pacientes en la UCI son los más susceptibles de presentarlas. Como se mencionó previamente, los cocos grampositivos son importantes agentes etiológicos de estas infecciones; diferentes autores reportan una mayor prevalencia de estas bacterias en la UCI 19,20,21, al igual que lo reportado en esta investigación en la que el 54,8 % de los estafilococos aislados derivan de este servicio; también se encontró asociación significativa entre el tipo de servicio y la especie de Staphylococcus. Con respecto a la resistencia a la meticilina en S. aureus, la tendencia fue constante como sucedió en un hospital de Medellín (Colombia) y en la zona metropolitana de esa ciudad entre 2007 y 2012 22.
El uso excesivo e indiscriminado de antibióticos favorece la aparición de resistencia e incrementa los costos sanitarios y la mortalidad 19. En el caso de los estafilococos es frecuente encontrar cepas resistentes a meticilina. De hecho, altos porcentajes de resistencia a meticilina se encontraron en S. aureus (47,5 %) y S. epidermidis (68,7 %). Los resultados de resistencia a la meticilina en S. aureus son acordes a los datos presentados en diferentes estudios de ciudades colombianas, encontrando porcentajes de resistencia a meticilina en S. aureus entre 30,0 y 50,0 % 16,23,24. Con respecto a SERM, la frecuencia de resistencia a meticilina va de un 70,0 a un 80,0 % en hospitales de diferentes lugares del mundo 25,26; porcentajes mayores a los encontrados en esta investigación.
La resistencia a la meticilina en S. aureus y S. epidermidis se debe al gen mecA que se encuentra en el cassette estafilocócico cromosomal ―SCCs― que contiene también genes de resistencia a otros antibióticos tales como quinolonas, lincosamidas y macrólidos, produciéndose cepas multirresistentes 27,28. En este estudio se encontraron cepas SARM resistentes a ciprofloxacina, eritromicina y clindamicina. Igualmente las cepas SERM fueron resistentes a múltiples antibióticos como eritromicina, vancomicina y gentamicina. Se encontró asociación estadística entre la resistencia a meticilina y resistencia a algunos de estos antibióticos. En esta investigación se identificó un 46,6 % de cepas SARM multirresistentes, menor a lo reportado por Gómez et al. en cepas SARM de un hospital de Maracaibo (Venezuela) 29. Con respecto a las cepas SERM multirresistentes se presentó un porcentaje mayor a lo reportado por Laspina et al. en un hospital de Asunción (Paraguay) 30.
Al analizar el índice MRA tanto para las cepas SARM como SERM se encontró un mayor porcentaje de cepas SERM multirresistentes. S. epidermidis es considerado un potencial reservorio de genes de resistencia para otras bacterias como S. aureus, incrementando su potencial de colonizar y resistir a tratamientos con antibióticos; por ello la presencia de estas cepas SERM multirresistentes puede contribuir con la aparición de cepas SARM multirresistentes, lo cual limitaría las opciones para tratar infecciones en el hospital estudiado 5,6.
CONCLUSIÓN
Adicional a los reportes de multirresistencia a antibióticos detectados en los aislamientos clínicos de S. aureus y S. epidermidis se observó un comportamiento constante en sus perfiles de resistencia durante el período de estudio, excepto en los dos últimos años en los que se identificó una reducción significativa en la meticilino resistencia. En conjunto, estos hallazgos sustentan la vigilancia constante de los perfiles de resistencia antimicrobiana de S. aureus y S. epidermidis en pro de fortalecer y ajustar las medidas de prevención y de control de las infecciones asociadas a estos agentes bacterianos.














