Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biotecnología en el Sector Agropecuario y Agroindustrial
Print version ISSN 1692-3561
Rev.Bio.Agro vol.10 no.2 Popayán July/Dec. 2012
DIVERSIDAD DE LEVADURAS ASOCIADAS A INFLORESCENCIAS DE MANGO Y FLORES DE 'LULO ARBÓREO'
YEAST DIVERSITY ASSOCIATED TO INFLORESCENCES OF MANGO AND FLOWERS OF 'LULO ARBOREO'
DIVERSIDADE DE LEVEDURAS ASSOCIADAS A INFLORESCÊNCIAS DE MANGA E FLORES DE 'LULO ARBÓREO'
1 Grupo de investigación en biología molecular de microorganismos Universidad del Valle.
2 Profesor titular sección de Genética, Departamento de Biología, Universidad del Valle.
Correspondencia: jackegaviria@gmail.com
Recibido para evaluación: 12/05/2012 Aprobado para publicación: 22/07/2012
RESUMEN
En Colombia, el conocimiento de levaduras ha sido limitado a especies de interés clínico. El micro-hábitat aéreo de las flores, presenta un área de gran interés para el aislamiento de levaduras con importancia en el campo industrial, por su potencial uso como controlador biológico. El objetivo de este trabajo es analizar la diversidad de levaduras asociadas a inflorescencia de Mango (Mangífera índica) y flores de Lulo arbóreo (Solanum wrightii). Para dicha finalidad se trabajó con 96 aislados, a los cuales se les realizó caracterización fenotípica y posteriores pruebas de MSP-PCR Fingerprinting. Estas pruebas fueron complementadas con amplificación parcial y secuenciación del segmento 26S del ADNr para identificación a nivel de especie. El patrón de diversidad encontrado refleja una dominancia de la especie Candida leandrae en flores de lulo arbóreo, también se presentaron especies como Cryptococcus laurentii, Candida parazyma, Aeurobasidium pullulans y Pseudozyma tsukubaensis. Las inflorescencias de mango presentaron especies como Sydowia eucalypti, Cryptococcus flavescens, C. laurentii, C. nemorosus, C. heveanensis, Pichia kluyveri, Pseudozyma tsukubaensis y Candida asparagi. Se logró identificar hasta el nivel taxonómico de especie el total de los aislados encontrados, además se hallaron especies con potencial uso biotecnológico.
PALABRAS CLAVES: Levaduras, MSP-PCR Fingerprinting, Secuenciación, Región D1/D2.
ABSTRACT
The knowledge of the communities of yeast in Colombia has been limited to species of yeasts from clinical interest. The aereo microhabitat of flowers, has an area of great interest for the isolation of yeasts in industrial importance for potential use as biocontrol agents. The objective of this study was to analyze the diversity of yeasts from the inflorescences of Mango (Mangifera indica) and flowers of Lulo 'Arboreo' (Solanum wrightii). For this, worked with 96 isolated, to which characterization is realized to them phenotypic and later tests of MSP-PCR Fingerprinting complemented with amplification and sequencing of the partial region for the 26S rDNA species level identification. The pattern of diversity found reflects a predominance of specie Candida leandrae in the flowers of lulo'arboreo' also species are as Cryptococcus laurentii, Candida parazyma, Aeurobasidium pullulans and Pseudozyma tsukubaensis. The inflorescences of mango presented species as Sydowia eucalyptus, Cryptococcus laurentii, C. flavescens, C. nemorosus, C. heveanensis, Pichia kluyveri, Pseudozyma tsukubaensis and Candida asparagi. We identified to the taxonomic level of species total isolates found, in addition to finding species with potential biotechnological use.
KEYWORDS: Yeast, MSP-PCR fingerprinting, Sequencing, Region D1/D2.
RESUMO
Na Colômbia, o conhecimento das leveduras é limitado a espécies de interesse clinico. O micro-habitat aéreo das flores apresenta um interessante campo para o isolamento de leveduras com importância na indústria pelo seu potencial uso em controle biológico. O objetivo deste trabalho foi analisar a diversidade de leveduras associadas a inflorescências de Manga (Mangifera indica) e flores de 'lulo' (Solanum wrightii). Para isso, foram analisados 96 isolados, que foram caracterizados com testes fenotípicos e MSP-PCR Fingerprinting complementadas com sequenciamento parcial da região 26S do rDNA para identificação no nível de espécie. O padrão de diversidade reflete a dominância da espécie Candida leandrae nas flores de 'lulo', além disso, foram encontradas espécies como Cryptococcus laurentii, Candida parazyma, Aeurobasidium pullulans e Pseudozyma tsukubaensis. As inflorescências de manga apresentaram espécies como Sydowia eucalypti, Cryptococcus flavescens, C. laurentii, C. nemorosus, C. heveanensis, Pichia kluyveri, Pseudozyma tsukubaensis e Candida asparagi. Foi possível identificar até o nível taxonômico de espécie a totalidade dos isolados, além de encontrar espécies com potencial uso biotecnológico.
PALAVRAS-CHAVE: Leveduras, MSP-PCR Fingerprinting, Sequenciamiento, Região D1/D2.
INTRODUCCIÓN
Los microorganismos epifitos, como las levaduras encontrados en flores, se desarrollan en un ambiente hostil, con rápidas fluctuaciones físicas y nutricionales, resultado de hábitats dinámicos con variables ambientales cíclicas y no cíclicas [1], ello ocasiona que permanezcan básicamente, a expensas de los pocos nutrientes que exuda la planta. En consecuencia, estas levaduras presentan adaptaciones concretas [2], que les permiten desarrollarse en hábitats donde hay altas concentraciones de azúcares, vivir en simbiosis con insectos (de los cuales dependen para su dispersión); tener temperaturas de crecimiento comprendidas entre 5 y 37°C; desarrollarse según las condiciones de oxígeno del medio en el que se encuentren y soportar amplias variaciones de pH comprendidas entre 2,8 y 8,5 [3].
Dado el importante papel que cumplen las levaduras aéreas en el desarrollo y producción de la misma planta, identificar las poblaciones de microorganismos asociados a ambientes aéreos de una planta particular sería equivalente a describir el nicho específico que estos microorganismos pueden potencialmente ocupar [2]; y comprender con ello la ecología particular del microorganismo; siendo de gran relevancia, especialmente si se pretende que estos puedan ser empleados como agentes de control biológico. Estos ambientes hostiles generan un importante factor de selección al momento de identificar levaduras con alto potencial biotecnológico.
A pesar de que las levaduras presentes en las flores se conocen desde hace aproximadamente un siglo, se han realizado muy pocos estudios referentes a conocer las poblaciones de microorganismos asociados a ellas y la ecología particular de los mismos. La mayoría de investigaciones acerca de comunidades de levaduras presentes en flores se basan en el néctar de la flor y en la interacción levadura-flor-polinizador [4, 5, 7]. En Colombia, los estudios sobre levaduras se centran principalmente en especies de interés clínico.
El estudio de la diversidad de especies en micohabitáts aéreos de las flores podría ampliar el conocimiento de estos ambientes, y a largo plazo, brindar posibles avances y descubrimientos en industria y biotecnología. El propósito de esta investigación es evaluar la diversidad de levaduras encontradas en dos tipos diferentes de flores, asimismo conformar una colección de cepas para del banco de levaduras de la Universidad del Valle. La identificación de dichas levaduras, no se llevó a nivel meramente morfológico, debido a que éste causa a menudo imprecisiones al estar fuertemente influenciado por condiciones del cultivo. Por ello, en el presente estudio la caracterización morfológica se complementa con datos obtenidos por técnicas de biología molecular, tomando como base la reacción en cadena de la polimerasa (PCR fingerprinting), la cual permite caracterizar genéticamente cepas de levaduras en forma rápida y económica.
METODO
Sitio y diseño experimental
El experimento se llevó a cabo en las instalaciones del campus principal de la universidad del Valle (sede Meléndez) ubicado en el municipio de Cali, Valle (3°22' N y 76°32' W). El experimento se realizó completamente al azar seleccionando 5 árboles de cada una de las dos especies a estudiar, se trabajó con un total de 15 flores o inflorescencia por especie y un total de 96 cepas obtenidas de las muestras de flores de mango y lulo arbóreo con diluciones seriadas (10-1-10-4) y una repetición por dilución.
Recolección de materia vegetal
Las muestras fueron tomadas en los meses de Febrero- Marzo (reportado como época de floración tardía para el mango [6]), de cada uno de los 5 árboles seleccionados se tomaron 3 muestras representativas en el estrato bajo (1,20 m) y/o medio (2-3 m) para flores de mango y en el estrato alto para flores de lulo (5 m). Las muestras fueron tomadas con tijeras estériles y guardadas en bolsas estériles para ser trasladadas al laboratorio de Biología Molecular de Microorganismos de la Universidad del Valle, donde fueron procesadas en las 2 a 3 horas siguientes después de ser recolectadas.
Aislamiento de levaduras
Las muestras de flores fueron homogenizadas por maceración, previo lavado con solución de agua destilada e hipoclorito de sodio comercial al 10%, por 10- 15 minutos.
El macerado se realizó en un matraz estéril con 2 ml de agua mili-Q estéril, de la solución madre resultante se tomaron 500 µl. Se realizó recuentos mediante diluciones seriadas (10-1 – 10-4), de cada dilución se tomó 100 µl para siembra por dispersión en medio de cultivo YPDA (1% extracto de levadura, 2% peptona micológica, 2% de glucosa, 2% agar) suplementado con 25mg/mL de Ampicilina y cloranfenicol. Las cajas Petri se incubaron a 30°C, monitoreando el crecimiento de los cultivos por 72 horas y realizando conteo a simple vista de las colonias conseguidas en placa, de esta manera se aislaron las colonias más representativas por cada muestreo. Las colonias aisladas fueron crio-conservadas a -20 °C en medio YPD-glicerol al 30%.
Caracterización fenotípica
La caracterización macroscópica se lleva a cabo de acuerdo a las descripciones realizadas por Boekhout [7]. Durante los primeros tres días post sembrado se realizó la caracterización morfológica de los aislados, teniendo en cuenta: forma, brillo, tamaño, elevación, bordes, superficie, consistencia y color de cada una de las colonias.
Se determinaron las características microscópicas relacionadas con la forma de las células, presencia de filamentos, germinación y tipo de gemación.
A partir de la caracterización tanto macroscópica como microscópica, se agruparon los aislados para posteriores análisis moleculares.
Extracción de DNA
La extracción del ADN genómico de levaduras se realizó utilizando la metodología descrita por Osorio-Cadavid [8]. La extracción se verificó en geles de agarosa al 1%, usando TAE 1X a 9v/cm durante 1h revelados con Bromuro de Etidio. La cuantificación del ADN se realizó mediante espectrofotometría a 260nm y se determinó la pureza mediante la relación espectrofotométrica A260/A280nm con el uso de Nanodrop TM 2000C (Termo Scientific) en el laboratorio de Genética Molecular Humana de la Universidad del Valle.
MSP-PCR Fingerprinting
Para el análisis MSP-PCR (Minisatellite-primed) se siguió la metodología propuesta por Silva-Filho en el 2005 [9], usando el primer (GTG)5. Las reacciones de PCR fueron realizadas en un volumen de reacción de 25µl, que contenía 5uL (aproximadamente 1ng/µL) de ADN genómico total, 10X PCR buffer, 1 mM dNTPs Mix; 3,5mM de cloruro de magnesio; 0,8 mM de iniciador, y 1 U Taq ADN polimerasa (Fermentas).
El ADN fue amplificado en un termociclador (M.J. Research, USA) bajo las siguientes condiciones: paso inicial de desnaturalización de 5 min a 94 °C, 34 ciclos de: 15 s (segundos) a 93 °C, 45 s a 3.55 °C y 90 s a 72 °C, y paso final de extensión de 6 min a 72 °C. Los fragmentos de ADN amplificados fueron separados por electroforesis en geles de agarosa al 1,5% (Gibco-BRL) en 0,5 X TBE buffer (Buffer tris-Borato-EDTA) a 60 V por 4,5 h y revelados en solución de Bromuro de Etidio (2 mg/ml).
En cada gel fue incluido un marcador de peso molecular como referencia (marcador de 250 pb). Todas las reacciones se realizaron mínimo por duplicado, a fin de evaluar la reproducibilidad de los perfiles de bandas obtenidos.
El tamaño de los fragmentos obtenidos en cada una de las reacciones realizas fueron estimados con el software Total Lab Quant overview ®, posteriormente fueron formados los agrupamientos moleculares con similitudes de 100%.
Secuenciación de la región D1/D2
Se amplificó el dominio D1/D2 de la Subunidad grande del gen ribosomal rDNA 26S. Se analizó una muestra de cada grupo principal conformado. Los primeros fueron: NL1 (5´-GCATATCAATAAGCGGAGGAAAAG-3´) y NL4 (5´-GGTCCGTGTTTCAAGACGG-3´).
Las reacciones de amplificación (PCR) fueron realizadas en un termociclador (Multigene- Labnet, USA) bajo las siguientes condiciones: desnaturalización inicial a 94°C por 5minutos, 30 ciclos de desnaturalización a 94°C por 1 min, apareamiento a 55°C por 30seg, y extensión a 72°C por 1 min, con una extensión final de 10 min a 72°C. Para la amplificación, se tomaron 7µL (aproximadamente 1ng/µL) de ADN Genómico extraído y se resuspendió en 28µL de mezcla de PCR: 0.5µM NL1, 0.5µM NL4, 10µM dNTPs, 1X NH4+, 1.5mM MgCl2, y 1U de Taq Polimerasa. La calidad de la amplificación fue evaluada en geles de agarosa al 1.5% y visualizadas con Bromuro de Etidio a 240nm.
Los productos de PCR fueron purificados y secuenciados por MACROGEN (USA) bajo condiciones estandarizadas por la misma empresa. Las secuencias fueron ensambladas, editadas mediante el programa Chromas Pro v. 1.42 ® y comparadas con las secuencias reportadas en el GenBank usando el algoritmo 'Basic Local Alignment Search Tool (BLAST)'. Posteriormente, se realizaron los árboles filogenéticos correspondientes, empleando el Software MEGA 5.
Análisis estadístico
Con el fin de verificar los grupos moleculares conformados con los pesos de las bandas obtenidas se realizó un análisis Cluster con algoritmo UPGMA y el índice de correlación de PEARSON, usando el Software estadístico PAST versión 2.14 [10].
RESULTADOS
Aislamiento
Se obtuvo un total de 96 aislados de levaduras a partir de flores de mango y lulo arbóreo. De estos, el 56 % pertenecen a los aislados obtenidos para mango y el 44 % restante para lulo. El conteo aproximado de unidades formadoras de colonia para las levaduras encontradas fue 7,12x102 UFC/ml.
Del total de levaduras obtenidas se conformaron 10 agrupamientos con características morfológicas similares para flores de lulo arbóreo y 10 para inflorescencias de mango, donde el morfotipo dominante fue la textura mantequillosa, apariencia lisa, color blanco, forma circular, margen entero y elevación convexa, también se encontraron 5 aislados con características morfológicas únicas.
MSP-PCR Fingerprinting
El promedio de concentración obtenido para la extracción de DNA fue 58,87± 5,8 ng/µl y pureza de 1,99 ± 0,02. Posterior a la extracción del DNA se realizó la técnica MSP-PCR fingerprinting, los patrones de bandas obtenidos con esta técnica dieron lugar a bandas de pesos comprendidos entre 250 y 2500 pb (Figura 1). Con los datos del peso de las bandas de cada perfil se realizó una matriz de datos binarios basados en la presencia/ausencia de bandas, sobre la cual se hicieron los análisis de Cluster (UPGMA).
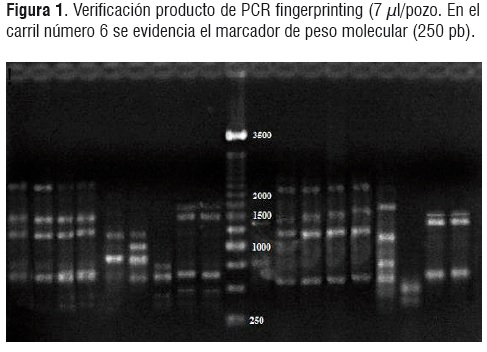
La reproducibilidad de las bandas obtenidas con la técnica MSP-PCR depende de las condiciones utilizadas para su empleo (Concentraciones de DNA exactas y previamente cuantificadas). Las diferencias en intensidades de bandas entre miembros de una misma especie encontradas en esta investigación, pueden ser atribuidas a variaciones entre condiciones de reacción o diferencia en concentración de DNA.
Agrupamientos
Para inflorescencias de mango se obtuvo un total de 9 agrupamientos con patrones similares en un 100% y un total de 4 cepas que no presentan similitud de 100% con ningún agrupamiento como indica la Figura 2.
Para cepas obtenidas de flores de lulo arbóreo se obtuvo un total de siete agrupamientos con patrones similares en un 100%, y un total de cuatro cepas que no presentan similitudes de 100% con ningún agrupamiento. Se registraron relaciones entre algunos grupos mayores al 80%, y relaciones entre grupos de 75 % como muestra la Figura 3.
Secuenciación dominio D1/D2 rDNA 26S
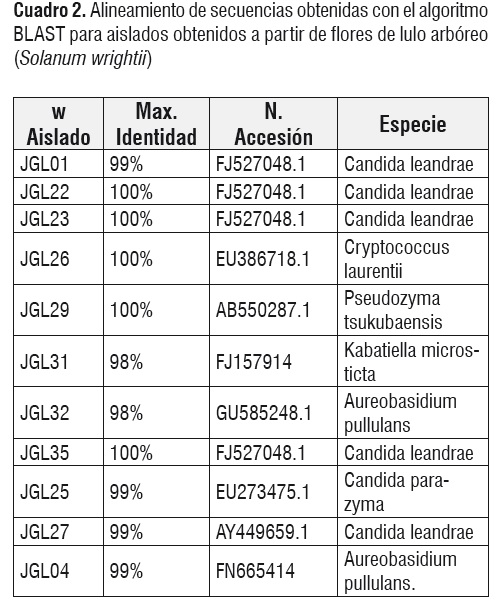
Se secuenció la región del dominio D1/D2 del gen ribosomal 26S para un aislado representativo de cada agrupamiento. Los datos obtenidos para cada una de las secuencias aisladas a partir de inflorescencias de mango y flores de lulo arbóreo se muestran en los cuadros 1 y 2, respectivamente.
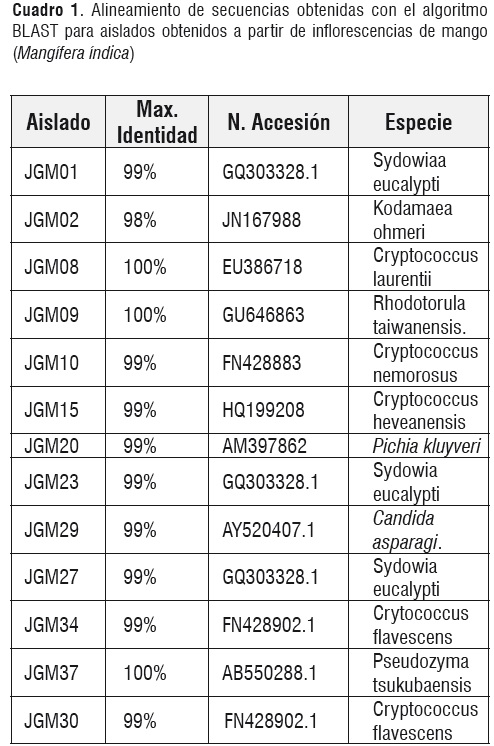
Las especies evaluadas a partir de inflorescencias de mango, muestran valores de 100% de identidad Máxima, para Cryptococcus laurentii, Rhodotorula taiwanensis y Pseudozyma tsukubaensis. Los aislados restantes muestran valores de Max. Identidad iguales a 99%, este valor de acuerdo a Kurtzman y Robnett, [11], es considerado correcto para asignar un aislado hasta el nivel de especie.
Los aislados JGL22, JGL23, JGL26, JGL29 y JGL35, presentaron una similitud del 100% con respecto a las secuencias depositadas en el Genbank, los aislados restantes presentaron identidad del 98-99%. Estos resultados corroboran con el máximo nivel de confianza la asignación del grupo a la especie. Los aislados JGL01, JGL22 y JGL23 pertenecientes a la especie Candida leandrae, muestran una similitud del 75% y 80% en análisis Cluster (Figura 3).
Del total de especies identificadas se reportaron en el Genbank las secuencias de los aislados que presentaron mayor abundancia y son recientemente descritos (JGM01, JGM09 Y JGL01) con los números de accesión JQ514555, JQ514556 y JQ514557 respectivamente.
Abundancia de especies obtenidas
El patrón de diversidad obtenido para especies aisladas a partir de flores de lulo arbóreo muestra una dominancia de la especie Candida leandrae con el 32% del total de aislados (figura 3). Este patrón de diversidad concuerda con trabajos realizados por Herrera, 2008, quien encontró una marcada dominancia de una única especie en néctar floral (Helleborous foetidius) y en aislados de abejorro Bombus, mismo género reportado como polinizador en flores de lulo. Además esta dominancia puede verse reflejada por características particulares presentes en esta especie para soportar el crecimiento en micro hábitats cambiantes o por la eficiente dispersión de esta levadura por medio de insectos (generalmente abejorros) o viento [5].
La especie Sydowia eucalypti presentó la mayor abundancia en inflorescencias de mango representando el 17% del total de especies obtenidas, esta es una especie de microhongo encontrada en hojas y hojarasca de especies de Eucalipto en el sur de Africa, Portugal y Australia [15], en condiciones de cultivo se presenta como 'yeast-like' y sus características morfológicas son similares a los géneros Aureobasidium y Hormonema. Esta especie es capaz de crecer en una amplia gama de especies de plantas no relacionadas con eucalipto [16].
Del total de especies identificadas los generos Cryptococcus y Candida, estuvieron mayormente presentes, lo que concuerda con lo publicado por Atlas, 2002, quien reportó que estos generos se encuentran frecuentemente asociados a plantas, ya sea flores u hojas [13].
Algunos géneros de levaduras encontradas tales como Cryptococcus, Pichia y Rhodotorula tienen la capacidad de formar capsulas [12] que actuan como escudo y les confiere ventajas adaptativas a los ambientes adversos, la presencia de pigmentos carotenoides en el caso de Rhodotorula les confiere proctección contra radiación UV [14]. Dichas ventajas adaptativas generan un importante factor de selección al momento de identificar levaduras con potencial biotecnológico como controladores de fitopatogenos en estos ambientes.
La especie Aureobasidium pullulans representa solo el 4% del total de aislados lo que concuerda a lo reportado por Lachance et al., 2001, quienes encontraron que las poblaciones de Aureobasidium estaban presentes en número muy bajo para flores de diferentes plantas [5]. Esta especie presenta un alto grado de diversidad intra-específica, reportándose diferentes variedades de la misma especie y presenta un alto potencial biotecnológico por su importancia como productor de Polisacarido extracelular biodegradable (EPS) pullulan (poly-α-1,6-maltotriose). Este componente es un promisorio biomaterial y es utilizado actualmente para el envasado de alimentos y medicamentos [17].
La especie Pseudozyma tsukubaensis encontrada tanto para aislados obtenidos a partir de flores de lulo arbóreo como para los obtenidos de inflorescencias de mango, presenta un amplio rango de distribución en plantas, se ha reportado para hojas de Perilla frutescens (Lamiaceae) en Japón, como productoras de biosurfactantes glicolípidos, P. tsuckubaensis es además capaz de producir gran cantidad de diastereoisómeros MEL-B a partir de aceites vegetales, por lo que son consideradas especies de gran interés biotecnológico [18].
Otros géneros como Rhodotorula, Cryptococcus y Pichia son ampliamente empleados en biotecnología por la capacidad de producir mananos lineales con enlaces alternados, heteropolisacáridos ácidos con alto contenido de xilosa y fosfomanos a partir de glucosa [19].
Las especies del género Cryptococcus reportadas para esta investigación corresponden a haveanensis, laurentii y flavescens, este género mostró una alta frecuencia de levaduras. Caso paradójico a lo publicado por Lachance en el 2001, quien reportó que varias especies de levaduras Basidiomycetas como Crytococcus y Pseudozyma son poco frecuentes en las flores, concluyendo que estos son miembros generalistas del filoplano y son simplemente alóctonos de los ecosistemas levadura-flor-polinizador [5]. Las especies pertenecientes al género Cryptococcus son de gran interés en el campo industrial, siendo productores de exopolisacaridos (C. flavescens) o controladores biológicos como C. laurentii, reduciendo la adhesión y germinación de conidias de Botrytis cinérea, un hongo causante de la declinación de post-cosechas en muchas especies vegetales [20].
Por último las especies Candida parazyma y Rhodotorula taiwanensis han sido descritas como especies recientes C. parazyma es una especie descrita en el 2002, encontrada en flores de Ipomoea indica en Hawai y relaciona con el insecto Drosophila ?oricola, el hábitat de esta especie son flores e insectos asociados a flores, de climas cálidos principalmente [5]. R. taiwanensis descrita en el 2010, fue obtenida a partir de tallos de Artemisia argyi, estando más cercanamente relacionada a Rhodosporidium sphaerocarpum [21]
CONCLUSIONES
La identificación del 100% de las especies halladas en este estudio, se logró gracias al empleo de las técnicas moleculares implementadas.
El patrón de diversidad encontrado tanto para flores de lulo arbóreo como para inflorescencias de mango varia ampliamente, presentando en este último un mayor número de especies, siendo la especie de mayor frecuencia Sydowia eucalyti con el 17% del total de aislados.
La mayor parte de las especies encontradas en esta investigación no habían sido reportadas para Colombia, contribuyendo de esta manera con el conocimiento de levaduras en microhabitats naturales de las flores.
Especies como A. pullulans, C. laurentii, P. tsukubaensis y C. flavencens, se han reportado como especies de interés biotecnológico, lo que permite un panorama interesante para futuras investigaciones en industria biotecnológica.
REFERENCIAS
[1] LINDOW, S.E. and LEVEAU, H.J. Phyllosphere microbiology. Curr Opin Biothec., 13, 2002, p. 259-265. [ Links ]
[2] MEDINA, C.M, CRISTANCHO D. and URIBE, D. Physiological Responses and Antagonistic Capacity of Yeast Phyllospheric: Isolates Obtained in Blackberry Crops (Rubus glaucus). Acta biológica Colombiana., 14(3), 2009, p. 181-198. [ Links ]
[3] URIBE, G. L. Caracterización fisiológica de levaduras aisladas de la filosfera de mora [tesis de pregrado]. Bogota-Colombi: Pontificia Universidad Javeriana, facultad de ciencias, 2007, 153 p. [ Links ]
[4] CANTO, A., HERRERA, C.M., MEDRANO, M., PÉREZ, R. and GARCÍA, I.M. Pollinator foraging modi?es nectar sugar composition in Helleborus foetidus (Ranunculaceae): an experimental test. American Journal of Botany, 2008, p. 315–320. [ Links ]
[5] LACHANCE, M. A., STARMER, W.T., ROSA, C.A., BOWLES, J.M., BARKER, J.S. and JANZEN, D.H. Biogeography of the yeasts of ephemeral flowers and their insects. FEMS Yeast Research, 2001, p.1–8. [ Links ]
[6] HUETE, M and ARIAS, S. Manual para la producción de mango. Usaid-red proyecto de diversificación económica rural, 2007, p 1-42. [ Links ]
[7] BOEKHOUT, T., ROBERT, V., SMITH, M., STALPERS, J., YARROW, D., BOER, P., BUIS, R., GIJSWIJT, G., KURTZMAN, C.P., FELL, J.W., GUÉHO, E., GUILLOT, J. and ROBERTS, I. Yeasts of the World: Morphology, Physiology, Sequences and Identification. 2 ed. Amsterdam, Netherlands, 2004. [ Links ]
[8] OSORIO-CADAVID, E., RAMÍREZ, M., LÓPEZ, A., W. and MAMBUSCAY, L.A. Estandarización de un protocolo sencillo para la extracción de ADN genómico de levaduras. Revista Colombiana de Biotecnología., 11(1), 2009, p. 125-131. [ Links ]
[9] SILVA-FILHO, E. A., DOS SANTOS, S. K., RESENDE, A. M., DE MORAIS, J. O., DE MORAIS, M. A. and SIMOES, D. A. Yeast population dynamics of industrial fuel-ethanol fermentation process assessed by PCR-fingerprinting. Springer Antonie Van Leeuwenhoek., 88, 2005, p.13-23. [ Links ]
[10] HAMMER, D.A., HARPER, T. and RYAN, P.D. PAST: Paleontological Statistics Software Package for Education and Data Analysis. Palaeontologia Electronica., 4(1), 2001, 9 p. [ Links ]
[11] KURTZMAN, C.P. and ROBNETT, C.J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S) ribosomal DNA partial sequences. Antonie van Leeuwenhoek., 73(4), 1998, p. 331-371. [ Links ]
[12] MARTIN-MAZUELOS, E. and VALVERDE-CONDE, A. Criptococosis: diagnostico microbiologico y estudio de la sensibilidad in vitro. Hospital Universitario de valme, 2006, 5 p. [ Links ]
[13] ATLAS, R. Ecología microbiana y microbiología ambiental. 4 ed. Madrid, (España): Addison Wesley, 2002, 677p. [ Links ]
[14] LIBKIND, D., MOLINÉ M. and VANBROOCK, M. Posibles mecanismos de fotoprotección en levaduras. Revista electrónica de Radiobiología., 4, 2004, p. 84-88. [ Links ]
[15] CHEEWANGK, R., SUMMERELL, B.A., HYDE, K.D., TO-ANUN, C. and CROUS, P.W. Myrtaceae: a cache of fungal biodiversity. Antonie van Leeuwenhoek, 23, 2009, p. 55-85 [ Links ]
[16] CROUS, P.W., PHILLIPS, A. J. and BAXTER, A. P. Phytopathogenic fungi from South Africa. South Africa, 2000, 358p. [ Links ]
[17] SINGH, R.S, SAINI, G., KENNEDY, J. Pullulan: microbial sources, production and applications. Carbohydrate Polymers, 2008, p. 515–531. [ Links ]
[18] TUKUOMA, F., M. TOMOTAKE, K. MASAAKI I. TOMOHIRO and K. DAI. A basidiomycetous yeast, Pseudozyma tsukubaensis, efficiently produces a novel glycolipid biosurfactant. The identification of a new diastereomer of mannosylerythritol lipid-B. Journal of Bioscience and Bioengineering., 112(2), 2008, p.137-144. [ Links ]
[19] KURTZMAN, C.P. and FELL, J. The yeast a taxonomic study. 4 ed. Elsevier, 1999, p. 16-105. [ Links ]
[20] FILONOW, A. Butyl acetato y yeasts interact in adhesion and germination of Botrytis cinerea conidia in vitro and in fungal decay of golden delicious apple. Chem Ecol, 27, 2001. p. 831- 844. [ Links ]
[21] HUANG, C.H., LEE, F.L., TIEN, C.J. and HSIEH, P.W. Rhodotorula taiwanensis sp. nov., a novel yeast species from a plant in Taiwan. Antonie Van Leeuwenhoek., 99(2), 2010, p. 297-302. [ Links ]













