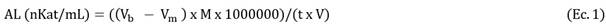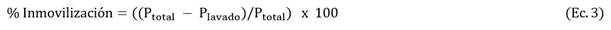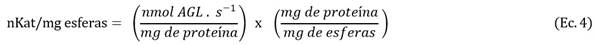INTRODUCCIÓN
Las lipasas (E.C. 3.1.1.3) son enzimas altamente versátiles muy utilizadas en procesos biotecnológicos como la producción de detergentes, surfactantes, biopolímeros, aromatizantes, cosméticos (Patel et al., 2019; Chandra et al., 2020) y fármacos quirales (Ehlert et al., 2019); también, en la bioconversión de alimentos (Akshita et al., 2020) y en la síntesis de biocombustibles (Lv et al., 2021). Después de las proteasas, las lipasas son las enzimas más utilizadas en la industria, por lo que hay un gran interés en la búsqueda de nuevas fuentes que satisfagan las necesidades de un mercado en crecimiento (Fatima et al., 2020).
Comparada con la catálisis química convencional, la catálisis mediada por lipasas presenta algunas ventajas que han llamado la atención de la industria biotecnológica, como son: mayor economía en el gasto del catalizador, condiciones de reacción suaves (temperatura y pH), baja generación de desechos y mayor capacidad de resolución de mezclas racémicas (Pereira-Cipolatti et al., 2019). Si bien las lipasas de origen microbiano (Ej. Pseudomonas cepacia, Rhizopus oryzae y Candida antarctica) son las más usadas en biotecnología (Chandra et al., 2020; Fatima et al, 2020), la producción de algunas de ellas es limitada por los altos costos que demanda el cultivo de microorganismos y los procesos de extracción y purificación. Los esfuerzos para solucionar estos problemas se han enfocado en el uso de cepas modificadas genéticamente para ser altamente productoras (Jares-Contesini et al., 2020), la optimización de condiciones de fermentación (Bharathi et al., 2019) y el desarrollo de procesos aguas abajo más eficientes (Khoo et al., 2020). Por otra parte, también se investigan fuentes alternativas de lipasas, como las plantas (Seth et al., 2014) a partir del látex de Carica papaya (Nhat et al., 2019) y algunas lipasas de semillas oleaginosas en germinación han sido exploradas (Kawinski et al., 2021).
Las especies del género Pachira (Malvaveae: Bombacoideae) son plantas tropicales, nativas de Sudamérica. Aunque en Colombia se consideran endémicas de la región Amazónica (Guevara-Andino et al., 2018), estas se encuentran distribuídas en varias zonas, donde se utilizan principalmente como plantas ornamentales. Pachira speciosa Triana and Planch y P. aquatica Aubl pertenecen al mismo grupo de especies con frutos en forma de cápsula leñosa y semillas oleaginosas grandes y desnudas (Bernal et al., 2015). Perez-Polizelli et al. (2008), mencionan las características de la lipasa de P. aquatica, que son deseables en procesos de biotransformación de lípidos, como su alta estabilidad a pH alcalinos y en diferentes solventes orgánicos, así como afinidad elevada por ésteres de ácidos grasos de cadena larga; no obstante, las enzimas que se encuentran en estado libre poseen baja estabilidad estructural, lo cual limita su reutilización y encarece los procesos biocataliticos. La inmovilización de enzimas en diferentes tipos de soportes ya sea de naturaleza orgánica, inorgánica o mixta, ha mostrado ser efectiva para mejorar características funcionales como la estabilidad y la reusabilidad (Alnoch et al., 2020), sin embargo, los soportes pueden afectar las propiedades de la enzima, de aquí la necesidad de escoger el material más apropiado.
El propósito de la presente investigación fue producir un sistema biocatalítico reciclable a partir de la lipasa extraída de semillas de P. speciosa inmovilizada en tres soportes a base de quitosano. El nuevo biocatalizador se constituiría en una alternativa económica en procesos de transformación de lípidos de interés para la industria biotecnológica nacional.
MÉTODO
Material biológico
Los frutos de P. speciosa se colectaron en el mes de marzo de 2018 en el Campus de la Universidad del Atlántico, municipio de Puerto Colombia, departamento del Atlántico (Coordenadas geográficas: 11°01′N y 74°52′O; temperatura: 28 °C; humedad relativa: 85 %). La identidad botánica de la planta fue establecida en el Herbario del Instituto de Ciencias Naturales de la Universidad Nacional de Colombia (No. COL576764).
Extracción de la lipasa
Se realizó siguiendo el protocolo descrito por Peres-Polizelli et al. (2008), con modificaciones; las semillas desnudas se mezclaron con un tampón de lisis (EDTA 0,001M; DTT 0,003M; K2S2O5 0,016 M; Tris-HCl 0,05 M, pH 8), usando la relación 0,3:1 (p/v). La mezcla se homogenizó en un procesador Osterizer® Blender durante 15 min, seguida de agitación (320 rpm) por 24 h a 15 °C, en un equipo Velp Scientifica ARE 230 V/50-60 Hzd y centrifugación a 4000 rpm por 40 min y 4 °C. El sobrenadante (extracto crudo) se almacenó a 4 °C.
La concentración de proteína total en el extracto se determinó mediante ensayo de Bradford (Kielkopf et al., 2020), usando el estuche comercial QuickStart™ Bradford Protein Assay (Bio-Rad) y una curva estándar de albumina sérica bovina.
Caracterización de la lipasa de P. speciosa
Cromatografía de filtración en gel. Se realizó la purificación parcial de la lipasa de P. speciosa en una columna Sephadex® G-100 (Sigma-Aldrich), usando el método descrito por Kumar et al. (2020) con modificaciones. Se agregaron a la columna 5 mL del extracto crudo de P. speciosa, la elución se realizó con buffer PBS 0,01 M (pH 7,4)/NaCl 0,015 M. Se colectaron alícuotas de 0,5 mL en viales eppendoff, monitoreando las proteínas a longitud de onda de 280 nm en un espectrómetro GENESYS 10S UV/Vis (Thermo Scientific™). Los eluídos se almacenaron a - 20 °C.
Actividad lipolítica (AL). Se determinó en los eluídos de la columna Sephadex G-100, mediante método volumétrico (Urquijo-Rodriguez et al., 2020). Brevemente, se preparó una emulsión compuesta por aceite de canola (1,25 g), agua destilada (1,25 g), Tris 40 mmol/mL, pH 8,3 (2 mL) y Tritón X-1000 (125 g). La hidrólisis del aceite emulsificado se realizó en matraces de 125 mL, en un baño termostático Dubnoff TPA-304 (Ethik Technology) con movimiento recíproco a 200 rpm. Después de 10 min de agitación se agregaron a la emulsión 950 µL de la lipasa. La reacción se detuvo con 10 mL de etanol:acetona en relación 1:1 (v/v). Para determinar los ácidos grasos liberados en la reacción, se adicionaron 10 mL de NaOH 0,05 M y tres gotas de fenolftaleína, luego se valoró el exceso de NaOH con una solución estandarizada de HCl 0,05 M. Se incluyó un blanco de reacción por tratamiento (emulsión con la enzima desnaturalizada a 100 °C). La AL se expresó en unidades de nKatal por mililitro de extracto (nKat/mL) y en unidades actividad enzimática específica (AL e ), de acuerdo con las ecuaciones 1 y 2.
Dónde,
V b = Volumen de HCl gastado en la titulación del blanco.
V m = Volumen de HCl gastado en la titulación de la muestra.
M = Molaridad del HCl.
t = Tiempo de incubación en segundos.
V = Volumen de enzima en la reacción.
Electroforesis en gel de poliacrilamida (SDS-PAGE). Se realizó de acuerdo con el protocolo de Zienkiewicz et al. (2015), en geles de poliacrilamida (gel separador 12 %T y gel concentrador 4 %C) y un equipo Mini-PROTEAN® Tetra Cell (BioRad®). Brevemente, 10 µL de muestra (1 µg/µL de proteína) se mezclaron con 10 µL del buffer Laemmli-2X (Bio-Rad®), con 2-mercaptoetanol (condición reductora) y sin 2-mercaptoetanol (no reductora), luego se cargaron en el gel. El corrido electroforético se ejecutó a 100 V por 2 h. Los geles se tiñeron con azul brillante de Coomassie R-250 (Merck., S.A., Darmstadt, Germany), luego se visualizaron en un equipo UVP ImagingSys EC3410 LMS-26 97-0279-01 (Bio-Rad®).
Efecto de la temperatura y el pH en la AL
Se aplicó un diseño experimental 22 para establecer las condiciones de temperatura y pH que maximicen la AL de la lipasa parcialmente purificada de P. speciosa. Se evaluaron dos niveles de pH usando un tampón de acetato de sodio 0,1 M/EDTA 0,01M (pH 5) o tampón Tris 0,03 M (pH 9); también se evaluaron dos niveles de temperatura (20 y 60 °C). Estos parámetros se eligieron tomando como referencia el estudio de Peres-Polizelli et al. (2008), e tanto que la AL se evaluó siguiendo el método volumétrico descrito arriba con un tiempo de reacción de 90 min.
Soportes de inmovilización
Se sintetizaron tres soportes de inmovilización, a saber: Esferas de quitosano (Q), de acuerdo con el protocolo de Biró et al. (2018); esferas magnéticas de quitosano-Fe(OH)3 (Q-Fe), de acuerdo con Pospiskova and Safarik (2013) y, esferas de alginato recubiertas con quitosano (Alg-Q), según Bai and Wu (2021).
Caracterización de los soportes
EL tamaño y forma de los soportes se determinó en un microscopio Dinolite AD 413t I2V con iluminación azul (400 nm), infrarroja (940 nm) y magnificación óptica de 10X. Adicionalmente, la superficie de los soportes se analizó en un Microscopio Electrónico de Barrido (MEB) TESCAN® modelo Vega 3, de acuerdo con el protocolo descrito por Santos-Duarte et al. (2019).
Inmovilización de la lipasa de P. speciosa
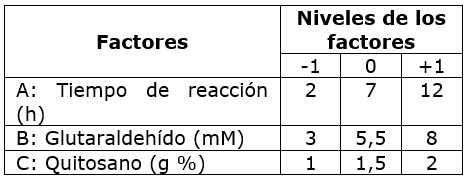
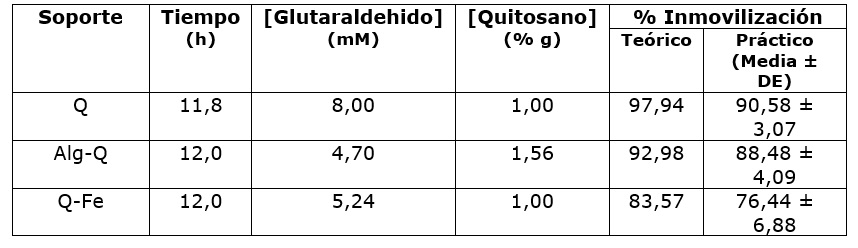
Se realizó mediante el método de entrecruzamiento covalente (Pal and Khanum, 2011). Inicialmente, las esferas se funcionalizaron con glutaraldehído y se analizaron mediante espectroscopía de infrarrojo medio con transformada de fourier (FT-MIR) en un equipo Perkin Elmer Modelo Spectrum Two 93881, midiendo el espectro de absorción de cada muestra (16 barridos/muestra) en el rango de 450 a 4000 cm-1. Luego, se optimizaron las condiciones de inmovilización de la lipasa mediante un diseño experimental de superficie de respuesta Box-Behnken con dos bloques (Cuadro 1). En cada reacción de inmovilización se utilizaron 0,2 g de esferas y 10 mL de glutaraldehído. Todas las reacciones se mantuvieron en agitación a 150 rpm por varios minutos (de acuerdo con el diseño). Finalizado el tiempo de reacción, las esferas se separaron del medio y el exceso de glutaraldehído se lavó varias veces con agua destilada. Cada soporte funcionalizado se mezcló con 5 mL de la lipasa parcialmente purificada de P. speciosa (1 mg/mL) disuelta en Tris 0,03 M (pH 9), seguido de incubación a 4 °C y agitación constante (100 rpm) durante 12 h. Finalmente, las esferas se filtraron y lavaron varias veces con el buffer Tris y se almacenaron a 4 °C. El porcentaje de proteína inmovilizada (variable respuesta) se calculó mediante la ecuación 3.
Donde, P total corresponde a la concentración de proteína total en la reacción y P lavado a la concentración de proteína en las aguas de lavado.
ALe y estabilidad de la lipasa inmovilizada
La AL e se determinó usando el método volumétrico, mezclando 3 g de la enzima inmovilizada (esferas) con 10 mL del aceite de canola emulsificado. Los resultados se expresaron en unidades nKat/mg de esferas (Ecuación 4), donde:
Para establecer la estabilidad de la lipasa inmovilizada, el sistema biocatalítico se separó de la mezcla de reacción, se lavó con agua ultrapura y solución Tris (pH 9), luego se utilizó en un nuevo ciclo de reacción hasta completar 5 ciclos. Para el cálculo de la actividad residual de la lipasa se tuvo en cuenta un 100 % de rendimiento de la AL e para el primer ciclo.
Análisis estadístico
Los ensayos de AL se realizaron por triplicado. Los resultados se presentan como valores de la media ± desviación estándar. Las diferencias entre tratamientos se evaluaron mediante ANOVA de una vía (significancia del 95 %); también, se usó la prueba post-hoc de diferencia significativa honesta de Tukey (I.C. 95 %), en el paquete estadístico RStudio versión 1.4.1106. El diseño de superficie de respuesta se realizó y analizó con el paquete estadístico Statgraphics Centurion (versión XVI 16.1.18), mientras que los gráficos se realizaron en RStudio.
RESULTADOS
Caracterización de la lipasa de semillas de P. speciosa
El rendimiento de extracción y la concentración de proteína total de las semillas de P. speciosa fue de 64±1,53 % p/p y 1,92±0,46 g/mL, respectivamente. Este resultado fue ligeramente menor al presentado por Peres-Polizelli et al. (2008) con la lipasa de semillas de P. aquatica (2,62 g/mL), usando condiciones de extracción similares, lo cual puede atribuirse a diferencia bióticas y abióticas entre las plantas utilizadas en los dos estudios.
En general, las semillas poseen alto contenido de proteínas de almacenamiento, las cuales tienen poca o ninguna actividad catalítica, por lo que es necesario reducir su proporción en el extracto y obtener fracciones ricas en lipasa para el proceso de inmovilización. La cromatografía en Sephadex G-100 logró separar dos fracciones (4 y 11) con alto contenido de proteínas (mayor absorbancia a 280 nm) y AL (Figura 1A). El análisis por SDS-PAGE de la fracción 4 con mayor AL, detectó una banda mayoritaria de 60 kDa en condición no reductora y, dos bandas de 32 y 28 kDa, en condición reductora (Figura 1B), sugiriendo que esta lipasa presenta dos cadenas polipeptídicas unidas mediante enlaces disulfuros, lo cual deberá confirmarse con análisis estructurales. Adicionalmente, el tamaño observado es cercano al reportado para otras lipasas de semillas oleaginosas (Mukherjee, 1994), incluida la lipasa de P. aquatica (55 kDa) (Peres-Polizelli et al., 2008).
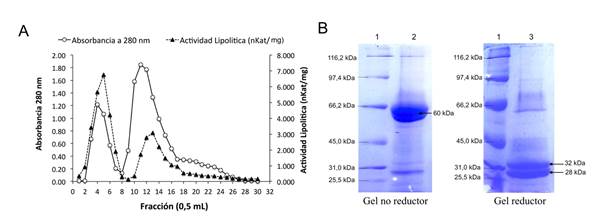
Figura 1 Caracterización de la lipasa de P. speciosa. A. Cromatografía de filtración en Sephadex G-100). B. SDS-PAGE. Carril1. Marcador de peso molecular (Broad range, Bio-Rad®); Carril 2. Gel no reductor; Carril 3: Gel reductor.
Efecto del pH y la temperatura
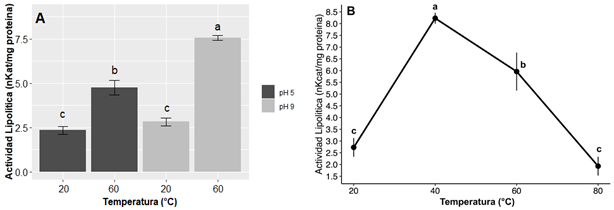
La máxima AL (18,33 ± 0,25 nKat/mL; AL e = 7,57 ± 0,33 nKat/mg) fue detectada a pH 9 y 60 °C a los 90 min de reacción (ANOVA: Razón F= 147,9; Valor p= 2,4 x 10-7). Cuando el experimento se llevó a cabo en tiempos de reacción mayores a 90 min se produjo reducción de la actividad (dato no mostrado), lo cual puede atribuirse a la acidificación del medio de reacción debido a la acumulación de ácidos grasos libres y a la posible desestabilización de la interfaz sustrato-enzima (Albayati et al., 2020). El pH del medio de reacción es uno de los factores críticos en la catálisis enzimática, debido a su influencia en el estado de ionización de las cadenas laterales de los aminoácidos y las constantes de ionización (pKa) de los aminoácidos catalíticos, relacionadas con la conformación del sitio activo y con las velocidades de reacción del enzima (Swagata et al., 2019). Por otra parte, la capacidad de esta lipasa para actuar a pH alcalino podría ampliar su abanico de aplicaciones a la biodegradación de las películas de lípidos en aguas residuales urbanas, contribuyendo a la reducción de la contaminación ambiental (Hu et al., 2018). También, como otras lipasas alcalinas, esta enzima podría usarse en la síntesis de ésteres para la industria de alimentos procesados (Cong et al., 2019).
Respecto a la temperatura, se realizó un experimento ampliando el rango de operación entre 20 y 80 °C, con intervalos de 20 °C, manteniendo fijo el pH en 9, con el propósito de establecer con mayor exactitud la temperatura optima de reacción. El análisis de ANOVA mostró que existe diferencia significativa en la AL de los tratamientos (Valor p= 1,12 x 10-6). El cambio de temperatura de 20 a 40 °C incrementó 3 veces la AL e de la enzima, no obstante, cuando la temperatura se fijó en 60 ºC la actividad decayó en aproximadamente 18 % y continuó disminuyendo a los 80 °C, resultado que permitió establecer la temperatura óptima de reacción en 40 °C con una AL e = 8,23 ± 0,23 nKat/mg (Figura 2B).
Soportes de inmovilización
Se lograron sintetizar tres soportes de inmovilización a base de quitosano. Q y Alg-Q presentaron forma esférica con diámetro de 1,5±0,14 y 2±0,2 mm, respectivamente. En el caso del Q-Fe, se obtuvo un material de color oscuro compuesto por partículas finas y agregadas de óxido de hierro con diámetros entre 30 y 120 μm. La MEB evidenció presencia de poros en la superficie de los tres soportes, los cuales fueron de forma y tamaño irregular, siendo el soporte Q-Fe el material más poroso (Figura 3). El tamaño de las esferas es uno de los parámetros importantes en la inmovilización de las enzimas; se considera que a menor diámetro, habrá mayor actividad catalítica debido a que se reduce la resistencia a la transferencia del sustrato y se incrementa el área de superficie funcional del soporte (Xie et al., 2009). Por otra parte, la porosidad y el tamaño de poro de la superficie del soporte tiene mayor influencia en los métodos de inmovilización por atrapamiento fisíco.
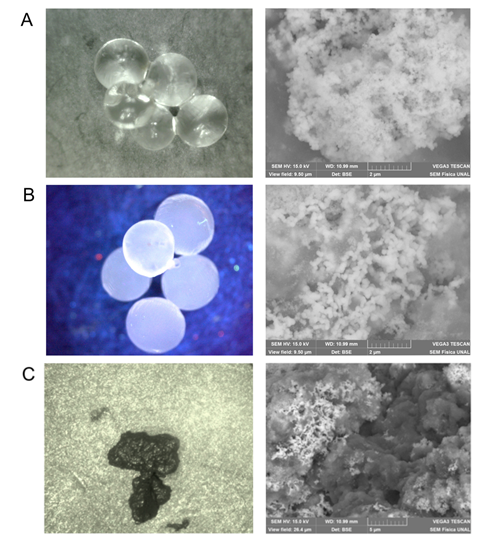
Figura 3 Soportes de inmovilización de la lipasa de P. speciosa. A. Esferas de quitosano; B. Esferas de alginato de calcio-quitosano; C. Esferas magnéticas de quitosano-Fe(OH)3.
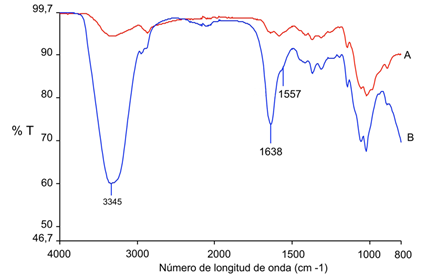
En cuanto al proceso de funcionalización, se pudo comprobar mediante FT-MIR formación de enlaces imino (C = N) y etilenico (C = C) entre el glutaraldehído y el quitosano (bandas a 1638 cm-1 y 1557 cm-1, respectivamente) en los tres soportes. La señal intensa a 3345 cm-1 corresponde al grupo OH de las moléculas de agua (Figura 4).
Inmovilización de la lipasa de semillas de P. speciosa
El cuadro 2 presenta los porcentajes de inmovilización de la lipasa de P. speciosa en los tres soportes, para cada tratamiento del diseño Box-Behnken. Los porcentajes máximos fueron de 79,70 ± 0,57 en Q; 77,1 ± 2,35 en Alg-Q y 78,60 ± 2,47 en Q-Fe.
Cuadro 2 Porcentaje de inmovilización de la lipasa parcialmente purificada de semillas de P. speciosa.
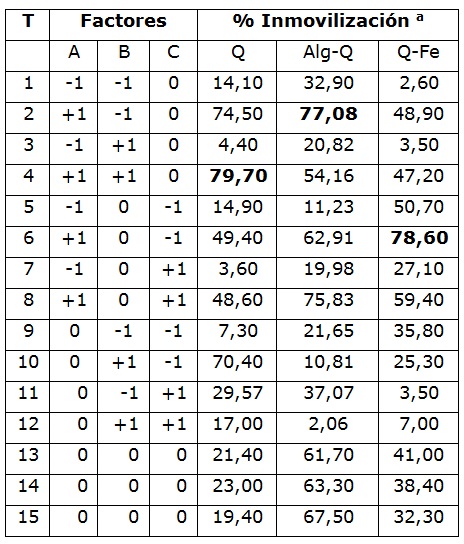
a Valores de la media de dos determinaciones para cada tratamiento; los tres últimos corresponden a los puntos centrales del modelo. A = tiempo de reacción (min); B = Concentración glutaraldehído (mM); C = Concentración quitosano (% p).
El ANOVA mostró que, para los tres soportes, el tiempo de reacción tuvo efecto significativo en la inmovilización, así: Q (ANOVA: Razón F= 35,11; Valor p= 0,0020), Alg-Q (ANOVA: Razón F= 123,45; Valor p= 0,0001), Q-Fe (ANOVA: Razón F= 86,34; Valor p= 0,0002). Las concentraciones de glutaraldehído y quitosano influyeron significativamente en la inmovilización de la enzima al Alg-Q (ANOVA: Razón F= 23,57; Valor p= 0,0047) y Q-Fe (ANOVA: Razón F= 33,39; Valor p= 0,0022), respectivamente. Con el propósito de observar el efecto de la interacción de estos factores en la inmovilización, se obtuvo el gráfico de superficie de respuesta del modelo, fijando la variable “tiempo de reacción” en el valor teórico optimizado por el modelo (12 horas), lo que evidenció que el efecto de estas variables es diferente para cada tipo de soporte (Figura 5), por lo que deberán optimizase para cada sistema biocatalítico particular.
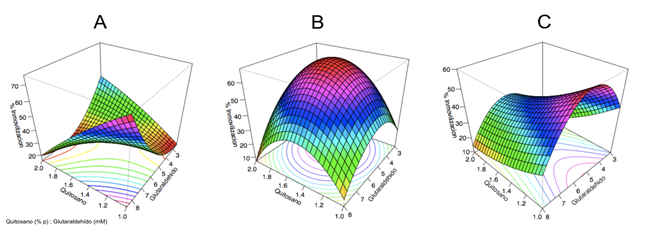
Figura 5 Superficie de respuesta para la reacción de inmovilización de la lipasa de P. speciosa en esferas de A: Quitosano; B: Alginato-quitosano; C: Quitosano-Fe(OH)3
El ajuste del modelo de superficie de respuesta se verificó a través del coeficiente de correlación (R2) que fue mayor a 0,91 (Q = 91,42 %; Alg-Q = 98,16 %; Q-Fe = 97,9 %); un R2 elevado indica que el modelo es capaz de explicar el mayor porcentaje de la variabilidad de la respuesta. Adicionalmente, el modelo arrojó una predicción de la combinación de niveles en los factores evaluados, que maximizan el porcentaje de inmovilización de la enzima en los tres soportes; estas condiciones fueron reproducidas experimentalmente produciendo resultados ligeramente más bajos a los predichos por el modelo (Cuadro 3), siendo el mayor porcentaje de inmovilización experimental el de las esferas de quitosano (90,58 ± 3,07 %).
AL de la lipasa inmovilizada
Las AL de los sistemas biocatalíticos, obtenidas bajo condiciones de inmovilización optimizada, fueron Lipasa-Q= 3,74±0,3 nKat/mg; Lipasa-Alg-Q= 3,62±0,1 nKat/mg y Lipasa-Q-Fe= 2,88±0,1 nKat/mg. El análisis de varianza mostró que existe diferencia significativa en la AL de los biocatalizadores (Valor p= 0,00019); sin embargo, la prueba post-hoc HSD Tukey (IC 95 %) indica que no hay diferencia significativa entre Lipasa-Q y Lipasa-(Alg-Q) (Valor p= 0,1225), indicando que la mayor actividad lipolítica se obtuvo con el sistema biocatalítico Lipasa-Q.
La inmovilización en materiales biopoliméricos es una estrategia que permite mejorar la actividad, selectividad, estabilidad y especificidad de las enzimas libres; no obstante, la eficacia de inmovilización depende tanto del método de inmovilización como de caracteristicas del soporte, tales como la porosidad, el área y funcionalidad superficial. En este estudio, el mayor porcentaje de inmovilización y AL se logró en esferas de Q, lo cual se atribuye al método de unión por entrecruzamiento entre los grupos -NH2 y -COO- de los residuos de aminoácidos del enzima y la superficie del quitosano funcionalizado con glutaraldehído (grupos -NH2 y -OH), que incrementa la estabilidad de la lipasa. Así mismo, el menor diámetro de la esfera, comparado con Alg-Q, pudo contribuir a la inmovilización al aumentar la superficie de contacto entre la enzima y el soporte. Eficiencias comparables (> 90 %) fueron reportados con la lipasa parcialmente purificada de Rhizopus oryzae R1 usando esferas de quitosano funcionalizadas con glutaraldehido y método de entrecruzamiento (Helal et al., 2021).
El soporte Alg-Q representa un complejo poli-electrolítico, donde las moléculas de alginato interaccionan con los grupos aminos del quitosano (Nuraliyah et al., 2021); esta interacción limita los grupos funcionales del quitosano disponibles para interactuar con el glutaraldehído y la enzima, esto podría explicar la ligera reducción de la actividad catalítica del sistema Lipasa-(Alg-Q). En contraste, los efectos de pérdida de enzima por lixiviación, ampliamente reportados en matrices de alginato (Alnoch et al., 2020), se pudieron disminuir con el recubrimiento de quitosano, lográndose porcentajes de inmovilización cercanos al obtenido con el sistema Lipasa-Q. De manera similar, Bonine et al. (2014) reportaron mejores eficiencias catalíticas de la lipasa de P. aquatica inmovilizada en esferas de alginato recubiertas con alcohol polivinílico (PVA), comparada con las esferas de alginato.
Por otra parte, el desarrollo de lipasas inmovilizadas en soportes magnéticos ofrece la ventaja de una separación rápida del enzima después de cada ciclo de reacción, lo cual reduciría el costo del biocatalizador en aplicaciones industriales; sin embargo, se ha reportado baja capacidad de reutilización de estos soportes, asociado a los métodos de inmovilización por adsorción y atrapamiento físico que permiten fuga de la enzima (Spelmezan et al., 2020). En el presente estudio se uso el método de unión covalente con glutaraldehído para reducir pérdida de la lipasa del soporte magnético. Si bien se obtuvieron buenos porcentajes de inmovilización (> 75 %), fueron inferiores a los obtenidos con Q y Alg-Q, lo que se atribuye a la agregación de las partículas de hierro magnético, causada por la mayor relación entre el área superficial y el volumen, que limita el acceso del enzima a tales interfaces causando disminución de la actividad lipolítica. Para evitar la agregación Xu et al. (2014) han propuesto el uso de tensioactivos, dispersantes naturales o distintos revestimientos poliméricos durante o después de la síntesis del soporte.
Actividad residual del sistema Lipasa-Q
Se evaluó la actividad residual del sistema biocatalítico Lipasa-(Q), debido a que este presentó el mayor rendimiento de inmovilización y AL, observándose la conservación del 85,29 % de la actividad enzimática (AL = 3,48±0,084 nKat/mg) hasta el tercer ciclo de reutilización; en el ciclo 4 fue del 63,23 % (AL = 2,58±0,09 nKat/mg) y en el 5 fue del 50,02 % (AL = 2,05±0,12 nKat/mg). Estos resultados son favorables, ya que se demuestra que un alto porcentaje de la lipasa se une al soporte en la orientación apropiada para preservar el sitio activo de la enzima, responsable de su estabilidad operativa y reusabilidad. Resultados comparables fueron descritos con la lipasa de P. aquatica inmovilizada en esferas de quitosano-PVA (Bonine et al., 2014) y con la lipasa de semillas de laurel inmovilizadas en esferas de quitosano (Yagar and Balkan, 2017). El siguiente paso será evaluar el efecto de la inmoviliación en la estabilidad a diferentes valores de pH y temperatura.
CONCLUSIONES
En este estudio se presenta un procedimiento eficaz para la obtención de un sistema biocatalítico reciclable formado por una lipasa de semillas oleaginosas de P. speciosa inmovilizada en esferas de quitosano funcionalizado con glutaraldehído, cuyas características funcionales son prometedoras a nivel biotecnológico. Teniendo en cuenta que las enzimas son catalizadores biodegradables y de menor toxicidad, el nuevo biocatalizador inmovilizado tiene el potencial de usarse en la bioconversión de aceites aplicables a la industria de los alimentos, así como también en procesos de degradación de lipidos en aguas residuales. Por otra parte, en el macrosector agroindustrial, las oleaginosas, grasas y aceites representa uno de lo sectores de mayor productividad (aproximadamente el 9 % del PIB de agroindustria en Colombia), por lo que las estrategias encamnaidas a la optimización de los procesos de bioconversión de estas materias primas serían de gran impacto para el sector. No obstante, para establecer la utilidad real del biocatalizador en procesos tanto a nivel de laboratorio como a escala industrial se requieren estudios sobre el comportamiento cinético de la enzima (libre e inmovilizada). Finalmente, el desarrollo de este biocatalizador permitirá dar un valor agregado a los frutos de P. speciosa en Colombia, con miras a generar oportunidades para el desarrollo del sector agrícola en comunidades vulnerables, debido a los pocos requerimientos del cultivo.