Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista Ciencias de la Salud
versión impresa ISSN 1692-7273versión On-line ISSN 2145-4507
Rev. Cienc. Salud v.4 supl.1 Bogotá oct. 2006
Determinación de anomalías cromosómicas y secuencias de ADN amplificadas en cáncer de mama
Establishment Of Chromosomic Abnormalities And Amplified Dna Sequences In Breast Cancer
Milena Rondón L.*, José Caicedo M.**, José Fernando Robledo
Proyecto financiado por aportes resultantes de convocatoria interna realizada en la Pontificia Universidad Javeriana. 2001.
* Química y Bióloga. MSc. Genética Humana. Unidad de Citogenética. Instituto de Genética Humana. Pontificia Universidad Javeriana.
** Cirujano de mama. Cirugía Oncológica. Clínica del Country.
† Cirujano de mama. Cirugía Oncológica. Clínica del Country. Correspondencia: Sandra Milena Rondón Lagos. Laboratorio de Biología Celular y Molecular. Universidad del Rosario. Calle 63D N 24-31. Bogotá. Colombia. E-mail: sandra.rondonla@urosario.edu.co.
Recibido: 08 de agosto de 2006. Aceptado: 29 de agosto de 2006.
Resumen
El cáncer de mama en Colombia, es la tercera causa de muerte en la población en general y la segunda en mujeres. En el año 2002 el 40.5% de los casos se presentaron en mujeres menores de 50 años (Pardo, et al. 2003). El cáncer de mama resulta de múltiples factores, entre los que se incluyen cambios sucesivos en el genoma de células epiteliales originalmente normales, que pueden conducir a la activación de oncogenes, inactivación de genes supresores de tumor y pérdida de función de genes reparadores de daños al ADN. Estas alteraciones pueden también ser producto de anomalías cromosómicas tales como monosomías, trisomías, translocaciones, inversiones, pérdida de material genético y amplificaciones que también afectan la expresión de genes (1) (2) (3) (4). Sin embargo, el orden de aparición de los diferentes eventos no está completamente dilucidado.
En este estudio se determinaron las anomalías cromosómicas y secuencias de ADN amplificadas en pacientes con cáncer de mama, tanto en muestras de sangre periférica como de tumor de mama de 30 pacientes. En las dos líneas celulares analizadas se observó una alta frecuencia de monosomías principalmente de los cromosomas X, 6, 7, 9, 17, 19 y 22. Hay una asociación entre las monosomías de los cromosomas 17 y 22 con el estado negativo para los receptores de estrógenos y progestágenos (p=0.027, p=0.050). También se encontró asociación entre la monosomía del cromosoma 19 con edad avanzada (p=0.034), observándose formas más agresivas de la enfermedad cuando ésta estuvo presente. Las monosomías fueron características de carcinomas ductales infiltrantes de todos los grados. En los demás tipos de carcinoma su frecuencia fue más baja. En el presente estudio se encontró una asociación significativa entre algunas anomalías cromosómicas y la enfermedad, no reportadas anteriormente, como fueron algunas monosomías, fragilidades y roturas cromosómicas y cromatídicas. La alta frecuencia de fragilidades encontradas tanto en sangre periférica (fra 9q12 p=0.001 y fra 3p14 p= 0.38) como de fragilidades expresadas espontáneamente (no inducidas por el uso de reactivos específicos) en muestras de tumor de mama (fra 1p11 p= 0.001, fra 2q11 p= 0.002), pueden ser el reflejo de una alta inestabilidad cromosómica en el genoma de estos pacientes, mostrando la utilidad de los estudios de fragilidad en la determinación de individuos en alto riesgo de desarrollar cáncer de mama. En ensayos de FISH no se observaron amplificaciones de los genes ERBb2 y c-myc en los pacientes analizados. Esto concuerda con lo encontrado en la literatura en donde se ha reportado, para este tipo de tumores, una sobre expresión de la proteína sin amplificación del gen, explicada por desregulación de la expresión del gen, a su vez posiblemente debida a mutaciones en la región promotora o a alteraciones, que conducen a un aumento de la tasa de transcripción (5) (6) (7). Los resultados obtenidos, aunque preliminares, aportan nuevos marcadores cromosómicos que pueden orientar el diagnóstico, pronóstico y tratamiento de esta patología.
Palabras Claves: Cáncer de mama, Anomalías cromosómicas, Citogenética, FISH, ERBb2, C-MYC.
Abstract
Breast cancer is the third cause of death in the general population and the second one in Colombian women. In 2002, 40.5% of the cases were of women younger than 50. Breast cancer originates as a result of multiple and consecutive changes in the genome of normal epithelial cells, such as mutations, oncogene activation, inactivation of tumor suppression genes and of DNA damage repairing genes. The latter, can have its origin in chromosomal abnormalities such as monosomies, trisomies, translocations, inversions, loss of genetic material and amplifications, which may lead to over-expression of oncoproteins related with a greater risk of tumor progression (1) (2) (3) (4). However, very little is known about the sequence in which the different types of alterations of the genome appear. In the present study, the chromosomal abnormalities and amplified DNA sequences were established in breast cancer patients in both samples of peripheral blood and of tissue from the breast tumor of 30 patients. A high frequency of monosomies, specially those of the chromosomes X, 6, 7, 9, 17, 19 and 22 were observed, with statistically meaningful differences between the monosomies of the chromosomes 17 and 22 and the negative STATE for the estrogen and progesterone receptors (p=0.027, p=0.050) and between the monosomy of the chromosome 19 with an advanced age (p=0.034), indicating more aggressive forms of the disease. The monosomies were characteristic of ductal infiltrating carcinomas of any stage and they were observed in a low frequency in other types of carcinomas. Although relationship between the monosomies, fragilities and BREAKINGS of the chromosome 9 with breast cancer has not been reported by previous studies, these chromosomal abnormalities were found in our study in a representative manner and this finding could constitute a new risk marker in this type of cancer. The high frequency and significance of the fragilities found in peripheral blood and (fra 9q12 p=0.001 y fra 3p14 p= 0.38) those of fragilities spontaneously expressed (not induced by the use of specific REACTIVOS) in breast tumor samples (fra 1p11 p= 0.001, fra 2q11 p= 0.002) may somehow confirm the high chromosomal instability in the genome of these patients, probably allowing these fragility tests to be useful in the early determination of the individuals with a high risk of developing breast cancer. Amplification of the ERBb2 and c-myc genes was not observed in FISH assays in any of the analyzed patients; this agrees with related research, in which an over-expression of the protein without an adequate amplification of the gene has been found in this type of tumors, probably due to mutations in the promoter region and an increase in the transcription rate without there being an amplification of the gene (5) (6) (7). The obtained results, although preliminary, are important due to the fact that they contribute to the search of new chromosomal markers and are also important to the orientation of the diagnosis, prognosis and treatment of this disease.
Keywords: Breast cancer, chromosomical abnormalities, Cytogenetics, FISH, ERBb2, CMYC.
1. INTRODUCCIÓN
El cáncer de mama es una enfermedad que representa uno de los mayores problemas de salud en Colombia, siendo la tercera causa de muerte por cáncer en la población general y la segunda en mujeres. Según estadísticas establecidas por el Instituto Nacional de Cancerología, en el 2002 se registraron 608 casos de cáncer de mama, de los cuales 2 casos se presentaron en hombres y 606 en mujeres, el promedio de edad de aparición de la enfermedad fue de 53,9 años donde el 40,5% de las mujeres eran menores de 50 años (8). Los cambios genéticos que derregulan los mecanismos de la muerte celular o apoptósis, como la adhesión celular, la proliferación celular, este último muy relacionado con el ciclo celular, se asocian frecuentemente con el desarrollo del cáncer. Entre los genes más frecuentemente alterados están los proto-oncogenes, los genes supresores de tumor y los genes de reparación de daños en el ADN. La activación, inactivación o pérdida de función de estos genes puede ser el resultado de una predisposición genética combinada con factores ambientales y/o de alteraciones cromosómicas como monosomías, trisomías, translocaciones, inversiones, deleciones o amplificaciones génicas que incrementan la inestabilidad genómica. Estudios citogenéticos muestran que las anomalías numéricas más comunes en tejido tumoral de pacientes con cáncer de mama, son: la trisomía de los cromosomas 3, 7, 8, 18, 12, 20, X y la monosomía de los cromosomas 1, 8, 9, 13, 14, 17, X, 2, 6, 18, 12, 19 y 22 (9), siendo las monosomías identificadas como eventos tempranos y comunes en cáncer de mama. Los carcinomas intraductales con neoplasia invasiva presentan un alto grado de monosomías comparadas con carcinomas ductales in situ (10). Recientes investigaciones señalan a los cromosomas 1 y 17 como portadores de biomarcadores de estados premalignos (4) (11) (12), debido a que en éstos se localizan genes supresores de tumor y genes reparadores de daños al ADN. Ambos han sido implicados tanto en el desarrollo de cáncer de seno como en asociaciones con cambios numéricos específicos para esta neoplasia, tales como ARHC, P73, MUC1 y KISS1 en el cromosoma 1 y en el cromosoma 17 el oncogen ERBb2 (13) (14) (15), los genes supresores de tumor BCPR y NME1 y los genes reparadores de daños al ADN: p53 y BRCA1. De allí la importancia de estos genes en el desarrollo y/o progresión del cáncer de mama, ya que al no estar presentes, los puntos de chequeo o de control del ciclo celular no se llevan a cabo correctamente, permitiendo el crecimiento celular descontrolado.
2. MATERIALES Y MÉTODOS
2.1 Población de Estudio
Se recolectaron muestras de tejido tumoral y sangre periférica de 30 pacientes con cáncer de mama diagnosticado sin previa caracterización histopatológica, sin restricción de edad, sexo, raza y sin tratamiento previo (quimioterapia, radioterapia o terapia hormonal). Los pacientes fueron remitidos de las clínicas del Country, Reina Sofía, Palermo y el Bosque de la ciudad de Bogotá. Previamente se les determinó el estado de los receptores hormonales (estrógenos y progestágenos positivos o negativos) y la presencia de la oncoproteína ERBb2 (sobre muestras de tejido) por inmunohistoquímica. Como grupo control se congregaron 24 mujeres sanas entre los 19 y 35 años de edad, con estudio de fragilidad en muestras de sangre periférica.
2.2 Procedimiento
2.2.1 Análisis por Citogenética Convencional
Los análisis citogenéticos para la determinación de anomalías cromosómicas se realizaron en sangre periférica y en muestras de tejido tumoral.
Se tomaron 5 ml de sangre con heparina como anticoagulante. Las muestras fueron cultivadas por duplicado a 37°C por 72 horas en medio RPMI 1640, suplementado con 10% de suero fetal bovino y 50 ul de fitohemaglutinina. La expresión de sitios frágiles se indujo mediante el uso de metotrexate por 17 horas. Para bloquear la citocinesis de las células en división se adicionó colchicina (0.0001 grs/ml). Los linfocitos fueron cosechados después de 72 horas de cultivo. Las células se trataron con solución hipotónica (KCl 0.075 M) para lisar los eritrocitos, fueron prefijadas en solución 3:1 de metanol: ácido acético (fijador carnoy), lavadas tres veces con fijador carnoy y extendidas sobre láminas de vidrio. Después de secar al aire, la mitad de las láminas se almacenó a –20°C para los análisis posteriores con FISH. Las demás láminas fueron teñidas con solución colorante giemsa para el análisis de fragilidad y anomalías cromosómicas.
Las muestras de tejido tumoral fueron disgregadas mecánicamente y sembradas en medio Leibovitz L15, enriquecido con suero fetal bovino al 20% y llevadas a 37ºC y 5% de CO2, hasta su crecimiento. Los preparados cromosómicos se obtuvieron usando métodos de rutina en el laboratorio (Laboratorio de Citogenética, Instituto de Genética Humana, Universidad Javeriana). La mitad de las placas se almacenaron a - 20ºC para los análisis con FISH, las demás fueron teñidas con giemsa para el correspondiente análisis cromosómico.
2.2.2 Análisis por FISH
En los preparados cromosómicos, almacenados a -20°C, se escogieron zonas que tuvieran buen número de metafases y de núcleos interfásicos para hibridación con las respectivas sondas. Para analizar el gen ERBb2 se usó una sonda dual LSI Her 2-neu /CEP 17 (Vysis 30-161060) específica para el centrómero y la región 17q12 del cromosoma 17 (PathVysion). También se evaluó el gen c-myc usando una sonda de secuencia única LSI c-myc (Vysis 32-190006) específica para la región 8q24. La sonda dual CEP 17 (marcada con espectro verde) y la sonda LSI Her 2 – neu (marcada con espectro naranja), así como la sonda LSI c-myc (marcada con espectro naranja) fueron incubadas con las láminas durante una noche, sobre las zonas seleccionadas. La hibridación y los lavados post-hibridación fueron realizados de acuerdo con las instrucciones de la casa comercial.
Después de los lavados post hibridación, las señales verdes y naranjas fueron visualizadas por espectroscopia de fluorescencía usando un filtro triple V2 para visualización simultánea de ambas señales.
Para la evaluación de las señales se usaron los siguientes criterios: núcleos no superpuestos, señales claramente separadas unas de otras y señales con la misma intensidad de fluorescencia. Se contaron las señales fluorescentes presentes en 100 núcleos interfásicos y 50 metafases seleccionadas al azar, donde se considero a un paciente como positivo para la amplificación cuando al menos el 40% de los núcleos y/o metafases presenten más de 2 señales.
2.3 Análisis Estadístico
Se aplicó la prueba de independencia de ji (X2) cuadrado a las anomalías numéricas, anomalías estructurales, fragilidades y roturas de más alta frecuencia para muestras de sangre periférica y tejido tumoral, así como para establecer la correlación entre las mismas con algunas características clínico patológicas tales como estado positivo o negativo de los receptores hormonales (estrógenos y progestágenos), edad, grado de desarrollo tumoral (Clasificación del grado nuclear de Bloom y Richardson en 1957) (16) y estado de la oncoproteína ERBb2. Valores de p por debajo de 0,05 fueron considerados como significativos en todas las pruebas.
3. RESULTADOS
3.1 Citogenética Convencional
Tanto en las muestras de sangre periférica como en las de tejido tumoral en los 30 pacientes analizados, se encontraron anomalías de tipo numérico, tales como monosomías, trisomías, poliploidías y endorreduplicaciones, anomalías estructurales tales como deleciones, derivados, inversiones, duplicaciones y asociaciones de satélites, así como fragilidades, roturas cromosómicas y cromatídicas.
3.1.1 Anomalías Numéricas:
La monosomía más frecuente en muestras de sangre periférica fue la del cromosoma X (45, X, -X) identificada en 14 pacientes (46.6%). En 8 pacientes (26.6%) se observaron 22 metafases con trisomías, siendo la más frecuente la del cromosoma X (47, XX, +X).
En tejido tumoral en 10 pacientes (33.3%) se identificaron monosomías de los cromosomas X y 19. En 4 pacientes (13.3%) se encontraron 53 trisomías, más frecuentemente las de los cromosomas 8 y 10. Las monosomías en general, fueron muy frecuentes tanto en muestras de sangre periférica como de tumor y se presentaron principalmente en los pacientes que tenían carcinomas ductales infiltrantes de todos los grados. La monosomía del cromosoma X fue la más frecuente en todos los estadios de desarrollo de la enfermedad y las poliploídias sólo se observaron en estadios tempranos G1 y G2 (Clasificación del grado nuclear de Bloom y Richardson en 1957) (16) pero no en tardíos. En total se encontraron 114 poliploidías: en 25 pacientes (83.3%) se identificaron 74 – 92, XXXX <4n> (111), 60 – 67, XXX <3n> (3), y en 10 pacientes (33.3%) se describieron 21 endorreduplicaciones (end 46, XX). Las monosomías de los cromosomas X, 4, 6, 7, 9, 10, 12, 17, 19 y 22 fueron más frecuentes en muestras de tejido tumoral que en muestras de sangre periférica siendo esta diferencia significativa estadísticamente. Cuando se analizó el grupo control y el grupo de pacientes se observaron diferencias significativas: en los pacientes se presentan con mayor frecuencia monosomías de los cromosomas 4, 9, 10, 17, 19 y 22, y trisomías de los cromosomas 8 y 10, así como endorreduplicaciones y poliploidías. Todas las alteraciones se presentaron en mayor número en muestras de tejido tumoral respecto a las muestras de sangre periférica (Tabla 1. Figura 1.).
3.1.2 Anomalías Estructurales:
Tanto en muestras de tejido tumoral como de sangre periférica se encontraron deleciones, derivados, duplicaciones, translocaciones y cromosomas dicéntricos. También se observó una gran proporción de anomalías únicas. El tipo de anomalía más frecuente fue la deleción en un 60.37%, seguida por los derivados en un 26.41%. Aunque no se observó la misma anomalía estructural en más de un paciente, se observaron regiones similarmente afectadas tanto en el tejido tumoral como en sangre periférica. En el tejido tumoral fue más frecuente la deleción Xq23 (p=0.036), descrita anteriormente como una anomalía estructural característica de este tejido (Figura 2).
3.1.3 Fragilidades:
Además de las anomalías cromosómicas encontradas se observó una alta frecuencia de fragilidades y roturas tanto en muestras de sangre periférica (inducidas mediante el uso de medios deficientes en ácido fólico) como en el tejido tumoral (expresadas espontáneamente).
En sangre periférica se encontraron un total de 141 fragilidades, las más frecuentes fueron 9q12, 3p14, 1p11 y 16q24. Ningún paciente presentó fragilidades en los cromosomas 14, 15, 20 y 21. Los demás cromosomas presentaron fragilidades en muy baja frecuencia. En muestras de tejido tumoral, las fragilidades más frecuentes fueron 1p11, 1q11, 2q11, 6p11 y 19p11.
El análisis de fragilidades muestra que hay diferencias significativas entre el número de afectados que portan las fragilidades 1p11, 1q11, 1q12, 2q11, 9p11 y 9q12. Fragilidades a nivel del cromosoma 9(9q12) son más frecuentes en muestras de sangre periférica, mientras que las fragilidades en los cromosomas 1(1p11-q11-q12), 2(2q11) y 19(19p11) son más frecuentes en muestras de tejido tumoral. (Tabla 2. Figura 3.).
En el grupo control no se observaron anomalías numéricas ni estructurales, se encontró un bajo porcentaje de fragilidades comparadas con las observadas en sangre periférica de pacientes. Por análisis de regresión lineal se mostró que no hay asociación entre la edad de las pacientes y del grupo control y las anomalías cromosómicas. Pero si se evidenció que hay diferencia significativa (sobre mitosis analizadas) para la fragilidad en el cromosoma 9 (9q12) entre el grupo de pacientes y de controles siendo éste un sitio frágil específico para muestras de sangre periférica de los pacientes con cáncer de mama. (Tabla 3).
Se obtuvo una correlación, entre el estado negativo del receptor para estrógenos con la monosomía del cromosoma 17 (p =0.0272) y con la monosomía del cromosoma 22 (p =0.05). Igualmente se encontró correlación estadísticamente significativa entre la monosomía del cromosoma 22 con el estado del receptor negativo para progestágenos (p=0.05). El grupo etáreo entre 60 y 70 años presentó con mayor frecuencia (p=0.0349) la monosomía del cromosoma 19.
Al evaluar la expresión de la oncoproteína ERBb2, no se obtuvo correlación entre el grado de desarrollo tumoral y la presencia de las monosomías de los cromosomas X, 17, 19 y 22. (Tabla 4.)
3.2 Citogenética Molecular
No se observó amplificación de los genes que codifican para ERBb2 (en 18 de pacientes) y cmyc (10 pacientes) en 100 núcleos y 50 metafases analizadas con las respectivas sondas por FISH. (Figuras 4 y 5).
4. DISCUSIÓN
En la población colombiana estudiada, la frecuencia de anomalías encontradas en muestras de tejido tumoral de mama muestra que las monosomías de los cromosomas X, 17, 19 y 22 pueden ser posibles candidatos a marcadores cromosómicos asociados con el desarrollo temprano de la enfermedad.
La presencia de monosomías de los cromosomas X, 17, 19 y 22 tanto en sangre periférica como en tejido tumoral fue característica en los pacientes con cáncer de mama, lo que permite pensar que éstas pueden llegar a ser marcadores cromosómicos de la enfermedad relacionados con el desarrollo temprano, diferenciación y comportamiento biológico del cáncer de mama debido a que estas monosomías se encontraron en tipos específicos de carcinomas y en grados tumorales tempranos. Teniendo en cuenta además que estos cromosomas contienen genes supresores de tumor como por ejemplo en el cromosoma X, el gen ODZ1 (Xq23), que participa en la activación de la apoptosis y en el cromosoma 17 el gen supresor de tumor LDOC1 (Xq23), el oncogen ERBb2 (17q11), y los genes p53 (17p13) y BRCA1 (17q21) que participan en procesos de reparación del ADN. También en esta región se ubican los genes supresores de tumor BCPR (17p13.3) y NME1 (17q21.3). En el cromosoma 19 (19p13.3) están los genes supresores de tumor PJS, SAFB y en el cromosoma 22 el gen CHK2 (22q12.1), que también contribuye a la reparación de daños del ADN y el gen supresor de tumor EP300 (22q13).
Todos los genes anteriores han sido relacionados con la neoplasia estudiada y su alteración podría estar confiriendo a estas células tumorales ventajas en el crecimiento y por ende favoreciendo la aparición de la misma. Al no estar presentes o no ser funcionales el chequeo o control del ciclo celular y los mecanismos celulares relacionados con la carcinogénesis, puede producirse el crecimiento celular descontrolado. De allí probablemente la alta frecuencia de éstas en los pacientes, por lo que podría tener un valor de pronóstico en el desarrollo del cáncer de mama (17) (18) (19). De la misma forma, podrían constituirse como marcadores de diagnóstico citogenético tanto en muestras de sangre periférica como de tumor de mama, para detectar tempranamente individuos en alto riesgo de desarrollar neoplasias quienes podrían beneficiarse de intervenciones preventivas.
El análisis de la asociación de la presencia de monosomías de los cromosomas 17, 19 y 22 con la ausencia de receptores hormonales (estrógenos) y edad, mostraron una correlación positiva, concordando totalmente con investigaciones realizadas en cáncer de mama, en las que se encontraron correlaciones positivas entre las monosomías del cromosoma 17 con el estado negativo para el receptor de estrógenos (4), y en las que no hubo correlaciones positivas entre la monosomía del cromosoma 17 con la sobre-expresión de la oncoproteína ERBb2 (13). También estos resultados son acordes, en su gran mayoría, con otras estudios donde se relaciona la presencia de monosomías de los cromosomas X, 17, 19 y 22 con pronósticos pobres en el transcurso de la enfermedad, y el papel de ciertos genes en el desarrollo y progresión tumoral con cambios numéricos en pacientes afectados (18) (3) (21) (12) (19) (20) (3).
De la misma forma otras anomalías a tener en cuenta como posibles marcadores cromosómicos de progresión tumoral son las poliploidías y las endorreduplicaciones. Ambas fueron encontradas en el 76.6% de los pacientes en muestras de tumor de seno y en 33.3% de los pacientes en sangre periférica en todos los tipos de carcinomas y en todos los grados tumorales pero en mayor frecuencia en grados avanzados (G3) (Clasificación del grado nuclear de Bloom y Richardson en 1957), por lo que podrían estar implicadas en el desarrollo tumoral y ser indicativas de mal pronóstico relacionado con la progresión tumoral. Lo anterior debido talvez a que el aumento del número de copias de cromosomas podría facilitar la expresión de oncogenes que promueven el crecimiento celular descontrolado.
Aunque la mayoría de investigaciones reportan acumulación de anomalías cromosómicas en estadios tumorales más avanzados y en nuestro estudio los resultados obtenidos señalan lo contrario, estos hallazgos se podrían explicar por la heterogeneidad de la enfermedad, la cual se puede originar por diferentes vías donde pueden originar de igual manera su progresión. (18) (Figuras 6 y
).
Los resultados relacionados con la ausencia de amplificaciones de los genes ERBb2 y c-myc, por FISH, muestran gran concordancia con estudios de sobre expresión (2+) de la proteína ERBb2 con inmuno-histoquímica y la amplificación de este gen, en donde se ha encontrado que algunos pacientes presentan sobre expresión de la proteína sin amplificación del gen (5) (6) (7) (8) similar a nuestro estudio donde no se encontró amplificación del gen ERBb2 en ninguno de los 7 pacientes con sobre expresión de la proteína determinada por inmunohistoquímica, en 5 de los cuales la sobre expresión de la proteína fue de 2+ (2) (14) (22) (23) (24) (25) (7), siendo esto muy frecuente en estos tumores, debido posiblemente a mutaciones en la región promotora de este gen que conlleva a la alteración en la proteína sin alteración del gen. La alta frecuencia de fragilidades y roturas observadas en nuestro estudio pueden ser consideradas como indicadores de inestabilidad genética y como posibles marcadores de predisposición y de progresión tumoral debido a que además de ser observadas en las dos líneas celulares estudiadas se encontraron, en su mayoría en regiones donde se localizan genes supresores de tumor y genes reparadores de daños al ADN responsables de la adquisición del fenotipo neoplásico siendo significativas las fragilidades en los cromosomas 1p11, 2q11, 9q12, 2 p11 y 3p14, donde las fragilidades en los cromosomas 9q12 y 3p14 en sangre periférica y 1p11 en tumor de seno se encontraron en grados tumorales avanzados. La expresión de sitios frágiles ya ha sido reportada específicamente en muestras de tumor de mama y la importancia de estudios en este tipo de neoplasia ha sido muy bien argumentada (23). Otro hallazgo interesante, es la alta expresión espontánea de sitios frágiles en muestras de tumor de mama, ya que su frecuencia fue similar a la encontrada en sangre periférica de pacientes donde fueron inducidos por exposición de las células a medio deficiente en ácido fólico. La expresión espontánea de estos sitios frágiles puede conducir a la pérdida de material cromosómico o a mutaciones de genes localizados en estas regiones y podrían ser útiles en la determinación de la predisposición genética al cáncer de mama y en el diagnóstico temprano del mismo. La alta expresión de fragilidades y la presencia de roturas encontradas en el cromosoma 9 en muestras de sangre periférica de los pacientes comparada con el grupo control, constituyen otro nuevo hallazgo que podría explicar la alta inestabilidad cromosómica en pacientes con cáncer de mama y ser considerado entonces como un marcador de predisposición a esta neoplasia. En el cromosoma 9 se encuentran los genes supresores de tumor CDKN2A (9p21), PIPSK1B, BTEB1, RECK y BAG1, este último con funciones antiapoptóticas y sobre expresado en carcinomas invasivos de seno.
Los resultados obtenidos en este estudio relacionados con la correlación entre anomalías cromosómicas y características clinicopatológicas, como tipo de carcinoma y grado tumoral, constituyen una base importante de investigación en Cáncer de Seno en Colombia, por esta razón es importante realizar el estudio en muestras poblacionales mayores para relacionar los hallazgos encontrados con el diagnóstico, pronóstico y tratamiento de esta patología.
AGRADECIMIENTOS
Los autores expresan sus agradecimientos al personal de las clínicas del Country, Palermo y Reina Sofía por su colaboración en la consecución de las muestras, a la Fundación Santafé y a Cecolfes por su apoyo en el uso de equipos y a los pacientes quiénes con su participación hicieron posible la realización del presente estudio.
Tabla 1. Prueba de X2 para anomalías numéricas en muestras de Sangre Periférica (SP) y Tumor de Seno (TS) determinadas sobre número de pacientes
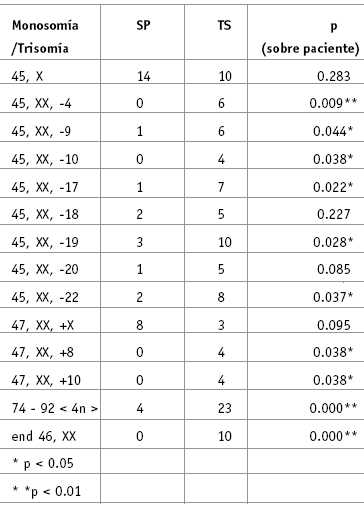
Tabla 2. Prueba de X2 para fragilidades más frecuentes en muestras de Sangre Periférica (SP) y Tumor de Seno (TS) determinada sobre número de pacientes afectado
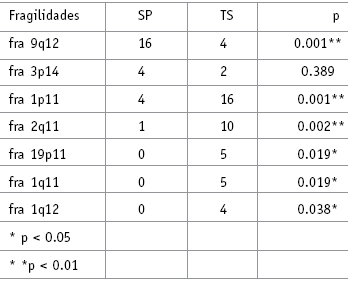
Tabla 3. Prueba de X2 para fragilidades más frecuentes en muestras de Sangre Periférica (SP) de pacientes con cáncer de seno e individuos control
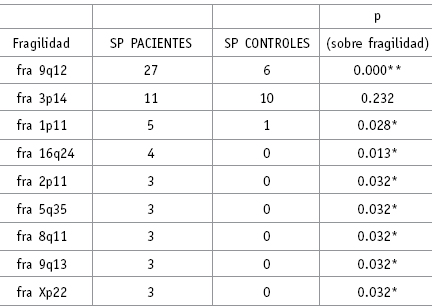
Tabla 4. Correlaciones entre Características Clinicopatológicas y Anomalías Cromosómicas en Muestras de Tumor de Seno.
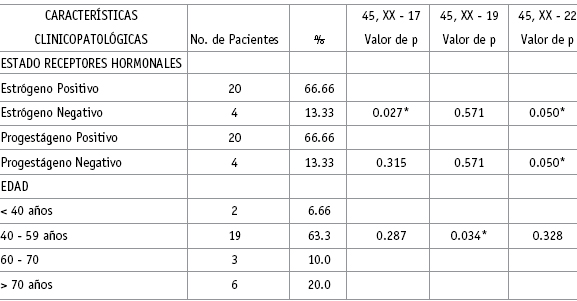
Figura 1. Cariotipo representativo con Monosomía del Cromosoma X (45, X,-X)
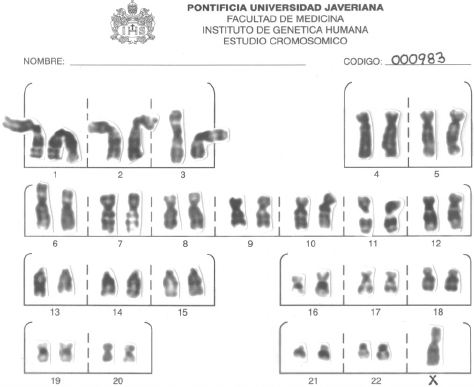
Figura 2. Cariotipo representativo con deleción del cromosoma X (46, X, del Xq23)
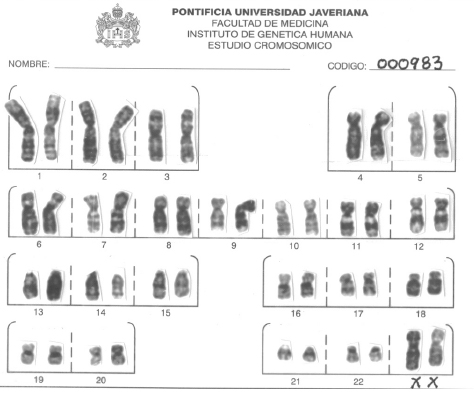
Figura 3. Cariotipo representativo con fragilidad del cromosoma 9 (fra 9q12)

Figura 4. Núcleos Interfásicos con FISH sonda dual (LSI ERBb2/CEP 17) para el gen ERBb2 (17q21-q22) (señal roja) y centrómero del cromosoma 17 (señal verde). Sin amplificación
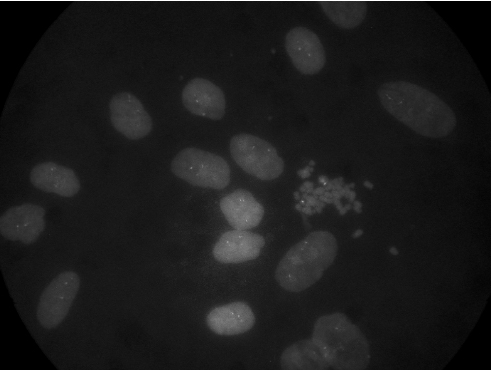
Figura 5. Núcleos interfásicos con FISH. Sonda LSI c-myc para el gen c-myc (8q24). Sin amplificación
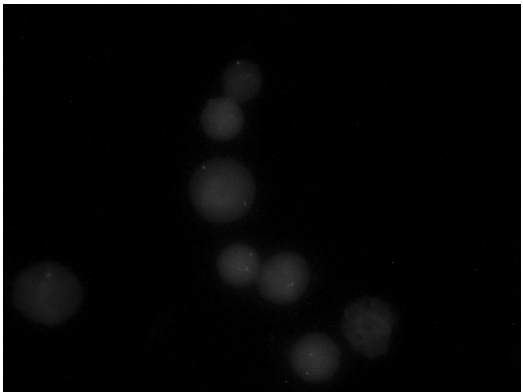
Figura 6. Porcentaje de anomalías cromosómicas, fragilidades y roturas en los diferentes tipos histológicos (CDI: Carcinoma Ductal Infiltrante; CLI: Carcinoma Lobular Infiltrante; CCI: Carcinoma Canalicular Infiltrante; CMA: Carcinoma Medular Atípico; CPI: Carcinoma Papilar Infiltrante) y grado tumoral (G1: Grado tumoral 1; G2: Grado tumoral 2; G3: Grado tumoral 3) en muestras de Tumor de seno
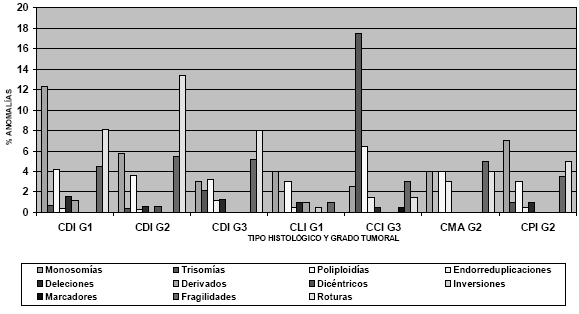
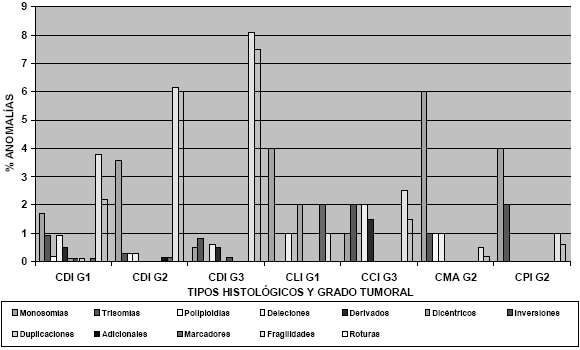
Figura 7. Porcentaje de anomalías cromosómicas, fragilidades y roturas en los diferentes tipos histológicos (CDI: Carcinoma Ductal Infiltrante; CLI: Carcinoma Lobular Infiltrante; CCI: Carcinoma Canalicular Infiltrante; CMA: Carcinoma Medular Atípico; CPI: Carcinoma Papilar Infiltrante) y grado tumoral (G1: Grado tumoral 1; G2: Grado tumoral 2; G3: Grado tumoral 3) y grado tumoral en muestras de Sangre periférica.
REFERENCIAS
1. Teixeira, M., Pandis, N. and Heim, S. Cytogenetic Clues to Breast Carcinogenesis. Genes, Chromosomes and Cancer. 2002; 33: 1-16. [ Links ]
2. Pérez, E., Roche, P., Jenkins, R., Reynolds, C., Halling, K., Ingle, J. and Wold, l. HER2 Testing in patients with Breast Cancer: Poor Correlation Between Weak Posivity by Immunohistichemistry and Gene Amplification by Fluorescence In Situ Hybridization. Mayo Clin Proc. 2002; 77: 148-154. [ Links ]
3. Tanja, F., Morrison, I., Saboorian, H., Hyan, l., Tucker, T. and Uhr, J. Patterns of aneusomy for three chromosomes in individual cells from breast cancer tumors. Breast Cancr Research. 2002; 75:227-239. [ Links ]
4. Watter, A., Going, J., Cooke,T. and Bartlett, J. Chromosome 17aneusomy is associated with poor prognostic factors in invasive breast carcinoma. Breast Cancer Research and Treatment. 2003; 77: 109-114. [ Links ]
5. Press, MF., Pike, MC., Chazin, VR., Hung, G., Udove, JA., Markowica, M. HER-2/neu expression in node negative breast cancer; direct tissue quantitation by computerized image analysis and association of over-expression with increased risk of recurrente disease. Cancer Research. 1993; 53: 4960-4970. [ Links ]
6. Robertson, KW., Reeves, JR., Smith, G., Keith, WN., Ozanne, BW., Cooke, TG. Quantitative estimation of growth factor receptor and c-erbB-2 in human breast cancer. Cancer Research. 1996; 56: 3823-3830. [ Links ]
7. Riou, G., Mathieu, M., Barrois, M., Le Bihan, M., Ahomadegre, J., Benard, J. and Le, M. C-erbB-2 (HER- 2/NEU) gene amplification is a better indicator of poor prognosis than protein over-expression in operable breast cancer patients. Int. J. Cancer. 2001; 95: 266-270. [ Links ]
8. Pardo, Constanza, Murillo, Raúl, Piñeros, Mario y Castro, Miguel Ángel. Casos Nuevos de Cáncer en el Instituto Nacional de Cancerología, Colombia, 2002. Revista Colombiana de Cancerología. 2003; 7 (3): 4-19. [ Links ]
9. Mark, HF., Browon, S., Taylor, W. Study of chromosome 12 copy number in breast cancer using fluorescence in Situ hibridization. Cancer Genet Cytogenet. 1999; 108: 26-31. [ Links ]
10. Visscher, DW., Wallis, TL., Crissman, JD. Evaluation of chromosome aneuploidy jn tissue sections of preinvasive breast carcinomas using intherphase cytogenetics. Cancer. 1996; 77: 315-320. [ Links ]
11. Botti, C., Pescatore, B., Mottolese, M., Sciarretta, F., Greco, C., Difilippo, F., Gandolfo, G., Cavaliere, F., Bovani, R., Varanese, A., Cianciulli, A. Incidence of Chromosomes 1 and 17 aneusomy in breast cancer and adjacent tissue and intherface cytogenetic study. J Am Coll Surg. 2000; 190: 530-9. [ Links ]
12. Fehm, T., Morrison, L., Saboorian, H., Hynan, L., Tucker, T. and Uhr Jonathan. Patterns of aneusomy for three chromosomes in individual cells from breast cancer tumors. Breast Cancer Research. 2002; 75: 227-239. [ Links ]
13. Bankfalvi, A., Simon, R., Brandt, B., Burguer, H., Vollmer, I., Dogckhorn – Dwornczak, B., Lellé, R., Boecker, W. Comparative Methodoligical Analysis of erbB – 2/ Her – 2 gene dosage, chromosomal copy number and protein overexpression in breast carcinoma tissues for diagnostic use. Histopathology. 2000; 37: 411-418. [ Links ]
14. Carr, J., Havstad, S., Zarbo, R., Divine, G., Mackowiak, P. and Velanovich, V. The Association of HER- 2/neu Amplification UIT Breast Cancer Recurrence. Arch Surg. 2000; 135: 1469-1474. [ Links ]
15. CM, Ellis, MJ, Dyson, TJ, Stepheson, EL, Maltby. HER-3 amplification status in breast cancer: a comparison between immunohistochemical staining and fluorescence in situ hybridisation using manual and automated quantitative image analysis scoring techniques. J.Clin. Pathol. 2005; 58: 710-714. [ Links ]
16. Bloom, HJG, Richardson, WW. Histologic grading and prognosis in breast cancer: A study of 1709 cases of which 359 have been followed for 15 years. Br J Cancer 1957; 2: 353-77. [ Links ]
17. O¢conell Peter. Genetic and Cytogenetic analyses of breast cancer yield different perspectives of a complex disease. Breast Cancer Research and Treatment. 2003; 78: 347-357. [ Links ]
18. Buerger, H., Mommers, E., Littmann, R., Simon, R., Dialio, R., Poremba, C., Dockhom, M., Diest, P. and Boecker, W. Ductal Invasive G2 and G3 carcinomas of the breast are the stages of at least two different lines of genetic evolution. J Pathol. 2001; 194: 165-170. [ Links ]
19. Isola, J., Visakorpi, T., Holli, K. and Kallioniemi, O. J. Natz Cancer Inst. 1999; 84: 1109. [ Links ]
20. Piao, Z. and Malkhosyan, S. Frequent Loss Xq25 on the Inactive X chromosome in Primary Breast Carcinomas Is Associated With Tumor Grade and Axillary Lymph Node Metastasis. Genes, Chromosomes and Cancer. 2002 ;33: 262-269. [ Links ]
21. Hans-Anton, L., Jacobs, W., Yaziji, H., Schnitt, S. and Gown, A. Quantitative Evaluation of Her-2/neu Satatus in Breast Cancer by Fluorescence In Situ Hybridization and by Immunohistochemistry With Image Analysis. Anatomic Pathology. 2001; 115: 814-822. [ Links ]
22. Pauletti, G., Dandekar, S., Rong, HM., Ramos, L., Penh, H., Seshadri, R. Assessment of methods for tissue based detection of the HER-2/neu alteration in human breast cancer: a direct comparasion of fluorescence in situ hybridization and immunohostochemistry. J Clin Oncol. 2001; 18: 3651-3664. [ Links ]
23. Cecener, G., Egeli, U., Tsdelen, I., Tunca, B., Duman, Hakan. and Kizil, A. Common Fragile Site Expresión and Genetic Predisposition to Breast Cancer. Teratogenesis, Carcinogenesis and Mutagenesis. 1998; 18: 279-291. [ Links ]
24. J, Zidan, I, Dashkovsky, C, Stayerman, W, Basher, C, Cozacov, A, Hadary. Comparison of HER-2 overexpression in primary breast cancer and metastatic sites and its effect on biological targenting therapy of metastatic disease. British Journal of Cancer. 2005; 93: 552-556 [ Links ]
25. S, Meng, D, Tripathy, S, Shete, R, Ashfaq, B, Haley, S, Perkins, P, Beitsch, A, Khan, T, Fehm, T, Tucker, N, Lane, J, Wang, J, Uhr. HER-2 gene amplification can be acquired as breast cancer progress. PNAS. 2004; 1001: 9393-9398. [ Links ]














