Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Ciencias de la Salud
Print version ISSN 1692-7273On-line version ISSN 2145-4507
Rev. Cienc. Salud vol.6 no.1 Bogotá Jan./Apr. 2008
Teoría de conjuntos aplicada a la caracterización matemática de unión de péptidos al HLA clase II
Set Theory Applied to the Mathematical Characterization of HLA Class II Binding Peptides
Javier Rodríguez Velásquez, MD, esp1.
1. Profesor Facultad de Medicina, Universidad Nacional de Colombia. Director del grupo Insight. Correo electrónico: Grupoinsight2025@yahoo.es
Recibido: 16 de septiembre de 2007 Aceptado: 7 de noviembre de 2007
Resumen
Las bases moleculares para el reconocimiento y la respuesta inmune están en la presentación de péptidos antigénicos. Se utilizaron la teoría de conjuntos y los datos experimentales para realizar una caracterización matemática de la región central de unión del péptido mediante la definición de 8 reglas asociadas a la unión al HLA clase II. Estas reglas se aplicaron a 4 péptidos promiscuos, 25 secuencias peptídicas naturales de la región central, de las cuales 13 presentaron unión, mientras que los demás no, y 19 péptidos sintéticos buscando diferenciar los péptidos. A excepción de uno, todos los péptidos de unión y no unión fueron caracterizados acertadamente. Esta metodología puede ser útil para escoger péptidos clave en el desarrollo de vacunas.
Palabras clave: teoría de conjuntos, unión al HLA clase II.
Summary
Antigen presentation contains the molecular basis for antigenic identification and immune responses. The set theory and experimental data were used in order to develop an union core region mathematic characterization through the definition of 8 laws associated to HLA class II binding. The laws were applied to 4 promiscuous peptides, 25 natural peptides sequences of core region: 13 binding peptides and 12 no binding peptides; and 19 synthetic peptides looking to differentiate peptides. Only one peptide was not rightly characterized. This methodology may be used to choose key peptides in the development of vaccine.
Key words: Set theory, HLA class II binding.
INTRODUCCIÓN
La teoría de conjuntos permite diferenciar elementos particulares a partir de conjuntos generales al realizar operaciones como la unión, la intersección, la diferencia y la diferencia simétrica [1].
Las moléculas presentadoras de antígenos (HLA) clase II son glicoproteínas de membrana que se encuentran exclusivamente en las células presentadoras de antígeno (APCs) cuya función es la presentación de antígenos para su reconocimiento por las células T. Las moléculas de HLA clase II presentan péptidos a las células T CD4+ [1,2]. Cada molécula de HLA está anclada a la célula mediante dominios transmembrana y citoplasmáticos y posee una hendidura extracelular de unión al péptido [3].
La hendidura de unión del HLA clase II es capaz de unir péptidos de tamaño variable, 10 a 24 aminoácidos, debido a que sus extremos se encuentran abiertos [4-7], un tamaño de 13 a 16 aminoácidos ha sido observado con mayor frecuencia en los péptidos de unión [8-11]. Análisis de unión sugieren que únicamente una región central compuesta por 9 aminoácidos es esencial para la unión péptido-HLA [12]. Estudios de dicha región han relacionado aminoácidos específicos en posiciones específicas, denominados motivos de unión [8], construidos a partir de grupos seleccionados de librerías M13 de péptidos presentados.
El propósito de la presente investigación es construir una metodología general, basada en la teoría de conjuntos, para caracterizar matemáticamente la unión de péptidos a moléculas de HLA clase II.
DEFINICIONES
Conjunto: selección de elementos que cumplen una característica común definida previamente.
A = {x | x Є P(x)}
Donde P(x) representa una característica particular [1].
Sea S un conjunto que contiene dos conjuntos A y B.
Subconjunto: dados A y B dos conjuntos, A es subconjunto de B si para todo elemento x, que pertenezca a A, entonces x también pertenece a B.
Unión: la unión de A y B (A U B) es el conjunto, el cual está formado por todos los elementos x tales que pertenecen a A o a B, o a ambos.
Intersección: la intersección de A y B (A ∩ B) es el conjunto de todos los elementos x, tales que pertenecen a los conjuntos A y B.
METODOLOGÍA
Se estudiaron 48 regiones centrales nonaméricas, partiendo de cuatro péptidos promiscuos: el HA, antígeno del virus de la influenza (MAT) y el CLIP, cadena invariante unida a las moléculas presentadoras antes de la presentación antigénica y la secuencia IAKMKEKAS, péptido antigénico de la malaria que se une a varios alelos [9], 13 péptidos que se unen al HLA clase II y 12 que no presentan unión, más 19 secuencias peptídicas artificiales. Se dividieron en dos grupos: los de unión y no unión, cada uno de estos grupos con 24 péptidos. Las medidas de unión fueron realizadas con las técnicas convencionales como el equilibrio de diálisis y las bases de datos establecidas en las que se recopilan estas medidas.
Los péptidos son escogidos en este trabajo por varias razones: la primera, para poder comparar péptidos naturales con sintéticos que no se unan o que sí lo hagan. Otra es la de escoger péptidos promiscuos que se sabe se pueden unir a más de un tipo de molécula de HLA, lo que posiblemente podría orientar respecto a las capacidades de unión de estas moléculas. Precisamente, el CLIP es seleccionado en este estudio por estar unido a la hendidura en todas las moléculas HLA clase II para que pueda ser comparado con los que se unen o no, naturales o sintéticos. Todas las secuencias fueron obtenidas de estudios de Hammer et al [10].
Para el análisis de secuencias peptídicas se definieron 6 reglas asociadas a motivos de unión, reconocidos según los resultados experimentales [9]; en la primera posición se consideran 7 aminoácidos hidrofóbicos característicos de la unión como motivo [10]; adicionalmente motivos reportados para las posiciones 2 y 3 [10]; además se definió una regla matemática independiente de los resultados experimentales.
Se construyeron conjuntos de péptidos asociados a cada una de estas reglas definidas. Las reglas para la determinación de los conjuntos son:
1. Presencia de Y, W, F, V, L, I o M en la posición 1.
2. Presencia de R en la posición 2.
3. Presencia de G o P en la posición 3.
4. Presencia de V, L, M o A en la posición 4.
5. Presencia de A, G, S, T, R o K en la posición 6.
6. Presencia de L, Q, M o N en la posición 7.
7. Presencia de L, M o A en la posición 9.
8. Presencia de Y, W o F en la posición 1 y la presencia de N y L en cualquier otra posición.
A partir de las reglas anteriores se definieron los siguientes conjuntos:
Sea S el conjunto de todas las posibles cadenas peptídicas de 9 aminoácidos.
A = {x Є S |Todos los x tales que cumplen la primera regla}
B = {x Є S |Todos los x tales que cumplen la segunda regla}
C = {x Є S | Todos los x tales que cumplen la cuarta regla}
D = {x Є S | Todos los x tales que cumplen la séptima regla}
E = {x Є S | Todos los x tales pertenecen a C y no pertenecen a D}
F = {x Є S | Todos los x tales que cumplen la octava regla}
Posteriormente se evaluaron los 48 de péptidos mencionados, divididos en los dos grupos, determinando primero, cuáles, y segundo cuantas reglas cumplía cada péptido para, finalmente, encontrar cuáles reglas se cumplen simultáneamente para los péptidos que presentan unión en el lenguaje de la teoría de conjuntos: restas, intersecciones y uniones. Por tratarse de una metodología matemática, ésta no requiere de análisis estadísticos ni el uso de una gran cantidad de datos experimentales.
RESULTADOS
De 48 péptidos evaluados, 25 péptidos están contenidos en el conjunto que señalamos de unión y 23 en el conjunto de no unión; un solo péptido reportado como de no unión, pertenece al conjunto de unión.
Así, los péptidos que se unen están contenidos en el conjunto K de unión:
K = (A ∩ B) U (A ∩ D) U (A ∩ E) U (B ∩ F).
De forma simplificada:
K= (A ∩ (B U D U E)) U (B ∩ F)
Observando los datos experimentales, se evidencia que, para péptidos de unión, el aminoácido hidrofóbico seleccionado en la primera posición, junto con el aminoácido cargado Arginina R en la segunda (A ∩ B) es característico del fenómeno de unión. Se podría confirmar con las secuencias sintéticas de unión y no unión, pero se observa que aún sin cumplir con los motivos de las 2 primeras reglas, puede darse la unión, o sea, el incumpliendo la segunda, pues de los 14 péptidos naturales de unión más los cuatro promiscuos, 13 no cumplen con la segunda regla. Si no se da la segunda regla, el conjunto de unión es ampliado por la regla A ∩ D, en el cual se toman los motivos encontrados experimentalmente en la posición 1 y 9, aunque no todos los péptidos la cumplen. Sólo la cumplen 8 secuencias de los 14 pépticos naturales y la mitad de los promiscuos. Respecto a A ∩ E, se observa que la cumplen 3 secuencias de los péptidos naturales y sólo una de los promiscuos.
Observar los que se unen podría hacer pensar que la clave es la primera regla, pero de 3 péptidos que no se unen,2 cumplen con las reglas 1 y 5, y 1 con la 1.
Las reglas definidas para las posiciones 3, 4, 6 y 7 no permiten diferenciar los péptidos que se unen de los que no, pues tanto péptidos de unión como de no unión pertenecen indistintamente a los conjuntos asociados a estas reglas. Los peptidos sintéticos de no unión cumplen con las reglas 2, 5, 6 y 7 y no se unen, mientras que la mayoría de los elementos de los conjuntos asociados a las reglas 1,2 y 7 son péptidos de unión. La caracterización para los péptidos que no se unen fue del 95,7%, mientras que para aquellos que se unen, la caracterización fue del 100%.
En la tabla 1 se enuncian los péptidos estudiados y se señalan con puntos en las casillas cuántas reglas cumplen. Para los péptidos sintéticos ver tabla 2.
Tabla 1. Reglas que satisfacen las cadenas peptídicas naturales de unión y no unión y las promiscuas
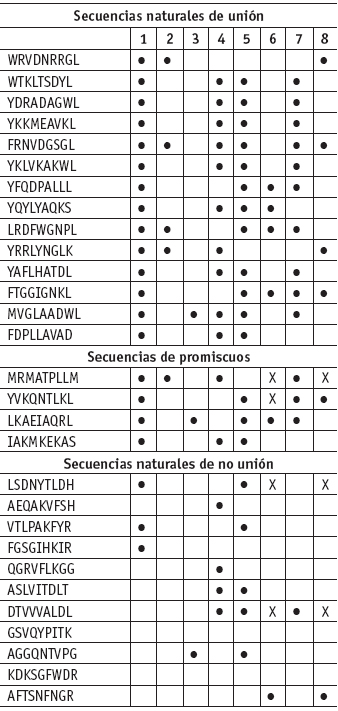
Tabla 2. Reglas que satisfacen las cadenas peptídicas sintéticas

DISCUSIÓN
Éste es el primer trabajo en el que se analiza la unión de péptidos al HLA clase II a través de teoría de conjuntos. Se encontró un comportamiento caracterizable a partir de reglas halladas experimentalmente en cualquier péptido nonamérico, diferenciando con operaciones matemáticas los péptidos de unión de los que no se unen. La inTeoría de conjuntos aplicada a la caracterización matemática de unión de péptidos al HLA clase II tersección y unión de los conjuntos definidos para reglas específicas permitió caracterizar matemáticamente regiones centrales de unión al HLA clase II, obviando la especificidad alélica de los péptidos que se unen. Se caracterizaron correctamente los péptidos de unión, logrando una especificación para todos los que se unen y desarrollando una metodología aplicable a cualquier secuencia de aminoácidos. La secuencia LRDFWGNPL cumple con 5 reglas; sin embargo, experimentalmente no presenta unión, constituyendo una excepción a la caracterización desarrollada.
Existen secuencias peptídicas que cumplen con la mayoría de las reglas y, sin embargo, no se unen al HLA, lo que probablemente muestra que no hay una relación causa consecuencia entre la cantidad de reglas cumplidas y la unión, sino un orden matemático subyacente, en este caso, con la mirada de la teoría de conjuntos que aporta relaciones acausales para entender el fenómeno. Sin embargo, el resultado de todas formas privilegia al conjunto A como eje fundamental de la unión. El predominio de este conjunto y su combinación con otros, a excepción de (B ∩ F), explica el resultado hallado. Por ejemplo, es interesante notar que se pueden cumplir la mayoría de las reglas sin que se presente unión, como en las secuencias sintéticas de no unión. Se generó una comprensión y operatividad matemática, basada en la teoría de conjuntos que da un sentido de versatilidad e integración a los motivos experimentales que, por separado, difícilmente explicarían el fenómeno de unión. La distribución heterogénea en algunos de los péptidos promiscuos y naturales, comparada con los sintéticos, puede ser explicada por la mayor probabilidad de hacer puentes de hidrógeno respecto a los sintéticos y a la versatilidad de la combinación de motivos, comparada con la búsqueda artificial sintética para comprender el fenómeno.
Trabajos anteriores en predicción de unión de péptidos han utilizado Artificial Neural Networks [13], programación lineal, motivos o vectores de máquina [14-16]. Las aplicaciones computacionales requieren de una gran cantidad de datos para su entrenamiento y las predicciones de estas metodologías generalmente están condicionadas a la población de péptidos estudiada.
Este trabajo es independiente de la población y del análisis aislado de las reglas experimentales para corroborar la caracterización desarrollada. Por ejemplo, con las reglas para las posiciones 3, 4, 6 y 7 no se encontraron diferencias entre los grupos. La intersección de todos los conjuntos formados por las distintas reglas asignadas a las cadenas peptídicas es un conjunto vacío y no da cuenta del fenómeno de unión, lo cual sugiere que por más reglas experimentales que se establezcan, el fenómeno de unión no se aclarará sino que, probablemente, la construcción de una teoría enmarcada en una serie de leyes podría explicar el fenómeno.
En trabajos experimentales anteriores [10] se presenta la dependencia de la energía de unión con el tamaño del péptido que generalmente varía entre 13 y 16 aminoácidos; dicha energía disminuye si se reduce la cantidad de aminoácidos en la cadena peptídica. Para compensar esta pérdida en secuencias más cortas, se introducen aminoácidos específicos en posiciones de anclaje. A partir de estos resultados experimentales sería posible diseñar antígenos sintéticos que se presenten por moléculas de HLA clase II con las longitudes consideradas en este trabajo.
Esta metodología se basa en una generalización, una relación para las reglas empíricas halladas, encontrando con 5 de 8 reglas, y sus nuevas relaciones, la caracterización de los péptidos que se unen y los que no. En las teorías fundamentales de la física [17] no hay causas sino órdenes físicos y matemáticos acausales para la descripción y comprensión del fenómeno estudiado; en este trabajo, el fenómeno de unión es estudiado de igual manera. Esta metodología puede caracterizar cualquier posible secuencia nonamérica, caracterizando de forma objetiva y reproducible la unión, lo cual, probablemente, pueda ser útil en el diseño matemático de vacunas eficientes, por ejemplo, para el cáncer, entre otras. De esta manera, se racionalizarían los recursos en su desarrollo, dejando de lado el método de ensayo-error en la experimentación actual. Adicionalmente, se agilizaría este proceso y, posiblemente, constituiría un primer paso en la comprensión de la interacción, ligando un receptor específico de la biología molecular, lo que sería de vital importancia para cualquier aplicación médica y biológica en el futuro.
Éste es un primer acercamiento con un diseño teórico que busca caracterizar lo esencial y la simplicidad con muy pocos casos, proyectando la posibilidad o no de construir una teoría predictiva para este fenómeno, como funciona la física teórica. Esta caracterización, aplicada a la unión de estas moléculas, podría tener la ventaja de generar una medida general para cualquier péptido frente a los actuales métodos de bioinformática que se están especializando por alelos, mejorando esta metodología. Más importante, respecto a la bioinformática, es ver el fenómeno acausal teóricamente y permitir una aproximación desde la física y la matemática para poder, tal vez, lograr predicciones desde la construcción de una teoría.
Aplicaciones futuras: se diseñará una prueba para medir la posibilidad predictiva de esta metodología con bases de datos experimentales.
CONCLUSIONES
1. La teoría de conjuntos permite representar simultáneamente, de manera sistemática, la presencia o ausencia de motivos combinados para caracterizar matemáticamente la versatilidad del fenómeno de unión.
2. El fenómeno de unión puede ser comprendido de una manera más sintética y general por esta metodología, a diferencia del tratamiento particular por el número de motivos aislados en diferentes posiciones específicas.
AGRADECIMIENTOS
A Signed Esperanza Prieto, investigadora grupo Insight; Pedro Alfonso Bernal, estudiante ingeniería de sistemas, Universidad Nacional de Colombia, Investigador grupo Insight; Catalina Correa, PSY, investigadora grupo Insight; Liliana Ortiz; delineante de arquitectura, investigadora grupo Insight; Laura Bibiana Pinilla, MD, investigadora grupo Insight, por su dedicación en el trabajo y su apoyo constante.
REFERENCIAS
1. Hrbacek K, Jech T. Introduction to set theory. 3ra ed. New York: Marcel Dekker, Inc; 1999. [ Links ]
2. Wang JH, Reinherz EL. Structural basis of T-cell recognition of peptides bound to MHC molecules. Mol Immunol 2002 May 38(14):1039-49. Review. [ Links ]
3. Abbas A, Lichtman A, Pober J. Inmunología celular y molecular. 4ª ed. Madrid: McGraw Hill-Interamericana de España; 2002. [ Links ]
4. Madden DR. The three-dimensional structure of peptide-MHC complexes. Annu Rev Immunol 1995;13:587-622. [ Links ]
5. Fremont DH, Hendrickson WA, Marrack P, Kappler J. Structures of an MHC class II molecule with covalently bound single peptides. Science 1996 May 17;272(5264):1001-4. [ Links ]
6. Brown JH, Jardetzky TS, Gorga JC, Stern LJ, Urban RG, Strominger JL, Wiley DC. 3-Dimensional structure of the human class-II histocompatibility antigen HLADR1. Nature 1993;364:33-39. [ Links ]
7. Stern LJ, Brown JH, Jardetzky TS, Gorga JC, Urban RG, Strominger JL, Wiley DC. Crystal structure of the human class II MHC protein HLA-DR1 complexed with an influenza virus peptide. Nature 1994 Mar 17;368(6468):215-21. [ Links ]
8. Hammer J, Takacs B, Sinigaglia F. Identification of a motif for HLA-DR1 binding peptides using M13 Display Libraries J Exp Med 1992 Oct 1;176(4):1007-13. [ Links ]
9. Hammer J, Valsasnini P, Tolba K, Bolin D, Higelin J, Takacs B, Sinigaglia F. Promiscuous and allele-specific anchors in HLA-DR-Binding Peptides. Cell 1993 Jul 16;74(1):197-203. [ Links ]
10. Hammer J, Belunis C, Bolin D, Papadopoulos J, Walsky R, Higelin J, Danho W, Sinigaglia F, Nagy ZA. High-affinity binding of short peptides to major histocompatibility complex class II molecules by anchor combinations. Proc Natl Acad Sci U S A 1994 May 10;91(10):4456-60. [ Links ]
11. Busch J, Strang G, Howland K, Rothbard J. Degenerate binding of immunogenic peptides to HLA-DR proteins on B cell surfaces. Int Immunol 1990;2:443-448. [ Links ]
12. Rammensee HG, Friede T, Stevanoviíc S. MHC ligands and peptide motifs: first listing. Immunogenetics 1995;41(4):178-228. [ Links ]
13. Zhao Y, Pinilla C, Valmori D, Martin R, Simon R. Application of support vector machines for T-cell epitopes prediction. Bioinformatics 2003 Oct 12;19(15):1978-84. [ Links ]
14. Southwood S, Sidney J, Kondo A, del Guercio MF, Appella E, Hoffman S, Kubo RT, Chesnut RW, Grey HM, Sette A. Several common HLA-DR types share largely overlapping peptide binding repertoires. J Immunol 1998 Apr 1;160(7):3363-73. [ Links ]
15. Brusic V, Rudy G, Honeyman M, Hammer J, Harrison L. Prediction of MHC class II-binding peptides using an evolutionary algorithm and artificial neural network. Bioinformatics 1998;14:121-30. [ Links ]
16. Dönnes1, P., & Elofsson, A. Prediction of MHC class I binding peptides, using SVMHC. BMC Bioinformatics 2002;3:25. [ Links ]
17. Fernández-Rañada A. Orden y caos. Introducción. Barcelona: Prensa Científica. Investig Cienc 1990. [ Links ]














