Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Ciencias de la Salud
Print version ISSN 1692-7273
Rev. Cienc. Salud vol.14 no.1 Bogotá Jan./June 2016
https://doi.org/10.12804/revsalud14.01.2016.01
Doi: http://dx.doi.org/10.12804/revsalud14.01.2016.01
Artículos de investigación clínica o experimental
Prevalencia de Staphylococcus aureus que coloniza el personal de salud de un hospital de la ciudad de Cali
Prevalence of Staphylococcus Aureus Colonizing the Health Care Personnel of a Hospital in the City of Cali
Prevalência de Staphylococcus aureus que coloniza o pessoal de saúde de um hospital da cidade de Cali
Leidy Catherine Arteaga Delgado1, Yuli Espinosa López1, Mónica Chávez Vivas2
1 Hospital San Juan de Dios, Cali. Programa de Medicina, Facultad de Salud, Universidad Santiago de Cali. Cali, Colombia
2 Departamento de Ciencias Biomédicas, Facultad de Salud, Centro de Estudio e Investigación en Salud (CEIS), Universidad Santiago de Cali. Cali, Colombia. Correo electrónico: monikchavez@gmail.com
Recibido: 13 de abril de 2015 • Aceptado: 30 de agosto de 2015
Para citar este artículo: Arteaga-Delgado LC, Espinosa-López Y, Chávez-Vivas M. Prevalencia de Staphylococcus aureus que coloniza el personal de salud de un hospital de la ciudad de Cali. Rev Cienc Salud. 2016;14(1):9-19. doi: dx.doi.org/10.12804/revsalud14.01.2016.01
Resumen
Objetivo: El objetivo de este estudio fue determinar la prevalencia de las cepas de Staphylococcus aureus (S. aureus) en los trabajadores de un hospital de la ciudad de Cali. Materiales y métodos: Se realizó un estudio descriptivo, con muestras de rastreo nasal y frotis de piel a 30 trabajadores de salud. La caracterización se basó en el análisis del antibiograma y la amplificación por PCR de los genes mecA y agr. El origen del aislamiento se estableció con el análisis del gen agr, al identificar los grupos agr. Resultados: 11 (26,7 %) trabajadores estuvieron colonizados por S. aureus. La frecuencia de S. aureus fue mayor en el personal de salud que se encontraban en la sala de cirugía (20%; OR = 2,077; P>0,05). Se identificaron cuatro antibiotipos, esta característica es compatible con los clones comunitarios que han demostrado ser altamente diversos con una gran capacidad de diseminación en la comunidad. El 36,4% de los aislamientos presentaron resistencia a cefotaxima y/o oxacilina, fenotipo sugerente de S. aureus resistente meticilina (SARM), en estos aislados se identificó el gen mecA. El grupo agr i se encontró, principalmente, entre los aislamientos sensibles a meticilina (SASM), compatible con un origen comunitario y en los aislamientos SARM pertenecen al grupo agr ii, siendo de origen hospitalario. Conclusión: Se demostró la prevalencia de S. aureus con resistencia a los antibióticos que colonizan al personal de salud, principalmente los que laboran en la sala de cirugía. Se debe mantener el control regular del personal para impedir la diseminación de patógenos.
Palabras clave: Staphylococcus aureus, personal de salud, antibióticos, resistencia bacteriana, epidemiología.
Abstract
Objecitve: The aim of this study was to determine the prevalence of isolates of Staphylococcus aureus in workers at a hospital in Cali. Materials and methods: A descriptive study was conducted with samples of nasal swabs, skin smears to 30 health care workers. Phenotypic characterization of isolates was based on susceptibility antibiogram testing and PCR amplification of the identified mecA and agr genes. The origin of isolates was established by analysis of agr gene, identifying the AGR groups. Results: Eleven (26.7 %) workers were colonized with S. aureus. The frequency of S. aureus was higher in health care personnel who were in the operating room (20 %; OR = 2.077; P> 0.05). Four antibiotypes were identified, this feature is compatible with community clones that have proven to be highly diverse with a large capacity to spread in the community. 36.4 % of the isolates were resistant to cefotaxime and/or oxacillin, suggestive MRSA phenotype in these isolated the mecA gene was identified. Agr i was found primarily among isolates metillicin-sensitive S. aureus (MSSA), compatible with community origin, and MRSA isolates belong to AGR ii, with hospital waste. Conclusion: The prevalence of S. aureus resistant to antibiotics that colonize health care personnel was demonstrated, mainly in those working in the operating room. Regular monitoring of personnel should be regularly conducted to prevent the spread of pathogens.
Keywords: Staphylococcus aureus, Health care personal, Antibiotics, Bacterial resistance, Epidemiology.
Resumo
Objetivo: o objetivo deste estudo foi determinar a prevalência das cepas de Staphylococcus aureus nos trabalhadores que trabalham em um hospital da cidade de Cali. Materiais e métodos: realizou-se um estudo descritivo, com amostras de rastreamento nasal e esfregaços da pele de 30 trabalhadores de saúde. A caracterização se baseou na análise do antibiograma e da amplificação por PCR dos genes mecA e agr. A origem do isolamento se estabeleceu com a análise do gene agr, identificando os grupos agr. Resultados: 11 (26,7%) trabalhadores estiveram colonizados por S. aureus. A frequência de S. aureus foi maior no pessoal da saúde que se encontrava na sala de cirurgia (20%; OR=2,077; P>0,05). Identificaram-se quatro antibióticos, esta característica é compatível com os clones comunitários que têm demonstrado ser altamente diversos com uma grande capacidade de disseminação na comunidade. O 36,4% dos isolamentos apresentaram resistência a cefotaxima e/ou oxacilina, fenótipo sugestivo de SARM, nestes isolados se identificou o gene mecA. O grupo agr i encontrou-se principalmente entre os isolamentos sensíveis a meticilina (SASM), compatível com uma origem comunitário e nos isolamentos SARM pertencem ao grupo agr ii, sendo de origem hospitalar. Conclusão: demonstrou-se a prevalência de S. aureus com resistência aos antibióticos que colonizam ao pessoal de saúde, principalmente os que trabalham na sala de cirurgia. Deve-se manter o controle regular do pessoal para impedir a disseminação de patógenos.
Palavras-chave: Staphylococcus aureus, pessoal de saúde, antibióticos, resistência bacteriana, epidemiologia.
Introducción
El Staphylococcus aureus es el agente más frecuentemente identificado como causa de infecciones asociadas con el cuidado de la salud, particularmente S. aureus resistente a meticilina (SAMR) (1, 2). Estas cepas también se detectan en la comunidad en pacientes, principalmente jóvenes sin patologías previas, causan infecciones de piel y tejidos blandos y también casos de infecciones graves, como neumonía necrotizante y osteomielitis (3-5).
La resistencia en las cepas de SARM es conferida por la presencia del gen mecA que codifica por una segunda proteína con capacidad de unir penicilina, conocida como PBP2a (6). Estas cepas pueden adquirir, simultáneamente, genes de patogenicidad que las hacen desarrollar infecciones más agresivas (7). Además, son resistentes a múltiples antibióticos, lo que deja como alternativas terapéuticas el empleo de antibióticos de espectro más reducido como la vancomicina, teicoplanina y el linezolid (8).
Los portadores nasales de S. aureus se consideran como uno de los principales reservorios y responsables de la diseminación de infecciones estafilocócicas nosocomiales y de la comunidad (9, 10).
Este microorganismo coloniza, con frecuencia, la piel y membranas mucosas, sin causar infección en personas sanas, no invade la piel indemne, pero mínimas lesiones de la barrera cutánea-mucosa le permiten penetrar en los tejidos y causar una gran variedad de infecciones (10).
En la comunidad, algunos grupos de personas son más propensos a estar colonizados, como el personal sanitario (11, 12). Se ha observado que puede existir hasta un 50 % de portadores nasales en el personal médico de los hospitales (13).
La propagación de cepas SARM se produce, en gran medida, cuando los trabajadores de salud se convierten en portadores transitorios, al llevarlos en las manos y no aplicar las barreras de contención necesarias (11-13).
En este trabajo se determinó la prevalencia de las cepas de S. aureus en los trabajadores de las salas de cirugía, postoperatoria y de cuidados intermedios en un hospital de Segundo Nivel de la ciudad de Cali en 2013. Este hallazgo es importante de establecer porque el personal colonizado puede actuar como portador asintomático e infectar al paciente.
Es importante considerar si los aislamientos de S. aureus que colonizan al personal de salud son de origen comunitario o asociado con el cuidado de la salud, a fin de elaborar estrategias que impidan la diseminación descontrolada de los patógenos en el ambiente hospitalario.
Materiales y métodos
Se trató de un estudio descriptivo de corte transversal que fue evaluado y aprobado por el Comité de Ética y Bioética de la Facultad de Salud de la Universidad.
Este estudio incluyó un total de 30 individuos que estaban laborando en el hospital y cumplieron los criterios de inclusión y exclusión, entre los que se destaca no haber recibido tratamiento antibiótico en los últimos tres meses y no presentar enfermedades respiratorias o de piel.
El personal de salud estuvo constituido por 20 mujeres y 10 hombres, con un promedio de edad de 33 años (Ds = 12,247; min. = 21 años, máx. = 59 años), distribuidos de la siguiente forma: sala de cuidados intermedios (3 individuos), sala de cirugía (19 individuos), sala de posquirúrgicas (8 individuos).
Condiciones de cultivo
Las muestras se tomaron con hisopos de algodón estériles, mediante frotis de la mucosa nasal y de piel en cada participante, y se procesaron inmediatamente en el laboratorio de microbiología. Los aislamientos se manejaron por códigos, respetando de esta manera la confidencialidad.
Para el aislamiento de especies del género Staphylococcus, las muestras se sembraron en agar salino manitol rojo de fenol (Oxoid Ltd., Hampshire, United Kingdom) y se incubaron de 24 a 48 horas a 37°C. La identificación de S. aureus se efectúo por la fermentación del manitol en el agar selectivo (coloración amarilla del medio), la reacción positiva de la prueba de la coagulasa, y se corroboró con la observación en microscopio de cocos Gram positivos en racimos, a partir de un extendido directo con tinción de Gram. El S. aureus se diferenció del Staphylococcus coagulasa negativo con el empleo de la prueba de la Dnasa.
Prueba de sensibilidad a los antibióticos
La prueba de sensibilidad antimicrobiana se realizó en muestras apareadas, al emplear el método de difusión en agar con discos. Para llevar a cabo este ensayo, se inoculó una cantidad estandarizada (estándar 0,5 de Mc Farland) de S. aureus en un medio de agar Mueller-Hinton (Scharlau Chemie S.A.) y, posteriormente, se colocaron los sensidiscos: Penicilina (PEN, 10 U), oxacilina (OXA, 1 μg), cefoxitina (FOX, 30 μg), gentamicina (GEN, 10 μg), ciprofloxacina (CIP, 5 μg), eritromicina (ERI, 15 μg), clindamicina (cli, 2 μg), trimetoprima/sulfametoxazol (SXT 1,25/23,75 μg), tetraciclina (TET, 30 μg), cloranfenicol (CHL, 30 μg) (Oxoid). Para verificar la acción de los sensidiscos, en el análisis de sensibilidad se empleó la cepa ATCC 25923 de S. aureus como control. Los aislamientos se clasificaron como sensibles, sensibilidad intermedia o resistentes, de acuerdo con las recomendaciones del Clinical and Laboratory Standards Institute (CLSI) (14).
La presencia de asilamientos SARM se realizó de acuerdo con los resultados obtenidos en la evaluación a los antibióticos oxacilina y cefoxitina, y mediante la detección del gen mecA (15).
Análisis moleculares de aislamientos
El ADN de las cepas referencia y de los aislamientos bacterianos se extrajo al emplear el protocolo modificado de Cheng et al., que se basa en la lisis bacteriana al utilizar solución de sacarosa al 25 %, 10 mg/ml de lisozima y 1 mg/ml de proteinasa K, a 56°C (16).
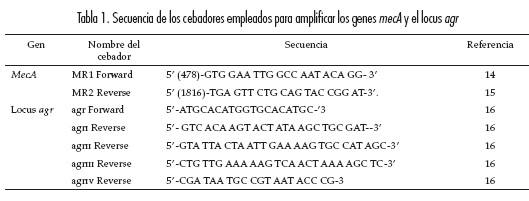
Para establecer la resistencia a meticilina, se amplificó el gen mecA, siguiendo el protocolo reportado por Tokue et al. (17). Para esta amplificación, se realizó un ciclo de denaturación a 94°C por 5 minutos, seguido de 30 ciclos a 94°C por un minuto, 52°C por un minuto y 72°C por un minuto, la extensión final se realizó a 72°C por 10 minutos, para amplificar una banda de 1334 pb del gen mecA con los cebadores MR1 y MR2 (tabla 1).
Para determinar los grupos agr I, agr II, agr III y agr IV, se amplificaron, de manera independiente, los fragmentos de 440 bp, 572 bp, 406 bp y 588 pb, respectivamente, empleando un juego de cebadores señalados en la tabla 1.
Las reacciones de PCR se realizaron de acuerdo con el protocolo establecido por Azimian et al., en un volumen de 50 μl de una mezcla de reacción compuesta por MgCl2 25 mM, 200 μM de los cuatro dNTP's, 0,5 U de Taq DNA polimerasa (Invitrogen®), 10 pmol de cada cebador y 5 μl de solución de ADN en un termociclador GeneAmp PCR System 2400® (18). Como control positivo, se empleó la cepa ATCC 25923 de S. aureus y el control negativo, la cepa ATCC 25922 de Eschericchia coli. La reacción de amplificación se llevó a cabo al realizar un ciclo de denaturación a 94°C por 5 minutos, seguido de 30 ciclos a 94°C por un minuto, 55°C por 45 segundos y 72°C por un minuto, y una extensión final a 72°C por 10 minutos.
Análisis estadísticos
La unidad de análisis fue el aislado bacteriano obtenido del hisopado nasal, del cual se registraron las características microbiológicas y moleculares y se relacionaron con las características sociodemográficas de los portadores, como el género y semestre académico. Las variables microbiológicas fueron la presencia de S. aureus y el grado de sensibilidad o resistencia a cada antibiótico evaluado, categorizadas en diferentes grados así: resistencia (a), sensibilidad intermedia (b) y sensibilidad (c), de acuerdo con las recomendaciones del CLSI (14). Las variables moleculares fueron presencia de los genes mecA, y las variantes del gen agr. Se construyó una base de datos con las variables de interés, empleando el programa ExcelTM.
En el grupo de colonizados por S. aureus se realizó un análisis de asociación entre las distintas variables, como el fenotipo de resistencia a los diferentes antibióticos, la presencia del gen mecA y la variante genética de agr, teniendo en cuenta la sala donde labora el personal de salud. Se determinó la prevalencia de las diferentes variantes del gen agr en los fenotipos SARM y S. aureus sensible a meticilina (SASM).
La significancia en la frecuencia de las variables entre los grupos establecidos fue determinada por análisis estadístico, empleando la prueba de chi-cuadrado. La significancia estadística fue asignada para valores de p<0,05, considerando un nivel de confianza del 95 % (alfa) y un error (beta) de 5 %. Los análisis estadísticos se realizaron empleando el paquete estadístico (sPSS versión 20.0, SPSS, Inc., Chicago, IL, USA).
Resultados
Las especies del género Staphylococcus colonizaron a 28 trabajadores (93,3 %) que se encontraban rotando en las tres salas evaluadas, 26 de ellos (86,7 %) presentaron colonización nasal y 8 trabajadores (26,7 %) estuvieron colonizados en piel.
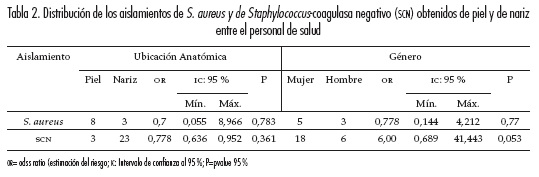
Un total de 11 aislamientos (18,3 %) de S. aureus fueron obtenidos del personal de salud, 8 aislamientos de piel (26,7 %) y 3 aislamientos de colonización nasal (10 %); 8 de los trabajadores colonizados por S. aureus fueron mujeres, sin representar un valor estadísticamente significativo (p>0,05) (tabla 2).
En el caso de los aislamientos de Staphylococcus coagulasa negativo (SCN), 26 aislamientos (43,3 %) fueron obtenidos del personal de salud, 21 trabajadores fueron portadores nasales y cinco portadores asintomáticos en piel. Tres de estos individuos presentaron simultáneamente colonización nasal y en piel. La distribución de estos aislamientos entre los trabajadores del hospital no fue significativa (p>0,05) (tabla 2).
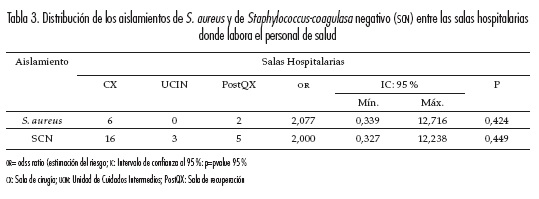
El personal de salud que se encontraba en la sala de cirugía presentó el mayor número de personas colonizadas por S. aureus (20 %; OR = 2,077; P>0,05) (tabla 3). Igualmente, los aislamientos de SCN se encontraron en mayor frecuencia en esta sala (53,3 %; OR = 2,000; P>0,05) y se presentaron con mayor frecuencia en mujeres (90 % frente a 20 %; OR = 6,00, y P = 0,053) (tabla 2).
Análisis fenotípico de los aislamientos de S. aureus
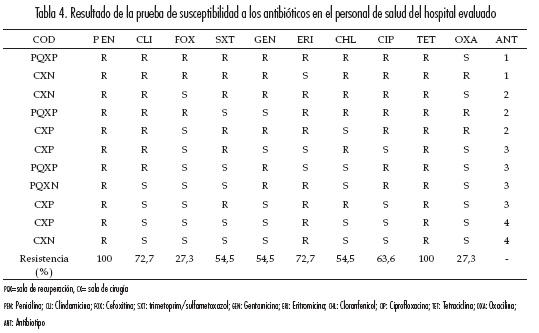
Todos los aislamientos de S. aureus que colonizaron al personal de salud presentaron resistencia a la penicilina, el 27,3 % de ellos presentaron resistencia a la cefoxitina, el 54,5 % a la gentamicina, el 63,6 % a la clindamicina, el 54,5 % al cloranfenicol, el 72,7 % a la eritromicina y el 63,6 % al ciprofloxacina. Los aislamientos de S. aureus con resistencia a oxacilina (saro) correspondieron al 27,3 % (tabla 4).
Asimismo, cuatro aislamientos (36,4 %) presentaron resistencia a cefotaxima y/o oxacilina, de acuerdo con este criterio, este fenotipo es sugerente de SARM. Los aislamientos SARM presentaron resistencia simultánea a casi todos los antibióticos, lo que los hace sugerentes a un SARM de origen hospitalario (SARM-AH), dos aislamientos se presentaron en cirugía y dos en la sala de recuperación.
Basado en el perfil de sensibilidad a los antibióticos, se determinaron cuatro (4) perfiles denominados antibiotipos. El antibiotipo 1 está representado por aislamientos con resistencia a los antibióticos evaluados, excepto a oxacilina o eritromicina y se encontró en dos trabajadores de las salas de cirugía y recuperación.
El antibiotipo 2, con sensibilidad únicamente a dos antibióticos, se presentó en los aislamientos de tres trabajadores, dos de ellos de la sala de cirugía y uno de la sala de recuperación. El antibiotipo 3, con resistencia simultánea a cinco antibióticos, se detectó en los aislamientos de dos trabajadores de las salas de cirugía y recuperación.
Los aislamientos del antibiotipo 4 presentaron resistencia simultánea a la penicilina, eritromicina y tetraciclina, y se detectó solo en los trabajadores que laboraban en la sala de cirugía (tabla 4).
Análisis molecular de los aislamientos de S. aureus
La detección molecular de la resistencia a meticilina se halló mediante la amplificación del gen mecA, en este caso, en los cuatro aislamientos de S. aureus que fueron considerados fenotípicamente SARM. Los aislamientos SARM se encontraron en el personal de salud que labora en las salas de cirugía y de recuperación (figura 1)
Caracterización de los grupos agr
La amplificación del gen agr permitió identificar dos grupos agr entre los aislamientos de S. aureus que se encontraban colonizando al personal de salud. El grupo agr i se determinó en los aislamientos de SASM y el grupo agr ii se presentó en los cuatros aislamientos SARM.
Discusión
Los portadores nasales de S. aureus del personal de salud tienen gran relevancia en la transmisión del microorganismo en los hospitales; en muchos casos, se ha determinado que la colonización nasal de trabajadores de la salud y pacientes normalmente precede a la infección intrahospitalaria por esta bacteria (9, 10). En este estudio, se detectó el S. aureus en el personal de salud en el 26,7 % de los casos. Estos resultados son coherentes con los obtenidos en otras ciudades de Colombia, con reportes de hasta un 72 % de colonización y en países de Asia con reportes entre 27 % al 37 % (19-23). Sin embargo, la prevalencia reportada en países de América Latina es más baja, con valores que oscilan entre el 12 % y 17,1 % (24, 25).
Por otra parte, la colonización por SARM en el personal de salud encontrada en este estudio (36,4 %) fue más alta que la registrada en países de Asia y América Latina (21-26).
En los Estados Unidos, para el año 1975, se reportaba una prevalencia del 2,4 %, en el personal de salud, la cual ascendió a un 29 % para el año 1991 (27). Sin embargo, en 2004, se estimó que menos del 6 % del personal de salud estaba colonizado (27, 28). Esta disminución reportada estuvo dada por la intensificación en el empleo de medidas de control, como el lavado de manos y el uso de barreras de contención. Esta medida debe ser aplicada de forma rigurosa para disminuir el aumento en la colonización bacteriana en los hospitales de la región.
El análisis de la prueba de sensibilidad a los antibióticos en los aislamientos SARM evidenció resistencia simultánea a eritromicina, clindamicina, ciprofloxacina y gentamicina, fenotipo compatible con cepas de origen hospitalario. Esta característica también la encontraron Olarte et al. En un estudio realizado en hospitales de Bogotá en el año 2010, donde determinó que los aislamientos SARM eran compatibles con el clon chileno (29). Este clon solo se encuentra entre los aislamientos hospitalarios (SARM-AH). En este sentido, el análisis molecular del locus agr, en los cuatro aislamientos de SARM detectados en este estudio, evidencia que pertenecen al grupo agr II, este grupo es compatible con aislamientos de origen nosocomial, lo que confirma los hallazgos microbiológicos (18).
Es importante destacar la resistencia a clindamicina entre los aislamientos SASM. Esta es una característica que se ha hecho notable desde finales del año 2000 con el incremento de S. aureus resistente a meticilina asociado con la comunidad (SARM-AC). Se ha establecido que la presencia de plásmido pUSA300 (clon usa300) en aislamientos obtenidos en usa es la responsable de la resistencia a clindamicina (30). En este estudio, todos los aislamientos SASM se ubicaron en el grupo agr I, genotipo compatible con un origen comunitario. Los resultados están acordes con la idea de que los aislamientos SASM están estrechamente relacionados con el clon usa300 que circula en Colombia (31, 32).
Escobar et al., establecen que la diseminación del SASM entre portadores asintomáticos es un factor importante para el establecimiento del SARM comunitario en nuestro país (CLON USA 300) (33). El clon usa300 tiene un alto poder de diseminación y, además, ha demostrado ser altamente virulento con capacidad de ocasionar infecciones más agresivas, favorecidas, quizá, por su origen, a partir de aislamientos SASM que se encuentran en portadores asintomáticos.
Es importante considerar la colonización por SCN en el personal de salud, porque, a pesar de que varias de estas especies hacen parte de la flora normal, son consideradas potenciales patógenos humanos relacionadas con infecciones de pacientes con enfermedades de base y en los que reciben terapias de inmunosupresión, además, se ha determinado capacidad de producir infecciones superficiales y profundas en humanos sanos (34).
Conclusión
El presente estudio demostró la presencia de S. aureus en las fosas nasales y la piel del personal de salud. Estas cepas son fenotípicamente diversas (4 antibiotipos) y algunas de ellas son portadoras del gen mecA. Esta característica es compatible con los clones comunitarios, que son altamente diversos con una gran capacidad de diseminación en la comunidad. Esta ventaja probablemente le permita dominar sobre los clones hospitalarios. La condición de portador asintomático es un factor de riesgo para desarrollar infecciones adquiridas en el hospital. La constante movilidad del personal de salud entre las salas del mismo hospital y, probablemente, entre hospitales, plantea la necesidad de generar estrategias de prevención de diseminación de SARM y SASM de forma institucional y en red.
Agradecimientos
A la Dirección General de Investigación (DGI) de la Universidad Santiago de Cali, por el apoyo económico para llevar a cabo este estudio, a Bellazmin Arenas, directora del Centro de Investigación y Estudios de la Facultad de Salud (CEIS), por su apoyo.
Referencias
1. Valaperta R, Tejada MR, Frigerio M. Staphylococcus aureus nosocomial infections: the role of a rapid and low-cost characterization for the establishment of a surveillance system. New microbiol. 2010;33(3):223-32. [ Links ]
2. Febrero-Peray P, Sotto A, Defez C, Cazaban M, Molinari L, Pinède M, et al. Mortality attributable to nosocomial infection: a cohort of patients with and without nosocomial infection in a French university hospital. Infect Control Hosp Epidemiol. 2007; 28:265-72. [ Links ]
3. Mediavilla JR, Chen L, Mathema B, Kreiswirth BN. Global epidemiology of community-associated methicillin resistant Staphylococcus aureus (CA-MRSA). Cur Op Microbiol. 2012;15(5):588-95. [ Links ]
4. González BE, Teruya J, Mahoney DH Jr, Hulten KG, Edwards R, Lamberth LB, et al. Venous thrombosis associated with Staphylococcal osteomyelitis in children. Pediatrics 2006; 117:1673-9. [ Links ]
5. Martínez-Aguilar G, Avalos-Mishaan A, Hulten K, Hammerman W, Mason EO Jr, Kaplan SL. Community-acquired, methicillin-resistant and methicillin-susceptible Staphylococcus aureus musculoskeletal infections in children. Pediatr Infect Dis J. 2004;23:701-6. [ Links ]
6. Appelbaum PC. Microbiology of Antibiotic Resistance in Staphylococcus aureus. Clin Infect Dis. 2007 [citado 2007 sep 15; 45(Sup 3):S165-70] [ Links ].
7. Tseng C, Kyme P, Low J, Rocha M, Alsabeh R, Miller L. Staphylococcus aureus Panton-Valentine leukocidin contributes to inflammation and muscle tissue injury. PLoS One. 2009;27:e6387. [ Links ]
8. De Lencastre H, Oliveira D, Tomasz A. Antibiotic resistant Staphylococcus aureus: a paradigm of adaptive power. Curr Opin Microbiol. 2007;10:428-35. [ Links ]
9. Huang S, Platt R. Risk of methicillin-resistant Staphylococcus aureus infection after previous infection or colonization. Clin Infect Dis. 2003;36:281-5. [ Links ]
10. Safdar N, Bradley E. The risk of infection after nasal colonization with Staphylococcus aureus. Am J Med. 2008;121:310-5. [ Links ]
11. Ibarra M. Prevalence of methicillin-resistant Staphylococcus aureus nasal carriage in healthcare workers. Pediatr Infect Dis J. 2008;27(12):1109-11. [ Links ]
12. Eveillard M, Martín Y. Carriage of methicillin-resistant Staphylococcus aureus among hospital employees: prevalence, duration and transmission to households. Infect Control Hosp Epidemiol. 2004;25(2):114-20. [ Links ]
13. Shakya B, Shrestha S, Mitra T. Nasal carriage rate of methicillin resistant Staphylococcus aureus among at National Medical College Teaching Hospital, Birgunj, Nepal. Nepal Med Coll J. 2010;12(1):26-9. [ Links ]
14. Clinical and Laboratory Standards Institute. Performance Standards for Antimicrobial Susceptibility Testing: Twenty-third Informational Supplement. Wayne, PA.: CLSI; 2013. M100-S23. [ Links ]
15. Datta P, Gulati N, Singla N, Vasdeva HR, Bala K, Chander J. et al. Evaluation of various methods for the detection of meticillin-resistant Staphylococcus aureus strains and susceptibility patterns. J Med Microbiol. 2011;60:1613-6. [ Links ]
16. Cheng HR, Jiang N. Extremely rapid extraction of DNA from bacteria and yeasts. Biotech Lett 2006;28:55-9. [ Links ]
17. Tokue Y, Shoji S, Satoh K, Watanabe A, Motomiya M. Comparison of a Polymerase Chain Reaction Assay and a Conventional Microbiological Method for Detection of Methicillin-Resistant Staphylococcus aureus. Antimicrob Agents Chemother. 1992;36:1:6-9. [ Links ]
18. Azimian A, Najar-pirayeh S, Mirab-Samiee S, Naderi M. Ocurrence of Methicillin Resistant Staphylococcus aureus (MRSA) among clinical samples in Tehran - Iran and its correlation with polymorphism of specific accessory gene regulator (AGR) groups. Braz. J. Microbiol. 2012; 43(2):779-85. [ Links ]
19. Londoño JF, Ortiz GM, Gaviria AM. Prevalencia de Staphylococcus aureus resistente a meticilina en personal de la unidad de terapia intensiva de la Clínica Universitaria Bolivariana. Infect. 2006;10(3):160-6. [ Links ]
20. Espinosa CT, Romero MK, Rincón, G. Portadores nasales de Staphylococcus aureus en personal que labora en un Hospital de Santander. Rev Univ Ind Santander Salud. 2011;43(2):111-7. [ Links ]
21. Goyal R, Das S, Mathur M. Colonization of methicillin resistant Staphylococcus aureus among health care workers in a tertiary care hospital of Delhi. Indian J Med Sci. 2002;56(7):321-4. [ Links ]
22. Shrestha B, Pokhrel BM, Mohapatra TM. Staphylococcus aureus nasal carriage among health care workers in a Nepal hospital. Braz J Infect Dis. 2009;13(5):322. [ Links ]
23. Shibabaw A, Abebe T, Mihret A. Nasal carriage rate of methicillin resistant Staphylococcus aureus among Dessie Referral Hospital Health Care Workers; Dessie, Northeast Ethiopia; Antimicrob Res Infec Control. 2013;2:25. [ Links ]
24. Cimera D, Pérez F. Prevalencia de portadores nasales asintomáticos de Staphylococcus aureus meticilino-resistente y su relación con factores de riesgo y protectores en el personal de salud del Hospital General de las Fuerzas Armadas. Rev. Mex Patol Clin. 2010;57(4):196-204. [ Links ]
25. Montalvo R, Huaroto L, Álvarezcano J, Ticona E, García Y. Prevalencia de portadores nasales por Staphylococcus aureus meticilino resistente en personal de salud del servicio de cuidados intensivos, Hospital Nacional Dos de Mayo. Rev Peruana Epi. 2009;13(2):1-5. [ Links ]
26. Cáceres M. Frecuencia de portadores nasales de Staphylococcus aureus resistente a meticilina en personal de salud de hospitales de Nicaragua. Rev Panam Salud Pública. 2011;30(6):610-4. [ Links ]
27. Marjolein F, VandenBergh MFQ, Yzerman EPF, van Belkum A, Boelens HAM, Sijmons M, et al. Follow-up of Staphylococcus aureus nasal carriage after 8 years: Redefining the persistent carrier state. J Clin Microbiol. 1999;37(10):3133-40. [ Links ]
28. Atsushi K, Eiko Y, Goro T, Zensei M, Tomokazu M, Takashi U et al. Prevalence rate of nasal disease among MRSA nasal carriers. Jap. J. Rhinol. 2005;44(2):127-30. [ Links ]
29. Olarte NM, Valderrama IA, Reyes KR, Garzón MI, Escobar JA, Castro BE, et al. Colonización por Staphylococcus aureus resistente a la meticilina en una unidad de cuidados intensivos de adultos de un hospital colombiano: caracterización fenotípica y molecular con detección de un clon de circulación en la comunidad. Biomédica 2010;30:353-61. [ Links ]
30. Diep BA., Chambers HF, Graber CJ, Szumowski JD, Miller LG, Han LL, et al. Emergence of multidrug-resistant, community associated methicillin-resistant Staphylococcus aureus clone USA300 in men who have sex with men. Ann. Intern. Med 2008; 148:249-58. [ Links ]
31. Reyes J, Rincón S, Díaz L, Panesso D, Contreras GA, Zurita J, et al. Dissemination of methicillin-resistant Staphylococcus aureus USA300 sequence type 8 lineage in Latin America. Clin Infect Dis. 2009;49:1861-7. [ Links ]
32. Diep BA, Gill SR, Chang RF, Phan TH, Chen JH, Davidson MG, et al. Complete genome sequence of USA300, an epidemic clone of community-acquired meticillin-resistant Staphylococcus aureus. Lancet. 2006;367:731-9. [ Links ]
33. Escobar-Pérez JA, Castro BE, Márquez-Ortiz RA, Gaines S, Chavarro B, Moreno J, et al. Aislamientos de Staphylococcus aureus sensibles a meticilina relacionados genéticamente con el clon USA300, origen de los aislamientos SARM de genotipo comunitario en Colombia. Biomédica 2014; 34(Supl.1):124-36. [ Links ]
34. Rodríguez-Gascón M, Roig P, Montagud JB, Merino J. Acute Staphylococcus lugdunensis endocarditis with septic cerebral and pulmonary emboli, showing favorable evolution. Enferm Infecc Microbiol Clin. 2003;21:465. [ Links ]