Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Nova
Print version ISSN 1794-2470
Nova vol.10 no.18 Bogotá July/Dec. 2012
El ataque de las bacterias: cómo prevenirlo sin morir en el intento
The bacteria attack: how to prevent it and not die trying
Gladys Pinilla MSc1, Liliana Muñoz MSc1, Jeannette Navarrete MSc1, Paola Arévalo2
1Programa de Bacteriología y Laboratorio Clínico, Universidad Colegio Mayor de Cundinamarca. Bogotá, Colombia
2Programa Jóvenes Investigadores, COLCIENCIAS convocatoria 2012-2013. Bogotá, Colombia
Correspondencia: gpinillab@unicolmayor.edu.co / gpinillab@gmail.com
Recibido: 02-05-2012 Aceptado: 15-06-2012
RESUMEN
La diversidad de poblaciones microbianas que colonizan de forma permanente o transitoria la piel, depende de la topografía y fisiología de cada zona del cuerpo y cada área provee las condiciones necesarias para el desarrollo de microorganismos simbióticos, inofensivos e incluso beneficiosos, al mismo tiempo que regula las condiciones necesarias para evitar la colonización de agentes perjudiciales o patógenos. Alteraciones en este equilibrio dinámico, pueden causar infecciones locales o generalizadas por la diseminación de la flora normal de un sitio a otro.
El proceso evolutivo de la resistencia bacteriana ha sido corto y variable, condicionado en parte, por la presión selectiva ejercida frente a la terapia antimicrobiana. En respuesta, los microorganismos se han adaptado a las condiciones adversas mediante mecanismos de persistencia y resistencia que generan problemáticas a nivel epidemiológico, terapéutico y en la salud pública.
El fenómeno de la multiresistencia causa un impacto directo en la morbimortalidad e incrementa los costos en la atención en salud, por lo cual, además del correcto, apropiado y racional uso de los antimicrobianos, se requiere la prevención de las infecciones asociadas a la atención en salud mediante el control de la contaminación ambiental y el lavado de manos.
Finalmente, de las bacterias aprendemos que su interacción y organización comunitaria se constituyen en su “verdadera inteligencia” y podríamos morir en el intento de contener su ataque, si seguimos fortaleciendo las armas del “enemigo”.
Palabras clave: microorganismos, flora normal, antibióticos, resistencia bacteriana, prevención.
ABSTRACT
The enormous diversity of microbial populations that colonize the human skin in permanent or temporar way depends on the topography and physiology of each area of the body. Each area of which are harmless and even beneficial. At the same time that regulate the basic conditions to prevent pathogenic bacteria colonization. Alterations in this dynamic equilibrium could spread the normal flora to a different place generating local or systemic infections.
After the penicillin discovery for the treatment of infectious disease caused by bacteria, the microorganisms have developed strategies to survive through different mechanisms resulting in potentially ineffective antibiotics. Parallel to bacteria resistance has been the creation hundreds of antibiotics for clinical use.
The multi-drug resistance phenomenon has direct impact on morbidity, mortality rates and in the health care, increased cost, thus it is essential to have a correct, appropriate and a rational use of antimicrobials. One key tools to prevent infection, is the environmental pollution control and hand washing, not less important a new approach to the understanding of the pathophysiology, the prevention of spreading using natural products and the creation of innovative molecules such as antimicrobial peptides.
Key words: microorganisms, normal flora, antibiotic resistance, prevention.
INTRODUCCIÓN
En ambientes bióticos y abióticos existe gran variedad de bacterias, cuya supervivencia se relaciona con la capacidad para adaptarse gracias a la plasticidad de su genoma, sus características metabólicas y fenotípicas, su composición celular, la rápida respuesta que desarrollan frente a la presión selectiva y la formación de nichos ecológicos (1,2).
En el hombre, las bacterias se pueden encontrar como organismos comensales, es decir, como parte de la flora normal, o como agentes patógenos, asociados principalmente a infecciones. La diversidad de bacterias que componen la microbiota y su proceso de colonización se encuentra supeditado a la fisiología y factores ambientales como las áreas húmedas, secas y el microambiente sebáceo (3,4).
¿Cómo interactúan las bacterias con la piel?
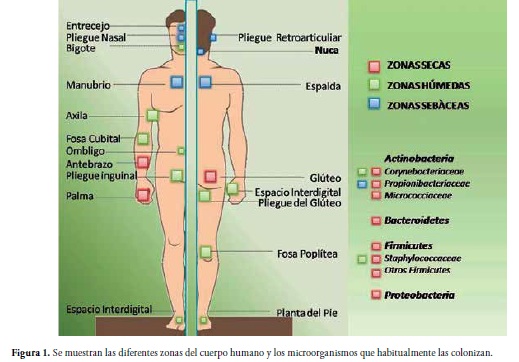
La piel es el órgano más grande del cuerpo humano y se encuentra colonizado por diversos microorganismos que se comportan de manera inofensiva, simbiótica e incluso beneficiosa para el huésped. La mayoría de las bacterias que la habitan se clasifican en cuatro phylum: Actinobacteria, Firmicutes, Bacteroidetes y Proteobacteria (5,6).
La piel posee características físicas y químicas que permiten la adaptación de un conjunto de microorganismos en cada zona. En general, mediante la excreción de agua, electrolitos y la acidificación, la superficie de la piel favorece el crecimiento de Staphylococcus coagulasa negativa (SCN) y Corinebacterias, mientras se inhibe la colonización de patógenos comunes, tales como S. aureus y S. pyogenes; por lo tanto, la oclusión de los poros de la piel, al favorecer la elevación del pH, promueve el crecimiento de estos últimos microorganismos (5-7).
La variabilidad de la flora microbiana del epitelio humano esta mediada por factores como: la ocupación, el uso de cosméticos, la exposición a rayos ultravioleta, el medio ambiente e incluso el uso de antibióticos; además influyen factores específicos de cada individuo como la edad, el género y la zona del cuerpo (5,6). La colonización de microorganismos se asocia directamente con la topografía, la temperatura, la humedad y el microambiente sebáceo de un individuo, lo cual permite la siguiente clasificación:
• Las zonas sebáceas están colonizadas principalmente por Propionibacterium sp., el cual mediante hidrolisis de los triglicéridos presentes en el sebo, obtiene los ácidos grasos requeridos para adherirse y colonizar el conducto auditivo externo, el pliegue retroarticular, el pliegue nasal, la cara, el pecho, la espalda y el entrecejo, Figura 1 (5,6).
• En las zonas húmedas, los microorganismos más abundantes son Corynebacterium sp. Y Staphylococcus sp. que utilizan la urea presente en el sudor como fuente de nitrógeno, lo cual, en asociación con otros microorganismos, genera el mal olor característico de zonas como la planta del pie, el ombligo, la axila, la ingle, el glúteo, la fosa poplítea y cubital, Figura 1 (5,6).
• La flora microbiana en las zonas secas es más variada que la encontrada en el intestino o en la cavidad oral. El antebrazo, los glúteos y algunas partes de las manos se encuentran colonizadas por abundantes microorganismos Gram negativos provenientes del tracto gastrointestinal, que en el pasado únicamente se consideraban contaminantes de la piel (5,6).
Como estrategia de sobrevivencia el hombre y las comunidades microbianas, conviven en un frágil equilibrio y cuando se altera esta relación, un microorganismo que hace parte de la flora normal de un sitio, coloniza e invade otras zonas. Así por ejemplo, Staphylococcus epidermidis puede sufrir cambios metabólicos indefinidos y complejos, que favorecen procesos infecciosos que, en ocasiones se asocian a la formación de biopelícula en dispositivos medicos mostrando resistencia a la meticilina y multirresistencia frente a otras clases de antimicrobianos. Otra característica que puede presentar es el efecto inhibitorio en la colonización de S. aureus en la fosa nasal, mediante la secreción de una serin proteasa Esp (7,8).
¿Qué pasa en los neonatos?
Durante la edad gestacional, el feto se encuentra en un ambiente estéril y en el momento del nacimiento se expone a gran variedad de bacterias presentes en el canal de parto y en el ambiente externo, como es el caso de los SCN, adquiridos de forma exógena proveniente de la microbiota de otros recién nacidos y las manos de los adultos, y/o endógena de la flora del mismo neonato. Durante el primer año de vida, algunas áreas de la cabeza, las fosas nasales y las axilas de los niños albergan un gran número de Staphylococcus sp., Propionibacterium sp., y Malassezia furfur (9-11).
La flora intestinal del neonato se encuentra influenciada por la microbiota de la madre y se desarrolla con mayor rapidez en el intestino de los bebes que nacen de forma natural en comparación con los que nacen por cesárea. La colonización regulada por los linfocitos T, dependerá de cada área del cuerpo humano y será modificada hasta llegar a la edad adulta, donde permanecerá constante mientras no se presente ninguna alteración inmunológica ni ambiental (10-13).
Medidas simples para un problema complejo.
En la práctica clínica es constante el riesgo de infección para el paciente y el profesional de salud, debido al contacto permanente con fluidos potencialmente infecciosos, que a su vez favorecen la diseminación de microorganismos en forma directa o indirecta, por medio de gotas, aerosoles, objetos inanimados, instrumentos y equipos contaminados, así como el contacto directo entre paciente y el profesional de salud. De esta manera, la adquisición de una infección nosocomial esta principalmente ligada a la contaminación del entorno y a las características del agente biológico (14,15).
Existen áreas sanitarias con mayor susceptibilidad a contaminación continua, en cada escenario varía de acuerdo a la ejecución de los protocolos de control de riesgo biológico establecidos. Sin embargo, toda superficie favorece la colonización de microorganismos, los cuales se establecen en poblaciones heterogéneas con un predomino de bacterias Gram positivas (16-18).
Las infecciones cruzadas se producen, en mayor medida, por microorganismos que colonizan las superficies que entran en contacto con el paciente y/o por las manos del personal de salud (18). Existen métodos de control del riesgo biológico que, de ser usados correctamente, garantizan la reducción y/o eliminación del contacto con microorganismos de interés clínico, entre los que se incluyen:
• La limpieza: consiste en la remoción de todo tipo de residuos visibles en las diferentes áreas, con lo cual se consigue una reducción significativa de la contaminación microbiana inicial; se realiza con agua, detergentes y productos enzimáticos y es considerado un paso vital, previo a la desinfección y/o esterilización, ya que su omisión evita el contacto directo de los agentes desinfectantes con las bacterias (19, 20).
• La desinfección: tiene como finalidad la destrucción de microorganismos que permanecen en las superficies después de la limpieza. La desinfección de alto nivel favorece la eliminación de todos los microorganismos, excepto los esporulados. La desinfección de nivel intermedio consigue eliminar formas vegetativas de bacterias, hongos y virus, pero no siempre las esporas bacterianas. Finalmente, la desinfección de bajo nivel consigue eliminar la mayoría de las bacterias, algunos virus y algunos hongos, sin contrarrestar microorganismos esporulados (20, 21).
• La esterilización: consiste en la eliminación de todas las formas vivientes patógenas, no patógenas, bacterias esporuladas y encapsuladas, por medio de métodos como radiación, calor seco y calor húmedo (19-21).
La relación existente entre los microorganismos presentes en el aire y las superficies no ha sido claramente dilucidada. Se sabe que las bacterias presentes en el aire eventualmente se depositan como contaminantes en las superficies y permanecen viables en el ambiente. Errores en el uso de las técnicas de remoción y eliminación de la contaminación hacen que los microorganismos sean capaces de sobrevivir y adaptarse al medio ambiente, mediante la formación de biopelículas en superficies inertes e incluso al interior del cuerpo humano (17,19, 21).
El vehículo ideal para la diseminación de la contaminación son las manos del personal de salud. Los espacios interdigitales, las unas y las palmas alojan un estimado bacteriano de 3.9 x 104 UFC a 4.6 x 106 UFC, conformado por la flora residente, que coloniza constantemente la superficie de la piel, es de baja virulencia y proporciona “resistencia a la colonización” al promover la defensa contra microorganismos oportunistas y por la flora transitoria, que es adquirida por contacto con otras personas, objetos, superficies y/o ambientes, tiene un grado de virulencia mayor y su permanencia en el ambiente es prolongada (22-23-24).
La eliminación de la flora bacteriana transitoria y la reducción de la flora residente se logran mediante el lavado de manos, la herramienta más importante, en la prevención de la contaminación cruzada, que debe considerarse como un procedimiento indispensable para el personal que se desempeña en el ámbito sanitario. Se debe realizar antes y después del contacto con pacientes y objetos inanimados, ya que, a pesar del uso de guantes, existe la posibilidad de la penetración de líquidos por agujeros no visibles (efecto wicking) (25-27).
¿Cómo ejercen su acción inhibitoria los antibióticos contra las bacterias?
La prevención de las infecciones surge desde los tiempos bíblicos, así lo demuestran los escritos del patriarca Moisés a los judíos y el tratamiento de estas data de la época de los antiguos egipcios, Babilónicos e Incas mediante el uso de sustancias naturales (28,29).
Uno de los avances relevantes del siglo XX, fue el surgimiento de los antibióticos. En 1928 el científico escoses Alexander Fleming, descubrió la penicilina; sus hallazgos fueron complementados por las investigaciones de Howard Walter Florey y Ernst Boris Chain, quienes descubrieron las propiedades químicas y biológicas producidas por el Penicillum notatum (actualmente antibióticos betalactamicos) (28-30).
A partir de los años cuarenta, con el descubrimiento de la penicilina y otros antibióticos, se da inicio a la denominada “era antibiótica”, cuya finalidad fue la desaparición de los agentes causantes de las infecciones. Poco tiempo después, las bacterias demostraron tener una organización comunitaria efectiva al desarrollar mecanismos que contrarrestaban el efecto de los antibióticos, con lo cual, hacia los años cincuenta, se empezó a manifestar rápidamente la resistencia microbiana.
A pesar de la síntesis de nuevas sustancias, para el año 1970, termina por considerarse la resistencia a fármacos como un problema de salud pública (31,32). A partir de 2006, se estima que más del 70% de las bacterias causantes de infecciones nosocomiales son resistentes al menos a uno de los fármacos comúnmente utilizados para contrarrestarlas.
En los últimos cuarenta años, tan solo dos nuevas clases de antibióticos (oxazolidinonas y lipopeptidos) han sido descubiertos y frente a las bacterias multidrogoresistentes, la vancomicina y sus derivados, continúan siendo el tratamiento de vanguardia, sin embargo ya existen cepas resistentes (28,31). Debido a ello, actualmente se continua con la búsqueda de nuevos compuestos antimicrobianos, tanto biológicos como semi-sinteticos y sintéticos, que favorezcan el desarrollo de novedosos mecanismos de acción de los antibióticos (33). Para comprender el fenómeno de la resistencia, es preciso conocer los mecanismos de acción de los antibióticos:
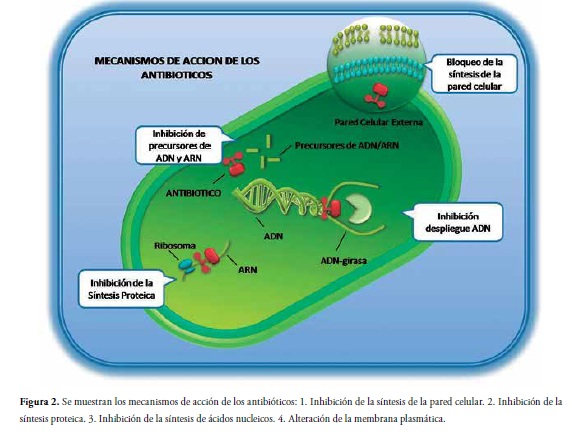
• Los antimicrobianos que inhiben la síntesis de la pared celular actúan inhibiendo la síntesis, transporte y organización del peptidoglicano, un componente esencial y especifico de la pared que contribuye en los procesos de crecimiento bacteriano. La ausencia de esta estructura causa la destrucción del microorganismo al permitir el aumento considerable de la osmoralidad del citoplasma bacteriano (28,34,35).
• Los antibióticos que inhiben las fases de síntesis proteica actúan directamente sobre las dos subunidades ribosomales 30S y 50S. Si el antimicrobiano se une con la subunidad 30S, se desata la incorporación errónea de aminoácidos durante la elongación de péptidos, dando como resultado proteínas alteradas funcionalmente; si la unión se da con la subunidad 50S, se bloquea el inicio de las transducción de las proteínas, se evita la transpeptidacion y el elongamiento de la cadena polipeptídica naciente (1,34,35).
• El grupo de antimicrobianos que altera la membrana plasmática actúa sobre la membrana interna (citoplasmatica) y la membrana externa (periplasmica), que contienen numerosas proteínas esenciales en el transporte de nutrientes y en la eliminación de sustancias toxicas, entre otras. Los antibióticos que alteran esta estructura modifican el metabolismo bacteriano, distorsionan la permeabilidad de la membrana con lo cual se altera el equilibrio iónico y se permite la entrada de sustancias a altas concentraciones, Figura 2, (1,28,34,35).
¿Tendrán los microorganismos la última palabra?
Las bacterias han desarrollado alternativas de adaptación y supervivencia, su gran velocidad para replicarse y la alta tasa de mutación han facilitado este proceso de microevolucion. A través de la presión selectiva las bacterias desarrollan la denominada tolerancia a un fármaco, es decir, son capaces de sobrevivir aun sin continuar la división celular mediante la formación de biopelículas, definidas como comunidades “inteligentes”, las bacterias coactuan proactivamente para evitar la destrucción masiva de un nicho, logrando persistir la acción de los antimicrobianos. Tanto la tolerancia como la persistencia son considerados precursores de la resistencia (36,37).
Han sido reconocidos diversos mecanismos de resistencia bacteriana que no muestran especificidad por ninguna especie, ni son exclusivos contra un tipo particular de antibiótico, entre ellos se destacan:
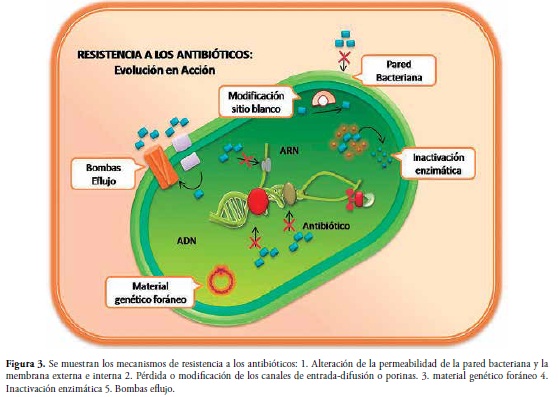
1. Mecanismos naturales o intrínsecos: hacen referencia a la resistencia natural de las bacterias, que es permanente en estas por determinación genética y que no se relaciona con la dosis del antibiótico. Debido a la disminución de la permeabilidad de la pared bacteriana y la membrana externa e interna y/o la pérdida o modificación de los canales de entrada-difusión o porinas, las bacterias son capaces de resistir a la acción del antibiótico al disminuir su entrada (28,38).
2. Mecanismos genéticos: es el tipo de resistencia adquirida, inducida en bacterias sometidas a un stress continuo, como el causado por los antibióticos, que deriva en diferentes mecanismos de defensa como la modificación en su genoma (mutación) o la denominada resistencia transmisible. Este último tipo de resistencia se encuentra mediada por la adquisición de material genético foráneo como plásmidos, transposones o integrones, elementos móviles que pueden ser diseminados a bacterias no patógenas por transferencia horizontal o lateral de genes (THG), en cepas de la misma especie o entre diferentes especies o géneros bacterianos, siendo este mecanismo el mayor determinante en la evolución de la patogenia bacteriana (38-41).
3. Mecanismos Bioquímicos
• Inactivación del antibiótico: mediante la acción de enzimas específicas, las bacterias logran resistir la acción antimicrobiana al inactivar o degradar el compuesto activo de un antibiótico. Estas enzimas son codificadas por genes del cromosoma bacteriano o por plasmidos extracromosomales y su síntesis puede ser constitutiva o inducible, es decir, luego de la exposición de la bacteria al antibiótico.
Principalmente, la modificación enzimática se da por hidrolisis, como sucede con las betalactamasas; además ocurren modificaciones no hidroliticas como acetilaciones, adenilaciones o fosforilaciones inactivantes de antibióticos aminoglucósidos (39,40).
• Modificaciones en el sitio blanco: consiste en la modificación de algunos sitios específicos de la estructura celular debido a mutaciones espontaneas en los genes del cromosoma bacteriano, específicamente a nivel de la RNA polimerasa, la DNA girasa, entre otras, que alteran los sitios blancos del antibiótico. A su vez, este cambio puede darse por THG principalmente por conjugación, y en menor medida por transducción y transformación (41,42).
• Bomba eflujo: es un sistema basado en la expulsión del antibiótico -sin modificarlo- del interior de la bacteria, llegando a reducir sus concentraciones a niveles subtóxicos. Las proteínas de membrana especializadas, cuya función normalmente es el transporte de metabolitos, pueden ser modificadas a través de mutaciones para promover la expulsión del antibiótico antes de llegar al sitio diana, mediante el eflujo activo, Figura 3, (43,44).
Mecanismo de persistencia por formación de biopelículas: consiste en un consorcio o comunidad bacteriana que se establece cuando las bacterias persisten unidas a superficies bióticas o abióticas. Se compone de una matriz de polímero extracelular que el mismo microorganismo produce, el cual consta principalmente de agua, polisacáridos proteínas y ADN extracelular. La formación de biopelicula resulta en un eficiente mecanismo para evadir la acción de los antibióticos, la fagocitosis y la acción de otros componentes del sistema de defensa innato, adaptativo e inflamatorio del organismo hospedero (45-47).
Los mecanismos de resistencia bacteriana se encuentran asociados al uso indiscriminado y/o abuso de antibióticos, a la dosis o duración inadecuada de la terapia antimicrobiana, al desconocimiento de los perfiles de sensibilidad de los diferentes gérmenes y a la difusión de cepas resistentes que se da principalmente por los inadecuados procedimientos de asepsia antes, durante y después de la atención del paciente en las áreas de salud. La asepsia se constituye en un punto crítico de control para prevenir la difusión de cepas patógenas o no patógenas, pero con potencialidad de convertirse en agentes infecciosos (oportunistas) (48-50).
CONCLUSIONES
Se hace evidente la necesidad de capacitación y actualización constante entre el personal sanitario para que, mediante la ejecución de los protocolos de control del riesgo biológico, se establezca un enfoque hacia el mantenimiento de la salud con actos preventivos, monitoreos ambientales, vigilancia epidemiológica y la prevención del riesgo de adquirir infecciones en la práctica clínica general.
La imparable diseminación de la multirresistencia evidencia la necesidad del desarrollo de nuevas alternativas antimicrobianas efectivas a corto, mediano y largo plazo, capaces de responder a los desafíos propuestos para el siglo XXI, que incluye el aumento de pacientes inmunocomprometidos, la aparición de nuevos agentes patógenos y la reemergencia de agentes con mayor virulencia.
Definitivamente, las bacterias nos ensenan, a través de la compleja interacción y organización en comunidades como las biopelículas, su verdadera inteligencia para interferir con la respuesta inmune del huésped y los tratamientos con antibióticos. Quizá hoy no sea exagerado afirmar que, ante la imposibilidad de saber el tiempo que nos tomara conocer y descubrir nuevas herramientas antimicrobianas, estamos a punto de sucumbir nuevamente en la era pre-antibiótica. Para no morir en el intento del ataque bacteriano es imperante entender como lo menciona el proverbio chino «Matar al ladrón no es cerrar la puerta» y que solo nos queda limitar la derrota, porque mientras usemos solo la terapia antimicrobiana, incluso de manera correcta, la microevolución en las bacterias, se asegurara de seguir su curso.
REFERENCIAS
1. Andersson, et al. Persistence of antibiotic resistance in bacterial populations. FEMS Microbiol Rev. 2011; 35(5):901-911. [ Links ]
2. Garmendia, L. et al. Metagenomics and antibiotics. Clinical Microbiology and Infection. 2012; 18: 27-31. [ Links ]
3. Clemente, J. et al. The Impact of the Gut Microbiota on Human Health: An Integrative View. Cell. 2012; 148 (6): 1258-1270. [ Links ]
4. Costello, E. et al. Bacterial Community Variation in Human Body Habitats Across Space and Time. Science. 2009; 326 (5960): 1694-1697. [ Links ]
5. Grice, E. et al. Topographical and temporal diversity of the human skin microbiome. Science 2009; 324:1190-1192. [ Links ]
6. Grice, E. et al. The skin microbiome. Nature Reviews Microbiology. 2011; 9: 244-253. [ Links ]
7. Peters, B. et al. Polymicrobial Interactions: Impact on Pathogenesis and Human Disease. Clin. Microbiol. 2012; 25 (1): 193-213. [ Links ]
8. Iwase, et al. Staphylococcus epidermidis Esp inhibits Staphylococcus aureus biofilm formation and nasal colonization. Nature 2010; 456: 346-349. [ Links ]
9. Park, H. Molecular Analysis of Colonized Bacteria in a Human Newborn Infant Gut. The Journal of Microbiology. 2005; 43 (4): 345-353. [ Links ]
10. Dominguez, M. et al. Delivery mode shapes the acquisition and structure of the initial microbiota across multiple body habitats in newborns. Proc. Natl Acad. Sci. USA. 2010; 107:11971-11975. [ Links ]
11. Reid, G. et al. Microbiota restoration: natural and supplemented recovery of human microbial communities. Nature Reviews Microbiology. 2011; 9: 27-38. [ Links ]
12. Brandt, K. et al. Establishment of the bacterial fecal community during the first month of life in Brazilian newborns. Clinics. 2012; 67 (2): 113-123. [ Links ]
13. Chia-Hui Y. et al. Host-microbial interactions and regulation of intestinal epithelial barrier function: From physiology to pathology. World J Gastrointest Pathophysiol. 2012; 3(1): 27-43. [ Links ]
14. Szymanska, J. Dental bioaerosol as an occupational hazard in a dentist's workplace. Annagric environmental medical. 2007; 14: 203-207. [ Links ]
15. Pasquarella, C. et al. Microbial environmental contamination in Italian dental clinics: A multicenter study yielding recommendations for standardized sampling methods and threshold valuesScience of The Total Environment. Elservier Science. 2012; 420, 289-299. [ Links ]
16. Zambrano, M. et al. Monitoreo bacteriológico de áreas clínicas odontológicas: estudio preliminar de un quirófano. Acta odontológica Venezolana. 2007; 45: 12-18. [ Links ]
17. Kramer, A. et al. How long do nosocomial pathogens persist on inanimate surfaces? A systematic review. BMC Infectious Diseases. 2006; 6, 130-137. [ Links ]
18. Álvarez, C. et al. Guías de práctica clínica para la prevención de infecciones intrahospitalarias asociadas al uso de dispositivos médicos. ACIN Capitulo Central. 2011; 3-96. [ Links ]
19. Just, M. et al. Hygiene in Medical Technology. Springer Handbook of Medical Technology. 2012; Part A, 11-34. [ Links ]
20. Szymanska, J. et al. Concentration and species composition of aerobic and facultatively anaerobic bacteria released to the air of a dental operation area before and after disinfection of dental unit waterlines. Annagric environ med. 2008; 2:301-307. [ Links ]
21. Castiglia, P. et al. Italian multicenter study on infection hazards during dental practice: control of environmental microbial contamination in public dental surgeries resins used at graduation clinics. BMC PublicHealth. 2008; 8:187. [ Links ]
22. Heinz-Michael, J. et al. Hygiene in Medical Technology. Springer Handbook of Medical Technology. 2012, Part A, 11-34. [ Links ]
23. CDC. Boyce J. et al. Manual Guía para la Higiene de manos en Centros Sanitarios. 2002; 1: 3-100. [ Links ]
24. Landers, T. et al. Patient-centered hand hygiene: The next step in infection prevention. American Journal of Infection Control.2012; 40 (4): S11-S17. [ Links ]
25. Lins, L. et al. Prevalence of hepatitis A, B and C and use of infection control procedures by dental health care workers in Salvador, Bahia, Brazil. Gazeta Medica da Bahia. 2009; 79 (2):9-12. [ Links ]
26. Chun, C. et al. The effectiveness of an air cleaner in controlling droplet/aerosol particle dispersion emitted from a patient's mouth in the indoor environment of dental clinics. J R Soc Interface. 2010; 8 (58): 699-710. [ Links ]
27. Buchter, A. et al. Development of a validated process for manual preparation of dental transmission instruments. Clinical Oral Investigation. 2011; 15(5): 605-608. [ Links ]
28. Navarrete, J. et al. Evolución histórica de los antibióticos. En Cátedra abierta - Diario de campo. 2012; 4:77-90Nueva versión internacional. La Biblia. Deuteronomio 14:3-8. [ Links ]
29. Lorenzo, Z. et al. History of Antibiotics. From Salvarsan to Cephalosporins. Journal of Investigative Surgery. 2012; 25 (2): 67-77. [ Links ]
30. Clardy, J. et al. The natural history of antibiotics. Curr Biol. 2009; 19 (11): R437-R441. [ Links ]
31. Álvarez, C. Uso prudente de antimicrobianos basado en el plan obligatorio de salud. Infectio. 2012; 16: 3-5. [ Links ]
32. Cortes, J. .Para donde vamos en investigación? Infectio. 2012; 16 (S1): 3-4. [ Links ]
33. Wright, G. et al. Antibiotics: A New Hope. Chemistry & Biology. 2012; 19 (1): 3-10. [ Links ]
34. Brandl, K. et al. Vancomycin-resistant enterococci exploit antibiotic- induced innate immune deficits. Nature. 2008; 455: 804-07. [ Links ]
35. Calvo, J. et al Mecanismos de acción de los antimicrobianos. Enfermedades Infecciosas y Microbiología Clínica. 2009; 27(1): 44-52. [ Links ]
36. Poole, K. Bacterial stress responses as determinants of antimicrobial resistance J. Antimicrob. Chemother. 2012; 67 (7): 196-200. [ Links ]
37. Bjarnsholt, T. et al. The role of quorum sensing in the pathogenicity of the cunning aggressor Pseudomonas aeruginosa.Analytical and bioanalytical chemistry. 2007; 409-414. [ Links ]
38. Munoz, l. et al. Mecanismos de resistencia bacteriana. Colombia Genesis.2006; 1: 43-56. [ Links ]
39. Wright et al. Molecular mechanisms of antibiotic resistance. Chem Commun (Camb). 2011; 47(14):4055-61. [ Links ]
40. Andam, C. et al. Multilevel populations and the evolution of antibiotic resistance through horizontal gene transfer. FEMS Microbiology Reviews. 2011; 35: 756-767. [ Links ]
41. Hall, R. et al. Mobile gene cassettes and integrons: moving antibiotic resistence genes in Gram negative bacterie. Origins, evolution, selection and spread. Ciba Foundation Symposium. 1997; 207:192-202. [ Links ]
42. D'Costa, V. et al. Antibiotic resistance is ancient. Nature.2011; 477: 457-461. [ Links ]
43. Poole, K. Efflux-Mediated Antimicrobial Resistance. Antibiotic Discovery and Development. Biomedical and Life Sciences. 2012; 3: 349-395. [ Links ]
44. Nikaido, H. et al. Broad-specificity efflux pumps and their role in multidrug resistance of Gram-negative bacteria. FEMS Microbiology Reviews. 2012; 36: 340-363. [ Links ]
45. Hoiby, N. et al. The clinical impact of bacterial biofilms. Int J Oral Sci.2011; 3:55-65. [ Links ]
46. Phillips, P. et al. Molecular Mechanisms of Biofilm Infection: Biofilm Virulence Factors. Advances in Wound Care. 2012; 1(3): 109-114. [ Links ]
47. O'Toole, G. Biofilm formation as microbial development. Annual Review of Microbiology 2000; 54: 49-79. [ Links ]
48. Leung, E. et al. The WHO policy package to combat antimicrobial resistance. Bull World Health Organ, Genebra. 2011; 89 (5). [ Links ]
49. Sosa, A. Containment of Antimicrobial Resistance in Developing Countries and Lessons LearnedBiomedical and Life Sciences. Antimicrobial Resistance in Developing Countries. 2010; 5: 447-461. [ Links ]
50. Mosquera, M. No fortalezca su enemigo. El Tiempo Sección Salud. Mayo 27 de 2012. Pag. 20. [ Links ]