Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Nova
versión impresa ISSN 1794-2470
Nova vol.13 no.24 Bogotá jul./dic. 2015
Artículo original producto de la investigación
Determinación de los genes, 16S ADNr, polA, y TpN47, en la detección de Treponema pallidum subsp. pallidum para el diagnóstico de sífilis congénita
Determination of genes, 16S ADNr, polA, and TpN47, in the detection of Treponema pallidum subsp. pallidum in the diagnosis of congenital syphilis
Gladys Pinilla B.1, Bibiana Chavarro P.1 Natalia Moreno A.2 Jeannette Navarrete O.1, Liliana Muñoz M.1
1 Programa de Bacteriología y Laboratorio Clínico, Universidad Colegio Mayor de Cundinamarca, Bogotá, Colombia
2 Programa Jóvenes Investigadores e Innovadores Colciencias. Grupo REMA. Colombia
Correspondencia: gpinillab@unicolmayor.edu.co
Recibido: 07/04/2015 Aceptado: 05/06/2015
Resumen
Objetivo. Comparar el comportamiento de tres genes diana 16S ADNr, polA, y TpN47, para la detección de T. pallidum subsp. pallidum. Métodos. Se usaron técnicas moleculares como la reacción en cadena de la polimerasa en muestras de cordón umbilical. Mediante PCR convencional, PCR anidada y PCR en tiempo real se amplificaron blancos moleculares del microorganismo. Resultados. Se evidenció que con los tres genes por PCR convencional se obtienen similares resultados, pero por con PCR anidada y PCR en Tiempo Real, el gen TpN47 tiene mayor sensibilidad en comparación con los genes polA y 16S ADNr. Se concluye que el gen TpN47 se puede usar como blanco molecular para el diagnóstico oportuno de sífilis congénita por medio de PCR anidada y en tiempo real, ya que alcanzó la máxima sensibilidad y especificidad en este estudio.
Palabras clave: Sífilis congénita, Treponema pallidum subsp. pallidum, PCR anidada, PCR en Tiempo Real.
Abstract
Objective. To compare the behavior of the three target genes (16S rDNA, polA, and TpN47) for the detection of T. pallidum subsp. Pallidum. Methods. Molecular techniques such as polymerase chain reaction were used on samples of an umbilical cord. Molecular targets of the microorganism were amplified by means of conventional PCR, nested PCR and real-time PCR. Results. Similar results for the three genes were obtained by conventional PCR; but in the case of nested PCR and real-time PCR, the gen TpN47 exhibited higher sensitivity in comparison to the genes polA and 16S rDNA. In conclusion, the gen TpN47 can be used as a molecular target for the prompt diagnosis of congenital syphilis through nested PCR and real-time PCR due to its high sensitivity and specificity shown in this study.
Key Words: Congenital syphilis, nested PCR, real-time PCR, Treponema pallidum subsp. pallidum.
Introducción
La sífilis es una infección de transmisión sexual (ITS), considerada un problema de salud pública, causada por el T. pallidum subsp. pallidum, que en ausencia de tratamiento permanece latente indefinidamente en los pacientes infectados; adicionalmente, se ha reportado que existe una significativa asociación entre la sífilis y el riesgo de incrementar la infección por VIH (1,2).
La transmisión transplacentaria del patógeno o sífilis congénita (SC), se caracteriza por la ausencia de manifestaciones de la sífilis temprana en el feto. En estos casos, una vez el T. pallidum subsp. pallidum ha alcanzado el torrente sanguíneo, se evidencia la afectación de órganos, hematopoyesis extramedular y manifestaciones derivadas de la respuesta inmune local. La Organización Panamericana de la Salud (OPS) define el caso confirmado de SC teniendo en cuenta otros parámetros del "producto de la gestación" (aborto, mortinato o nacido vivo) (3,4).
La sífilis tiene un alto impacto socioeconómico ya que durante el embarazo provoca cerca de 305.000 muertes fetales y neonatales, y deja 215.000 lactantes en riesgo de muerte cada año (2). Entre los programas y políticas sanitarias implementadas por Colombia, donde es considera como una enfermedad venérea de notificación y declaración obligatoria (3), se encuentra la tamización para sífilis en mujeres gestantes durante el control prenatal y al momento del parto; sin embargo, la falta de diagnóstico precoz y la falta de cobertura nacional para el manejo adecuado de la sífilis gestacional (SG), ha ocasionado un aumento considerable de los casos de sífilis congénita (SC) y neurosifilis (NS).
El diagnóstico de SC se realiza de forma presuntiva y puede basarse en la aparición de características clínicas específicas, aunque la mayoría de los casos es asintomática. La reacción en cadena de la polimerasa (PCR) es una herramienta diagnóstica de alta sensibilidad y especificidad, utilizada durante las últimas dos décadas, en la detección del T. pallidum subsp. pallidum en muestras clínicas como sangre total, suero fetal neonatal y materno, LCR, ulceras genitales, chancros, humor acuoso, sangre total y líquido amniótico entre otros (5-9). Dentro de los genes que se han empleado como herramienta molecular diagnóstica, se encuentran los genes 16S ARNr, polA, tpr, arp, TpN47, tpf-1, bmp, tmpA, tmpB (6, 8). Algunos de los más usados son el gen TpN47, que codifica para la proteína de membrana de 47-kDa; el gen polA, que codifica para la ADN polimerasa I de T. pallidum subsp. pallidum, y el gen 16S ARN ribosomal que posee secuencias altamente conservadas que favorecen la tipificación de la espiroqueta (10-13).
El objetivo del presente trabajo fue determinar la presencia de los genes 16S ADNr, polA y TpN47, como dianas diagnósticas, en muestras de sangre de cordón umbilical inoculadas experimentalmente, mediante PCR convencional, PCR anidada y PCR en tiempo real, para su uso en la detección del T. pallidum subsp. pallidum.
Materiales y métodos
Tipo de muestra: el microorganismo T. pallidum subsp. pallidum se obtuvo a partir de exudados de testículos de conejos infectados con la cepa control Nichols, proporcionada por el Departamento de Inmunopatología de la Sífilis de la Universidad de Washington Seattle USA. Cada exudado fue resuspendido en Buffer de lisis 2X (Tris 10mM pH 8.0, SDS 0,5% y EDTA 0.1M pH 8.0).
Infección de muestras: las inoculaciones en muestras negativas de sangre de cordón umbilical (1 mL) fueron realizadas con exudados de testículo infectado de conejo (25 ul, 50 ul y 100 ul).
Extracción de ADN: para obtener el ADN del T. pallidum subsp. pallidum se realizó un pre tratamiento con 17.5 ul de Proteinasa K (20 mg/mL) y se incubó en baño seco a 56°C por 1 hora con la solución de lisis, teniendo en cuenta las recomendaciones suministrada en el kit Genomic DNA Purification (Promega®). Para las muestras de sangre de cordón umbilical inoculadas, se tomaron 500 ul de estas para realizar un pre tratamiento con 50 ul de Proteinasa K (20 mg/mL), mediante incubación en baño seco a 65°C por 1 hora; posteriormente se procedió con la extracción de ADN descrita anteriormente.
La cuantificación del ADN extraído se hizo en el equipo NanoDrop® y verificando la pureza a través de geles de agarosa al 1.8%.
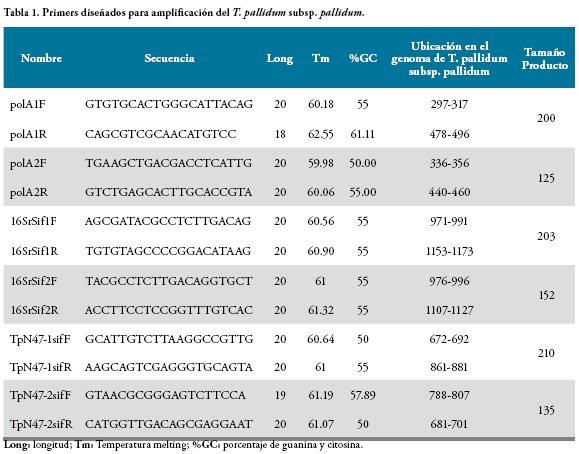
PCR convencional y anidada: para la amplificación de los tres genes diana (polA, 16S ADNr y TpN47), las condiciones de normalización para la amplificación de una reacción de 20 ul fueron: MgCl2 3 mM, dNTPs 200 uM, primers sentido y antisentido 0.25 uM polA1, 16Ssif1 y TpN471 (Tabla 1), Taq polimerasa (Promega®) 0.5 U y ADN de T. pallidum subsp. pallidum a diferentes concentraciones. La reacción de amplificación se realizó en un termociclador (Maxigene) a 94°C por 5 min; 94°C por 30 seg, anillaje que depende de cada gen (polA 63°C, 16S ADNr y TpN47 62°C) y extensión (72°C) por 35 ciclos. Los productos fueron analizados por geles de agarosa al 1.8% teñidos son GR SAFE®.
Los productos de amplificación obtenidos por PCR convencional, se re-amplificaron utilizando PCR anidada mediante una segunda pareja de primers internos (polA2, 16Ssif2 y TpN47-2); las condiciones usadas fueron las anteriormente mencionadas.
PCR en Tiempo Real (PCR - tr): para la detección del gen TpN47 por PCR -tr se usó el kit Platinum® SYBR® green qPCR SuperMix-UDG (Invitrogen) como agente intercalante y se llevó a cabo en el termociclador iQ5 (BioRad). Las condiciones utilizadas para una reacción de 25 ul fueron SYBR® Green 1X, primers sentido y antisentido 0.14 uM, Tabla 1.
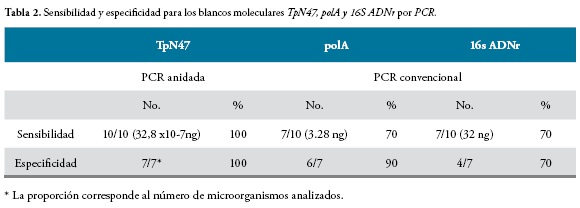
Sensibilidad y especificidad: para estos ensayos se determinó la mínima cantidad de ADN que se detecta con la amplificación de cada uno de los genes y teniendo en cuenta cada tipo de PCR utilizada. Para analizar la especificidad de las pruebas, se utilizó ADN humano de individuos sanos y 6 diferentes microorganismos con el fin de establecer la ausencia de reactividad cruzada con los mismos (S. enteritidis, K. oxytoca, P. mirabilis, E. coli, E. aerogenes, C. freundii).
Clonación: para realizar una curva de calibración con el número de copias detectado del T. pallidum subsp. pallidum, se clonó un inserto de 210 pb obtenido como amplicon por PCR a partir de la secuencia del gen TpN47, en el vector de clonación pJET1.2/blunt con el Kit CloneJET™ PCR Cloning (Thermo Scientific™) siguiendo las recomendaciones del fabricante y usando células E. coli TOP10 para la transformación; el rastreo de colonias se llevó a cabo por PCR convencional.
Resultados
PCR convencional y anidada
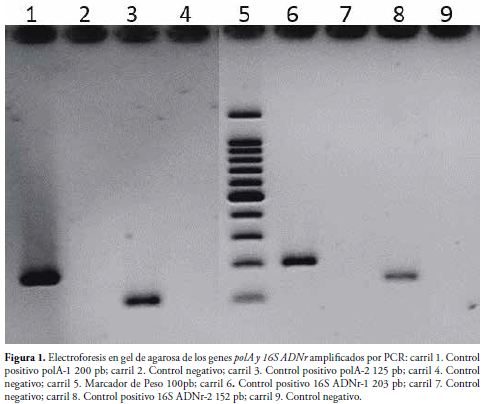
A partir de las extracciones de ADN de T. pallidum subsp. pallidum, las cuales fueron monitoreadas tanto por espectrofotometría como por electroforesis en geles de agarosa (datos no mostrados), se procedió a los ensayos de PCR convencional usando dos parejas de primers para cada uno de los genes con el objetivo de utilizarlos posteriormente en un ensayo de PCR anidada, Figura 1.
Con el gen TpN47 se evidenció una sensibilidad del orden de picogramos utilizando PCR anidada mientras que con los genes polA y 16S ADNr se llegó al orden de nanogramos por PCR convencional, Tabla 2.
PCR en Tiempo Real
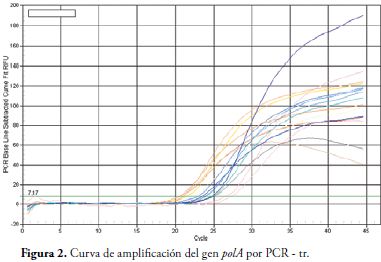
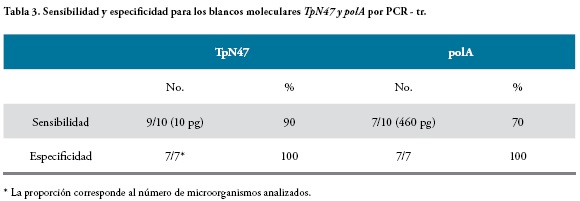
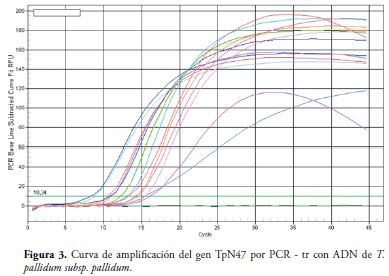
Diluciones seriadas (en base 2) de ADN de T. pallidum subsp. pallidum, se analizaron para la amplificación del gen polA; los valores de Ct obtenidos oscilaron entre: 20,74 y 25,05, considerado como un resultado positivo, Figura 2, y valores de Ct por encima de 32.0 se consideró como un resultado negativo, Tabla 3. Muestras de ADN humano y de los otros microorganismos como S. enteritidis, K. oxytoca, P. mirabilis, E. coli, E. aerogenes, C. freundii, se analizaron con el fin de evaluar la especificidad frente a estos, la cual fue del 100%, Tabla 3. Para el gen TpN47, diluciones seriadas de ADN de T. pallidum subsp. pallidum fueron analizadas, encontrando que los valores de Ct considerados positivos oscilan entre: 27.0 - 29.0 y resultados negativos por encima de 32.0, Figura 3.
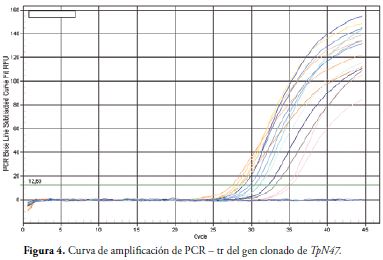
Para establecer con exactitud el número de copias detectadas por PCR en tiempo real, se realizaron 14 diluciones seriadas con ADN clonado del gen TpN47 determinando que los valores de Ct entre 10.52 y 16.13 son consideran como resultados positivos y valores por encima de 32.0 como resultado negativo, Figura 4. El análisis de TpN47 en muestras de ADN humano y de los otros microorganismos mostro un 100% de especificidad, Tabla 3.
Discusión
Se han descrito diversos tipos de PCR para el hallazgo de T. pallidum subsp. pallidum, mediante la amplificación de genes diana con potencial diagnóstico para sífilis congénita en muestras clínicas. Dentro de los genes que se han empleado están el 16S ADNr, polA, TpN47, utilizados en este estudio (6, 8, 10-13).
El genoma del T. pallidum subsp. pallidum tiene un tamaño relativamente pequeño: 1.138.006 pares de bases que contienen 1041 marcos de lectura abiertos (ORFs) (14). En el año 2013 se realizó la re-secuenciación completa del genoma y se detectaron aproximadamente un 11.5% de errores en los genomas secuenciados (119 genes afectados). Esta re-secuenciación mostró que hay dos subgrupos genéticos distintos que causan sífilis en los seres humanos. Por esta razón, para el diseño y uso de los primers en el presente estudio, se tomaron las secuencias reportadas como resultado de la resecuenciación del genoma de la espiroqueta (15).
El ARN ribosomal del T. pallidum subsp. pallidum posee secuencias altamente conservadas que además de favorecer la tipificación de la espiroqueta, ha permitido la comprensión de la evolución del genoma del microorganismo, así como las relaciones filogenéticas entre las especies del género. En nuestro trabajo se amplificó una región de 203 pb del 16S ADNr del T. pallidum subsp. pallidum, detectada mediante PCR convencional, alcanzando menos sensibilidad (32 ng) que la obtenida con el gen polA (3,28 ng) para la detección de ADN de la espiroqueta. Estos resultados contrastan con el uso de RT-PCR para amplificar el gen 16S ARN detectándose un único organismo en muestras control (16). Por otra parte, para un diagnóstico clínico, el protocolo usado en este último estudio, es más complejo, costoso y requiere de confirmación mediante el uso de Southern blot, lo cual dificultaría el empleo de esta tecnología en laboratorios clínicos no especializados.
En cuanto al uso del gen polA, que codifica para la ADN polimerasa I del T. pallidum subsp. pallidum, ha sido ampliamente estudiado debido a la homología de la enzima con las ADN polimerasas I de otros microorganismos. En este estudio, los resultados con este gen, muestran que se logra una sensibilidad del orden de nanogramos por PCR convencional y de picogramos con PCR-tr (ver tablas 2 y 3). Se ha reportado el uso del gen polA, con una sensibilidad de 95.8% y una especificidad de 95.7%, similar a nuestros resultados, lo que sugiere que se puede usar como método diagnóstico de rutina para la sífilis (17, 21). Es de anotar que para la cuantificación del número de espiroquetas obtenida en otros estudios con muestras de sangre total de pacientes adultos, se estableció una sensibilidad de 15 a 150 copias/ml (8).
Con el gen TpN47 con PCR anidada, se llega a una sensibilidad (100%) del orden de picogramos (0,032 pg) y a una especificidad del 100%. Cabe destacar que esta muestra corresponde a un ADN purificado y que en la práctica clínica, posiblemente no se llegue a este orden de sensibilidad. Por otra parte, este gen fue usado para el diagnóstico de NS en LCR, comparado con las pruebas diagnósticas establecidas por la Organización Mundial de la Salud (OMS), donde 0evidenció la capacidad del microorganismo para invadir el SNC durante las etapas primarias de la enfermedad (18). Asimismo, usando tejidos embebidos en parafina, muestras de líquido amniótico, suero y LCR neonatal, se obtuvo una sensibilidad de aproximadamente 0,01 pg de ADN purificado de T. pallidum subsp. pallidum, después de la amplificación por PCR (13); estos resultados llegan al mismo orden de sensibilidad al que se reporta en este estudio.
Debido a los hallazgos promisorios con el uso del gen TpN47, se ha diseñado una PCR-tr que detecta T. pallidum subsp. pallidum sin reactividad cruzada con treponemas de la flora común de la piel o con patógenos asociados a ITS; esta prueba se puede utilizar para detección y cuantificación del microorganismo en diferentes lesiones y etapas de la enfermedad, encontrándose que en la sífilis primaria hay gran cantidad de microorganismos en úlceras, pero un número de copias bajo en sangre; en la sífilis secundaria hay una mayor bacteremia, mientras que en la etapa latente, la bacteremia y la carga treponemica se detecta con menor frecuencia (20).
Aunque el gen TpN47 es el que presenta los mejores resultados en cuanto a sensibilidad y especificidad, los genes 16S ADNr y polA, podrían ser usados para la tipificación molecular que surge como herramienta importante para vigilar la aparición de cepas resistentes a macrólidos, evaluar los subtipos asociados con la afección del SNC, diferenciar procesos de infección y re-infección, así como la comprensión de la transmisión de T. pallidum subsp. pallidum y el comportamiento epidemiológico de la enfermedad. Para este mismo fin, se ha utilizado el gen arp (acidic repeat protein) y la familia de genes tpr subfamilia II (tprE, tprG y tprJ) (22, 23). Posteriormente, se adoptó el estudio conjunto con los genes rpsA y tp0548 para aumentar la capacidad discriminatoria de la tipificación de las cepas (24, 25).
Además de la sensibilidad, las técnicas moleculares requieren evidenciar especificidad analítica, ya que otras espiroquetas y agentes causales de ITS pueden ser detectadas en la misma muestra (10, 11, 28, 30). Por esta razón, se realizó un estudio in silico, previo a los ensayos experimentales, los cuales mostraron que la especificidad con el gen TpN47 fue del 100%.
Teniendo en cuenta estos resultados, el gen TpN47 sería el candidato como blanco diagnóstico para la detección efectiva y eficaz de sífilis congénita, aportando en la contención de este problema de alto impacto en salud pública, que se relaciona con otras ITS como el VIH, por lo cual es necesario el oportuno diagnóstico y tratamiento para así disminuir el índice de morbimortalidad de neonatos en Colombia.
Referencias
1. Organización Panamericana de la Salud. Iniciativa regional para la eliminación de la transmisión materno-infantil de VIH y de la sífilis congénita en América Latina y el Caribe: documento conceptual. Montevideo, Uruguay: OPS, 2009. Publicación Científica CLAP/SMR 1569. [ Links ]
2. Organización Mundial de la Salud. Infecciones de transmisión sexual. Suiza: Nota descriptiva Nº110, Noviembre de 2013 [consultado el 9 de Noviembre de 2015]. Disponible en http://www.who.int/mediacentre/factsheets/fs110/es/. [ Links ]
3. Ministerio de Salud y Protección Social. Guía de Práctica Clínica (GPC) basada en la evidencia para la atención integral de la sífilis gestacional y congénita. Bogotá, Colombia: MinSalud, 2014. GPC-2014-41. [ Links ]
4. Organización Panamericana de la Salud. Sífilis Materna y Sífilis Congénita: Definiciones de Caso, Boletin Epidemiologico. Estados Unidos: OPS, 2005. Vol. 26, No. 1. [ Links ]
5. Jethwa HS, Schmitz JL, Dallabetta G, Behets F, Hoffman I, Hamilton H, et al. Comparison of molecular and microscopic techniques for detection of Treponema pallidum in genital ulcers. J Clin Microbiol. 1995; 33(1): 180-183. [ Links ]
6. Grimprel E, Sanchez PJ, Wendel GD, Burstain JM, McCracken Jr, GH, Radolf JD, et al. Use of polymerase chain reaction and rabbit infectivity testing to detect Treponema pallidum in amniotic fluid, fetal and neonatal sera, and cerebrospinal fluid. J Clin Microbiol. 1991; 29(8): 1711-1718. [ Links ]
7. Genest DR, Choi-Hong SR, Tate JE, Qureshi F, Jacques SM, Crum C. Diagnosis of congenital syphilis from placental examination: comparison of histopathology, Steiner stain, and polymerase chain reaction for Treponema pallidum DNA. Hum Pathol. 1996; 27(4): 366-372. [ Links ]
8. Cruz AR, Pillay A, Zuluaga AV, Ramirez LG, Duque JE, Aristizabal GE, et al. Secondary Syphilis in Cali, Colombia: New Concepts in Disease Pathogenesis. PLoS Negl Trop Dis. 2010; 4(5): e690. [ Links ]
9. de Souza Campos Fernandes RC, Medina-Acosta E. Congenital neurosyphilis. Lancet Infect Dis. 2013; 13(6): 474-475. [ Links ]
10. Orle KA, Gates CA, Martin DH, Body BA and Weiss JB. Simultaneous PCR Detection of Haemophilus ducreyi, Treponema pallidum, and Herpes Simplex Virus Types 1 and 2 from Genital Ulcers. J Clin Microbiol. 1996; 34(1): 49-54. [ Links ]
11. Hay PE, Clarke JR, Strugnell RA, Taylor-Robinson D, Goldmeier D. Use of the polymerase chain reaction to detect DNA sequences specific to pathogenic treponemes in cerebrospinal fluid. FEMS Microbiol Lett. 1990; 56(3): 233-238. [ Links ]
12. Hay PE Clarke JR, Taylor-Robinson D, Goldmeier D. Detection of treponemal DNA in the CSF of patients with syphilis and HIV infection using the polymerase chain reaction. Genitourin Med. 1990; 66(6): 428-432. [ Links ]
13. Burstain JM, Grimprel E, Lukehart SA, Norgard MV, Radolf JD. Sensitive detection of Treponema pallidum by using the polymerase chain reaction. J Clin Microbiol. 1991; 29(1): 62-69. [ Links ]
14. Fraser CM, Norris SJ, Weinstock GM, White O, Sutton GG, Dodson R. Complete Genome Sequence of Treponema pallidum, The Syphilis Spirochete. Science. 1998; 281(5375): 375-388. [ Links ]
15. Pĕtrošová H, Pospíšilová P, Strouhal M, Čejková D, Zobaníková M, Mikalová L, et al. Resequencing of Treponema pallidum ssp. pallidum strains Nichols and SS14: correction of sequencing errors resulted in increased separation of syphilis treponeme subclusters. PLoS One. 2013; 8(9): e74319. [ Links ]
16. Centurion-Lara A, Castro C, Shaffer JM, Van Voorhis WC, Marra CM, Lukehart SA. Detection of Treponema pallidum by a sensitive reverse Transcriptase PCR. J Clin Microbiol. 1997; 35(6): 1348-1352. [ Links ]
17. Liu H, Rodes B, Chen C-Y, Steiner B. New Tests for Syphilis: Rational Design of a PCR Method for Detection of Treponema pallidum in Clinical Specimens Using Unique Regions of the DNA Polymerase I Gene. J Clin Microbiol. 2001; 39(5): 1941-1946. [ Links ]
18. Grange PA, Gressier L, Dion PL, Farhi D, Benhaddou N, Gerhardt P. Evaluation of a PCR test for detection of treponema pallidum in swabs and blood. J Clin Microbiol. 2012; 50(3): 546-552. [ Links ]
19. Chung KY, Lee MG, Lee JB. Detection of Treponema pallidum by polymerase chain reaction in the cerebrospinal fluid of syphilis patients. Yonsei Med J. 1994; 35(2): 190-197. [ Links ]
20. Tipple C, Hanna M, Hill S, Daniel J, Goldmeier D, McClure M, Taylor G. Getting the measure of syphilis: qPCR to better understand early infection. Sex Transm Infect. 2011; 87(6): 479-485. [ Links ]
21. Shields M, Guy RJ, Jeoffreys NJ, Finlayson RJ, Donovan B. A longitudinal evaluation of Treponema pallidum PCR testing in early syphilis. BMC Infect Dis. 2012; 12: 353. [ Links ]
22. Peng RR, Wang AL, Li J, Tucker JD, Yin YP, Chen XS. Molecular Typing of Treponema pallidum: A Systematic Review and Meta-Analysis. PLoS Negl Trop Dis. 2011; 5(11): e1273. [ Links ]
23. Pillay A, Liu H, Chen CY, Holloway B, Sturm AW, Steiner B, Morse SA. Molecular subtyping of Treponema pallidum subspecies pallidum. Sex Transm Dis. 1998; 25(8): 408-414. [ Links ]
24. Katz KA, Pillay A, Ahrens K, Kohn RP, Hermanstyne K, Bernstein KT, et al. Molecular epidemiology of syphilis--San Francisco, 2004-2007. Sex Transm Dis. 2010; 37(10): 660-663. [ Links ]
25. Marra C, Sahi S, Tantalo L, Godornes C, Reid T, Behets F, et al. Enhanced Molecular Typing of Treponema pallidum: Geographical Distribution of Strain Types and Association with Neurosyphilis. J Infect Dis. 2010; 202(9): 1380-1388. [ Links ]
26. Stamm LV, Bergen HL. A Point Mutation Associated with Bacterial Macrolide Resistance Is Present in Both 23S rRNA Genes of an Erythromycin-Resistant Treponema pallidum Clinical Isolate. Antimicrob Agents Chemother. 2000; 44(3): 806-807. [ Links ]
27. Michelow IC, Wendel GD Jr, Norgard MV, Zeray F, Leos NK, Alsaadi R, Sánchez PJ. Central nervous system infection in congenital syphilis. N Engl J Med. 2002; 346(23): 1792-1798. [ Links ]
28. García P, Grassi B, Fich F, Salvo A, Araya L, Abarzúa F, et al. Diagnóstico de la infección por Treponema pallidum en pacientes con sífilis temprana y neurosífilis mediante reacción de la polimerasa en cadena. Rev Chilena Infectol. 2011; 28(4): 310-315. [ Links ]
29. Koek AG, Bruisten SM, Dierdorp M, van Dam AP, Templeton K. Specific and sensitive diagnosis of syphilis using a real-time PCR for Treponema pallidum. Clin Microbiol Infect. 2006; 12(12): 1233-1236. [ Links ]
30. Heymans R, van der Helm JJ, de Vries HJ, Fennema HS, Coutinho RA, Bruisten SM. Clinical Value of Treponema pallidum Real-Time PCR for Diagnosis of Syphilis. J Clin Microbiol. 2010; 48(2): 497-502. [ Links ]
31. Limberger RJ, Slivienski LL, El-Afandi MC, Dantuono LA. Organization, transcription, and expression of the 5' region of the fla operon of Treponema phagedenis and Treponema pallidum. J Bacteriol. 1996; 178(15): 4628-4634. [ Links ]
32. Chen CY, Chi KH, Pillay A, Nachamkin E, Su JR, Ballard RC. Detection of the A2058G and A2059G 23S rRNA gene point mutations associated with azithromycin resistance in Treponema pallidum by use of a TaqMan real-time multiplex PCR assay. J Clin Microbiol. 2013; 51(3): 908-913. [ Links ]
33. Pandori MW, Gordones C, Castro L, Engelman J, Siedner M, Lukehart S, Klausner J. Detection of azithromycin resistance in Treponema pallidum by real-time PCR. Antimicrob Agents Chemother. 2007; 51(9): 3425-3430. [ Links ]
34. Ghanem KG. Neurosyphilis: A historical perspective and review. CNS Neurosci Ther. 2010; 16(5): e157-68. [ Links ]