Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Nova
Print version ISSN 1794-2470
Nova vol.14 no.25 Bogotá Jan./June 2016
Perfil de resistencia a antibióticos en bacterias que presentan la enzima NDM-1 y sus mecanismos asociados: una revisión sistemática
A profile of resistance in bacteria and the mechanisms associated due to the presence of the enzyme NDM-1: a systematic review
Jeimmy Castañeda1, Karen Gómez1, Lucía Corrales2, Sebastián Cortés3
1 Estudiantes Universidad Colegio Mayor de Cundinamarca, Bogotá, Colombia.
2 Docente investigador Universidad Colegio Mayor de Cundinamarca, Bogotá, Colombia.
3 Investigador eje de salud pública Fundación Santa Fé de Bogotá, Bogotá, Colombia.
ID Orcid 0000-0002-2398-348X Correspondencia: lcorrales@unicolmayor.edu.co
Recibido: 02-02-2016 Aceptado: 15-04-2016
Resumen
En 2008 se reportó la enzima NDM-1 que genera resistencia a carbapenémicos. Para determinar el perfil de resistencia que presentan las bacterias frente a esta enzima se adelantó una revisión sistemática. La búsqueda de artículos se realizó en diferentes bases de datos y para su selección se consideraron criterios de inclusión y exclusión obteniendo un total de 154 artículos.
Se identificaron 617 casos, presentados en trece géneros bacterianos, que codifican para los cuatro mecanismos de resistencia, principalmente a betalactámicos y aminoglucósidos. Cuando se presenta la enzima, la posibilidad que haya genes asociados para la producción de resistencias es alta, generando así que se presenten mecanismos que evitan la acción del antibiótico haciendo difícil implementar un tratamiento efectivo.
Palabras clave: NDM-1, métalo-beta-lactamasas, resistencia bacteriana.
Abstract
In 2008 the NDM-1 enzyme was first reported, the enzyme responsible for the resistance to carbapenems. We conducted a systematic review to determine the resistance profile due to the presence of this enzyme in bacteria. We searched academic articles in principal databases resulting in the selection of 154 articles given our inclusion and exclusion criteria.
617 cases and 13 bacterial genera were reported in our sample. We find 4 resistance mechanisms which are principally resistant to beta-lactams and aminoglycosides. Hence, we have that the presence of the NDM-1 increases the likelihood of having genes that improves the bacteria's resistance dramatically. The presence of the NDM-1 induces mechanisms which impacts the effectiveness of antibiotics and appropriate treatments are difficult to find.
Key Words: NDM-1, metallo-beta-lactamases, bacterial resistance.
Introducción
La resistencia antimicrobiana es el fenómeno por el cual los microorganismos disminuyen la acción de los antimicrobianos, de modo tal que los tratamientos no cumplen con su objetivo y como resultado las infecciones persisten. Dicha resistencia ha aumentado en los últimos años, especialmente a antibióticos como las quinolonas, carbapenémicos y cefalosporinas de tercera generación. El elevado consumo de antibióticos sin prescripción médica, además del flujo constante de especies patógenas en el entorno médico, favorece el desarrollo de la resistencia y amenaza el éxito de los tratamientos en todos los niveles de atención en salud, generándose así un entorno ideal para la propagación de bacterias resistentes y la transferencia de genes de resistencia. Por consiguiente, el manejo de las bacterias resistentes es un tema clave, y por esto se requiere la implementación de sistemas de vigilancia a nivel mundial para contrarrestar la disminución en la eficacia de los antibióticos (1-2). En el informe ''Antimicrobial resistance: global report on surveillance" publicado por la OMS en el año 2014 en Ginebra, se pone de manifiesto la gravedad de la situación actual frente a las consecuencias que está generando la resistencia a los antimicrobianos a nivel mundial, dado que las bacterias multire-sistentes surgen como un importante problema y amenaza para la salud pública (3).
Un claro ejemplo en la aparición de bacterias multiresistentes por el desarrollo o adquisición de diferentes mecanismos es el de la enzima NDM-1, la cual ocasiona en las bacterias que la presentan multirresistencia a una variedad de antibióticos incluidos los carbapenémicos. Se ha descrito que la enzima NDM-1 está presente en diferentes géneros y especies bacterianos, por cuanto el gen bla-NDM que codifica para la síntesis de la enzima se moviliza fácilmente entre diferentes cepas bacterianas generando preocupaciones considerables en el ámbito de la salud pública (4).
Actualmente existen reportes limitados respecto a la epidemiologia, diseminación, reporte y situación actual de la métalo-b-lactamasa NDM-1 en Colombia, pese a que es una grave problemática que se está presentando a nivel mundial. El objetivo de la revisión fue determinar el perfil de resistencia a antibióticos en bacterias que presentan la enzima NDM-1 y sus mecanismos de resistencia asociados, mediante una revisión sistemática de los reportes de caso que se han presentado en el mundo, con los siguientes objetivos específicos:
- Identificar los principales genes asociados a los mecanismos de resistencia en las bacterias productoras de NDM-1.
- Relacionar a partir de los reportes los principales antibióticos a los que presentan resistencia las bacterias productoras de NDM-1.
- Determinar las especies bacterianas que presentan mayor prevalencia en la producción de la enzima NDM-1 a nivel mundial.
- Comparar la situación de Colombia con otros países en términos de reportes de la enzima NDM-1.
En la revisión se tuvo como base los estudios primarios altamente potenciales que incluyeran como objeto de estudio la enzima NDM-1 y su resistencia y finalmente por medio de un meta-análisis como herramienta estadística, se realizó una síntesis de los resultados de las búsquedas realizadas logrando dar una estimación global en cuanto a los mecanismos genéticos utilizados por las bacterias y el perfil de resistencia que estas presentan.
Materiales y métodos
Búsqueda de la información y criterios de selección
Se realizó una revisión bibliográfica de manera sistemática sobre la presencia de la enzima Nueva Delhi Metallobelactamasa-1 y la resistencia que esta genera. Dicha revisión se efectuó en las bases de datos Medline, Cochrane, Embase y Lilacs, para esto se siguieron los lineamientos del libro Systematic Reviews in Health Care (5) y no se revisó literatura gris. El tiempo de publicación incluido fue de septiembre de 2009 a Enero de 2016 y el idioma que se consideró fue el inglés. Para la búsqueda realizada en las bases de datos se utilizó como palabra clave "NDM-1" y fueron incluidos artículos de acceso libre y de bases de datos privadas.
Se incluyeron artículos originales descriptivos (reportes de caso o vigilancia epidemiológica) que presentaran aislamientos de microorganismos de pacientes con la presencia de la enzima, se consideraron dos criterios de selección: que la identificación de la enzima se hubiera realizado por medio de reacción en cadena de la polimerasa (PCR) y que estuviera disponible el perfil de resistencia del microorganismo aislado con al menos 4 antibióticos evaluados. No se consideraron artículos en los que el aislamiento del microorganismo se diera del medio ambiente y/o animales, artículos en los que los dos criterios anteriormente nombrados no se cumplieran, o que el perfil de resistencia no estuviera completo y diferenciado para cada caso reportado.
Recolección y abstracción de datos
Para la recolección de los estudios, una vez seleccionadas las bases de datos a revisar y determinado el número de opciones mostradas al realizar la búsqueda con la palabra clave, se consideraron los títulos que expusieran palabras alusivas a la presentación de casos de NDM-1 como: brote, ocurrencia, aislamiento, aparición, detección, diseminación, reporte, adquisición, identificación, caso clínico y vigilancia o aquellos que contuvieran palabras referentes a la resistencia que la enzima genera como: productores de carbapenemasas, producción de betalactamasas, cepas multirresistentes o extremadamente resistentes. Una vez seleccionados los títulos se consideró para la selección de los artículos aquellos resúmenes que hicieran alusión a una descripción del caso, su aislamiento e identificación. En el caso de las cartas de investigación dado que estas no presentan resumen se revisó el documento completo. Para la selección final de los estudios se utilizaron los criterios de selección anteriormente nombrados.
Una vez completada la recolección de los estudios, para la extracción de datos se conformó una base de datos con información de los casos reportados donde se referenció cada uno de los antibióticos para los que el microorganismo presentaba resistencia, la identificación de la NDM-1 en el microorganismo por medio de PCR, además del año en el que se presentó el caso, el país, y los genes asociados a la resistencia del microorganismo diferentes del bla-NDM-1.
Determinación de los mecanismos asociados a la resistencia bacteriana y especies bacterianas reportados en la revisión
Con la base de datos conformada se determinó el mecanismo de resistencia asociado que cada gen generaba, realizando una búsqueda en diferentes artículos; de igual forma se aclara que un microorganismo puede presentar más de un gen asociado a resistencia y por lo tanto podrá presentar uno o más mecanismos de resistencia. Para establecer las especies bacterianas que producen la enzima NDM-1, se revisó cada uno de los casos y se identificó el microorganismo reportado en los 154 artículos.
Identificación del perfil de resistencia a los antibióticos de las bacterias identificadas en la revisión
A partir de cada uno de los casos reportados en los artículos, se realizó la datificación del perfil antibiótico realizado a los microorganismos identificados en la revisión; además, por medio de un modelo lineal generalizado multinivel con efectos mixtos se determinó la relación existente entre la resistencia generada respecto al género bacteriano y al mecanismo de resistencia asociado. Para esto se consideraron efectos aleatorios por país de procedencia del estudio y por cada estudio incluido en el análisis, dado que un artículo puede reportar múltiples casos.
Análisis estadístico
En el análisis de los datos presentados por los 617 casos se tuvo información sobre: fecha en la que se encontraron los casos, país de origen, microorganismo aislado, genes identificados en el microorganismo y perfil de resistencia. Se realizó un análisis descriptivo en el cual se conformaron tablas de contingencia con cada uno de los criterios a analizar en la revisión sistemática, para posteriormente, por medio de un modelo lineal generalizado de multinivel con efectos mixtos realizar el análisis. En este análisis de regresión se consideraron como variables independientes: géneros bacterianos, tomando como categoría base el género bacteriano Klebsiella spp. y mecanismos de resistencia asociados; y como variable dependiente, el número de antibióticos a los cuales el microorganismo fue resistente.
Resultados
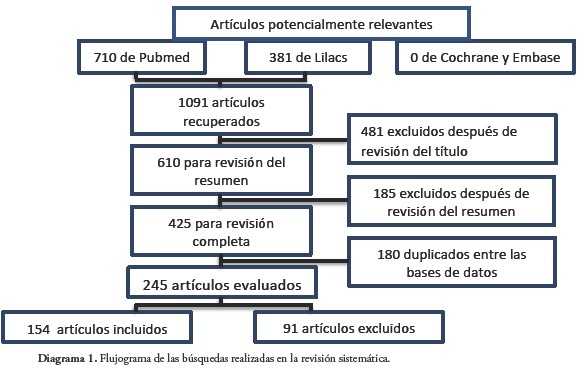
Fueron incluidos 154 artículos en el estudio en los que se reportan 617 casos de microorganismos aislados en los cuales se presenta la enzima NDM-U, Diagrama 1.
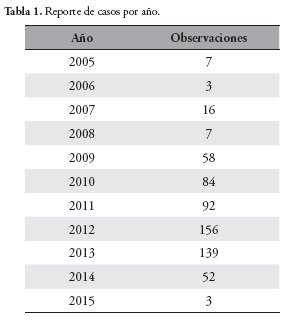
En los casos reportados, los aislamientos comienzan a presentarse desde el año 2005, aunque la publicación de los primeros casos no se dio inmediatamente, sino hasta años después al realizar estudios de vigilancia, como consecuencia del primer reporte realizado en 2008. Para el 2012 fueron encontrados 156 casos, siendo este el año que más presenta reportes, Tabla 1.
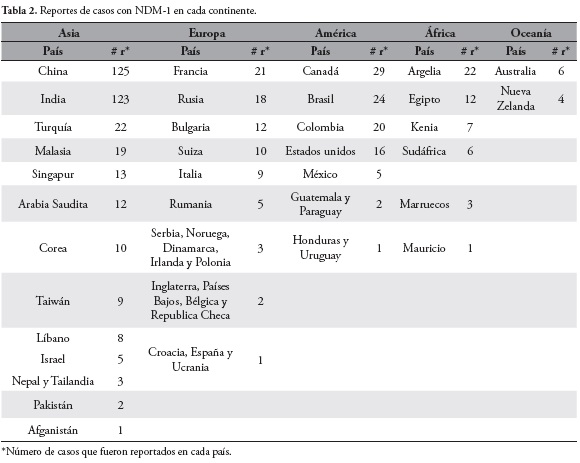
De los 617 casos, más de la mitad de los reportes corresponden al continente Asiático con 355 casos (57.5%), estos principalmente en China con 125 casos. En segundo lugar se encuentra Europa con 101 casos (16.4%), en este continente es donde hay mayor multiplicidad de países que han realizado reportes de microorganismos con la enzima, con un total de 19; el país con mayor número de reportes es Francia con 21 casos. En tercer lugar se encuentra América con 100 casos (16.2%), estos se presentaron principalmente en Canadá con 29 casos, en el caso particular de Colombia se presentan 20 casos reportados. El continente Africano se ubica en cuarto lugar con 51 casos reportados (8.3%), y sobresale Argelia con 22 casos. Finalmente, la menor proporción de casos reportados se encuentran en Oceanía con un total de 10 casos (1.6%), Tabla 2.
Géneros bacterianos que presentan reporte de la enzima NDM-1
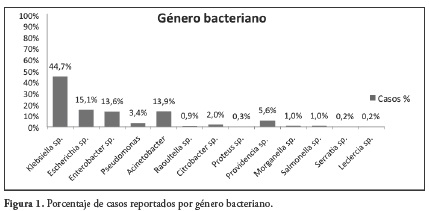
De los 617 casos reportados incluidos en el estudio el género bacteriano que fue aislado un mayor número de veces fue Klebsiella con 276 casos (44.7%), esto principalmente por K. pneumoniae que presento 271 casos (43.9%), y en menor media K. oxytoca; seguido por el género Escherichia en el cual todos los casos correspondieron a Escherichia coli con 93 casos (15.1%). Del género Enterobacter se encontraron reportes de las especies, E. cloacae siendo esta la reportada con mayor frecuencia con 70 casos (11.3%), además de E. aerogenes, E. ludwigii y E. hormaechei subsp. oharae.
En cuanto al género Acinetobacter se encontraron reportes de A. nosocomialis, A. calcoaceticus, A. soli, A. junii y A. pittii, reportándose en mayor porcentaje la especie A. baumannii que presento 64 casos (10.4%). En menor medida los aislamientos correspondieron a géneros como, Providencia donde la especie más reportada fue P. rettgeri con 31 casos (5.0%), además de P. stuartii; Routella para el cual los casos correspondieron a las especies R. planticola, R. ornithinolytica; para otros géneros bacterianos los casos identificados correspondieron a las especies Pseudomonas aeruginosa, Morganella morganii, Citrobacter freundii, Salmonella enterica, Proteus mirabilis, Serratia marcescens y Leclercia adecarboxylata, Figura 1.
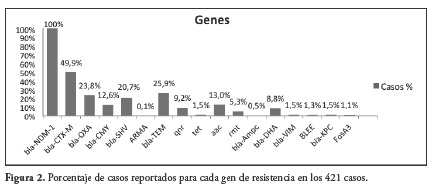
En las observaciones realizadas a los 617 casos fueron identificados 17 genes incluyendo el bla-NDM-1, el cual fue reportado en el 100% de los casos. Los genes asociado a NDM-1 que fueron encontrados con mayor frecuencia fueron bla-CTX-M en 308 casos (49.9%), seguido por bla-TEM en 160 casos (25.9%), bla-OXA se presentó en 147 casos (23.8%) y bla- SHV en 128 casos (20.7%), Figura 2.
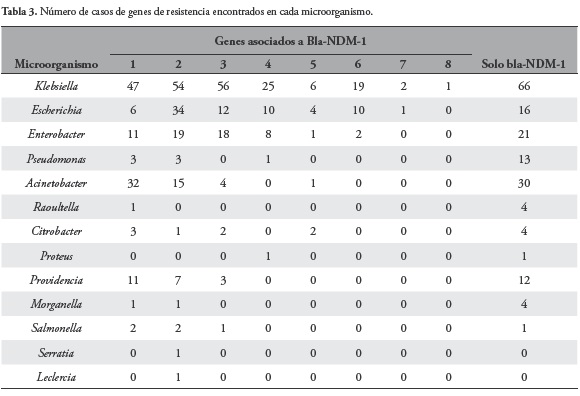
De las 617 bacterias identificadas en la revisión, 172 no presentaban un gen de resistencia diferente del bla-NDM-1, las 445 restantes presentaban de 1 a 8 genes de resistencia asociados al gen bla- NDM-1; en 138 de los casos (22.4%) el número de genes asociados correspondió a 2, siendo esta la asociación que se presentó con mayor frecuencia, Figura y Tabla 3.
Mecanismos asociados a la resistencia bacteriana
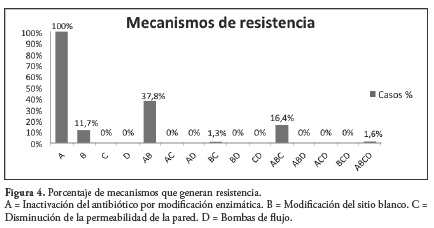
Con respecto a los mecanismos que generan resistencia asociados a los genes anteriormente nombrados, fueron identificados cuatro mecanismos, de los cuales la inactivación del antibiótico por modificación enzimática se presenta con mayor frecuencia en el 100% de los casos (dado a que este es generado por bla-NDM-1 (6), además de BLEE (7), armA (8), bla-CTX-M, tet (9), AmpC (10), DHA (11), rMTB (12), fosA3 (13), bla-VIM (14), bla-CMY (15), Rmt (16), seguido por modificación del sitio blanco con 425 casos (68.9%) (generado por los genes Bla-TEM, bla-SHV, bla-OXA, bla-CTX-M, qnr, tet (9), armA (8) y aac (17) en tercer lugar disminución de la permeabilidad de la pared con 119 casos (19.3%) (generado por los genes armA (8), aac (18) y AmpC (10) y finalmente bombas de flujo con 10 casos (1.6%) generado por Tet (9) y AmpC (10).
Como cada microorganismo puede presentar más de un gen de resistencia, así mismo puede presentar más de un mecanismo de resistencia al mismo tiempo, es así como la mayor presencia se dio entre los mecanismos inactivación del antibiótico por modificación enzimática y modificación del sitio blanco los cuales se presentaron de manera simultánea en 233 de los casos (37.8%), es de resaltar que los cuatro mecanismos se presentaron de manera simultánea en 10 casos (1.6%), Figura 4.
Perfil de resistencia en bacterias con presencia de la enzima NDM-1
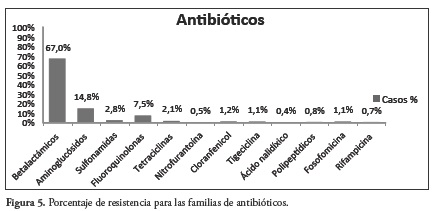
Dependiendo de la bacteria y el estado particular del paciente, en cada estudio fueron evaluados diferentes antibióticos. El total de antibióticos probados fue de 50 que corresponden a las familias: betalactámicos, aminoglucósidos, sulfonamidas, fluoroquinolonas, tetraciclinas, nitrofuranos, fenicoles glicinas, quino-lonas, polipeptídicos, fosfonatos y rifamicinas; de acuerdo a la revisión, se observa que en promedio el número de antibióticos testeado para cada bacteria fue de 13, y al sumar individualmente cada uno de los antibióticos utilizados en las pruebas de sensibilidad y resistencia realizadas a las 617 bacterias identificadas en la revisión dio un total de 6530 antibióticos.
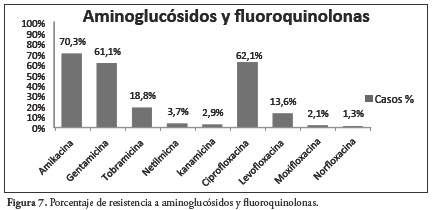
Se observa que el uso más frecuente en las pruebas está relacionado con los betalactámicos con 4374 antibióticos (67.0%), seguido por los amino-glucósidos con 968 antibióticos (14.8%) y las fluoroquinolonas con 488 antibióticos (7.5%). La frecuencia en los reportes fue menor para otras familias y antibióticos probados como: sulfonamidas (trimetoprim y trimetoprim-sulfametoxazol), tetraciclinas (tetraciclina, doxiciclina y minociclina), nitrofurantoína, cloranfenicol, tigeciclina, ácido nalidíxico, polipeptídicos (colistina y polimixina B), fosfomicina y rifampicina, Figura 5.
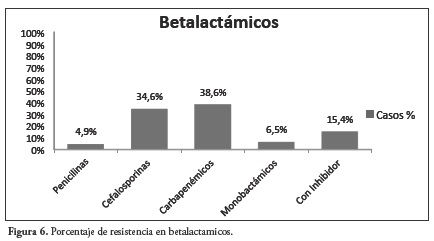
De los 4374 betalactámicos testeados, los más utilizados y que presentaron más resistencia corresponden a los carbapenémicos con 1687 antibióticos (38.6%), de estos particularmente imipenem, meropenem y ertapenem fueron resistentes en 556, 504 y 426 de los casos respectivamente, y en menor medida los carbapenémicos doripenem, biapenem y panipenem.
En segundo lugar se encuentran las cefalosporinas con 1512 antibióticos testeados (34.6%), de estas el mayor número de casos resistentes correspondió a ceftazidime con 313 casos. En tercer lugar la resistencia fue a betalactámicos con inhibidor con 675 antibióticos (15.4%); piperacilina / tazobactam mostró mayor resistencia con 349 casos, Figuras 6 y 7.
Situación de Colombia en términos de reportes de la enzima NDM-1
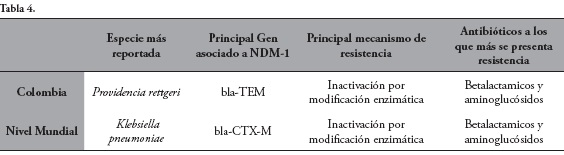
Al comparar los reportes obtenidos de Colombia con los obtenidos a nivel mundial, se observa cómo las diferencias son que el microorganismo que más se reporta con presencia de la enzima en Colombia es Providencia rettgeri y el gen de resistencia que más se asocia a bla-NDM-1 es la betalactamasa bla-TEM, Tabla 4.
Comportamiento de la resistencia generada por la enzima respecto a las bacterias y los mecanismos asociados
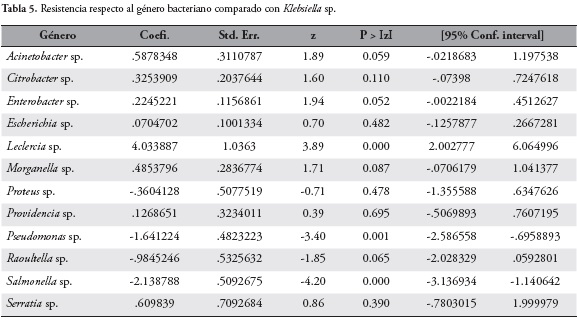
Respecto a la evaluación del comportamiento que está presentando la resistencia generada por la enzima, al realizar la regresión lineal de acuerdo al género bacteriano, los datos fueron evaluados frente a la resistencia que presenta el género Klebsiella dado que este género en la revisión es el que mayor número de casos presenta. De acuerdo con los resultados mostrados en la Tabla 5, el género y Leclercia (P-valor < 0.001) sobresale dado que presenta una resistencia mayor a Klebsiella en 4.03 antibióticos (aunque este no puede generar inferencia debido al bajo número de reportes); otros géneros bacterianos presentan una resistencia menor respecto a Klebsiella como: Pseudomonas (P-valor < 0.001) menor en -1.6 y Salmonella (P-valor < 0.001) menor en -2.1, Tabla 5.
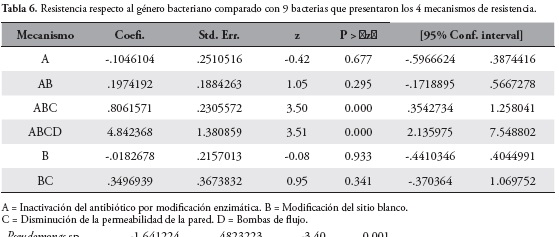
La evaluación del comportamiento de la resistencia respecto a los mecanismos que presentaba cada uno de los casos fue calculada en relación a las bacterias 172 que presentaron únicamente el mecanismo de resistencia inactivación del antibiótico por modificación enzimática como se observa en la tabla 5, esto dado a que este lo presentan los aislamientos al ser generado por la enzima NDM-1, Tabla 6.
Discusión
El fenómeno de resistencia bacteriana constituye una preocupación permanente en salud pública dado que genera interferencia y disminución en las opciones terapéuticas para el tratamiento de enfermedades infecciosas, causando así un aumento considerable en porcentajes de morbilidad y mortalidad, lo cual se ve reflejado en el informe emitido por la OMS y la OPS en marzo de 2014 sobre la situación actual en las Américas con respecto a las carbapenemasas tipo New Delhi metalobetalactamasas (NDM). En éste se hace énfasis sobre la rápida diseminación de la producción de la enzima entre los microorganismos constituyendo así una grave amenaza para la salud y vida de la humanidad (19). Es por esto que la OMS genera una resolución con la cual se busca la prevención de la infección, la sostenibilidad y la optimización del uso de los medicamentos antimicrobianos impulsando normas mundiales respecto a su administración, dado que la resistencia a los antimicrobianos es un problema en la eficacia de los tratamientos y al ser una amenaza para la salud pública se hace necesaria una acción global (20).
En la presente revisión sistemática se realizó un compendio de información acerca de la enzima NDM-1 a nivel mundial. Fueron analizados 421 casos reportados en 111 artículos; en los cuales se evidencia cómo el año de reporte no tiene relevancia con respecto al número y frecuencia de reportes, pese a que se observa un aumento de estos en el año 2012 (3,21). Con relación a los países se observa cómo la mayor frecuencia de los casos se centra en el continente asiático, esto se relaciona con el hecho de que el primer reporte de esta enzima fue en un paciente que había sido hospitalizado en Nueva Delhi, capital de India (22, 23), país en el cual se presenta el mayor número de reportes. De igual manera en India fue identificado el primer microorganismo con la presencia del gen-NDM-1 el cual corresponde a Acinetobacter baumannii (24), además de esto hay que tener en cuenta que este país tiene gran movimiento de personas hacia otras partes del mundo por lo cual facilita la diseminación del gen. De otro lado India posee condiciones especiales como sobrepoblación, pobreza extrema, desigualdad social, limitaciones del sistema sanitario y escasas condiciones higiénicas, propicias para una rápida propagación de infecciones y el contagio de enfermedades (25).
En el caso particular de Colombia, se observa cómo presenta un porcentaje de reportes menor en comparación con otros países del mundo (26-27) y de América como Canadá (país con el mayor número de reportes en América). Esto podría deberse a que este país fue uno de los primeros junto con Estados Unidos en los cuales fueron encontrados los primeros hallazgos de esta enzima en el continente en el año 2010 (28-29). Aunque cabe destacar que en América, Brasil está aportando considerablemente en el número de casos, después del primer aislamiento de Providencia rettgeri con presencia de la enzima en febrero de 2013 (30), así como el reporte de la enzima en otros géneros bacterianos como Morganella, Enterobacter y Acinetobacter (30-33).
Por otro lado, hay que tener en cuenta que los casos incluidos en esta revisión no son los únicos que se han presentado en Colombia. Se conoce que el primer estudio reportado en el país fue en el año 2012 en Bogotá de un brote causado por Klebsiella pneumoniae (34), y a partir de este momento el Instituto Nacional de Salud (INS), como ente regulador fomentó por medio de boletines la necesidad por parte de los laboratorios e instituciones prestadoras de servicio de salud de reforzar acciones destinadas a la prevención, vigilancia y control de infecciones por enterobacterias resistentes a carbapenémicos (enzimas de tipo KPC y NDM), haciendo mayor énfasis en aquellos lineamientos para la vigilancia de las metalobetalactamasas tipo NDM (35).
Es así como actualmente por medio de reportes sobre la vigilancia por el laboratorio de resistencia bacteriana de infecciones asociadas a la atención en salud (IAAS) en Colombia después del brote presentado en Bogotá, se han realizado nuevos aislamientos en los departamentos de Santander, Antioquia, Cundinamarca, Valle, Norte de Santander y Nariño, los cuales son remitidos al grupo de microbiología del INS para su confirmación, identificando diferentes especies bacterianas con la enzima como Providencia rettgeri, Morganella mor-ganii y Acinetobacter baumannii, lo que demuestra la rápida diseminación que este gen presenta (2, 36), con el fin de fortalecer la vigilancia y las estrategias para el control y prevención frente a la diseminación de esta resistencia (2).
Con respecto al reporte de las bacterias que presentan mayor diseminación del gen, se encuentran Klebsiella pneumoniae y Escherichia coli. Esto es consecuente con el hecho de que estas dos especies presentan una alta diseminación intrahospitalaria, además de poderse adquirir también en la comunidad (37); particularmente por encontrarse en aguas (38), lo que les permite diseminarse a diferentes ambientes. Tal es el caso de Klebsiella pneumoniae con la enzima NDM-1 que se encontró en el río Nguu Kim en 2011 en Vietnam (39), para el caso de E. coli se presentó reporte de un aislamiento de medio ambiente con presencia de la enzima en una casa (40), pero esta rápida diseminación no solo se ve evidenciada en casos humanos sino también en anímales de compañía. Este es el caso de 5 aislamientos encontrados de E. coli con presencia de la enzima NDM-1 en Estados Unidos en 4 caninos y un felino, lo cual aumenta la preocupación a nivel de salud pública, dado que los humanos además de poder contraer bacterias con esta enzima del medio ambiente también podrían adquirirlas de sus mascotas (41).
De igual manera, estos dos microorganismos además de ser los de mayor diseminación, también fueron los que más número de genes de resistencia asociados a NDM-1 presentaron (42-44). Por otro lado, han sido aislados de manera simultánea del mismo paciente (28, 45-46) (aunque hay que tener en cuenta que no solo estas dos bacterias con presencia de la enzima NDM-1 se están generando de manera simultánea, se han reportado otros casos como Raoultella planticola con Escherichia coli (47) y Klebsiella pneumoniae con Salmonella (48) esto sumado a su fácil y rápida diseminación, también facilita la adquisición de nuevos genes de resistencia haciendo muy difícil el tratamiento.
Otro género bacteriano que se asocia a enfermedades nosocomiales y que se puede transmitir en el ambiente es Salmonella enterica (un reporte en esta revisión) la cual fue encontrada en un ave silvestre "milano negro" en Alemania (49). Cabe destacar que el género Acinetobacter es en el que mayor número de especies se presenta la enzima, lo que podría explicarse por el hecho de que esta fue la primera bacteria en la cual fue reportada NDM-1, en muestras que fueron recogidos en 2005 de un hospital en Tamil Nadu, India, estos casos llevan a la hipótesis de que bla-NDM-1 podría haber sido difundido desde Acinetobacter a enterobacterias en el sur de Asia. (24). Así mismo, este género se hace importante al ser encontrado con presencia de la enzima en animales destinados al consumo humano como son los casos de Acinetobacter baumannii en cerdos (50) y Acinetobacter iwoffii en pollos (51), lo cual genera implicaciones a nivel de seguridad alimentaria.
En relación a los genes de resistencia encontrados con mayor frecuencia en los estudios identificados en la revisión sistemática diferentes de bla-NDM-1 fueron bla-CTX-M (44,52) bla-TEM (27-53) y bla-OXA (54,55) lo que muestra un aumento en la expresión de betalactamasas de este tipo. Así mismo, esta observación guarda estrecha relación con la idea de que estos genes codifican para mecanismos que generan resistencia por medio de inactivación del antibiótico dada por modificación enzimática y modificación del sitio blanco, mecanismos que en la revisión presentan una mayor frecuencia cuando están presentes simultáneamente en una bacteria. Lo anterior podría ser el resultado de la alta frecuencia en los reportes de las especies bacterianas Klebsiella pneumoniae y Escherichia coli, en las cuales se han encontrado betalactamasas tipo CTM-X y OXA (56) con importancia epidemiológica debido a su dispersión intra y extrahospitalaria.
La notable variación encontrada en el número de genes asociados en cada uno de los casos reportados por género bacteriano, demuestra la relación existente entre el mecanismo de transferencia genética horizontal por medio de plásmidos conjugativos y transposones de las bacterias reportadas y la rápida diseminación de la enzima NDM-1 (57). Un aspecto de gran interés es que la multiresistencia bacteriana que se está presentando es debida a que los genes que codifican para diferentes mecanismos de resistencia pueden generar resistencia cruzada; es decir que un gen produce resistencia a antibióticos que tienen mecanismos de acción similares o de la misma clase, incluso a compuestos de familia diferentes (34), lo cual se puede evidenciar con uno de los genes encontrados con mayor frecuencia: bla-CTX-M, que incluye 223 casos (53.0%), pues este codifica para enzimas tipo betalactamasas con actividad hidrolítica frente a la cefotaxima, ceftazidime, ceftriaxona y cefepime, antibióticos pertenecientes a cefalosporinas de tercera generación (35).
En la revisión se observa claramente que las bacterias que poseen la enzima NDM-1, presentan resistencia a los betalactámicos incluidos los carbapenémicos (imipenem, meropenem, ertape-nem, doripenem y biapenem), los cuales corresponden a los últimos recursos terapéuticos para el tratamiento de infecciones. Esto genera que como opción terapéutica para estos microorganismos se consideren antibióticos como colistina y tigecicli-na, pero hay casos en los cuales estos no se pueden implementar como es el caso de Proteus mirabilis, los cuales son intrínsecamente resistentes a estos antibióticos (58). Además de esto, se presentan casos en los cuales las bacterias con presencia de la enzima NDM-1 también presentan resistencia a estos (52, 59, 60). Adicionalmente en el caso particular de la colistina, se conoce que presenta efectos tóxicos renales en una tercera parte de las personas que la reciben en su tratamiento.
La resistencia a los aminoglucósidos es considerable ya que un alto número de casos presenta resistencia a amikacina o gentamicina y esto se encuentra relacionado con el hecho de que hay una disminución intracelular del antibiótico, dado a que entre los mecanismo de resistencia, presentan disminución de la permeabilidad de la pared generando modificación en la difusión de los antibióticos y que los aminoglucósidos no sean bien transportados a través de la membrana bacteriana (61, 62). En cuanto a las fluoroquinolonas ocurre algo similar dado que la dificultad del paso del antibiótico a través de la pared es uno de los principales mecanismos que genera resistencia a estas al disminuir la penetración intracelular (63-64). Además de esto la resistencia a estas dos familias de antibióticos se debe a la presencia de genes de resistencia como acc el cual genera la acetilación de los aminoglucósidos (39). Para el caso particular de las fluoroquinolonas algunas variantes de acc (como aac- 6-Ibcr) son capaces de acetilar no solo aminoglucósidos si no también fluoroquinolonas (9).
De igual manera hay que tener presente que los aislamientos de Escherichia coli y Klebsiella pneumoniae que se reportan actualmente son resistentes a por lo menos uno de los antimicrobianos sujetos a vigilancia, y muchas de estas bacterias identificadas en el estudio presentan resistencia combinada a cefalosporinas de tercera generación, aminoglucósidos y fluoroquinolonas, lo que confirma el generalizado aumento en los niveles de resistencias a dichos antimicrobianos en los últimos años (65), además de que actualmente con la aparición de NDM-1 se le suma la resistencia a carbapenémicos.
Limitaciones del estudio
Respecto a las limitaciones se considera que a pesar de la amplia búsqueda y recolección de información se puede presentar sesgo, dado que en los estudios incluidos no siempre se hace uso del mismo tipo y número de antibióticos en cada caso reportado; así mismo existió variación de los métodos de identificación de resistencia empleados. Los aspectos anteriormente mencionados se alteraban por factores como clínica del paciente, nivel de atención del hospital en el cual se realizó el estudio, tipos de protocolos que se manejan en cada país, ya que poseen diferentes sistemas de salud y vigilancia y generan que los datos no sean precisos en su totalidad dado que de acuerdo a la variación del estudio los reportes no son uniformes. Además de esta, al no ser considerada la literatura gris ni publicaciones en otros idiomas se reduce el número de casos que se presentan.
Conclusiones
La revisión pone de manifiesto que cuando se presenta la enzima NDM-1 la posibilidad que haya genes asociados para producción de resistencias es alta, lo que genera que se presenten uno o varios mecanismos de resistencia que evitan la acción del antibiótico y por lo tanto se hace difícil implementar un tratamiento terapéutico efectivo y oportuno. Además, el perfil de resistencia a antibióticos in vitro encontrado en las bacterias identificadas en esta revisión sistemática, se centra principalmente en las familias de antibióticos de betalactámicos, aminoglucósidos y fluoroquinolo-nas y esta resistencia está mediada esencialmente por los mecanismos de resistencia e inactivación del antibiótico por modificación enzimática y modificación del sitio blanco.
Respecto a los principales genes asociados a bla-NDM1 y por los cuales se está generando el perfil y los mecanismos de resistencia son de tipo beta-lactamasas como bla-CTX-M y bla-TEM, aunque no hay que descartar que se están presentando de manera simultánea con otros genes como aac, el cual genera resistencia a aminoglucósidos. En relación a los principales antibióticos a los que están presentado resistencia las bacterias productoras de NDM-1, correspondieron a los carbapenémicos imipenem, meropenem, ertapenem seguido de los aminoglucósidos gentamicina y amikacina y la fluoroquinolona ciprofloxacina; además de esto destaca la resistencia a las cefalosporinas de tercera y cuarta generación ceftazidime y cefepime.
Para el caso de las especies bacterianas se obtuvo que las que presentaron mayor prevalencia en la producción de la enzima NDM-1 a nivel mundial fueron Klebsiella pneumoniae siendo esta la que presenta mayor difusión del gen, seguida de Escherichia coli, Acinetobacter baumannii y Enterobacter cloacae, por lo que se sugiere tener especial cuidado y atención cuando se encuentre en la práctica clínica alguno de estos microorganismos con sensibilidad disminuida a los carbapenémicos.
En el caso particular de Colombia, al compararlo con los reportes de otros países se observa cómo el perfil de resistencia de las bacterias aisladas y los genes de resistencia asociados son similares; sin embargo la mayoría de casos se presentan en Providencia rettgeri.
Referencias
1. Roca I, Akova M, Baquero F, Carlet J, Cavaleri M, Coenen S, et al. The global threat of antimicrobial resistance: Science for intervention. New Microbes New Infect. Elsevier Ltd; 2015;6(April 2015):22-9. [ Links ]
2. Grundmann H. Towards a global antibiotic resistance surveillance system: a primer for a roadmap. Ups J Med Sci. 2014;119(2):87-95. [ Links ]
3. Organización Mundial de la Salud. Antimicrobial Resistance Global Report on Surveillance 2014. [Internet] Disponible en: http://www.who.int/drugresistance/documents/AMR_report_Web_slide_set.pdf?ua=1. [ Links ]
4. Rasheed JK, Kitchel B, Zhu W, Anderson KF, Clark NC, Ferraro MJ, et al. New Delhi Metallo-β-Lactamase-producing Enterobacteriaceae, United States. Emerg Infect Dis. 2013;19(6):870-8. [ Links ]
5. Egger M, Smith GD AD. Systematic reviews in health care. Meta-analysis in context. BMJ Books. 2001. [ Links ]
6. Nordmann P, Poirel L, Carrêr A, Toleman M a., Walsh TR. How to detect NDM-1 producers. J Clin Microbiol. 2011;49(2):718-21. [ Links ]
7. Arce-gil Z, Flores-clavo R. Deteccion del gen CTX-M en cepas de Escherichia coli productoras de betalactamasas de espectro extendido procedentes del hospital regional de lambayeque; Chiclayo-Perú: Noviembre 2012-Julio 2013. 2013;6(4):13-6. [ Links ]
8. González-Zorn B, Catalan A, Domínguez L, Teshager T, Porrero C, Moreno MA. Genetic basis for dissemination of armA. J Antimicrob Chemother. 2005;56(3):583-5. [ Links ]
9. Mosquito S, Ruiz J, Ochoa TJ. MECANISMOS MOLECULARES DE RESISTENCIA ANTIBIÓTICA EN Escherichia coli ASOCIADAS A DIARREA. Rev Peru Med Exp Salud Publica. 2011;28(4):9-11. [ Links ]
10. Jacoby G a. AmpC β-Lactamases. Clin Microbiol Rev. 2009;22(1):161-82. [ Links ]
11. Verdet C, Benzerara Y, Gautier V, Adam O, Ould-Hocine Z, Arlet G. Emergence of DHA-1-producing Klebsiella spp. in the Parisian region: Genetic organization of the ampC and ampR genes originating from Morganella morganii. Antimicrob Agents Chemother. 2006;50(2):607-17. [ Links ]
12. Yao Q, Zeng Z, Hou J, Deng Y, He L, Tian W, et al. Dissemination of the rmtB gene carried on IncF and IncN plasmids among Enterobacteriaceae in a pig farm and its environment. J Antimicrob Chemother. 2011;66(11):2475-9. [ Links ]
13. Hou J, Yang X, Zeng Z, Lv L, Yang T, Lin D, et al. Detection of the plasmid-encoded fosfomycin resistance gene fosA3 in Escherichia coli of food-animal origin. J Antimicrob Chemother. 2013;68(4):766-70. [ Links ]
14. Pournaras S, Tsakris A, Maniati M, Tzouvelekis LS, Maniatis AN. Novel Variant (bla VIM-4 ) of the Metallo- β-Lactamase Gene bla VIM-1 in a Clinical Strain of Pseudomonas aeruginosa. Society. 2002;46(12):4026-8. [ Links ]
15. Kang M, Besser TE, Call DR. Variability in the Region Downstream of the bla CMY-2 β-Lactamase Gene in Escherichia coli and Salmonella enterica Plasmids. 2006;50(4):1590-3. [ Links ]
16. Ma L, Lin CJ, Chen JH, Fung CP, Chang FY, Lai YK, et al. Widespread dissemination of aminoglycoside resistance genes armA and rmtB in Klebsiella pneumoniae isolates in Taiwan producing CTX-M-type extended-spectrum B-lactamases. Antimicrob Agents Chemother. 2009;53(1):104-11. [ Links ]
17. Aminoglucósidos RA, Cepas EN, M LM, B GP, Ruiz - Parra AI, C YC, et al. DETERMINACIÓN DEL GEN aac(6')-aph(2") asociado con resistencia a aminoglucósidos en cepas de staphylococcus coagulasa negativa en una unidad neonatal en Bogotá. 2009;55:326-33. [ Links ]
18. Lidia Yolanda Fuchs, M.C. PD. LC. Mecanismos moleculares de la resistencia bacteriana. Salud Publica Mex. 1994;36(4):428-38. [ Links ]
19. Organizacion Panamericana de la salud organizacion mundial de la salud. Actualización Epidemiológica, carbapenemasas tipo New Delhi metalobetalactamasas (NDM). 2014: 1-3. [ Links ]
20. Shallcross LJ, Davies SC. The World Health Assembly resolution on antimicrobial resistance. J Antimicrob Chemother. 2014;69(11):2883-5. [ Links ]
21. Qin S, Fu Y, Zhang Q, Qi H, Wen JG, Xu H, et al. High incidence and endemic spread of NDM-1 positive Enterobacteriaceae in Henan province, China. Antimicrob Agents Chemother. 2014;58(8):4275-82. [ Links ]
22. Ageevets V a., Partina I V., Lisitsyna ES, Ilina EN, Lobzin Y V., Shlyapnikov S a., et al. Emergence of carbapenemase-producing Gram-negative bacteria in Saint Petersburg, Russia. Int J Antimicrob Agents. Elsevier B.V. 2014;44(2):152-5. [ Links ]
23. Yong D, Toleman M a., Giske CG, Cho HS, Sundman K, Lee K, et al. Characterization of a new metallo-??-lactamase gene, bla NDM-1, and a novel erythromycin esterase gene carried on a unique genetic structure in Klebsiella pneumoniae sequence type 14 from India. Antimicrob Agents Chemother. 2009;53(12):5046-54. [ Links ]
24. Jones LS, Toleman M a., Weeks JL, Howe R a., Walsh TR, Kumarasamy KK. Plasmid carriage of blaNDM-1in clinical Acinetobacter baumannii isolates from India. Antimicrob Agents Chemother. 2014;58(7):4211-3. [ Links ]
25. Collantes F. India: colonialismo, pobreza y estrategias de desarrollo. 1-34 [Internet]. Disponible en http://www.unizar.es/departamentos/estructura_economica/personal/collantf/documents/India-Texto.pdf. [ Links ]
26. Gaibani P, Ambretti S, Berlingeri a., Cordovana M, Farruggia P, Panico M, et al. Outbreak of NDM-1-producing Enterobacteriaceae in northern Italy, July to August 2011. Euro Surveill Bull Eur sur les Mal Transm = Eur Commun Dis Bull. 2011;16(47):2002. [ Links ]
27. Teo J, Ngan G, Balm M, Jureen R, Krishnan P, Lin R. Molecular characterization of NDM-1 producing Enterobacteriaceae isolates in Singapore hospitals. West Pacific Surveill Response. 2012;3(1):1-1. [ Links ]
28. Mulvey MR, Grant JM, Plewes K, Roscoe D, Boyd D a. New Delhi metallo-β-lactamase in Klebsiella pneumoniae and Escherichia coli, Canada. Emerg Infect Dis. 2011;17(1):103-6. [ Links ]
29. Mochon a. B, Garner OB, Hindler J a., Krogstad P, Ward KW Lewinski M a., et al. New Delhi metallo-β-lactamase (NDM-1)-producing klebsiella pneumoniae: Case report and laboratory detection strategies. J Clin Microbiol. 2011;49(4):1667-70. [ Links ]
30. Carvalho-Assef APDA, Pereira PS, Albano RM, Berião GC, Chagas TPG, Timm LN, et al. Isolation of NDM-producing providencia rettgeri in brazil. J Antimicrob Chemother. 2013;68(12):2956-7. [ Links ]
31. Rozales FP, Ribeiro VB, Magagnin CM, Pagano M, Lutz L, Falci DR, et al. Emergence of NDM-1-producing Enterobacteriaceae in Porto Alegre, Brazil. Int J Infect Dis. 2014;25:79-81. [ Links ]
32. Carvalho-Assef APDA, Pereira PS, Albano RM, Berião GC, Tavares CP, Chagas TPG, et al. Detection of NDM-1-, CTX-M-15-, and qnrB4-producing enterobacter hormaechei isolates in Brazil. Antimicrob Agents Chemother. 2014;58(4):2475-6. [ Links ]
33. Barguigua A, Zerouali K, Katfy K, El Otmani F, Timinouni M, Elmdaghri N. Occurrence of OXA-48 and NDM- 1 carbapenemase-producing Klebsiella pneumoniae in a Moroccan university hospital in Casablanca, Morocco. Infect Genet Evol. 2015;31:142-8. 34. [ Links ]
34. Escobar Pérez JA, Olarte Escobar NM, Castro-Cardozo B, Valderrama Márquez IA, Garzón Aguilar MI, De La Barrera LM, et al. Outbreak of NDM-1-producing Klebsiella pneumoniae in a neonatal unit in Colombia. Antimicrob Agents Chemother. 2013;57(4):1957-60. [ Links ]
35. Instituto Nacional de la Salud. Circular 1000 - 0057. [Internet]. 2012. Disponible en: http://www.ins.gov.co/normatividad/Normatividad/circular_0057_de_2012.PDF. [ Links ]
36. IQEN. Circulación de Carbapenemasas tipo New Delhi Metalo-β-lactamasa (NDM), Colombia, 2011 a 2013. 2013;18(11):111-20. Disponible en: http://www.ins.gov.co:81/iqen/IQUEN/IQEN_vol_18_2013_num_11.pdf. [ Links ]
37. ANTIMICROBIAL RESISTANCE Global Report onSurveillance 2014.Organizacion Mundial de la Salud. 2014. [ Links ]
38. Organización Mundial de la Salud. Hojas de información microbiológica. Guías para la Calid del agua potable [Internet]. 2008;191-241.Disponible en: http://www.who.int/water_sanitation_health/dwq/gdwq3_es_11.pdf. [ Links ]
39. Isozumi R, Yoshimatsu K, Yamashiro T, Hasebe F, Nguyen BM, Ngo TC, et al. BlaNDM-1-positive Klebsiella pneumoniae from environment, Vietnam. Emerg Infect Dis. 2012;18(8):1383-5. [ Links ]
40. Kutumbaka KK, Han S, Mategko J, Nadala C, Buser GL, Cassidy MP, et al. Draft Genome Sequence of bla NDM-1 -Positive Escherichia coli O25b- ST131 Clone Isolated from an Environmental Sample. ASM. 2014;2(3):14-5. [ Links ]
41. Shaheen BW, Nayak R, Boothe DM. Emergence of a New Delhi metallo-β-Lactamase (NDM-1)-encoding gene in clinical Escherichia coli isolates recovered from companion animals in the United States. Antimicrob Agents Chemother. 2013;57(6):2902-3. [ Links ]
42. Barguigua A, El otmani F, Lakbakbi el yaagoubi F, Talmi M, Zerouali K, Timinouni M. First report of a Klebsiella pneumoniae strain coproducing NDM-1, VIM-1 and OXA-48 carbapenemases isolated in Morocco. Apmis. 2013;121(7):675-7. [ Links ]
43. Arpin C, Noury P, Boraud D, Coulange L, Manetti A, André C, et al. NDM-1-producing Klebsiella pneumoniae resistant to colistin in a French community patient without history of foreign travel. Antimicrob Agents Chemother. 2012;56(6):3432-4. [ Links ]
44. Castanheira M, Deshpande LM, Farrell SE, Shetye S, Shah N, Jones RN. Update on the prevalence and genetic characterization of NDM-1-producing Enterobacteriaceae in Indian hospitals during 2010. Diagn Microbiol Infect Dis. 2013;75(2):210-3. [ Links ]
45. Poirel L, Schrenzel J, Cherkaoui A, Bernabeu S, Renzi G, Nordmann P. Molecular analysis of NDM-1-producing enterobacterial isolates from Geneva, Switzerland. J Antimicrob Chemother. 2011;66(8):1730-3. [ Links ]
46. Stone NRH, Woodford N, Livermore DM, Howard J, Pike R, Mushtaq S, et al. Breakthrough bacteraemia due to tigecycline-resistant escherichia coli with New Delhi metallo-β-lactamase (NDM)-1 successfully treated with colistin in a patient with calciphylaxis. J Antimicrob Chemother. 2011;66(11):2677-8. [ Links ]
47. Li J, Lan R, Xiong Y, Ye C, Yuan M, Liu X, et al. Sequential Isolation in a Patient of Raoultella planticola and Escherichia coli Bearing a Novel ISCR1 Element Carrying blaNDM-1. PLoS One. 2014;9(3):e89893. [ Links ]
48. Savard P, Gopinath R, Zhu W Kitchel B, Rasheed JK, Tekle T, et al. First NDM-positive Salmonella sp. strain identified in the United States. Antimicrob Agents Chemother. 2011;55(12):5957-8. [ Links ]
49. Fischer J, Schmoger S, Jahn S, Helmuth R, Guerra B. NDM-1 carbapenemase-producing Salmonella enterica subsp. enterica serovar Corvallis isolated from a wild bird in Germany. J Antimicrob Chemother. 2013;68(12):2954-6. [ Links ]
50. Zhang WJ, Lu Z, Schwarz S, Zhang RM, Wang XM, Si W, et al. Complete sequence of the blaNDM-1-carrying plasmid pNDM-AB from acinetobacter baumannii of food animal origin. J Antimicrob Chemother. 2013;68(7):1681-2. [ Links ]
51. Wang Y, Wu C, Zhang Q, QiJ, Liu H, Wang Y, et al. Identification of New Delhi metallo-β-lactamase 1 in acinetobacter Lwoffii of food animal origin. PLoS One. 2012;7(5):3-8. [ Links ]
52. Lascols C, Hackel M, Marshall SH, Hujer AM, Bouchillon S, Badal R, et al. Increasing prevalence and dissemination of NDM-1 metallo-p-lactamase in India: Data from the SMART study (2009). J Antimicrob Chemother. 2011;66(9):1992-7. [ Links ]
53. Kumarasamy K, Kalyanasundaram A. Emergence of klebsiella pneumoniae isolate co-producing NDM-1 with KPC-2 from India. J Antimicrob Chemother. 2012;67(1):243-4. [ Links ]
54. Bonnin R. A., Cuzon G, Poirel L, Nordmann P. Multidrug-resistant Acinetobacter baumannii clone, France. Emerg Infect Dis. 2013;19(5):822-3. [ Links ]
55. Bakour S, Touati A, Bachiri T, Sahli F, Tiouit D, Naim M, et al. First report of 16S rRNA methylase ArmA-producing Acinetobacter baumannii and rapid spread of metallo-p-lactamase NDM-1 in Algerian hospitals. J Infect Chemother. 2014;20(11):696-701. [ Links ]
56. García CS, de la Gándara MP, García FJC. Betalactamasas de espectro extendido en enterobacterias distintas de Escherichia coli y Klebsiella. Enferm Infecc Microbiol Clin. Elsevier; 2010;28(SUPPL. 1):12-8. [ Links ]
57. Qu H, Wang X, Ni Y, Liu J, Tan R, Huang J, et al. NDM- 1-producing Enterobacteriaceae in a teaching hospital in Shanghai, China: IncX3-type plasmids may contribute to the dissemination of blaNDM-1. Int J Infect Dis. International Society for Infectious Diseases. 2015;34:8-13. [ Links ]
58. Williamson D a., Sidjabat HE, Freeman JT, Roberts S a., Silvey A, Woodhouse R, et al. Identification and molecular characterisation of New Delhi metallo-β-lactamase-1 (NDM-1)- and NDM-6-producing Enterobacteriaceae from New Zealand hospitals. Int J Antimicrob Agents. Elsevier B.V. 2012;39(6):529-33. [ Links ]
59. Gefen-Halevi S, Hindiyeh MY, Ben-David D, Smollan G, Gal-Mor O, Azar R, et al. Isolation of genetically unrelated blandm-1-positive providencia rettgeri strains in israel. J Clin Microbiol. 2013;51(5):1642-3. [ Links ]
60. Khajuria A, Praharaj AK, Kumar M, Grover N. Emergence of NDM - 1 in the clinical isolates of Pseudomonas aeruginosa in India. J Clin Diagnostic Res. 2013;7(7):1328-31. [ Links ]
61. Santiago MA, Esquirol JL, Fernández RN, Maletá PD. Aminoglucósidos: Mirada actual desde su historia. Rev Cubana Pediatr. 2007;79(2). [ Links ]
62. Grünbaum F. Resistencia a aminoglucósidos en ENTERO-BACTERIACEAE. Universidad Autonoma de Barcelona. [Internet]. 2011. Disponible en: http://www.tdx.cat/bitstream/handle/10803/42291/fg1de1.63. [ Links ]
63. Leyva S, Leyva E. Fluoroquinolonas. Mecanismos de acción y resistencia, estructura, síntesis y reacciones fisicoquímicas importantes para propiedades medicinales. Boletín la Soc Química México. 2008;2(1):1-13. [ Links ]
64. Rodríguez-Martínez JM. Mecanismos de resistencia a quinolonas mediada por plásmidos. Enferm Infecc Microbiol Clin. 2005;23(1):25-31. [ Links ]
65. Butler CC, Hood K, Verheij T, Little P, Melbye H, Nuttall J, Kelly MJ, Mölstad S GM, Almirall J, Torres A, Cals JWL, Butler CC, Hopstaken RM, Hood K DG. Informe Resistencia Bacteriana. [Internet]. 2013. Disponible en: http://www.portalfarma.com/Profesionales/comunicacionesprofesionales/informes-tecnico-profesionales/Documents/Informe_Resis-tencia_Bacteriana_PF79.pdf. [ Links ]