Introducción
Staphylococcus aureus es una bacteria de gran importancia debido a su participación en diferentes patologías 1, esto dado por la intervención de los distintos factores de virulencia y patogenicidad, codificados por los diversos genes que son expresados a lo largo de su ciclo de vida. Se conoce que este microorganismo es de difícil tratamiento y es capaz de colonizar e invadir las células de su hospedero, lo cual es posible debido a su fisiopatología, donde se encuentran mecanismos de resistencia como la formación de biopelícula las cuales crean una matriz extracelular conformada principalmente por proteínas, poli actualmente conocido como sacáridos y ácidos nucleicos. La formación de esta matriz causa que la interacción de los antibióticos con las bacterias no se dé de manera adecuada generando fallas en los tratamientos 2. Se describe también las funciones de las MSCRAMM conformadas por proteínas capaces de reconocer las sustancias presentes en la matriz extracelular de los seres humanos destacándose las fnBPs y el factor clumping encargados de reconocer la fibronectina y el fibrinógeno respectivamente 3,4, además se detalla el mecanismo que usa la bacteria para invadir las células del huésped y posteriormente producir enfermedad.
El propósito de este artículo es hacer una revisión de los mecanismos de patogenicidad de S. aureus con énfasis en los componentes de la superficie microbiana que reconocen moléculas de la matriz adhesiva (MSCRAMM) en la formación de biopelícula y en la importancia de cada uno de ellos en la virulencia de este microorganismo.
Generalidades de Staphylococcus aureus
Staphylococcus aureus es un microorganismo descubierto en 1880 por el cirujano escocés Alexander Ogston (1844-1929), quien encontró que el pus producido en las heridas quirúrgicas era generado por esta bacteria, al observar un absceso de uno de sus pacientes al microscopio. Posteriormente, en 1882, Ogston le dió el nombre de "Staphylococcus", del griego "Staphylo" que significa "racimo de uvas". Después, en 1884, el cirujano alemán Anton J. Rosenbach (1842-1923) identificó dos cepas de Staphylococcus y las nombró de acuerdo a las pigmentaciones que producían: Staphylococcus aureus, del latín "aurum" para el pigmento color oro, y Staphylococcus albus (actualmente conocido como Staphylococcus epidermidis), del latín "albus" para el pigmento blanco 5.
Esta bacteria está clasificada como un coco Gram positivo que se agrupa en racimos, β hemolítico, catalasa y coagulasa positivo. Se describe que este microorganismo hace parte de la flora normal de los seres humanos encontrándose principalmente en la piel, en la zona nasofaríngea, pliegues inguinales y axilas 6. Sin embargo, este patógeno se caracteriza por generar infecciones en piel y tejidos blandos (músculos, tendones, tejidos grasos, vasos sanguíneos), invasión a dispositivos médicos 7,8 y también ha sido relevante en las enfermedades transmitidas por alimentos (ETA) 9.
Se ha descrito a S. aureus como la principal causa de bacteriemia nosocomial en Norteamérica y Latinoamérica, y en Europa como la segunda causa de bacteriemia en hospitales 10. Aunque cualquier persona puede llegar a adquirir una infección por este microorganismo, hay algunos factores de riesgo en diferentes comunidades que son importantes; por ejemplo, los niños menores de dos años, los usuarios de drogas inyectadas, diabéticos, pacientes con infecciones pulmonares recientes, o de piel 11, o que hayan sido sometidos a hemodiálisis o a una cirugía reciente 9, entre otras, incrementan la posibilidad de sufrir una infección por S. aureus.
La colonización de Staphylococcus aureus puede evidenciarse en varios tejidos o sistemas del huésped; por ejemplo, en el endotelio cardiaco comienza, ya sea por un traumatismo directo o por inflamación que puede ser causada por una enfermedad de base. Estos factores contribuirán a que las células subendoteliales produzcan proteínas que serán expresadas en la matriz extracelular. A su vez las bacterias tienen distintos factores que les permitirán unirse a estas proteínas (proteínas de unión al fibrinógeno y proteínas de unión a la fibronectina). Estos factores son claves para la invasión y colonización e inflamación del tejido. En las infecciones en piel y tejidos blandos S. aureus evade la respuesta inmune mediada por neutrófilos y macrófagos al bloquear la quimiotaxis, secuestrando anticuerpos u ocultándose por medio de la formación de biopelícula 12.
Para la identificación de esta bacteria en el laboratorio, inicialmente se puede utilizar microscopia, donde se observan cocos Gram positivos. Para la confirmación de la especie se realizan pruebas como la de la enzima coagulasa, donde a diferencia de las demás especies de Staphylococcus, S. aureus es coagulasa positivo. Asimismo, se puede utilizar la prueba de catalasa, donde S. aureus por medio de esta enzima produce oxígeno, al interactuar con el peróxido de hidrogeno 13. También se utilizan diferentes medios de cultivo, el más comúnmente usado para la identificación y crecimiento de este microorganismo es el agar Baird Parker, en este medio las colonias de S. aureus se ven de color negro debido a la reducción del telurito y con un halo transparente a causa de la acción lipolítica sobre la yema de huevo. En el agar salado manitol, el cual posee agentes inhibidores para que solamente haya crecimiento de las diferentes especies de Staphylococcus, las colonias típicas de S. aureus son de color amarillo debido a la fermentación manitol, mientras que las especies de Staphylococcus coagulasa negativa producen colonias de color rojo. Para la identificación de cepas patógenas se utiliza el agar DNAsa ya que la actividad desoxirribonucleasa es indicadora de patogenicidad; en este agar se estudia la capacidad del patógeno de hidrolizar el ADN, lo cual se observa por la formación de halos transparentes en el medio 9.
Desde el descubrimiento de este microorganismo se han utilizado diferentes tratamientos. En 1940 la penicilina G era el antibiótico de elección para tratar estas infecciones; sin embargo, algunas cepas adquirieron resistencia no solo a este medicamento, sino a la meticilina y al grupo de los β-lactámicos, también se han descubierto cepas resistentes a macrólidos 14 y lincosamidas y en algunos países se ha reportado resistencia al trimetropim y a las sulfonamidas 15.
Fisiopatología
Dentro de la fisiopatología de S. aureus se pueden encontrar ciertos factores de virulencia siendo los más destacados los polisacáridos capsulares, la formación de biopelículas, las MSCRAMM (componentes de la superficie microbiana que reconocen las moléculas de la matriz adhesiva) entre las cuales se encuentran las proteínas de unión a la fibronectina, proteínas de unión al fibrinógeno (factor clumping) y toxinas (α y β).
En general, S. aureus presenta en su DNA secuencias de inserción (segmentos de DNA que puede moverse en una posición cromosómica a otra del mismo o diferente cromosoma), bacteriófagos e islas de patogenicidad (fracción de ADN que lo faculta como virulento y está contenido en plásmidos) 16, pero el genoma de este microorganismo puede variar de acuerdo a la cepa con la que se trabaje. En el caso de la cepa USA300, su genoma consiste en un cromosoma circular de 2 872 769 pares de bases (pb) y tres plásmidos (pUSA01, pUSA02, pUSA03). El plásmido pUSA01 no tiene una función identificable, en el plásmido pUSA02 está codificada la resistencia a tetraciclina, mediada por el gen tetK; mientras en el plásmido pUSA03 se encuentran los genes ermC y ileS que codifican la resistencia a macrólidos, lincosamidas estreptogramina B y mupirocina 17. Una de las características más importantes que presenta esta cepa es la existencia de islas de patogenicidad, la más importante de ellas es el elemento móvil catabólico arginina (ACME) que se encuentra únicamente en la cepa USA300 de S.aureus, la cual fue adquirida de S. epidermidis por transferencia horizontal de genes 18. La existencia de esta isla de patogenicidad le permite a la USA300 persistir en ambientes similares a la piel humana, además de prosperar en ambientes ácidos y con poliamidas similares al sudor, lo que contribuye a la virulencia de la cepa 19. Además, se encuentra la isla de patogenicidad SCCmec (Staphylococcal cassette chromosome mec), en la cual se encuentra el gen mecA 20 y algunos reguladores que en conjunto son responsables de la resistencia a la meticilina 21. También se encuentran las islas de patogenicidad vSaα y vSaβ, que codifican genes de virulencia como la leucocidina y superantígenos como enterotoxinas y exotoxinas 22.
Otros elementos móviles encontrados en la USA300 son, por una parte, el profago Sa2USA el cual posee los genes que codifican la toxina PVL (Panton- Valentine Leucocidina), la cual es una toxina sinergohimenotrópica que produce dos Proteínas la LukS-PV y LukF-PV, encargadas de provocar muerte celular por apoptosis o necrosis 23, y, por otra, el profago Sa3USA al cual se le atribuye tener los genes que codifican las estafiloquinasas y las proteínas inhibidoras de quimiotaxis, las cuales generan un desplazamiento bacteriano a través de la fibrina disuelta en sangre y la inhibición del reclutamiento de neutrófilos respectivamente 17.
Los polisacáridos capsulares contribuyen a la resistencia de la bacteria contra la fagocitosis, mediada por los neutrófilos polimorfonucleares 24. La encapsulación de S. aureus fue descrita por primera vez en 1931 por Gilbert 25, y actualmente se conocen dos tipos de polisacáridos capsulares CP5 y CP8, estos polisacáridos son los de mayor importancia y se encuentran en todas las cepas clínicas de esta bacteria 26.
Otro factor importante en la virulencia de S. aureus es su capacidad de formar biopelícula, generando que sea más complicado tratar infecciones causadas por este patógeno y contribuyendo a la resistencia bacteriana.
Biopelícula
La biopelícula fue observada por primera vez en 1683 por el microbiólogo Van Leeuwenhoek 27 quien la describió en la placa dental, esta se forma mediante conglomerados de células adheridas a una superficie, formando una matriz extracelular conformada principalmente por exopolisacáridos, proteínas y ácidos nucleicos esto la haca la bacteria mil veces más resistentes que las bacterias que viven en forma planctónica 28,29
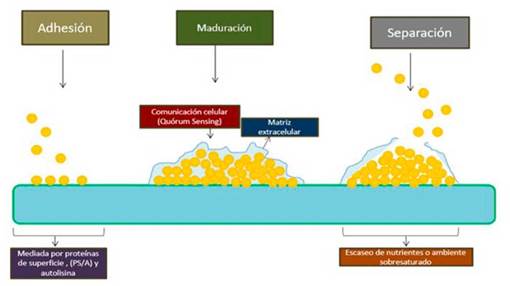
La adhesión que es la primera etapa de formación de la biopelícula, las condiciones ambientales como la temperatura, osmolaridad, pH, presencia de hierro y oxígeno, son importantes para generar una adecuada unión entre las células y la superficie que desean colonizar; también interviene la polaridad y las interacciones hidrofóbicas presentándose una mayor unión en superficies rugosas e hidrofóbicas como el látex y el plástico (Figura 1). Para que se dé la adhesión de estos microorganismos intervienen las proteínas de superficie, polisacárido/adhesinas capsulares (PS/A), la autolisina y fibrillas poliméricas extracelulares. Generalmente, después del proceso de adhesión las bacterias pierden sus flagelos para empezar a formar la biopelícula 30. En esta etapa el polisacárido de adhesina intercelular (PIA) que es catalizado por enzimas del operón ica, el cual posee los genes icaR (regulador) y icaADBC (biosintéticos). En presencia de glucosa 31,32 es importante en la adhesión intercelular 33.
Posteriormente se llega a la etapa de maduración, donde hay crecimiento, las células se dividen y estas células resultantes se diseminan por el lugar colonizado y forman una matriz extracelular 29. Se ha visto que estas etapas se llevan a cabo por medio de un mecanismo intercelular denominado quorum sensing (QS) que permite que haya un proceso de señalización, lo que da lugar a que la biopelícula actúe como una comunidad en lugar de un grupo de células independientes 34. El QS en S. aureus está mediado por el locus agr. El sistema agr está constituido por RNAII y RNAIII que a su vez son manejados por dos promotores, el P1 y el P3 35. El RNAII contiene 4 genes (agrB, agrD, agrC, y agrA) el gen agrB cumple funciones de controlador de densidad y activador de la fosforilación; asimismo, este junto con el gen agrD están implicados en la producción de autoinductores (AIs), que a su vez junto con la proteína agrC promueven la transcripción de RNAII y RNAIII. La importancia del sistema agr en el QS radica en que este regula los factores de virulencia de la bacteria, por lo tanto, cuando ocurre una mutación en este sistema, disminuye patogénesis o la severidad de la enfermedad 36.
La última etapa de formación de la biopelícula es la separación, que se genera cuando el ambiente se encuentra sobresaturado o escasean los nutrientes y debido a esto por medio de señales químicas comienza el desprendimiento de las bacterias para colonizar nuevas superficies 29.
La importancia de la biopelícula radica en su papel en la producción y degradación de materia orgánica, la degradación de contaminantes ambientales y la infección. En cuanto al papel que tienen estas estructuras en las enfermedades y considerando que las bacterias formadoras de biopelícula presentan una sensibilidad reducida a los tratamientos convencionales, el Instituto Nacional de Salud de EEUU reporta que más del 60% de todas las infecciones microbianas son causadas por biopelículas, de igual manera se les atribuye el 60% de las infecciones nosocomiales. También es bien conocida la capacidad de las biopelículas de colonizar dispositivos médicos como catéteres y válvulas cardiacas, que pueden conducir a bacteriemias o sepsis 37.
También se ha visto que la producción de biopelícula por diferentes microorganismos puede aplicarse en el tratamiento de aguas, gracias a que degrada la materia orgánica que se encuentra en aguas residuales 38; asimismo, se ha observado su efectividad en la eliminación de fósforo de estos ecosistemas, lo cual se logra exponiendo la biopelícula a ciclos anaerobio-aerobio, causando que los micoorganismos acumulen más cantidad de fósforo que el necesario para sus necesidades estequiométricas, los microorganismos más utilizados en estos procesos son Acinetobacter, Pseudomonas vesicularis y Klebsiella pneumoniae39.
Así como la biopelícula contribuye a la persistencia bacteriana y a la complicación en el tratamiento de las infecciones producidas por S. aureus, también hay otros factores propios de la bacteria que están implicados en esto, uno de ellos son las MSCRAMM, las cuales se mencionan a continuación.
MSCRAMM
Las MSCRAMM son componentes de la superficie microbiana que reconocen moléculas de la matriz adhesiva. Este grupo de proteínas media la adherencia de las bacterias a los componentes de la matriz extra-celular del huésped, tales como colágeno, fibronectina (Fn), fibrinógeno (Fg), elastina, factor de von Willebrand, vitronectina y sialoproteína ósea 40,41. Las proteínas que componen esta familia tienen una estructura general, en la cual hay un dominio A en el extremo amino terminal y una región R. En el dominio A se encuentran tres subdominios (N1, N2, N3), donde N2 y N3 son importantes para la formación del sitio de unión al ligando 42.
Factor clumping
Una de las características más importantes de S. aureus es su capacidad de formar grumos en presencia de plasma, esta habilidad está dada por proteínas de adhesión al fibrinógeno que permiten que se cree una red entre la bacteria y el fibrinógeno, lo que a su vez ocasiona aglutinación celular 43. La afinidad que presenta este factor es muy alta ya que se puede producir aglutinación en bajas concentraciones de fibrinógeno. Este factor está dado por dos proteínas la ClfA y la ClfB. Se ha visto que estas proteínas podrían tener una implicación directa en la endocarditis invasiva producida por S. aureus al unirse al fibrinógeno presente en el cuerpo del huésped 44.
Proteínas ClfA y ClfB
Son proteínas encargadas de reconocer el fibrinógeno presente en el plasma del huésped dando como resultado un acúmulo de células bacterianas. El fibrinógeno es una glicoproteína sintetizada en el hígado con un alto peso molecular, se estima que podemos encontrar en circulación concentraciones de 2 a 3,5 mg/ml. La estructura de esta proteína está dada por dos dominios D laterales, un dominio E central y el dominio αC, los cuales se encuentran unidos por tres cadenas polipeptídicas Aα (610 aminoácidos) la Bβ (461 aminoácidos) y la cadena Y la cual presenta dos isoformas la γA (411 aminoácidos) y la γ' (427 aminoácidos) 45. La importancia de esta proteína está dada por ser el precursor de la fibrina la cual interviene en los procesos de coagulación y su posible participación en enfermedades aterotrombóticas. Las proteínas de unión al fibrinógeno son expresadas en diferentes etapas del desarrollo bacteriano dándose la expresión de la proteína ClfA durante todo proceso de crecimiento, mientras que la proteína ClfB sólo es expresada en la fase temprana de crecimiento exponencial 24.
La proteína ClfA posee dos sitios de unión al fibrinógeno dado por el reconocimiento de las integrinas allβ3 y allβ2 las cuales son encontradas en la cadena polipeptídica Y del Fg, haciendo esto una diferencia con la proteína ClfB debido a que ésta tiene sus sitios de unión con el fibrinógeno en las cadenas polipeptídicas α y β 46.
Proteínas FnBPA y FnBPB
El reconocimiento de la fibronectina presente en la matriz extracelular de los humanos por parte de S. aureus es de gran importancia, dado que esto ayuda a la colonización e invasión de las células. Este reconocimiento está dado por las proteínas de adhesión FnBPA y FnBPB. La fibronectina es una glicoproteína que se puede encontrar en el plasma sanguíneo u otros fluidos (forma soluble), y tisularmente se ensambla en la matriz celular 47. Dentro de sus funciones en las superficies bióticas y abióticas se ha visto que juega un papel importante en la adhesión de las células a la matriz. Además, también actúa como guía de las migraciones celulares que tiene lugar en los embriones de los vertebrados. Se ha visto que la fibronectina ayuda a incrementar la coagulación de la sangre, así como también está presente en la cicatrización y en la fagocitosis 48. La fibronectina posee una estructura de 12 módulos de tipo I (FnI) y dos módulos de tipo II (FnII), también debido a la variación de empalmes puede tener de 15 a 17 módulos de tipo III (FnIII) 49.
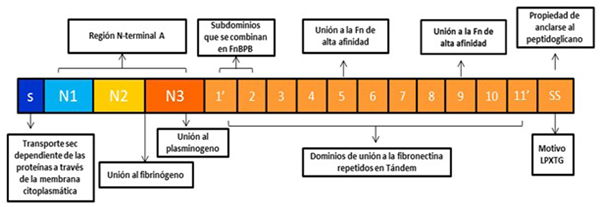
Cada proteína de unión a la fibronectina posee una región N-terminal que a su vez se divide en tres subdominios (N1, N2, N3). También posee dominios de unión a la fibronectina repetidos en tándem. La proteína FnBPA posee 11 dominios mientras que la proteína FnBPB posee 10 41,50. La razón de que la proteína FnBPB tenga un dominio menos es que los dominios 1 y 2 se combinan formando uno solo. También se ha visto que hay dominios que tienen más afinidad que otros, por ejemplo los dominios 5 y 9 tienen más afinidad que el resto.
Estas proteínas tienen un extremo SS que se ha relacionado con la propiedad de anclarse al peptidoglicano 51 (Figura 2).
Fuente. Elaboración propia.
Figura 2 Estructura general de las proteínas de unión a la fibronectina (FnBPA y FnBPB).
Aunque las proteínas FnBPA y FnBPB tienen una estructura muy similar existen algunas diferencias: en el caso de FnBPA hay dos repeticiones adicionales (repeticiones B) de 30 residuos de aminoácidos de función desconocida, estos se localizan en el extremo C-terminal de la región A 52.
Ambas proteínas tienen una región S ubicada en el extremo N-terminal. Esta región está implicada en el transporte Sec-dependiente (es necesario una secuencia señal en el extremo N-terminal) de las proteínas a través de la membrana citoplasmática. También se encuentra la región M, que está constituida por residuos hidrófobos. En estas proteínas se puede encontrar un motivo LPXTG que es el destino de la enzima sortasa, esta enzima se ancla covalentemente a la pared celular del peptidoglicano 50 por los grupos carboxilos de las dos proteínas 7.
La actividad de unión a la fibronectina se ha localizado en las repeticiones D, las cuales están ubicadas en el extremo C de las proteínas, estas repeticiones hacen parte de las regiones conservadas de las FnBPs. Las FnBPs también poseen una región A, cuya función desconocida, sin embargo, algunas investigaciones han sugerido que hay cierta similitud de esta región con la región A de la ClfA, lo que podría sugerir que las FnBPs tengan la capacidad de unirse a otras sustancias diferentes a la fibronectina, como el fibrinógeno 24.
Las fnBPs en S. aureus están codificadas por los genes fnbA y fnbB41. Estos genes son altamente homólogos y se encuentran separados por una región intergénica de 682 pb y su expresión está dada en la fase exponencial de crecimiento y se encuentra limitada en la fase estacionaria mediado por los locus reguladores sar y agr. El locus agr cumple un efecto regulador negativo frente a la expresión de los genes fnbA y fnbB esto debido a la presencia de RNA II y RNA III, los cuales limitan la expresión de estos genes. Sin embargo, el locus sar puede cumplir la función de regular positiva y negativamente la expresión de estos genes, este locus consta de tres genes que son sarA, sarB y sarC encontrándose este último en la fase estacionaria y los dos primeros en la fase de crecimiento exponencial. La expresión de los genes fnb regulada positivamente por el locus sar está dada mediante la unión del gen sarA a un promotor encontrado en la región A de las proteínas de unión a la fibronectina 53
Invasión de las células del huésped producida por las proteínas FnBPA y FnBPB
La invasión celular es un mecanismo de patogenicidad y persistencia del microorganismo debido a que puede permanecer oculto en las células del huésped evitando así la respuesta inmune y la interacción con los antibióticos 54. S. aureus tiene la capacidad de invadir células que normalmente no participan en la fagocitosis, por ejemplo, las células epiteliales o los osteoclastos 24.
Las FnBPs cumplen una función importante en la invasión de las células del huésped debido a la capacidad de estas proteínas de adherirse a la fibronectina. En la invasión celular por S. aureus la fibronectina actúa como un puente molecular entre la bacteria y la célula. El proceso de invasión celular empieza cuando las FnBPs se unen a los módulos de tipo I en el dominio N-terminal de la fibronectina por el mecanismo de cremallera en tándem, compitiendo con los enlaces intramoleculares, dando lugar a un cambio de conformación en la fibronectina. Las interacciones intramoleculares entre los módulos N-terminales de tipo I y los módulos C-terminales de tipo III dan lugar a la activación alostérica del módulo de tipo III, exponiendo un motivo RGD que es reconocido por la integrina a531 para promover la invasión por endocitosis. Teniendo en cuenta esto se podría decir que la fibronectina actúa como un puente, entre la célula huésped y la bacteria 51.
Hay que tener en cuenta que una sola proteína FnBP se puede unir a múltiples moléculas de fibronectina, por lo tanto al menos un dominio de unión de fibronectina de alta afinidad es necesario para la activación alostérica de fibronectina y la posterior invasión de las células epiteliales. Mientras tanto en la invasión de queratinocitos se requiere de tres FnBRs de alta afinidad, esto puede ser probablemente porque hay menos integrinas presentes 51.
Finalmente, el conjunto de estos mecanismos de patogenicidad y virulencia, le permiten a la bacteria persistir en el huésped y en el ambiente, sobreviviendo a factores adversos, al sistema inmune y a los antimicrobianos.