Introducción
En la última década se han desarrollado una variedad de estudios que giran en torno al uso de modelos de investigación, un ejemplo de ello es el uso de las líneas celulares. Los modelos celulares se establecen como una herramienta del conocimiento en relación con el nivel celular del desarrollo del organismo humano, ya que orienta la comprensión de tres características básicas: patrón particular en cuanto a la síntesis de proteínas, cambios morfológicos adaptativos e información genética contenida en las células, para así poder postular un modelo efectivo encaminado al desarrollo y producción de conocimiento en investigación. La técnica del cultivo celular ha permitido conocer el comportamiento in vitro de las células cancerosas y patologías en general, constituyendo una herramienta básica de aplicación fundamental en el campo médico o veterinario, en investigación y en la industria farmacéutica, ya que en las diferentes áreas se pretende analizar, describir, explicar, simular y entender un proceso fisiopatológico 1.
La línea celular HEp-2 (Human Epidermoid carcinoma cepa N° 2), también conocidas como Human Epithelioma, fue descrita por primera vez por Moore, Sabachewsky y Toolan en 1952 2. Esta línea celular se ha empleado para evaluar la infección con C. trachomatis, lo que permite realizar estudios en el comportamiento de la infección frente a diferentes áreas del conocimiento. Es una línea metabólicamente activa lo que favorece el cultivo de la misma para su posterior infección. C. trachomatis es una bacteria Gram negativa, intracelular obligada porque carece de habilidad para sintetizar ATP, se caracteriza por causar infecciones de transmisión sexual en humanos, especialmente en poblaciones que se encuentran en edades sexualmente activas 3. En Colombia, se ha reportado una prevalencia del 6% 4.
Las infecciones causadas por C. trachomatis están relacionadas con enfermedades como la artritis de inicio temprano e infecciones oculares como el tracoma. Por otro lado, la infección con este microorganismo es de gran importancia en salud pública, por el curso asintomático de la infección, produciendo graves secuelas en mujeres por falta de tratamiento, entre ellas: infertilidad, cervicitis, enfermedad inflamatoria pélvica (EPI), dolor pélvico crónico y la posibilidad de padecer un embarazo ectópico. La infección por C. trachomatis se trata con antibióticos; sin embargo, la resistencia al tratamiento ha aumentado, lo que ocasiona persistencia del microorganismo en algunos pacientes 5.
Los cultivos celulares para infectar células con C. trachomatis, en la investigación básica, permite estudiar fenómenos complejos como, por ejemplo, la actividad intracelular, específicamente la transcripción de ADN, síntesis de proteínas, metabolismo energético, ciclo celular, diferenciación, apoptosis, el flujo intracelular de biomoléculas, como el procesamiento del ARN, el movimiento de las proteínas hacia diversos orgánulos; y, en cuanto a genómica y proteómica, el análisis genético, transformación celular, inmortalización, senescencia, expresión génica, rutas metabólicas, entre otros 6. Por lo anterior, en este estudio se optimizó la infección de la línea celular HEp-2 con C. trachomatis serovar L2 (VR- 902B) como modelo de investigación básica para realizar estudios con este microorganismo, lo que permitirá evaluar diversos factores morfológicos, genéticos, inmunológicos y de tratamiento.
Metodología
Establecimiento del cultivo de la línea celular HEp-2
Las células HEp-2 fueron obtenidas de la Fundación Instituto de Inmunología de Colombia (FIDIC) con una confluencia del 80%. Se mantuvieron en medio Eagle's Modificado Dulbecco's (DMEM), suplementado con suero fetal bovino al 20% (Gibco) y agregando gentamicina, hasta alcanzar una confluencia del 90%. Luego se procedió a congelar las células utilizando tripsina + EDTA al 0,25% y suero fetal bovino al 90%. Durante los protocolos se realizó un conteo de viabilidad celular por medio del método de exclusión de Azul de Tripán. La coloración de Giemsa realizada presentó algunas modificaciones, siendo necesario ajustar los volúmenes para apreciar de manera óptima las células. Cabe aclarar que se mantuvo la relación de colorante de 30 partes de solución amortiguadora y 1 parte de solución madre, y el mismo tiempo de contacto. Todos los protocolos que se utilizaron fueron estandarizados por Jutinico y colaboradores 7.
Infección de células HEp-2 con EB’s de C. trachomatis serovar L2
El protocolo de infección optimizado fue tomado de Engstrom y col. 8. Inicialmente, las células HEp-2 fueron sembradas a diferentes concentraciones en láminas portaobjetos MilliCell EZ SLIDE, con el fin de determinar la concentración a la cual se obtiene una confluencia óptima del 90%, determinando que al 5% de CO2 5% de SFB a 37°C durante 24 horas se obtenía una confluencia del 90%.
Para determinar la concentración de cuerpos elementales necesarios para la infección, se retiró el medio DMEM del cultivo y se realizaron lavados con el buffer solución salina equilibrada de Hanks (HBSS) durante 1 minuto cada uno; esto con el fin de retirar detritos y células muertas. Una vez lavadas las células los EB’s diluidos en buffer estéril de Sucrose-Phosphate-Glutamate (SPG se distribuyeron en 6 de los 8 pozos de la lámina, se adiciono una concentración distinta de cuerpos elementales diluidos SPG), partiendo de 10µL, 20µL y 40µl, los 2 pozos restantes fueron empleados como controles negativos de infección. Se les agregó SPG estéril de manera tal que cada pozo se completó a un volumen de 100uL. Posteriormente, se llevó a incubar a 37ºC en CO2 al 5% durante 1 hora y 20 minutos, tiempo tras el cual se agregaron 300uL de medio DMEM suplementado con 20% de SFB a cada uno de los pozos, incluyendo los controles negativos, para un volumen final de 400uL. Esto se llevó nuevamente a incubar a 37ºC en CO2 al 5% y se evaluó a las 24 y 48 horas post-infección (h.p.i). Pasadas las horas post-infección, se eliminó el medio DMEM contenido en cada pozo, se lavaron las células 3 veces empleando HBSS y se fijaron con 2 gotas de metanol al 100% y se tiñeron con Giemsa.
Detección de la infección de HEp 2 con EBs de C. trachomatis por inmunofluorescencia directa
Para corroborar la infección de células HEp-2 con C. trachomatis serovar L2 se realizó la técnica de inmunofluorescencia directa, por medio de un anticuerpo primario policlonal de conejo: “Rb pAB to C. trachomatis (FITC) ab21211 dirigido contra la proteína de membrana MOMP de C. trachomatis”. Las células HEp2, una vez infectadas, fueron lavadas con PBS. Posteriormente, las células se fijaron a las 24 y 48h.p.i con metanol frío (mantenido a -20°C) al 100%. Luego se adicionarion 50uM de cloruro de amonio durante 5 minutos, con el fin de eliminar las trazas de metanol que no fueron removidas. Enseguida se agregó saponina al 0,1% por 10 minutos, para permeabilizar la membrana celular. Luego se procedió a realizar el bloqueo con una solución de PBS al 1% de SFB durante 20 minutos y se preparó una dilución del anticuerpo 1:100 para adicionar en cada pozo. Se mantuvo la lámina durante 1 hora en cámara húmeda a 37°C, se retiró el anticuerpo diluido realizando lavados con PBS y se observó en el microscopio de fluorescencia Leica DM2500. Finalmente, se adicionó colorante Hoechst, para tinción de núcleo y nuevamente se realizaron lavados para visualizar cada pozo al microscopio. Las imágenes se capturaron utilizando Leica DFC425 C, con los filtros I3 y A, con rangos de excitación de 565 +/-25 y 360 +/- 20 y luz de color Verde y Azul respectivamente.
Resultados
Para realizar la infección de las células HEp2 con EB’s de C. trachomatis, se partió de cultivos celulares óptimos que permitieran el desarrollo del microorganismo. La línea celular HEp-2 fue cultivada con medio DMEM + SFB (Gibco) en una atmósfera de 37°C con 5% de CO2, la infección se demostró con coloración de Giemsa e inmunofluorescencia directa.
Estructura y morfología de las células HEp-2 sin infectar
Las células HEp-2 sin infectar fueron visualizadas en un microscopio invertido (Leica) en objetivos de 40x y 100x respectivamente, apreciándose células alargadas de forma triangular (ver Figura 1A). Se observó claramente el citoplasma bien definido, núcleo de gran tamaño con 2 nucleolos prominentes (ver Figura 1B). En los cultivos iniciales se evaluó la confluencia en diferentes tiempos, a las 48 horas se obtuvo una confluencia del 70%, donde se distinguieron islotes de células y campos sin adherencia celular (ver Figura 1C). Posteriormente, a las 72 horas, se logró una confluencia óptima del 90% con pocos campos sin adherencia y sin presencia de detritos ni células muertas, criterios importantes para realizar una buena infección (ver Figura 1D).
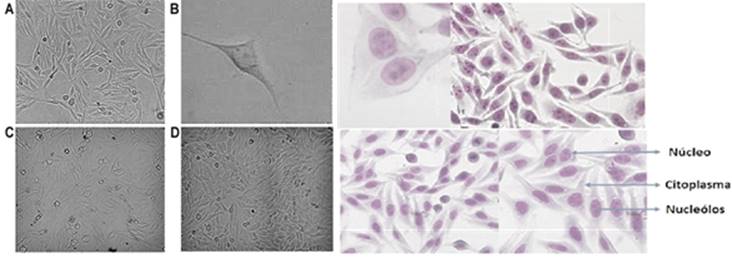
Figura 1 Estructura y morfología de las Células HEp-2 sin infectar. A. Se visualizan células HEp-2 observando su estructura (izquierda objetivo 40x). B. Se aprecia el citoplasma, núcleo y nucleolos en el microscopio invertido. C. Se observa una confluencia del 70% de la línea celular HEp-2, a las 48. D. Se observa una confluencia del 90% a las 72 horas. En la derecha se observan las células HEp-2 teñidas con colorante Giemsa y visualizadas al microscopio óptico para diferenciar su estructura, en distintos objetivos.
Las células fueron teñidas con coloración de Giemsa y visualizadas en un microscopio óptico (Olympus), observándose células triangulares típicas de HEp-2 en cultivo, con citoplasmas uniformemente teñidos de morado sin presencia de precipitados, núcleos fucsias bien delimitados y entre 2-3 núcleos oscuros.
Células HEp-2 infectadas con EB’s serovar L2 de C. trachomatis y visualizada con Giemsa
Las células de cultivo de tejidos en monocapa (HEp-2) se infectaron con una multiplicidad de infección (MOI) de 1 EB’s/célula huésped. Una vez infectadas las células se tiñeron con Giemsa y fueron visualizadas en microscopio óptico. En los pozos control (células HEp-2 sin EB’s), se observan células completas, con morfología definida, sin daños en las estructuras, sin precipitados ni formas anexas (ver Figura 2A). En los pozos con células infectadas con C. trachomatis se visualizaron los EB’s como formas ovoides con una fuerte coloración morada, intracelulares y próximos a las células (ver Figura 2B). Además, las células presentaron vacuolización y ruptura de las membranas, que sugieren daño celular, (ver Figura 2C).
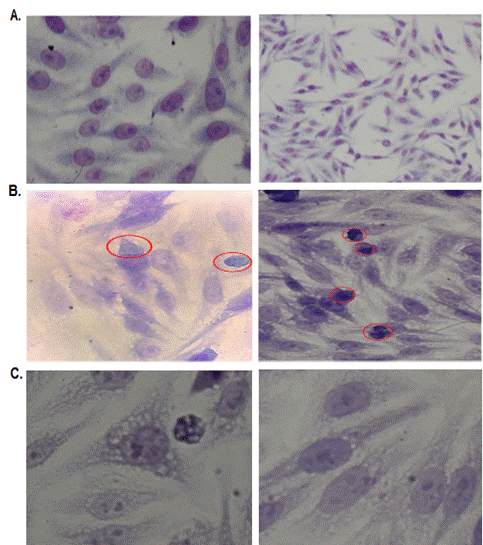
Figura 2 Las células HEp-2 infectadas y teñidas con coloración de Giemsa. A. Se observó la morfología celular de los pozos control, células con citoplasma completo, núcleos y nucleolos definidos, células adheridas en monocapa y sin cuerpos extraños. B. Células infectadas con cuerpos elementales de C. trachomatis a las 24 h.p.i. Se visualiza una forma de inclusión intracelular señalizadas con circunferencias rojas. C. Células vacuoladas post-infección.
Detección de la infección de HEp-2 con EB’s de C. trachomatis por Inmunofluorescencia directa
Al ser C. trachomatis un parásito intracelular obligado que desarrolla su ciclo de vida dentro del citoplasma, se comprobó la infección por inmunofluorescencia directa utilizando el anticuerpo policlonal ab 21211 dirigido contra C. trachomatis. La evaluación se hizo utilizando el microscopio de fluorescencia Leica DM2500 y se capturaron las imágenes con el software Leica DFC425C. Los pozos con células HEp-2 no infectadas (control) mostraron células con núcleos teñidos uniformemente de azul por el colorante fluorescente Hoechst (filtro A). No se observó la presencia de EB’s marcados con el anticuerpo policlonal (filtros I3 verde), confirmando con el merge la ausencia del microorganismo (ver Figura 3A). Por otro lado, las células infectadas, mostraron núcleos de tonalidad azul (Hoechst), se observaron estructuras verde fluorescentes de forma ovoide indicando la presencia de C. trachomatis (ab 21211). Finalmente, en el merge se evidenciaron formas próximas a los núcleos celulares correspondientes a EB’s de C. trachomatis serovar L2, confirmando la infección (ver Figura 3B).
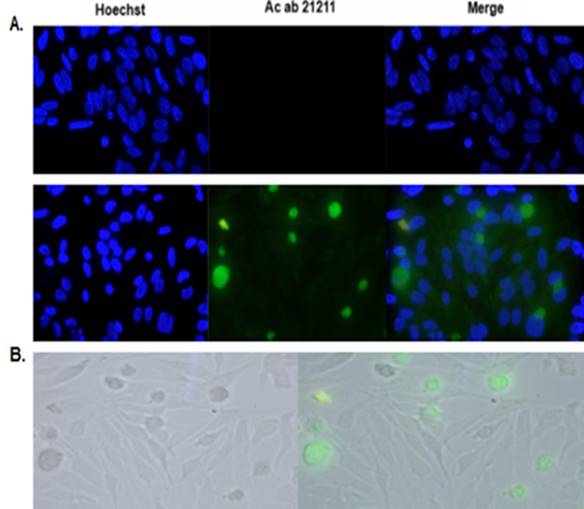
Figura 3 Detección de EB’s de C. trachomatis con Inmunofluorescencia directa sobre células HEp-2 utilizando el anticuerpo policlonal ab21211 . A. Control negativo: se observa ausencia de fluorescencia por parte del Anticuerpo ab21211, se observan únicamente núcleos teñidos de azul con Hoechst. En las células infectadas se observan estructuras verde fluorescentes sobre citoplasmas celulares y cercanos al núcleo, correspondientes a EB’s de C. trachomatis reconocidos con el anticuerpo ab21211. B. Imagen capturada sin filtros (con luz blanca), donde se observan células confluentes con formas de gran tamaño sobre y próximas a las mismas. El merge con el anticuerpo se observan estas estructuras clamidiales fluorescentes en el interior de las células.
La infección intracelular de C. trachomatis en células HEp-2 se confirmó por la captura de la imagen sin filtros, con luz blanca, donde se pudieron observar células confluentes con EB’s ovoides sobre y próximas a las mismas. Posteriormente, se realizó un merge de las células sin filtro y con el filtro I3 donde se observó la fluorescencia dada por el fluorocromo del anticuerpo dirigido a C. trachomatis (ver Figura 3C).
Discusión
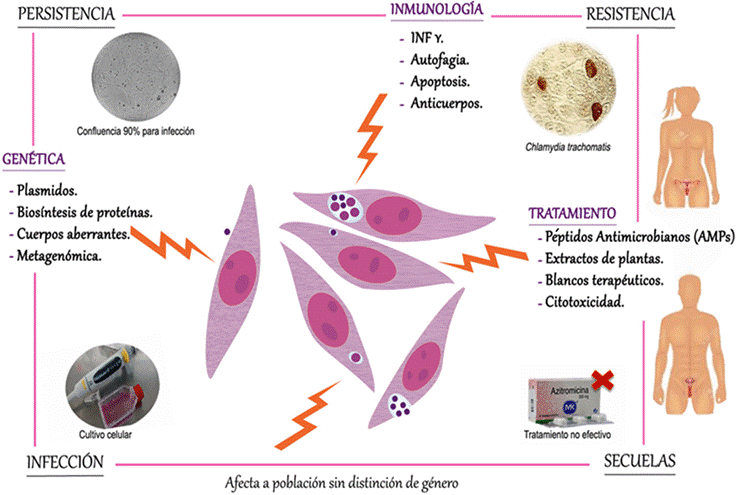
Los modelos de líneas celulares utilizados en investigación permiten simular procesos fisiológicos que dan información y ayudan a resolver diferentes interrogantes en áreas del conocimiento antes de requerir un modelo in vivo; así mismo, el establecer modelos de infección permiten aclarar diferentes interrogantes en investigación. En este estudio se realizó la infección de la línea celular HEp-2 con EB’s de C. trachomatis, con el fin de contar con un modelo de estudio para futuros proyectos que permitan generar conocimiento sobre los procesos que realiza esta bacteria intracelular y poder ser empleada en nuevos estudios en áreas como inmunología, genética, procesos de persistencia/resistencia y tratamiento (ver Figura 4).
En este estudio, la línea celular HEp-2 fue cultivada en medio DMEM modificando el porcentaje de SFB al 20% y agregando gentamicina a 37°C en una atmósfera de 5% CO2 y 95% aire para obtener una confluencia del 90% a las 24 horas. En el estudio realizado por Pajaniradje y cols. 9 se reportó que las células HEp-2 se pueden mantener en medio DMEM suplementado con 10% SFB, a 37°C en una incubadora humificada con una atmósfera de 5% CO2 y 95% de O2, condiciones similares a las establecidas en este estudio. Por otro lado, el estudio realizado por Shubach y cols. 10 mostró que las células HEp-2 pueden ser cultivadas en medio MEM Gibco® y DMEM: F12 en una concentración de SFB entre 5% y 10%, con presencia de glutamina al 1%. Estos estudios muestran cómo los requerimientos nutricionales son de importancia para establecer el cultivo de la línea celular y generar un crecimiento apropiado que sea requerido en cada estudio propuesto como se observó en los ensayos realizados en este estudio.
El uso de gentamicina como antibiótico, es indicado en cultivos para infección con C trachomatis. Antibióticos como la penicilina y la estreptomicina comúnmente utilizados inhiben el desarrollo de este microorganismo en cultivos. A su vez, es preciso destacar que la gentamicina se utiliza rutinariamente para inhibir el crecimiento bacteriano extracelular; en este caso, C. trachomatis al replicarse dentro de una vacuola única donde no se fusiona con organelos endocíticos o lisosómicos, no se encuentra expuesta a daño celular por dicho antibiótico. Por lo tanto, el medio de cultivo suplementado con gentamicina puede usarse sin ningún inconveniente, considerando que la gentamicina no puede cruzar las membranas de mamíferos y no entra en contacto con el cuerpo reticulado, y aunque pueda internalizarse en las células de los mamíferos por pinocitosis, las vesículas pinocíticas tampoco se fusionan con las vacuolas que contienen a C. trachomatis 11-13. Así mismo, es fundamental resaltar el trabajo de Coico 14, quien hace mención del uso de gentamicina.
Por otro lado, el uso de soluciones tales como SPG y HBSS no afectan la monocapa celular. La unión células-EB’s y la invasión a estas son clave para el desarrollo de C. trachomatis y su patogenia. Al presentarse una interrupción puede llegar a ser inhibida la infección, resaltando la importancia de un cultivo de células confluente y una infectividad EB’s-célula adecuada 14.
De igual manera, en estudios donde se realizan procesos de infección, se muestra el uso del SPG para la dilución de cuerpos elementales y como medio de transporte para C. trachomatis, y el HBSS para lavados de las monocapas 8,15,16 donde se emplean una o ambas soluciones durante el proceso de infección y para el mantenimiento de los EB’s, igual a lo realizado en el protocolo de infección en este estudio. No obstante, en otras investigaciones se omite el uso de estas soluciones y los protocolos de infección son modificados en algunos procesos como centrifugación asistida para favorecer la infección 11,17. Al añadir los cuerpos elementales, estos se incubaron a 37°C sin centrifugación para evitar algún efecto sobre el proceso infección-adhesión celular. Sin embargo, Al-Zeer y cols. 18, de acuerdo a la especie de Chlamydia describen el uso o no de centrifugación como requerimiento para la infección. En este caso, no influyó el realizar un proceso de centrifugación y optar por un proceso de incubación de EB’s con células HEp-2 a temperatura de 37°C, según se pudo comprobar al realizar la tinción de Giemsa a las monocapas de células HEp-2 infectadas con el microorganismo.
La infección celular con EB’s de C. trachomatis es un proceso en donde es importante mantener una infectividad cuerpo elemental-célula. Las inclusiones de C. trachomatis pueden fusionarse si dos bacterias infectan una sola célula huésped, por lo que la detección de C. trachomatis por Giemsa se ha utilizado para observar la presencia de inclusiones citoplasmáticas de líneas celulares infectadas con esta bacteria. En el trabajo de Giakoumelou y cols. 19, células estromales del endometrio fueron infectadas con C. Trachomatis y los signos de infección se revelaron a partir de inclusiones teñidas de color púrpura mediante la tinción de Giemsa, lo cual se asemeja a este ensayo, dado que los cuerpos elementales son observados de esta tonalidad, próximos a las células HEp-2.
Por otro lado, las células teñidas con Giemsa, tal como lo muestra el estudio realizado por Soysa y cols. 20, tras ser infectadas exhiben cambios característicos en la morfología celular que indican la muerte apoptótica y daño en células HEp-2, al observarse por microscopía óptica después de ser tratadas con el extracto de la planta semecarpus donde se añadió un volumen de Giemsa diluido (1: 9, pH7,2, 5μL) a las células durante 10 minutos 20. Lo que difiere para este caso, ya que tras probar las variables se determinó un tiempo de exposición de 60 minutos añadiendo un volumen de Giemsa (30:1, pH7,2, 300μL) 21. Comprobándose que la tinción de Giemsa es una herramienta útil para poder observar las estructuras celulares y evaluar la infección con C. trachomatis.
La técnica de inmunofluorescencia permitió confirmar la presencia de C. trachomatis en células HEp-2. Los resultados obtenidos se pueden comparar con la tinción de Giemsa en cuanto al tamaño de la partícula infecciosa. En estudios como el de Cochrane y cols. 21, donde evaluaron la infección en células HEC1-A con el serovar L2 (VR-902B) de C. trachomatis y la demostraron con inmunofluorescencia, los cuerpos de inclusión se observan de gran tamaño (análogo al núcleo celular) siendo similar a lo encontrado en este estudio. De igual manera, el trabajo de Nogueira y cols. 22 en células HaCaT con C. trachomatis serovar L2, donde las inclusiones de Chlamydia parecían maduras 22.
El establecer un modelo de infección de C. trachomatis abre las puertas para realizar nuevos ensayos en diferentes áreas de la investigación para contribuir al conocimiento científico, aplicándolo en estudios 23-26, como la reducción de tamaño de los RB’s al dividirse antes de su conversión a EB’s 26 y regulación de la extrusión como mecanismo de liberación de C. trachomatis 27.
Perspectivas en investigación sobre C. trachomatis
Establecer un modelo de infección de C. trachomatis permite realizar ensayos en diferentes áreas de la investigación con el fin de contribuir a dar solución a diferentes interrogantes. Este modelo de investigación básica permite realizar diferentes estudios sobre este microorganismo; así mismo, poder evaluar diversidad de factores morfológicos, genéticos, inmunológicos y de tratamiento. Considerando que este microorganismo afecta a la población sin distinción de género, sexualmente activa, relacionándose con enfermedades de importancia y secuelas dada su persistencia en el organismo y la resistencia a los antibióticos con los que actualmente es tratada (ver Figura 4).