Introducción
El uso de antibióticos se convirtió en una panacea en su momento, al revolucionar la medicina moderna, además de que su aplicación y masificación ha conllevado al tratamiento efectivo para múltiples enfermedades. Sin embargo, las infecciones causadas por bacterias resistentes a los antibióticos han aumentado de forma considerable en los últimos años y representan una causa importante de morbilidad y mortalidad a nivel mundial, incluso en países desarrollados 1. Por esto, se han considerado las infecciones causadas por patógenos multirresistentes (MDR) como una emergente enfermedad global y una problemática de salud pública, que requiere la acción mancomunada de los actores en el ámbito de la salud mundial 2.
Se ha llamado la atención acerca de la emergencia y difusión de organismos Multidrogo-Resistentes (MDRO, por sus siglas en inglés), tales como S. aureus resistente a meticilina 3, enterococos resistentes a la vancomicina, y bacterias Gram negativas resistentes a cefalosporinas de tercera generación 4. Se estima que para las próximas décadas, el número de muertes causado por patógenos multirresistentes será mayor que las muertes causadas por cáncer 5.
La resistencia a los antibióticos es un fenómeno natural 6, que ocurre en respuesta evolutiva a la fuerte presión selectiva dada principalmente por la exposición de los microorganimos a estos compuestos. Mutaciones puntuales de novo en una población de bacterias susceptibles (por ejemplo, mutaciones puntuales en sitios de unión al ribosoma, confieren resistencia a tetraciclinas) y la difusión horizontal de determinantes genéticos móviles de resistencia, probablemente sea la causa del uso a gran escala en la actualidad de estos fármacos tanto en clínica como a nivel de la agroindustria. Esta hipótesis es soportada por las bajas tasas en los porcentajes de resistencia a los antibióticos en grupos de cepas bacterianas patógenas antes de la era antibiótica 7.
Desde un punto de vista global, para comprender la evolución y el impacto de la resistencia microbiana, en el año 2007, surgió el concepto de "resistoma", que enmarca todos los genes de resistencia a los antibióticos y sus precursores, tanto en bacterias patógenas como no patógenas, para entender y estudiar los orígenes, evolución y aparición de la resistencia 8.
La resistencia a los antimicrobianos se atribuye parcialmente a bacterias ambientales naturales que no causan enfermedades humanas 9,10. Cuando éstas interactúan, producen y metabolizan pequeñas moléculas, teniendo como resultado el desarrollo de una amplia gama de mecanismos para modular las actividades de estos compuestos. Los genes asociados a menudo ofrecen una ventaja selectiva más amplia, facilitando la movilización y el traspaso horizontal a otros microorganismos que comparten un nicho ecológico determinado 11. La liberación de agentes químicos (desinfectantes, metales pesados y otros contaminantes) en el medio ambiente puede acelerar el traspaso de genes de resistencia en las poblaciones bacterianas circundantes 12, lo que conlleva al aumento de la presión selectiva, y el eventual incremento en el número de aislados multirresistentes con ventajas evolutivas y adaptativas.
Se ha permitido la mezcla entre bacterias asociadas a infecciones humanas y microorganismos ambientales, por ejemplo, por medio de plantas de tratamiento de aguas residuales 13, fábricas de producción química, y el uso de estiércol como materia prima para la fertilización de cultivos 14, proporcionando así condiciones que facilitan enormemente la movilización y trasmisión de genes de resistencia, lo que se traduce en un conjunto de escenarios perfectos para que patógenos humanos bacterianos e incontables generaciones microbianas adquieran un sinnúmero de elementos genéticos que les confieran ventajas adaptativas y evolutivas.
Por otro lado, existe una relación directa entre el consumo de antibióticos y las variaciones de resistencia a estos entre diferentes países 15. El uso en exceso de los antibióticos en el ser humano y los animales está acelerando la pérdida de su actividad y es una de las mayores amenazas para la salud mundial, la seguridad alimentaria y el desarrollo sostenible, en general. El número de infecciones resistentes a tratamientos convencionales va en aumento. Por ejemplo, tuberculosis 16, neumonía 17, meningitis 18 entre otras, requieren cada vez más tratamientos complejos combinando sinérgicamente varios compuestos antibióticos para poder combatirlas 19.
Los microorganimos para hacer frente a los antibióticos y protegerse de su acción poseen mecanismos que incluyen la inactivación enzimática del antibiótico, modificaciones sitio-específicas del objetivo del antibiótico, hasta la eliminación de concentraciones intracelulares tóxicas del antibiótico mediante la producción de bombas de eflujo. Lo anterior muestra la gran plasticidad genómica que estos microorganismos tienen para adatarse y sobrevivir. A pesar de la necesidad urgente de nuevos antibióticos que sean eficaces contra bacterias resistentes, muy pocos compuestos están en desarrollo, la mayoría de los cuales son análogos a las diferentes clases de antibióticos conocidos 20 Es por esto que se requieren nuevas estrategias para combatir las infecciones persistentes.
Péptidos antimicrobianos
Los péptidos antimicrobianos (PAMs) son parte esencial de la inmunidad innata, y por tanto, agentes antibióticos eficaces que se encuentran en plantas, animales y microorganismos 21. Estas moléculas tienen un amplio espectro de acción, siendo activas contra bacterias, hongos, y virus. La estructura anfipática que comparten los AMP facilita sus interacciones y la inserción en la pared celular aniónica y las membranas fosfolipídicas de los microorganismos 22. Frecuentemente, la actividad de los AMPs resulta de la alteración de la integridad de la membrana celular; sin embargo, estos pueden actuar en diferentes dianas celulares incluyendo ADN 23, ARN 24 y otras proteínas 25, apareciendo como una alternativa prometedora a los antibióticos convencionales 26.
PAMs funcionan como parte de la defensa del huésped a través de la muerte microbiana. Además, existe una sólida evidencia sobre su capacidad de inmunomodulación en organismos superiores 27. Los AMPs son considerados como una interesante estrategia para la lucha contra la multirresistencia microbiana 28. Debido a su selectividad por bacterias sobre células eucariotas los hacen candidatos atractivos para nuevos compuestos farmacéuticos. De hecho, se han realizado intentos para aprovechar su potencial, y algunos de esos péptidos han sido evaluados en ensayos.
Estos pequeños péptidos son multifuncionales como efectores de la inmunidad innata sobre la piel y superficies mucosas 29. Han demostrado una actividad antimicrobiana directa contra diversas especies bacterianas, virus, hongos y parásitos 26. Los AMPs existen en una amplia gama de estructuras secundarias, tales como estructuras tipo α-hélices, β-hebras con uno o más puentes disulfuro, estructuras tipo bucle y láminas extendidas 30. Sus múltiples formas estructurales les permiten un amplio espectro de actividad antimicrobiana. Además de estas propiedades, ciertos factores cruciales como el tamaño, carga, hidrofobicidad, anfipáticidad y las interacciones específicas con componentes de la membrana celular también se atribuyen a su amplio espectro de actividad 31. Una de las características más llamativas es su reducido tamaño, ya que les facilita la rápida difusión y secreción fuera de las células, lo cual es necesario para obtener una respuesta de defensa inmediata contra patógenos 32. La mayoría de los péptidos antimicrobianos son catiónicos a un pH fisiológico, gracias al alto contenido de residuos tipo arginina y lisina en relación con aminoácidos como ácido glutámico y ácido aspártico, residuos con carga negativa 22; y generalmente, una proporción substancial (≥30%) de residuos hidrofóbicos con una carga neta entre +2 y +9. Además, este carácter catiónico usualmente es reforzado por una modificación tipo amida en el extremo C-terminal de la secuencia 33. Asimismo, adoptan estructuras anfipáticas con caras hidrofóbicas y extremos hidrofílicos 34, lo que les confiere propiedades de unión hacia membranas bacterianas mediante interacciones electrostáticas entre las cadenas laterales de los aminoácidos catiónicos y las superficies polianiónicas de las paredes bacterianas 34 ya sea por los ácidos teicoicos y lipoteicoicos en bacterias Gram positivas ó los lipopolisacáridos en bacterias Gram negativas 35, lo que les permite eliminar células diana específicas, sin afectar a las células del huésped 36.
Mecanismos de acción
Los PAMs pueden variar en tamaño (desde 12 a 50 o más aminoácidos) y son en general catiónicos, debido a la presencia de un exceso de los aminoácidos lisina y arginina. Aproximadamente, 50% de los aminoácidos que componen la secuencia, son de tipo hidrofóbico 37, favoreciendo así su rápida acción. Su actividad incluye bacterias Gram positivas 38, Gram negativas 39, hongos 40, virus encapsulados 41, parásitos 42 e incluso células cancerosas 43; pero en contraste con los antibióticos, su mecanismo de acción es muy diferente, ya que estos últimos actúan como inhibidores específicos de vías esenciales en las células microbianas como síntesis de la pared celular (antibióticos filactámicos: penicilina y derivados); síntesis de proteínas (aminoglucósidos: estreptomicina y derivados); inhibición de ácidos nucleicos (quinolonas: fluoroquilononas) o en la inhibición de rutas metabólicas importantes (trimetoprim-sulfametoxazol, inhibición de la ruta de síntesis del tetrahidrofolato), entre otros. Mientras que en los AMPs, por su amplia variedad estructural, su actividad no está dirigida a blancos celulares concretos como enzimas o receptores, sino más bien a características comunes de las membranas bacterianas 44-46.
Existe evidencia de que la permeabilización de la membrana inducida por los péptidos es el resultado de su interacción con la matriz lipídica de la membrana celular 47,48. En las bacterias Gram negativas, tanto la membrana externa como la interna poseen moléculas aniónicas orientadas hacia el exterior de la célula 49. Su interacción con fosfolípidos cargados negativamente explicaría su especificidad por las membranas bacterianas y no por los lípidos zwitteriónicos de la capa extracelular de las células eucarióticas 50. Respecto al mecanismo de cómo los PAMs destruyen la membrana, es posible que induzcan lisis completa de la bacteria, o que la perturben de una manera tal que permita la salida de componentes celulares esenciales, a la vez que se disipa el potencial de membrana. El mecanismo inicial de lisis consiste en el reconocimiento de los fosfolípidos mediante interacciones electrostáticas y una vez unidos a la membrana, los péptidos sufrirían una reorganización estructural, pasando de un estado desnaturalizado a una estructura de carácter anfipático, estabilizada por la interface lipídica con el medio acuoso 37.
Producción de nuevos PAMs
Como los PAMs naturales se derivan en gran medida de secuencias codificadas por genes, se han aplicado métodos bioinformáticos para crear bases de datos de PAMs conocidos, así como herramientas para predecirlos a partir de genomas que aún no han sido anotados. Desde la publicación de las bases de datos APD 51 y ANTIMIC 52, varias bases de datos han sido creadas para enfatizar ciertas características de éstos, agrupándolos en diferentes categorías; por ejemplo, en naturales, sintéticos o recombinantes (péptidos circulares, defensinas y tiopéptidos) 53,54, o por su diferente origen (humanos, bacterias, plantas, insectos y anfibios) 55,56. La base de datos DADP contiene únicamente péptidos de anfibios 57. Más recientemente, también se crearon las bases de datos YA-DAMP 58, CAMP 59 y LAMP 60.
Actualmente, el enfoque para el desarrollo de nuevos PAMs está en sus aplicaciones biomédicas, terapéuticas y biotecnológicas. Los métodos actuales para buscar en la cantidad casi ilimitada de secuencias de péptidos conocidas o predichas, bien sea empíricamente o computacionalmente nuevas funciones o características, están evolucionando continuamente. Se conocen tres enfoques claros de investigación en esta área: modificación de secuencias de PAM conocidas (denominadas como plantillas 61), modelado biofísico 62 para entender la actividad del péptido y el cribado virtual 63.
Las metodologías actuales usadas para la construcción de bibliotecas de PAMs presentan ventajas y desventajas, en cuanto al diseño de secuencias concierne; por ejemplo, la longitud de las secuencias ó el tamaño de la biblioteca. Técnicas basadas en la Reacción en Cadena de la Polimerasa (PCR), tales como el sitio de saturación por mutagénesis dirigida 64 y el barajado de ADN 65, donde las bibliotecas de ácido nucleico generadas aleatoriamente que codifican para PAMs se expresan en un huésped biológico, tienen gran complejidad y la longitud del péptido no es restringido en la mayoría de los casos, dado que las mutaciones se introducen de forma aleatoria; sin embargo, el control por parte del usuario sobre el diseño de la secuencia es muy limitado en estas técnicas. Los métodos combinatorios sintéticos, por otro lado, permiten el diseño de la secuencia personalizando una variedad de características de la secuencia per se; por lo tanto, esta última se ha empleado con éxito para generar bibliotecas combinatorias de PAMs 66. No obstante, estos sistemas siguen siendo limitados por el tamaño de la secuencia del péptido (longitud óptima hasta 20 aminoácidos), así como la inmensa dimensión de la biblioteca, debido al intenso trabajo asociado de producción y al alto costo relacionado con la complejidad de la química sintética 67.
Varios PAMs han sido desarrollados con éxito para aplicaciones farmacéuticas y comerciales. La información sobre la estructura y secuencia de aproximadamente 2846 de estos, de diversas fuentes, se pueden encontrar en las bases de datos en todo el mundo 68. Sin embargo, para que estos compuestos logren cumplir su propósito terapéutico y superar los infortunios clínicos, se necesitan más estudios para comprender sus mecanismos de acción (ver Figura 1) y reducir el potencial de citotoxicidad no deseado, así como conferirles más resistencia a la degradación por proteasas, mejorar la vida media en sangre periférica y generar un medio de fabricación a gran escala consistente y rentable.
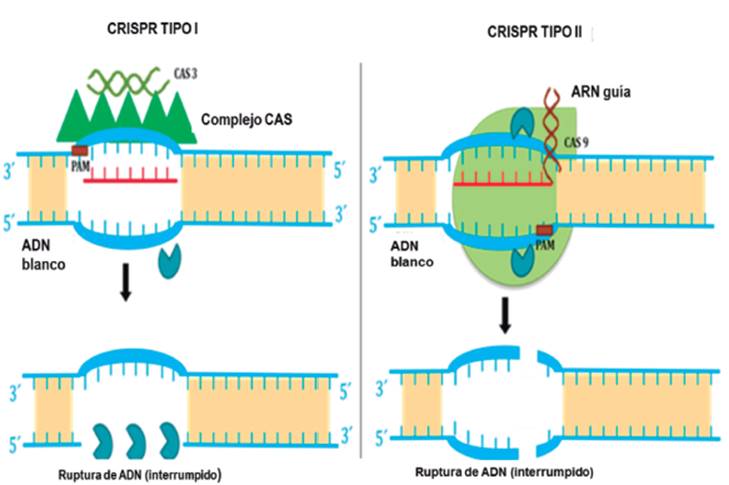
Figura 1 Características generales del CRISPR tipo I y tipo II. Imagen modificada a partir de http://www.dciencia.es/crispr-antimicrobianos/
Muchos de los PAMs ya han sido probados como terapia profiláctica y agentes terapéuticos contra las biopelículas tanto in vitro como in vivo, incluyendo bacterias de lento crecimiento y de no-crecimiento. Su acción en las diferentes etapas en la formación de la biopelícula y la baja frecuencia de selección de cepas resistentes son características muy atractivas para su uso generalizado. Por otro lado, la utilización como agentes inmunomoduladores de la respuesta inmune innata, al impulsar la resolución de la infección a través de la estimulación de la propia inmunidad del hospedador 69,70, mientras al mismo tiempo, controlan los potenciales daños pro-inflamatorios.
Alternativas terapéuticas no convencionales
Las terapéuticas no tradicionales que están resurgiendo son generadas en su gran mayoría a partir de técnicas de ingeniería genética y biología sintética. La exploración de nuevas alternativas a los antibióticos convencionales se ha convertido en un importante objetivo de investigación, debido al incremento en la tasa de multirresistencia de patógenos bacterianos, especialmente en entornos hospitalarios. Así mismo, ha habido una disminución paulatina en el descubrimiento de nuevos compuestos antibióticos 70.
La terapia con fagos se refiere a la utilización de bacteriófagos (virus que infectan bacterias), para tratar infecciones bacterianas 71. Los bacteriófagos son muy abundantes y se cree que cada bacteria tiene sus propios virus específicos que podrían utilizarse como agentes antibacterianos 72,73. Históricamente, los fagos se utilizaron terapéuticamente a principios del siglo XX 74. Sin embargo, el descubrimiento de antibióticos ampliamente eficaces llevó al detenimiento del desarrollo de la terapia fágica en los países occidentales y sólo cuando los antibióticos empezaron a mostrar inconvenientes, se retomó el desarrollo de dicha herramienta. Sin embargo, la segunda venida de la terapia fágica enfrenta desafíos relacionados con las estrictas pautas regulatorias y el desarrollo de prácticas terapéuticas efectivas 74.
Es importante señalar que los fagos infectan bacterias de manera muy selectiva. A menudo, la estrecha gama de huéspedes se considera una ventaja sobre los antibióticos tradicionales, ya que el tratamiento con fagos puede centrarse con precisión en el patógeno, sin dañar, por ejemplo, la flora bacteriana comensal. Sin embargo, las bacterias pueden desarrollar resistencia rápidamente también a fagos, por lo que el efecto antibacteriano conseguido puede ser transitorio. Cuando se usa un conjunto de fagos diferentes simultáneamente en un cóctel de fagos, el desarrollo de resistencia es menos probable. Sin embargo, es difícil obtener un conjunto de fagos que sea eficaz contra todas las variantes de un patógeno determinado 75.
Puede existir una compensación entre la gama de dianas bacterianas y la eficacia terapéutica de un cóctel de fagos para una especie específica de bacteriana. Esto se debe a que cuando el número de fagos en un coctel se incrementa en un intento por ampliar el número de bacterias target, el número de fagos para una cepa específica microbiana puede ser reducida 76. Por lo tanto, la especificidad de los fagos a células microbianas, aunque en teoría es beneficioso, plantea un problema práctico cuando se combina para tratar fenotipos resistentes que emergen rápidamente. Por esta razón, el uso terapéutico de fagos se considera como una posible alternativa a los antibióticos convencionales 76.
Repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas o CRISPR (clustered regularly interspaced short palindromic repeats).
Las secuencias CRISPR, que se encuentran en un 40% de las bacterias y en el 90% de las arqueas, consiste en otro mecanismo de defensa que los procariotas han desarrollado para protegerse de los agentes virales. Funciona como un sistema inmune adaptativo que memoriza infecciones previas integrando secuencias cortas de genomas invasores denominados espaciadores en el locus CRISPR. Está constituido por repeticiones palindrómicas cortas agrupadas y espaciadas entre sí, junto con sus proteínas asociadas que hacen este sistema funcional (Cas) 77. Estas proteínas Cas9, tienen la capacidad de cortar con precision el ADN basado en una secuencia de ARN guía.
Existen dos clases de sistemas CRISPR-Cas dependiendo de sus módulos efectores y del tipo de nucleasa asociada. Los sistemas CRISPR-Cas de clase 1 se caracterizan por un módulo efector con múltiples subunidades de ARNcr e incluyen los sistemas CRISPR-Cas de tipo I, III y IV. Todos los sistemas CRISPR-Cas de tipo I contienen el gen Cas3 o su variante Cas3', la cual codifica una helicasa. Frecuentemente, el dominio helicasa se fusiona con un dominio endonucleasa situado en la proteína Cas3 que es responsable de la ruptura del ADN diana (ver Figura 1).
Los sistemas CRISPR-Cas de tipo III poseen el gen Cas10, que codifica para proteínas que muestran una gran variación de secuencia. Todos los loci de tipo III, al igual que los de tipo I, codifican para una proteína Cas5 y varias proteínas Cas7. Los genomas bacterianos también poseen los sistemas de tipo IV que por lo general se presentan en plásmidos y poseen una pequeña subunidad csfl única 78.
Los sistemas CRISPR-Cas de clase 2 cuentan con un módulo efector de una sola subunidad de ARNcr. Esta clase incluye los sistemas de tipo II y V. Los tipo II son los más simples en cuanto al número de genes. El gen característico de este es Cas9 (ver Figura 1), el cual codifica una proteína multidominio que combina las funciones de complejo efector de ARNcr con la de escisión del ADN blanco.
Debido a su menor complejidad el sistema CRISPR-Cas de tipo II es el que se utiliza en edición dirigida de genes, ya que su actividad depende de una sola proteína Cas9 de tipo endonucleasa que funciona con el ARNcr y el ARNtracr, formando un complejo que va examinando el ADN invasor para encontrar una secuencia diana de unos 20 nucleótidos, al igual que el correspondiente AMP, y entonces corta dicha secuencia 79-80.
El sistema CRISPR-Cas9 abrió nuevos campos de investigación en la edición del genoma en una amplia gama de células y organismos con alto potencial en diversas aplicaciones, que incluyen la generación de materiales sintéticos, tales como las diatomeas de sílice derivadas de algas para la administración oral de fármacos y la generación de precursores de drogas en bacterias reduciendo significativamente el costo y facilitando la accesibilidad de terapias útiles. Por otra parte, se está probando el diseño de antimicrobianos basados en nucleasas CRISPR-Cas programables, contra Staphyloccus aureus resistentes a antibióticos. Es así como construyendo fagémidos contra los plásmidos pUSA01, pUSA02 de la cepa USA 300 de S. Aureus se consiguió que el 99,99% de las bacterias fueran sensibles a la tetraciclina, debido a la pérdida de pUSA02; es decir, que el uso de esta nucleasa Cas9 específica contra una bacteria altamente resistente, no elimina a la bacteria, pero sí reduce su actividad virulenta, al disminuir el contenido plasmídico en una población bacteriana. De esta manera el sistema CRISPR, podría usarse para eliminar la resistencia a antibióticos de bacterias patógenas, causantes de infecciones asociadas a la atención en salud, como en ambientes comunitarios 81-82.
Conclusiones
La resistencia microbiana ha llevado a la búsqueda de innovadoras alternativas para su contención y dentro de las más promisorias están el uso de péptidos, no sólo debido a sus características intrínsecas antimicrobianas, sino también a las interacciones sinérgicas y antagónicas que presenta con otros mediadores inmunológicos. Estas propiedades han permitido crear péptidos reguladores de defensa innata que representan un nuevo enfoque inmunomodulador para el tratamiento de infecciones. Sin embargo, a pesar de los múltiples intentos ensayados, sólo los diseñados con alto score antimicrobiano han demostrado eficacia en estudios clínicos de fase 3. No obstante, dado su excepcional amplio espectro de actividad, un único péptido puede tener actividad contra bacterias Gram negativas, Gram positivas, hongos incluso virus y parásitos, aumentando el interés por investigar estas dinámicas moléculas. Muchos de estos péptidos han sido probados en terapias profilácticas y como agentes antibiopelícula, inhibiendo su formación in vitro e in vivo, su efecto prometedor y la poca resistencia que se ha reportado son características importantes para considerarlos por sí solos y en sinergia con antibióticos comunes, como una alternativa ante la baja eficiencia actual de los antibióticos.
Por otra parte, la terapia con fagos, usando coctel para aumentar el espectro de acción de estos y el sistema CRISPR, que permite la edición de genomas bacterianos, permitiría reducir su actividad virulenta y diseñar antimicrobianos basados en nucleasas CRISPR-Cas programables contra dianas específicas, lo que permite pensar que representan un promisorio camino en el estudio de nuevas alternativas con alto potencial para eliminar la resistencia a antibióticos de bacterias altamente patógenas.
En nuestros días quizás no sea exagerado afirmar que estamos a punto de sucumbir nuevamente en la era pre-antibiótica, ante la imposibilidad de saber el tiempo que nos tomará conocer y descubrir nuevas herramientas antimicrobianas efectivas y oportunas. No obstante, "para no morir en el intento del ataque bacteriano" 81, es imperante entender como lo menciona el proverbio chino, que «matar al ladrón no es cerrar la puerta» y que sólo nos queda limitar la derrota, porque mientras se usen como principal medio de contención, tratamientos convencionales antimicrobianos, incluso de manera correcta, la microevolución en las bacterias, se asegurará de seguir su curso.














