Introducción
La resistencia bacteriana a antibióticos constituye un grave problema de salud pública que cada vez se hace más complejo de tratar. La problemática se agudiza aún más con la aparición de organismos ambientales y de la microbiota humana portadoras de resistencia 1-3. Entre las cepas de Streptococcus del grupo viridans (EGV) se ha detectado una prevalencia creciente de resistencia a macrólidos, β-lactámicos y clindamicina 4-6. Otro grupo importante de bacterias que fácilmente desarrollan resistencia a los antibióticos lo constituye el grupo de bacilos Gram negativos de la familia Enterobacteriaceae. Este grupo tiene capacidad de colonizar y proliferar en la cavidad bucal haciendo parte de la microflora transitoria 2,3,7-9.
Numerosos estudios señalan que las bacterias de la microbiota oral permanente y transitoria adquieren genes de resistencia en gran medida debido a la transferencia intermicrobiana a partir de patógenos a la flora normal 10-12. La elevada exposición de los EGV y de los bacilos Gram negativos a la penicilina y otros antibióticos β-lactámicos ha producido las condiciones ideales para la adquisición de enzimas β-lactamasas que alteran y confieren la resistencia a estos antibióticos y son codificadas por los genes bla11,13. La antibioterapia con antibióticos de amplio espectro, como la eritromicina, también ha ejercido una presión selectiva sobre las bacterias orales. La resistencia a este antibiótico está mediada por la alteración del sitio de unión de la eritromicina al ribosoma por metilación de una enzima codificada por el gen emr, que induce un cambio conformacional que impide la unión a su lugar de acción, tanto de los macrólidos como de las lincosamidas y estreptograminas B. Este patrón fenotípico se denomina resistencia MLSB. La expresión del gen erm puede ser constitutiva o inducible 14,15. Otros mecanismos de resistencia a la eritormicina lo constituyen la inactivación del antibiótico por la adición de un grupo fosfato al hidroxilo de posición 2' del amino azúcar por la acción de las fosfotranferasas codificadas por los genes mphA. También se reporta una resistencia mediada por bombas de eflujo codificadas por los genes mefA y mefE 16.
El propósito del presente estudio fue investigar la prevalencia de bacilos entéricos Gram negativos resistentes a los antibióticos β-lactámicos y de los EGV con resistencia a eritromicina, y determinar si los genes que confieren resistencia a estos antibióticos son detectados en la cavidad oral sana de pacientes que consultaron una clínica odontológica de la ciudad de Cali durante el año 2018.
Métodos
Se realizó un estudio descriptivo de corte transversal, por lo cual se tomaron muestras de saliva y de placa dental de 178 adultos (96 mujeres y 82 hombres) con un promedio de edad de 44 +13.56 años, que asistieron la Clínica Odontológica de la ciudad de Cali y sometidos a profilaxis oral. Se incluyeron pacientes que no habían recibido tratamiento con antibióticos en los últimos 3 meses y no sufrían de alguna enfermedad oral. Una muestra de saliva (2 ml) se recolectó en un recipiente estéril y las muestras de la placa dental se tomaron de frotis con un hisopo estéril de alginato de calcio. En el caso de las muestras de placa subgingival se tomaron de cuatro sitios diferentes con puntas de papel estériles y se agruparon en 4 ml de solución de Ringer y se mezclaron con la muestra de saliva.
Métodos de Microbiología Clásica Aislamiento de bacterias entéricas y de EGV e identificación
Las muestras se resuspendieron en caldo Todd-Hewitt (Oxoid) y se sembraron en cada medio. Para el aislamiento de bacterias entéricas las diluciones se sembraron en agar Mc Conkey (Oxoid, Hampshire, United Kingdom) por 24 horas a 37oC. El estudio microscópico se basó por el método de Gram para establecer morfología y tinción bacteriana y la posterior identificación se realizó con pruebas bioquímicas empleando el sistema automatizado Vitek GNI+ (bioMeriex Vitek Inc., Hazelwood, MO).
Para aislar los EGV se sembraron las muestras en medio selectivo agar mitis salivarius y agar chocolate base tripticasa-soya y se incubaron en una atmósfera de CO2 durante 2 días a 37°C. Se seleccionaron las bacterias por su morfología celular con coloración de Gram y la identificación bioquímica se realizó por el sistema Api 20 Strep (Biomerieux). La confirmación de las cepas del grupo viridans se realizó por el método de dilución en caldo por Microscan System (Becton Dickison), la cual emplea un panel de 27 pozos para la identificación bioquímica con pruebas como pruebas cristal violeta, nitritos, Novobiacina, PGR, B-D Glucoronidasa, Indol Fosfatasa, Voges Proskawer, Optoquina, Fosfatasa, Bilis Esculina, Pirridolina, Arginina, Galactosidasa, Urea, Manitol, Lactosa, Trehalosa, Mannosa, Cloruro De Sodio, Sorbitol, Arabinosa, Ribosa, Inulina, Raffinosa, Bacitracina y Piruvato.
Estudio de sensibilidad a los antibióticos y detección fenotípica de la producción de β-lactamasas
La evaluación de la sensibilidad antimicrobiana de las bacterias entéricas se realizó mediante el método de difusión del disco sobre placas con agar Müller Hinton (Oxoid Ltd., Hampshire, United Kingdom) utilizando un estándar de 0.5 Mcfarland. Los sensidiscos correspondieron a los siguientes antibióticos: cefotaxima (CTX 30 µg), ceftazidime (CAZ 30 µg), cefepime (FEP 30 µg), cefoxitina (FOX 75 µg), ceftriaxona (CRO 30 µg), piperacillina-tazobactam (TZP 10 µg/30 µg), aztreonam (ATM 30 µg), imipenem (IMP 10 µg), meropenem (MEM 10 µg), gentamicina (GEN10 µg), trimetoprim/sulfametoxazol (SXT 25 µg), levofloxacina (LEV 5 µg) (Oxoid Ltd., Hampshire, United Kingdom) de acuerdo con los lineamientos establecidos por el Clinical and Laboratory Standards Institute (CLSI) 17.
La susceptibilidad intermedia a cada antibiótico se consideró como resistencia. El control de calidad se realizó utilizando cepas estándar de Escherichia coli (ATCC 25922) y Pseudomonas aeruginosa (ATCC 27853).
Los aislamientos que fueron resistentes a aztreonam, cefotaxime, ceftazidime ó ceftriaxona fueron sometidos a la confirmación de presencia de β-lactamasa de espectro extendido (BLEE) mediante la prueba de sinergia de doble disco. En esta prueba, se colocó un disco de amoxicilina/ácido clavulánico (20 µg/10 µg) en el centro de la placa y a 20mm (centro a centro) lejos de un disco de aztreonam (30 µg) y de un disco de ceftazidima (30 µg). La placa se incubó a 35 °C durante 24 horas. La prueba se consideró positiva cuando hubo un aumento o distorsión de la zona de inhibición entre cualquier marcador antimicrobiano y el disco de amoxicilina/ácido clavulánico.
Los aislamientos que fueron sensibles a la cefoxitina en el antibiograma se sometieron a la prueba confirmatoria de β-lactamasa cromosómica inducible, AmpC, en el que se colocó un disco de cefoxitina (30 µg) en el centro de la placa, a 20mm (centro a centro) de un disco de ceftriaxona (30 µg) y de un disco de ceftazidima (30 µg). La placa se incubó a 35 °C durante 18-24 h. La cefoxitina funciona como un inductor de la enzima AmpC y la lectura se consideró positiva cuando se observó achatamiento del halo alrededor de la ceftriaxona y/o el disco de ceftazidima.
Análisis fenotípico de la resistencia a los antibióticos de los EGV
Para los EGV se evaluó la concentración inhibitoria mínima (CIM) de penicilina (PEN), ceftriaxona (CRO), cefotaxime (CTX), cefepime (FEP), imipenem (IPM), tetraciclina (TET), levofloxacina (LEV), vancomicina (VAN), eritromicina (ERY), clindamicina (CLI), linezolid (LZD) y gentamicina (GEN) (Oxoid Ltd., Hampshire, United Kingdom) por dilución seriada en agar Mueller-Hinton suplementada con un 5% de sangre de carnero según los parámetros de CLSI 18.
El fenotipo de resistencia a macrólidos se determinó mediante discos comerciales de eritromicina (15 µg) y clindamicina (2 µg) (Oxoid Ltd., Hampshire, United Kingdom) colocados a una distancia de 12mm sobre placas de agar Mueller-Hinton (Oxoid Ltd., Hampshire, United Kingdom) suplementada con un 5% de sangre de carnero e incubados en una atmósfera de CO2 a 37oC durante 2 días.
La resistencia en ambos discos se consideró una resistencia constitutiva (cMLSβ), mientras que el achatamiento de la zona de inhibición del disco de clindamicina en la zona próxima al disco de eritromicina indicó resistencia inducible (iMLSβ). La sensibilidad detectada a clindamicina sin achatamiento en la zona de inhibición se consideró un fenotipo M. El fenotipo L se definió cuando se observó resistencia en el disco de clindamicina y sensibilidad a eritromicina.
Aislamiento de DNA de las cepas referencia y de los aislados bacterianos
El DNA de las bacterias entéricas y los EGV se obtuvo empleando el kit "Easy-DNATM" (Invitrogen, life technologies). La calidad y concentración del ADN se determinó por espectrofotometría usando el equipo nano-drop (Thermo 2000®).
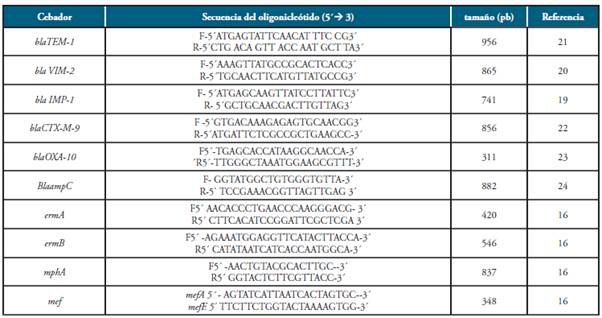
Amplificación por PCR de los determinantes genéticos que confieren resistencia a los antibióticos
Para la identificación de genes bla se amplificó 5 a 101~| g de ADN genómico con 2,5 pmol de cada cebador para amplificar los genes blaIMP-118, blaVIM-219, blaTEM-1 20, blaCTXM-9 21, blaOXA-10 22 y blaampC 23 y se mezcló con 0,2mM dNTPs (BioLine®), 1,5mM de MgCl2 y 0,6 U de Taq polimerasa (Invitrogen®) en un volumen final de 25 µl. Las condiciones de amplificación fueron: denaturación a 94oC por 4 minutos, seguidos por 30 ciclos a 94oC segundos, extensión a 72oC por 1 minuto y una extensión final a 72°C por 10 minutos. La temperatura de alineamiento para los genes blaCTX-M-9 y blaOXA-10 fue de 60oC.
La amplificación por PCR de los determinantes de resistencia a eritromicina se realizó empleando los conjuntos de cebadores ermA, ermB, mef y mphA de acuerdo con el protocolo establecido por Sutcliffe et al 14. La secuencia de los cebadores se encuentra en la Tabla 1.
Se realizó una electroforesis en geles de agarosa al 1,2% para distinguir los productos de la PCR. El tamaño de cada producto de PCR se estimó utilizando marcadores de peso molecular estándar (escala de ADN de 1kb de Gibco BRL, Gaithersburg, Md.) y se reveló empelando SYBR Green (Bio-Rad) bajo luz UV en el transiluminador.
Análisis estadísticos de los resultados
La frecuencia de aislamientos con resistencia a los antibióticos se calculó como la pre-valencia de pacientes con aislamientos resistente a los antibióticos. La diferencia en la frecuencia de las variables y su asociación fueron determinadas por análisis de X2, un valor de p ≤ 0,05 se consideró estadísticamente significativo. Para realizar este análisis se empleó el paquete estadístico SPSS Vs.23 (Inc, Chicago, IL).
Consideraciones éticas
Este estudio fue respaldado por el Comité de la Clínica Odontológica (Ley Núm. 05-013). Todos los pacientes estaban suficientemente informados sobre los objetivos y procedimientos. Se obtuvieron muestras y datos después de firmar el consentimiento informado por escrito. El estudio cumplió con las disposiciones de la Declaración de Helsinki y las normas científicas técnicas y administrativas para la investigación en salud mencionadas en la "Resolución 8430 de 1993" del Ministerio de Salud de Colombia.
Resultados
Un total de 193 aislamientos fueron obtenidos, 59 aislamientos correspondieron a bacilos entéricos y 134 fueron EGV.
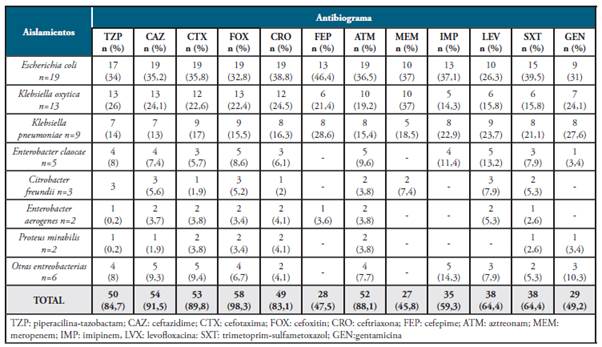
Entre los bacilos entéricos se encontró que el mayor número de aislamientos correspondió a E. coli con el 32,2% (19/59), seguido por Klebsiella oxytoca con el 22% (13/59) y K. pneumoniae con el 15,3% (9/59). También se aisló Enterobacter claocae, Citrobacter freundii, E. aerogenes y Proteus mirabilis en menor proporción (ver Tabla 2).
Tabla 2 Perfíl de sensibilidad a los antibióticos en los aislamientos de bacilos entéricos de cavidad oral. n=59.
Fuente: Autores.
Análisis de los perfiles de sensibilidad en los aislamientos de bacilos entéricos
Más del 47% de los aislamientos de los bacilos entéricos presentaron resistencia a los antibióticos evaluados. Cifras cercanas al 90% o más altas se encontraron de aislamientos resistentes a cefalosporinas de tercera generación como cefoperazona (98,3%), cefoxitin (91,5%) y cefotaxima (89,9%) y a aztreonam (88%). Los aislamientos resistentes a carbapenemes y a cefalosporinas de cuarta generación estuvieron por encima del 45% (ver Tabla 2). E. coli presentó el mayor número de aisalmientos con resistencia especialmente a cefepime (46,4%), ceftriaxona (38,8%) y carbapenemes (37,1%). Los aislamientos de K. oxytoca y K. pneumoniae mostraron también resistencia a los antibióticos β-lactámicos, especialmente a las cefalosporina y carbapenemes como se observa en la Tabla 2.
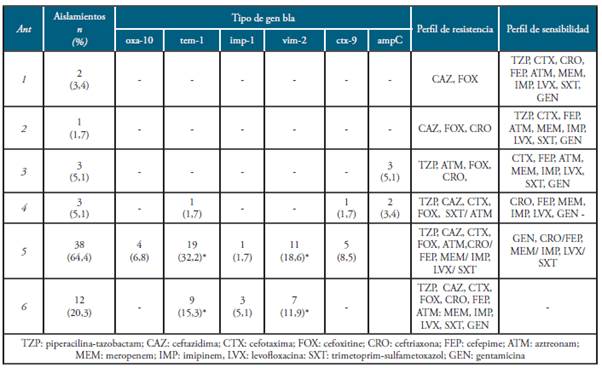
La Tabla 3 muestra los resultados del análisis de la prueba de sensibilidad a los antibióticos. Se determinaron 6 perfiles de sensibilidad (antibiotipos) entre los bacilos entéricos.
Tabla 3 Perfíl de sensibilidad a los antibióticos en los aislamientos de bacilos entéricos de cavidad oral. n=59.
Fuente: Autores.
De acuerdo con la definición propuesta por Magiorakos et al 24, la resistencia a tres o más clases de antibióticos se definió como multirresistente (MDR). La mayoría de los bacilos entéricos presentan perfiles de MDR a los antibióticos; así, el 64,4% de los aislamientos se ubicaron en el antibiotipo 5, con aislamientos MDR resistentes a 6 o 7 antibióticos. En este grupo se identificaron 15 aislamientos de E. coli y 9 aislamientos de K. oxytoca. Sin embargo, el antibiotipo 6 agrupó los bacilos con resistencia a todos los antibióticos evaluados (20,3%). En este caso, seis aislamientos correspondieron a E. coli, cuatro a K. oxytoca y dos a K. pneumaniae.
Los antibiotipos 3 y 4 con perfiles de resistencia a 4 y 5 antibióticos, respectivamente se determinaron en el 5,1% de los aislamientos. Los aislamientos con los antibiotipos 1 y 2 fueron lo menos prevalentes.
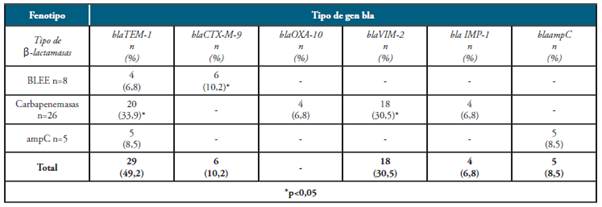
En este estudio se detectaron fenotípicamente 39 aislamientos portadores de β-lactamasas. Esta detección tuvo en cuenta tres tipos de estas enzimas: β-lactamasas de espectro extendido (BLEE), β-lactamasas del tipo AmpC y carbapenemasas.
Los aislamientos que presentaron el fenotipo compatible con BLEE correspondieron al 20,5% (8/39) y el fenotipo específico BLEE tipo CTX (detectado en seis aislamientos con resistencia a cefotaxima) correspondió al 15,4%.
La β-lactamasa tipo AmpC se encontró en el 12,8% (5/39) de los aislamientos, dos de ellos en K. Oxytoca, otros dos en K. pneumoniae y uno de E. aerogenes. Sin embargo, el 66,7%, de los aislamientos presentaron el fenotipo carbapenemasas, representados principalmente por E. coli (17/39) y K. pneumoniae (7/39) (ver Tabla 4).
Detección de los genes bla
La distribución de los genes bla entre los antibiotipos y los fenotipos detectados en los aislamientos de los bacilos entéricos se encuentra en la Tabla 3 y 4, respectivamente.
El mayor número de aislamientos presentaron el blaTEM-1 (49,2%). Este gen codifica para una β-lactamasa de amplio expectro (BLEA).
Es importante destacar que en el 30,5% de los aislamientos se detectó el gen bla-VIM-2, el cual codifica por una Metalo-β-lactamasa y confiere resistencia a carbapenemes. En concordancia con este resultado, se observó que la presencia de este gen fue significativa entre aquellos aislamientos que presentaron el fenotipo carbapenemasa (p<0,05).
En este estudio también fue significativa la presencia de blaCTX-M-9 en los aislamientos con el fenotipo BLEE (ver Tabla 4).
Los genes bla se detectaron sólo en aquellos aislamientos con un fenotipo MDR. Los bacilos entéricos con el antibiotipo 5 presentaron todos los genes bla, excepto el blaampC, siendo el gen blaTEM-1 con el 32,2%, el más abundante, seguido del gen blaVIM-2 (18,6%). Los bacilos entéricos con el antibiotipo 6 presentaron también abundancia de genes bla. El gen blaampC fue determinado sólo en los bacilos con los antibiotipo 3 y 4 (ver Tabla 3).
Al establecer la concordancia de los genes bla con el fenotipo determinado en la Tabla 4, encontramos que el 10,2% de los bacilos entéricos con el fenotipo BLEE presentaron el gen blaCTX-M-9 y el 6,8% blaTEM-1.
Todas las cepas con el fenotipo AmpC presentaron los genes blaTEM-1 y blaampC. En el caso de los bacilos entéricos con carbapenemasas, se encontró que la resistencia a estos antibióticos se debió principalmente por la presencia de genes que codifican para las metalo enzimas blaVIM-2 (30,5%) y blaIMP-1 (6,8%), siendo significativa la presencia de cepas con el gen blaVIM-2 (p<0,05). En el 6,8% de estas cepas se determinó que la resistencia a los carbapenemes estaría mediada por el gen blaOXA-10. Algunas cepas presento más de un gen bla.
Análisis fenotípico y molecular de los aislamientos de los EGV
Entre los aislamientos de los EGV, el S. salivarius fue el más común, seguido de S.anginosus, S. mitis, S. sanguinis, S. bovis, y S. mutans.
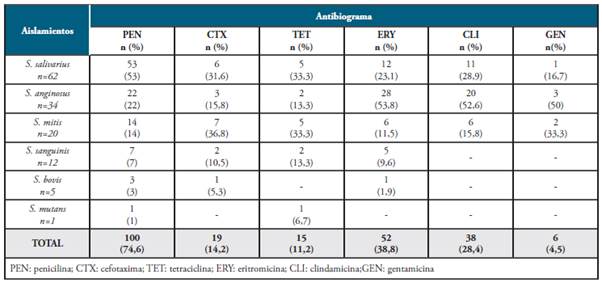
El 74,6% (100/134) fueron resistentes a penicilina, 14,2% (19/134) a ceftriaxona, 4,5% (6/134) a gentamicina, 11,2% (15/134) a tetraciclina, 38,8% (52/134) a eritromicina y el 28,4% (38/134) a clindamicina. El resto de los antibióticos evaluados presentaron 100% de actividad contra los aislamientos de EGV (ver Tabla 5).
Tabla 5 Perfil de sensibilidad a los antibióticos en los aislamientos de los Streptococcus del grupo viridans. n=134.
Fuente: Autores.
S. salivarius presentó el mayor número de aislamientos con resistencia a penicilina (53%) y S. anginosus a eritromicina y clindamicina (53,8% y 52,6%, respectivamente).
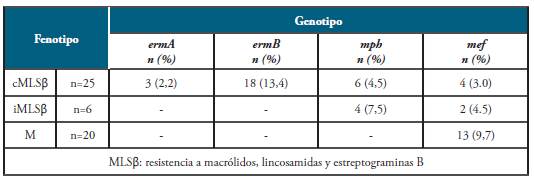
Los resultados de la evaluación fenotípica de la resistencia a eritromicina y clindamicina fueron los siguientes: 18,7% de los aislamientos (25/134) presentaron un fenotipo cMLSβ de resistencia constitutivo, el 4.5% (6/134) un fenotipo iMLSβ inducible, el 14,9% (20/134) un fenotipo M. Aislamientos de EGV con el fenotipo L no fueron detectados.
En los aislamientos cMLSβ, el gen ermB se determinó con mayor frecuencia (18/25), tres de estos aislamientos presentaron también el gen ermA y otros tres aislamientos el genes mef. Sin embargo, seis aislamientos cMLSP presentaron únicamente el gen mph y un aislamiento el gen mef.
Entre los aislamientos iMLSβ se determinó los genes mph y mef. Entre los aislamientos con el fenotipo M, sólo se logró determinar el gen mef en el 9,7% (13/134) de ellos como se muestra en la tabla 6.
Discusión
Los bacilos entéricos Gram negativos pueden habitar la cavidad oral bajo condiciones fisiológicas especiales. En este estudio, los aislamientos entéricos obtenidos de la saliva y placa dental de sujetos sanos fueron del 33,1%. Este valor fue superior al 23,3% y 18,7% encontrado en un estudio similar realizado con estudiantes 8 y trabajadores de un hospital en Sao Paulo-Brasil 9, respectivamente.
Bryskier et al, sugieren que la prevalencia de los bacilos entéricos en la boca variaba de acuerdo a las características de los individuos: en pacientes con dentadura postiza tienen una prevalencia hasta del 48%, en pacientes con hiatosis del 27,1%, en los pacientes de ortodoncia del 13% y en pacientes con una cavidad oral sana del 16,4% 10. Según este resultado, nuestros hallazgos sólo fueron menores que en el caso de los sujetos con dentadura postiza.
Ferreira et al, establecen que la colonización de este nicho por los bacilos entéricos comienza tempranamente. Los recién nacidos presentan un 8% de colonización por estas bacterias, siendo Enterobacter spp. la más prevalente y en algunas cepas ya se detecta resistencia, especialmente a ampicilina, gentamicina y cefalotina 25.
En este estudio se evidenció diversidad de especies de Enterobacteriaceae en la cavidad oral de personas sin enfermedad dental con resistencia a los antibióticos β-lactámicos por encima del 80% de prevalencia, especialmente a las cefalosporinas de tercera generación, aztreonam y piperacilina/tazobactam, siendo potenciales fuentes de infecciones oportunistas como lo han señalado algunos investigadores 7,26,27.
El estudio desarrollado en jóvenes estudiantes en Brasil encontró que el 57,8% y 45,3% de los bacilos entéricos mostraron resistencia a la amoxicilina/ácido clavulánico y cefoxitina, respectivamente; y fueron 100% sensibles a cefepime, ciprofloxacina, gentamicina, imipenem, meropenem y levofloxacina 8. En forma similar, Gaetti-Jardim et al reportan niveles significativos de resistencia para todos los β-lactámicos, excepto para imepenem y meropenem, que respectivamente presentaron 2,3% y 1,6% de resistencia 28.
Los resultados presentados en la Tabla 2 muestran que los carbapenemes y cefepime fueron los únicos β-lactámicos que tuvieron una actividad antimicrobiana en menos del 55% de los aislamientos. La resistencia al imepenem fue levemente más alta a la observada al meropenem.
Los antibióticos β-lactámicos constituyen los agentes antimicrobianos más tradicionales empleados en el tratamiento de infecciones de todo tipo en Colombia, esto explicaría la alta frecuencia de enterobacterias resistentes halladas en este estudio. El aumento de la resistencia a estos antibióticos se debe a la producción de enzimas β-actamasas principalmente. En este estudio encontramos que el 66,1% de los bacilos entéricos presentaron el fenotipo β-lactamasas, detectándose, tres tipos de ellas: BLEE, AmpC y carbapenemasas.
Los aislamientos productores de BLEEs detectados en la cavidad oral sana representaron el 13,6%. La presencia de estas cepas es un problema importante en infecciones adquiridas en pacientes hospitalizados y en la comunidad porque producen desde una infección urinaria no complicada adquirida en la comunidad hasta sepsis nosocomial potencialmente mortal.
Los aislamientos con el fenotipo AmpC representaron el 8,5% y fueron resistentes a la cefoxitina, lo que indica que son organismos mutantes y producen constantemente esta enzima independientemente de la exposición a un antibiotico inductor. Las infecciones debidas a este tipo de cepas, son difíciles de tratar debido al perfil MDR que presentan frente a los antibióticos β-lactámicos comúnmente empleados 29.
Es importante destacar que el más alto número de aislados fueron resistentes a los carbapenemes (44,1%). Este tipo de antibiótico se emplea como última elección para el tratamiento de infecciones complicadas en las cuales los otros antibióticos β-lactámicos no han sido efectivos. Los genes encontrados entre los aislados resistentes a los carbapenemes fueron aquellos que codifican para β-lactamasas de la clase B, tipo IMP y VIM.
Otro hallazgo importante fue la alta prevalencia del fenotipo MDR (84,7%) entre los aislamientos agrupados con los antibiotipos 5 y 6. Además, estos aislamientos fueron portadores de genes bla, con predominio significativo de los genes blaTEM-1, y bla-VIM-2.
Los reportes señalan que en la cavidad oral y en el intestino del humano predominan principalmente β-lactamasas de la clase A, clase C y clase D; mientras que las β-lactamasas de la clase B son más representativas de la microbiota del suelo 1,3. Sin embargo, en este estudio encontramos un alto predominio de genes bla que codifica para β-lactamasas clase B.
Estos resultados evidencian que la cavidad oral sana es un reservorio de cepas bacterianas capaces de producir diferentes tipos de β-lactamasas y potenciales fuentes de genes bla que pueden propagarse a otras poblaciones como lo han establecido ya otros autores 11,12,14,15.
En el caso de los aislamientos de EGV recuperados en el período de estudio, estos presentaron resistencia a algunos antibióticos β-lactámicos como la penicilina y ceftriaxona. También fue importante el número de aislamientos con resistencia a la eritromicina (38,8%) y clindamicina (28,4%). Estos resultados son concordantes con los reportados por otros estudios, con resistencias que van desde un 35%-70% para macrólidos y del 10-15% para penicilinas (naturales o sintéticas) y clindamicina 4-6.
La alta resistencia a los macrólidos entre los aislamientos de EGV se asocia frecuentemente a una alta resistencia a las tetraciclinas, la clindamicina y los azálidos 5,6.
En este estudio encontramos una correlación entre el fenotipo de resistencia a la eritromicina y el genotipo. Todos los aislamientos con el fenotipo cMLSβ presentaron los genes erm, principalmente el gen ermB lo que evidencia que la resistencia a eritromicina es mediada principalmente por metilación. En los Streptococcus spp. diversos investigadores han encontrado una relación importante entre la presencia del gen ermB y la expresión del fenotipo MLSB constitutivo 16,30,31.
En relación al gen ermA es frecuente entre los aislamientos de Staphylococcus aureus resistente a eritromicina, con prevalencia mayor al 80% 32. Esto indicaría, quizás, que el gen ermA detectado en algunos aislamientos de EGV podrían ser fuente de S. aureus presentes en la cavidad oral de estas personas.
Es de destacar que entre los aislamientos iMLSβ, cMLSβ y principalmente en los M, la resistencia a eritromicina se debió también a bombas de eflujo codificadas por el gen mef.
Los reportes indican el aumento en la expresión de la bomba de flujo codificada por este gen confiriendo resistencia a la acriflavina, aminoglucósidos, glicilciclina, macrólidos y β-lactámicos 33,34.
El tratamiento de la infección odontogénica genera una alta prescripción de antibióticos en la comunidad, aumentando la posibilidad de tener microrganismos resistentes. Sin embargo, algunos investigadores sugieren que la alta diversidad de organismos resistentes y la abundancia de genes de resistencia en la cavidad oral estaría siendo influenciada principalmente por factores ambientales generados durante los cambios estacionales y el tipo de alimentación como se ha demostrado en individuos japoneses 35, de países del suroeste de Europa como España, Francia e Italia 36 y Latinoamérica 37-38.
Conclusión
En este estudio se demostró la presencia de EVG y bacilos entéricos Gram negativos resistentes a los antibióticos y portadores de genes resistentes a eritromicina y genes bla, respectivamente. La presencia de estas bacterias en la cavidad oral sana representa un riesgo no solo para la salud de los individuos portadores, sino que contribuye a la creciente epidemia de resistencia bacteriana por varias implicaciones: 1) pueden considerarse como reserva genética a partir de la cual se puede transferir factores de resistencia a otras bacterias más patógenas; 2) tienen la potencialidad de producir enfermedades sistémicas difícil de tratar por el carácter de multirresistencia que presentan; y 3) tienen la potencialidad de desarrollar infecciones nosocomiales, si estos pacientes requieren en algún momento ser hospitalizados.