Introducción
En una línea de tiempo que llega hasta la actualidad, una epidemia de casos de infecciones respiratorias bajas detectadas en Wuhan se informó a la oficina de la OMS en China, en diciembre de 2.019 1. El Centro Chino para el Control y Prevención de Enfermedades (CDC) y los CDC locales organizaron un programa intensivo de investigación de brotes 2.
La etiología de esta enfermedad, denominada enfermedad por coronavirus 2019 (COVID-19), se atribuye a un nuevo virus perteneciente a la familia del coronavirus (CoV) llamado SARS-CoV-2 3. Este nuevo virus es altamente contagioso y se ha expandido rápidamente por todo el mundo. El brote fue declarado por la Organización Mundial de la Salud (OMS) como Emergencia de Salud Pública de Preocupación Internacional 4. El 11 de marzo, cuando el número de casos de COVID-19 fuera de China aumentó 13 veces y el número de países involucrados se triplicó con más de 118.000 casos en 114 países y más de 4.000 muertes, la OMS declaró al COVID-19 una pandemia 5.
Según la OMS, las enfermedades virales continúan surgiendo y representan un problema grave para la salud pública. En los últimos veinte años, se han producido dos epidemias adicionales causadas por CoV: el coronavirus del síndrome respiratorio agudo severo (SARS-CoV) en 2002 que provocó una epidemia a gran escala que comenzó en China e involucró a dos docenas de países con aproximadamente 8.000 casos y 800 muertes; y el coronavirus del síndrome respiratorio del Medio Oriente (MERS-CoV) en 2012 que comenzó en Arabia Saudita y ha causado aproximadamente 2.500 casos y 800 muertes y todavía causa casos esporádicos 6,7.
Los CoV se han convertido en los principales patógenos de brotes emergentes de enfermedades respiratorias 8,9. Son virus de ARN monocatenarios de polaridad positiva, pueden aislarse en diferentes especies animales y hacer salto de especie, causando enfermedades en humanos que van desde el resfriado común hasta enfermedades más graves como el MERS y el SARS 10,11. Estos últimos virus probablemente se originaron en murciélagos y luego se trasladaron a otros mamíferos: la civeta de palma del Himalaya para SARS-CoV y el camello dromedario para MERS-CoV, antes de saltar a los humanos 12-14. La dinámica del SARS-Cov-2 se desconoce actualmente, pero se especula que también tiene un origen animal.
Los gobiernos mundiales trabajan para establecer contramedidas para detener posibles efectos devastadores de esta pandemia. Las organizaciones de salud coordinan los flujos de información y emiten directivas y directrices para mitigar mejor el impacto de la amenaza. Al mismo tiempo, los científicos de todo el mundo trabajan incansablemente, y la información sobre los mecanismos de transmisión, el espectro clínico de la enfermedad, los nuevos diagnósticos y las estrategias preventivas y terapéuticas se están desarrollando rápidamente 15. Quedan muchas incertidumbres con respecto a la interacción virus-huésped y a la evolución de la epidemia, con referencia específica a los momentos en que la epidemia alcanzará su punto máximo.
Por el momento, las estrategias terapéuticas para tratar la infección son solo de apoyo, y la prevención dirigida a reducir la transmisión en la comunidad es nuestra mejor arma 16. Las medidas agresivas de aislamiento en China han llevado a una reducción progresiva de casos en los últimos días. En Italia, en regiones geográficas del norte, inicialmente, y posteriormente en toda la península, así como en España las autoridades políticas y sanitarias están haciendo esfuerzos increíbles para contener un tsunami que desplome los sistemas hospitalarios y la economía de estos países 17,18.
Desde su aparición en diciembre de 2019 y hasta la fecha se han registrado aproximadamente 12.000.000 de personas infectadas en el mundo con una tasa de mortalidad de 7%, y se espera que el número de casos de pacientes infectados aumente exponencialmente 19. En Colombia, el número de casos ya supera los 120.000 infectados, desde la aparición del primer caso a inicios de marzo, con más de 4200 muertes a la fecha 20.
Esta revisión describe los aspectos más importantes de SARS-CoV-2 desde el punto de vista bioquímico-estructural y presenta los métodos de detección y diagnóstico aprobados por la FDA hasta el mes de abril.
Transmisión
Los individuos sintomáticos son la fuente más frecuente de propagación del SARS-CoV-2 21. La posibilidad de transmisión antes de que aparezcan los síntomas parece ser poco frecuente, aunque no hay datos contundentes para excluirla 22. Además, es posible que las personas asintomáticas transmitan el virus 23. Estos datos sugieren que el aislamiento es la mejor manera de contener esta epidemia.
La transmisión se produce a través de gotitas respiratorias por toser y estornudar. La transmisión por aerosoles también es posible en caso de exposición prolongada a concentraciones elevadas de aerosoles en espacios cerrados 24,25. Se sugiere que otras vías de transmisión incluyen la vía fecal-oral y la superficie ocular 26,27. El análisis de los datos relacionados con la propagación del SARS-CoV-2 en China parece indicar que cerca que el contacto entre individuos es necesario. La propagación inicial, de hecho, se limita principalmente a miembros de la familia, profesionales de la salud y otros contactos cercanos 24.
Según los datos de los primeros casos en Wuhan y las investigaciones realizadas por el CDC de China y los CDC locales, el tiempo de incubación podría ser generalmente de 3 a 7 días y prolongarse hasta 2 semanas, ya que el tiempo más largo desde la infección hasta los síntomas fue de 12.5 días (95% IC, 9,2 a 18) 25,28. Estos datos también han mostrado que esta nueva epidemia se duplicó aproximadamente cada siete días, mientras que el número básico de reproducción (R0) es 2,68 29.
Prevención
Las medidas preventivas son la estrategia actual para limitar la propagación de casos. Debido a que una epidemia aumentará mientras R0 sea mayor que 1, las medidas de control deben enfocarse en reducir el valor a menos de 1.
Las estrategias preventivas se centran en el aislamiento de los pacientes y el control cuidadoso de la infección para mantener el R igual a 0, incluidas las medidas apropiadas que deben adoptarse durante el diagnóstico y la prestación de atención clínica a un paciente infectado. Por ejemplo, las gotas, el contacto y las precauciones en el aire en espacios cerrados deben manejar con extremo cuidado durante la recolección de muestras, y debe evitarse la inducción de esputo.
La OMS ha emitido las siguientes recomendaciones generales 16,30:
Evite el contacto cercano con sujetos que padecen infecciones respiratorias agudas.
Lávese las manos con frecuencia, especialmente después del contacto con personas infectadas o su entorno.
Evite el contacto sin protección con animales de granja o animales salvajes.
Las personas con síntomas de infección aguda de las vías respiratorias deben mantenerse a distancia, cubrirse la boca al toser o estornudar con pañuelos desechables o ropa y lavarse las manos.
Fortalecer, en particular, en los departamentos de medicina de emergencia, la aplicación de medidas estrictas de higiene para la prevención y el control de infecciones.
Las personas inmunocomprometidas deben evitar las reuniones públicas.
La estrategia más importante para los poblados es lavarse las manos con frecuencia y usar desinfectante de manos portátil y evitar el contacto con la cara y la boca después de interactuar con un entorno posiblemente contaminado.
Los trabajadores de la salud que atienden a personas infectadas deben utilizar precauciones de contacto y en el aire para incluir elementos de protección personal como máscaras N95 FFP3, protección para los ojos, batas y guantes para evitar la transmisión del patógeno 31.
Estructura de SARS-CoV-2
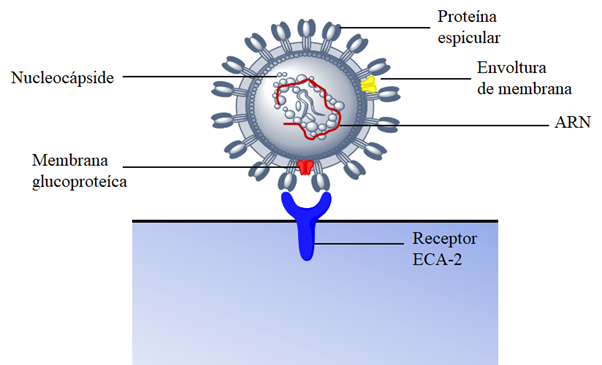
El SARS-CoV-2, conocido así por las espículas presentes en su cápside que forman una especie de corona alrededor de su superficie, es un virus de ARN monocatenario y polaridad positiva de entre 26 y 32 kilobases de longitud, es un virus envuelto y con una nucleocápside de simetría helicoidal, perteneciente a la familia Coronoviridae32,33. Actualmente, se conocen 4 géneros derivados de la familia principal: alfa-coronavirus, beta-coronavirus, gamma-coronavirus y delta-coronavirus. Se sabe que los géneros alfa y beta-CoV pueden infectar humanos mientras los otros dos géneros sólo infectan aves 10.
Las proteínas de SARS-CoV-2 incluyen proteínas tipo espícula (S), envoltura de membrana (E) y nucleocápside (N) 34. Particularmente, en el SARS-CoV-2, el reconocimiento por parte del receptor de la célula hospedadora se lleva a cabo vía endosoma mediante un dominio de unión que dirige la adherencia del virus al receptor celular, la enzima convertidora de angiotensina 2 (ECA-2) 35. La figura 1, muestra una ilustración de la estructura del SARS-CoV-2 y el reconocimiento por parte del receptor viral ECA-2 presente en la superficie de la célula hospedadora.
Ciclo infeccioso de SARS-CoV-2
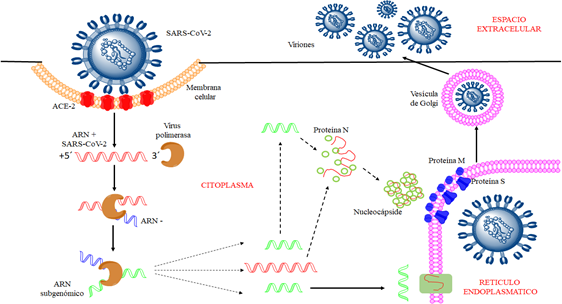
El ciclo infeccioso de SARS-CoV-2 inicia con la entrada endosomal del virus a la célula hospedadora mediante la interacción del dominio S1 de la proteína S con receptores ECA-2 presentes principalmente en neumocitos y enterocitos del sistema respiratorio 36. Una vez ingresa el virus pierde su envoltura y el genoma viral se libera en el citoplasma traduciéndose en poliproteínas virales de replicasa pp1a y 1ab, que luego se dividen en pequeños productos mediante proteinasas virales, como se observa en la figura 2 37.
El genoma de ARN de SARS-CoV-2 presenta una estructura metilada en el extremo 5' y una cola poliadenilada en el extremo 3'. Esta particularidad hace que al ARN se le adhieran los ribosomas citoplasmáticos para el proceso de traducción. SARS-CoV-2 presenta una proteína replicasa codificada en su genoma, que le permite producir nuevas copias de su ARN 38. Al igual que otros virus de la misma familia del SARS-CoV-2 esta replicasa es la primera proteína que se sintetiza ya que una vez que el gen que codifica la replicasa es traducido, el proceso se detiene por un codón de parada. Las nucleocápsides virales se ensamblan a partir del ARN genómico y la proteína N viral presente en el citoplasma, para ello utilizan una proteasa denominada 3CLpro25 que corta la poliproteína seguido de la gemación en el lumen del compartimiento intermedio Golgi-retículo endoplasmático. Finalmente, los viriones son liberados de la célula infectada por medio de exocitosis 39.
Estructura de la proteína S
La glicoproteína S es una proteína de membrana integral homo-trimérica tipo I, similar a las glicoproteínas: 160 de VIH (Env), hemaglutinina de la influenza (HA) y a la glicoproteína viral del Ébola 35,40. Está organizada en tres dominios: un dominio extracelular (EC), un dominio de anclaje transmembranal y un dominio tipo cola intracelular. El dominio EC contiene dos subdominios necesarios para el proceso de reconocimiento por parte de la célula hospedadora (S1) y fusión de la membrana (S2). El S1 presenta dos dominios independientes, un dominio N-terminal (S1-NTD) y un dominio de unión al receptor (RBD) (figura 3a), el cual desempeña un papel clave en los procesos de unión y reconocimiento. Durante el proceso de fusión hospedador-virus, la proteína S es cortada en el límite de los subdominios S1/S2 por las proteasas de la célula hospedadora, liberando un péptido de fusión el cual es indispensable para la entrada del virus 41.
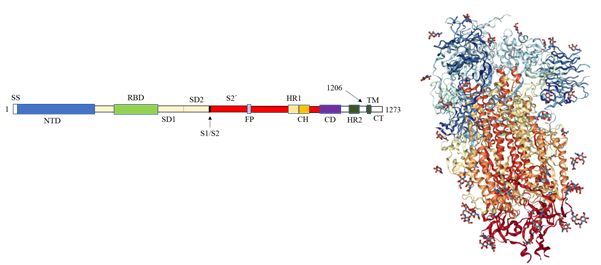
Fuente: Tomado y modificado de Liu Z, Xiao X y cols 35
Figura 3 Esquema coloreado por dominios de la estructura primaria de la proteína S de SARS-CoV-2. RBD, dominio de unión al receptor; SS, secuencia de las señales; S2´, sitio de escisión de proteasas; FP, péptido de fusión; HR1, repetición heptad 1; CH, hélice central; CD, dominio conector; HR2, repetición heptad 2; TM; dominio transmembranal; CT, cola citoplásmica. Las flechas indican los sitios de acción de las proteasas.
Actualmente se sabe que la proteína S es clave para el desarrollo de vacunas, tratamientos y métodos de diagnóstico 42. Recientemente, Wrapp y cols. elucidaron la estructura cuaternaria de la proteína S en la conformación de prefusión mediante criomicroscopía electrónica de alta resolución, técnica de microscopía electrónica, a temperaturas criogénicas, que permite observar estados conformacionales nativos de proteínas a resolución atómica 43. El estado conformacional predominante del trímero S tiene uno de los tres dominios de unión al receptor (DUR) rotados en una conformación accesible al receptor, como se puede observar en la figura 3.
Diferentes estudios han demostrado la interacción de diferentes tipos de CoV que comparten la misma afinidad por la ECA-2 de las células hospedadoras. En el año 2.003 Li y cols. identificaron una ECA de células Vero E6 infectadas con SARS-CoV que se enlazó eficientemente al dominio S1 de la proteína S del SARS-CoV. Además, se encontró que la enzima ECA-2 en su forma soluble no se enlaza a los receptores presente en células infectadas con VIH 44. Más adelante, precisamente en el año 2.020 Hoffman y cols. demostraron que el nuevo SARS-CoV-2 reconoció el receptor ECA-2 por medio de la proteasa celular TMPRSS2 (proteína transmembranal serina 2) 45. Mediante experimentos de resonancia de plasmón superficial (SPR) Wrapp y cols, estudiaron la interacción ECA-2 con la proteína S caracterizada con una constante de afinidad (KD) de 1.42 nM 43.
Es importante recalcar que no únicamente la ECA-2 es el único sitio de recepción identificado hasta ahora para el ingreso de SARS-CoV-2 a la célula hospedadora. Mediante estudios de docking molecular se sabe que probablemente son 4 los sitios que utiliza la proteína S para unirse a una proteína chaperona del retículo endoplasmático conocida como GRP78 para lograr su entrada a la célula hospedadora 42. La GRP78 es una proteína de choque térmico codificada por el gen HSPA5 que juega un papel importante en el plegamiento de proteínas 46.
Diagnóstico de COVID-19
Las infecciones emergentes por CoV humanos (CoVH) han propiciado el desarrollo de métodos de diagnóstico rápidos, sensibles y específicos. Desde pruebas moleculares tipo reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR) hasta dispositivos moleculares integrados, de acceso aleatorio y en el punto de atención (POCT, por sus siglas en inglés de Point-of-care testing), altamente seguros para el personal de salud y accesibles en hospitales y clínicas a nivel mundial.
Debe tenerse en cuenta que, a pesar de la precisión y rapidez de los métodos utilizados en el laboratorio, el diagnóstico oportuno de neumonías virales como la causada por el SARS-CoV-2 implica recolectar una óptima muestra del paciente: del lugar indicado, con los cepillos recolectores recomendados, en la cantidad necesaria y el momento adecuado.
Pruebas de laboratorio para SARS-CoV-2
1. RT-PCR
Tan pronto como se conoció la secuencia viral de SARS-CoV-2, la PCR acoplada a transcripción reversa, fue el primer método empleado para diagnosticar COVID-19, debido a que las sondas de hidrólisis y cebadores necesarios para la detección de genes diana específicos se pueden producir rápidamente (Figura 4E).
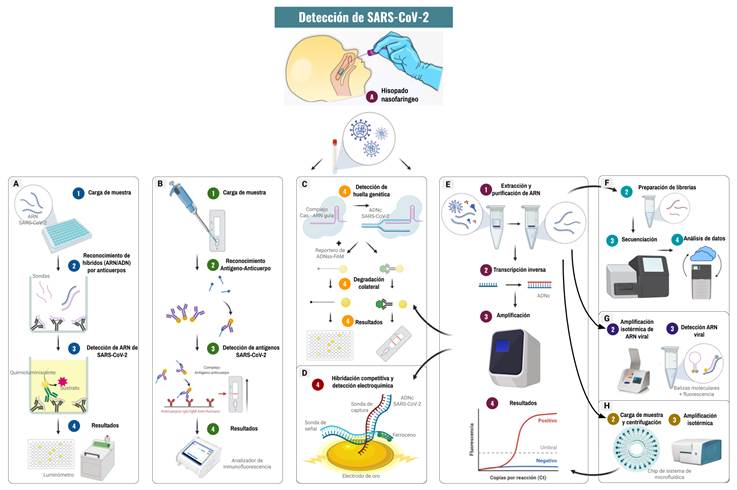
Fuente: autores
Figura 4 Esquema ilustrativo resumido de los métodos diagnósticos para la detección de SARS-CoV-2
En enero, la OMS se encargó del diseño y distribución de las primeras RT-PCR para detectar SARS-CoV-2. Este protocolo es complejo y costoso, tarda entre 4 a 6 horas y requiere el envío de las muestras clínicas a laboratorios de diagnóstico grandes y centralizados, por lo que requiere de personal profesional y el tiempo de respuesta es de máximo 24 horas 47.
2. Pruebas rápidas de antígeno
Ofrecen economía y obtención de resultados en un tiempo corto, aunque en ocasiones a expensas de la sensibilidad de detección, como se ha visto en otros casos, por ejemplo, con los virus de influenza 48,49. No obstante, el ensayo inmunocromatográfico de fluorescencia para el diagnóstico de COVID-19 propuesto por Diao et al, podría ser un método preciso para detectar en 10 minutos la proteína de la nucleocápside del nuevo coronavirus a partir de una muestra tomada con un hisopo nasofaríngeo. Esta proteína también se pudo detectar en la orina del 73% de los pacientes diagnosticados evaluados en este estudio (Figura 4B) 50.
Aparte de recolectar la muestra cuando los títulos virales son más altos, otra de las medidas que se pueden implementar en la práctica clínica ambulatoria para aumentar la sensibilidad diagnóstica de las pruebas rápidas de antígeno, y que ya ha funcionado antes en la detección de los virus de la influenza A y B, es la conjugación de anticuerpos monoclonales al oro coloidal como reactivo de detección rápida de virus respiratorios 51.
3. Ensayos serológicos
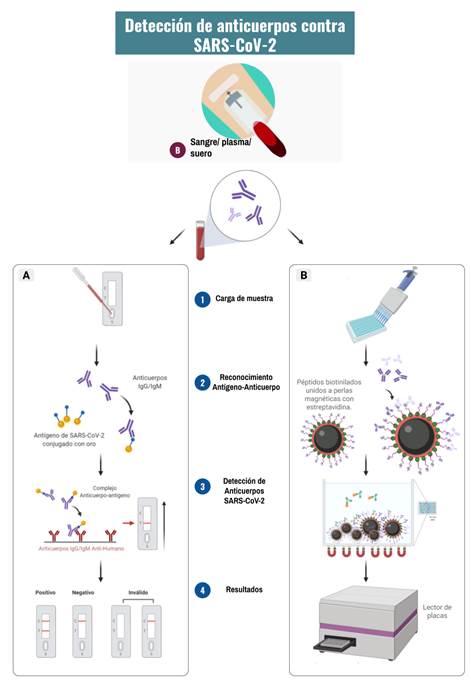
No se usan rutinariamente para diagnosticar infecciones por CoV, su importancia radica en la comprensión de la epidemiología de los CoVH emergentes y en el papel de las infecciones asintomáticas, que al parecer son más frecuentes de lo que se creía 52. Para el 12 de marzo, en China, seis ensayos serológicos ya tenían la aprobación urgente de la Administración Nacional de Productos Médicos (tabla 1, figura 5A).
Tabla 1 Ensayos serológicos autorizados en China para el diagnóstico de laboratorio de infecciones por SARS-CoV-2.
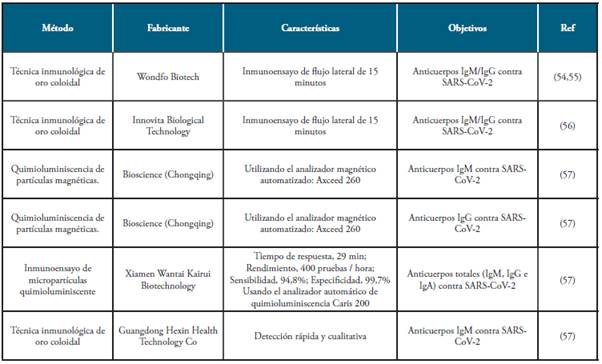
Fuente: Autores.
4. Secuenciación
La secuenciación genética es la forma más precisa y confiable de identificar el SARS-CoV-2, además, proporciona una base efectiva para el posterior desarrollo de kits de RT-PCR sensibles y específicos, es la única forma de rastrear las mutaciones del virus, puede brindar información valiosa para el desarrollo de vacunas, para realizar estudios sobre el mecanismo de patogenicidad, propagación y epidemiología genética de SARS-CoV-2 con el fin orientar de mejor manera las medidas de prevención.
Inicialmente, se utilizó el sistema de secuenciación DNBSEQ-T7 de ultra alto rendimiento de MGI Tech, cuya tasa de precisión puede alcanzar el 100% y puede completar todo el proceso de detección desde la extracción de ácidos nucleicos hasta el informe de resultados en 20 - 22 horas 58. Sin embargo, el costo es alto, la operación es complicada y no es adecuada para la detección de rutina en una clínica (Figura 4F).
5. Amplificación isotérmica en chip de sistema de microfluídica
La amplificación y las reacciones son isotérmicas en función del efecto sinérgico de la transcripción inversa y la transcriptasa, utilizando sondas fluorescentes específicas para la detección de fluorescencia en tiempo real.
Es un ensayo de alta precisión que emplea chips microfluídicos, controles de calidad, tecnología anticontaminación y tiene una alta sensibilidad en la detección simultánea de múltiples genes objetivo, incluyendo SARS-CoV-2, virus de la gripe A, virus de la gripe H1N1, virus de la gripe A H3N2, virus de la gripe B, virus sincitial respiratorio y otros patógenos, con el fin de descartar diferentes etiologías, optimizar los recursos del sistema de salud y conocer el porcentaje de coinfecciones.
Las muestras positivas crearán una curva de amplificación de aspecto sigmoideo similar a la PCR de fluorescencia en tiempo real, los resultados se interpretan con ayuda de un software y generalmente toma 1,5 horas. Sin embargo, algunos patógenos pueden pasarse por alto (Figura 4H) 59.
6. Inmunoensayo magnético quimioluminiscente
Este ensayo emplea péptidos sintéticos de SARS-CoV-2, sintetizados a partir de la secuencia genómica, para detectar anticuerpos contra este CoV. Para garantizar una alta especificidad, la región escogida debe tener una homología baja en relación con otros CoV. Los péptidos biotinilados y purificados están unidos a perlas magnéticas recubiertas con estreptavidina. Las perlas se hacen reaccionar con los anticuerpos conjugados y con el sustrato (Figura 5B) 60. Este método es altamente sensible, específico, reproducible y completamente automatizado, los resultados generalmente se producen en 30 minutos. Sin embargo, se requiere un equipo especial de detección de quimioluminiscencia.
7. Amplificación isotérmica de ácidos nucleicos
Ensayo automatizado que detecta ácidos nucleicos virales específicos de SARS-CoV-2 utilizando tecnología de amplificación a la misma temperatura. Las plantillas (similares a los cebadores) se dirigen al ARN de SARS-CoV-2 y amplifican una región única. Se utilizan balizas moleculares marcadas con fluorescencia para identificar específicamente cada una de las dianas de ARN amplificadas. En comparación con el método de PCR fluorescente, los requisitos del equipo son menores, proporciona calentamiento, mezcla y detección, y generalmente se obtienen resultados en 15 minutos (Figura 4G) 61.
8. Método de inmunofluorescencia de captura híbrida
Sistema de detección rápida in situ de los ácidos nucleicos de SARS-CoV-2 en un solo paso. No requiere extracción, purificación de ácidos nucleicos, ni amplificación por PCR. El ARN y la sonda específica forman híbridos (ARN/ADN) que son identificados por anticuerpos específicos y una solución quimioluminiscente. La fluorescencia emitida se mide en un luminómetro, obteniendo resultados cualitativos de la presencia del virus. Estos kits de prueba se pueden almacenar y transportar a temperatura ambiente, lo cual minimiza la logística necesaria de la cadena de frío, el tamaño del equipo de detección es pequeño, portátil y el tiempo de detección de una sola muestra es más corto, generalmente de 45 minutos a 1 hora (Figura 4A) 62.
9. Hibridación competitiva de ADN y detección electroquímica
Este método es altamente específico, no se basa en la detección fluorescente u óptica. Está diseñado para ser llevado a cabo por personal de laboratorio calificado y capacitado y requiere un equipo especial de detección.
La muestra y los reactivos se introducen en un cartucho y por medio de campos eléctricos se programa su desplazamiento para completar todas las partes del procesamiento de la muestra: extracción y purificación de ácidos nucleicos mediante extracción magnética en fase sólida; transcripción inversa seguida de PCR; y digestión con exonucleasa, obteniendo finalmente ADN monocatenario.
El ADN objetivo se hibrida con su sonda de señal complementaria marcada con ferroceno y con las sondas de captura, que están unidas a electrodos chapados en oro. El análisis electroquímico determina la presencia o ausencia de objetivos mediante voltamperometría, generando señales eléctricas específicas de la sonda de señal marcada con ferroceno (Figura 4D) 63.
10. Pruebas rápidas basadas en CRISPR para SARS-CoV-2
Método de detección de SARS-CoV-2 ultrasensible, rápido y portátil utilizando las enzimas Cas12 o Cas13 para la detección de huellas genéticas únicas. Una vez obtenido y amplificado el cDNA, se agregan los complejos de detección CRISPR, los cuales constan de una endonucleasa y un ARN guía dirigido a diferentes blancos de SARS-CoV-2, por ejemplo, a los genes RdRp, ORF1b y ORF1ab.
La detección se logra con reporteros de ADN monocatenario (ADNss) ya sea etiquetado con FAM en un lector de placas, o etiquetado con biotina para la detección portátil con tiras de papel. Después del reconocimiento del objetivo, la endonucleasa induce la degradación colateral indiscriminada de los reporteros de ADNss, que emiten una señal de fluorescencia o pueden detectarse por flujo lateral en menos de 60 minutos (Figura 4C) 64.
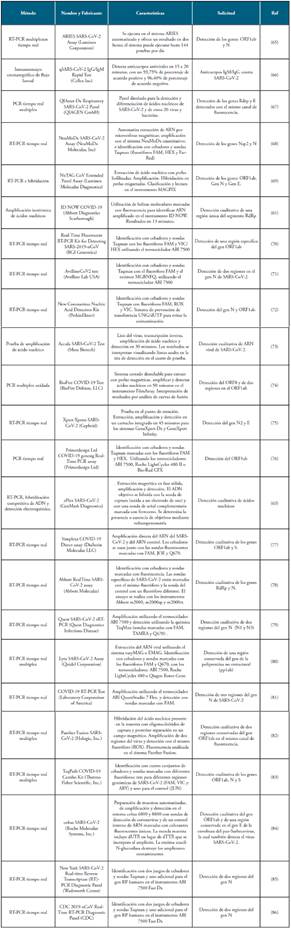
Teniendo en cuenta las tasas de mortalidad e infecciosidad asociadas a este nuevo CoV, la FDA emitió autorizaciones de uso de emergencia a las siguientes pruebas (tabla 2).
Conclusiones
Teniendo en cuenta que la OMS y el CDC han determinado que la RT-PCR es el estándar de oro actual para el diagnóstico de la infección por SARS-CoV-2, y que esta ha demostrado una baja positividad en muestras de hisopados de garganta de pacientes sintomáticos, una posible forma de aumentar la sensibilidad en la detección de SARS-CoV-2 es implementando la PCR digital 87.
La tecnología de PCR digital podría aumentar la sensibilidad diagnóstica de individuos con bajos títulos virales de SARS-CoV-2 que se encuentran bajo aislamiento y aquellos bajo observación que pueden no presentar síntomas clínicos, como se ha evidenciado en los infectados presintomáticos, asintomáticos y las personas adultas mayores que están en proceso de recuperación en quienes las pruebas moleculares actuales los detectan como recuperados pero todavía no han eliminado del todo al SARS-CoV-2 de su sistema. Recientemente, se ha evidenciado que la PCR digital podría disminuir el límite de detección inferior en al menos 500 veces en comparación con el de la RT-PCR, así como el porcentaje de falsos negativos de la RT-PCR estándar actual 88.
Adicionalmente, en vista de que la RT-PCR requiere de la extracción de ARN por medio de kits que actualmente escasean a nivel mundial y han causado un grave cuello de botella en las pruebas para diagnosticar COVID-19, se ha propuesto un protocolo en el que se detecta directamente el ARN del SARS-CoV-2 a partir de muestras nasofaríngeas a través de un ensayo directo de PCR omitiendo el paso de extracción de ARN y reduciendo el tiempo de obtención de resultados 89.
Considerando que, la práctica clínica actual dicta que el convaleciente por COVID-19 debe continuar en cuarentena durante 2 semanas, la alta sensibilidad de la PCR digital, ayudaría a disminuir el tiempo de aislamiento de los pacientes recuperados otorgando un resultado negativo más confiable para agilizar la atención, optimizar los recursos y evitar colapsar los sistemas de salud.