Metodologías POCT para el estudio de SARS-CoV-2 (2)
Serológicas
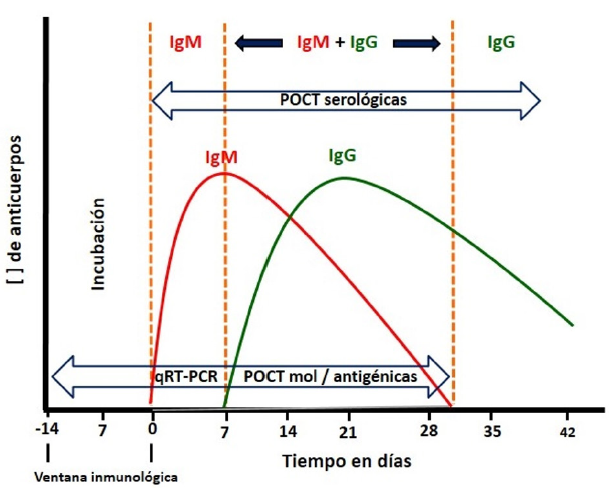
Estas pruebas detectan, en sangre, suero o plasma 2,3, los anticuerpos generados como respuesta a la infección por SARS-CoV-2 4,5,6. En pacientes jóvenes y sanos, la IgM y la IgA aparecen entre el cuarto y el sexto día del inicio de los síntomas, con pico en el día 14 (7,8). En pacientes inmunodeprimidos y crónicos la respuesta se da entre el séptimo y decimocuarto día. Por su parte, los anticuerpos IgG se retrasan en algunos casos y aparecen entre los días 15 y 21 8,9,10,11, de modo que aparece su pico entre los días 17 y 19. Por ello, una POCT serológica realizada demasiado temprano puede no caracterizar adecuadamente el estado de los pacientes y generar falsos negativos 7,12,13,14,15.
Las metodologías usadas son Elisa e inmunocromatografía de flujo lateral con partículas de oro coloidal 16. Las proteínas virales de la espiga (S) y la nucleocápside (N) actúan como antígenos. Otros formatos, como el de inmunoflorescencia y búsqueda de anticuerpos neutralizantes, solo están disponibles en laboratorios de referencia o investigación 17. La sensibilidad de los productos comerciales varía entre el 40 % y 93 %, y la especificidad entre 93 % y 100 % 5,8,10,12,18, por lo que la tasa de casos falsos negativos puede ser sustancial 8.
Asimismo, se ha reportado reactividad cruzada por la inmunidad adquirida contra otros coronavirus como el SARS-CoV 4,19,20. Por ello, la Organización Panamericana de la Salud (OPS) y la Organización Mundial de la Salud (OMS) recomiendan validar y evaluar el rendimiento de estas pruebas en términos de especificidad y sensibilidad, y han aconsejado que cada país realice sus propias validaciones antes de implementarlas 20,21. Igualmente, aunque estas pruebas no las indican para el diagnóstico clínico temprano, las consideran herramientas valiosas para la vigilancia, predicción del resultado de la enfermedad y la investigación epidemiológica 19,20,21. La lista de los ensayos en evaluación está disponible en el sitio web de la Foundation for Innovative New Diagnostics (FIND)22.
Para mejorar los criterios de calidad y el índice de detección se ha propuesto emplear la combinación rtRT-PCR e IgM/IgG en pacientes que seroconvierten 23,24; obtener muestras de suero emparejadas (en la fase aguda y convaleciente); utilizar como antígeno secciones de la proteína S que sean particularmente distintas entre los coronavirus, y recurrir a ensayos basados en la metodología de antígeno recombinante 3,19.
Rapidez en la obtención de resultados
Producción masiva y de bajo costo
Baja o nula carga viral de las muestras usadas
Adaptables a diferentes formatos de diagnóstico
Pueden utilizarse en el sitio de la toma de la muestra
Antigénicas
Con estas pruebas se detectan componentes estructurales del virus SARS-CoV-2 como la proteína de la nucleocápside (N) 26,27 y las subunidades S1 o S2 de la proteína espiga (S) 28,29, en muestras de esputo o hisopado nasofaríngeo u orofaríngeo, que se recolectan en etapas tempranas de la enfermedad 30,31,32,33.
Las metodologías disponibles para estos ensayos son la inmunocromatografía con incorporación de partículas de oro coloidal y los ensayos inmunofluoresecentes34. También se han desarrollado pruebas inmunocromatográficas de fluorescencia que correlacionan 100 % con la qRT-PCR27. Todas ellas utilizan anticuerpos monoclonales específicos contra el SARS-CoV-2 que son complejos. Por ello, para mejorar los valores diagnósticos se requiere concentrarlos o amplificar la fase de detección 35. La lista de los ensayos en evaluación está disponible en la página de la FIND 22.
Teóricamente el antígeno viral es el marcador específico del virus y precede la aparición de anticuerpos; por lo tanto, la detección del antígeno implica replicación activa del virus e indica infección actual por SARS-CoV-2 25. Sin embargo, aunque hay estudios que demuestran buena sensibilidad, especificidad y correlación de estas pruebas con la qRT-PCR 27,36, otros la refutan y plantean la posibilidad de obtener falsos negativos 6,21,25,30. De igual manera, mientras algunos laboratorios señalan que no hay reacción cruzada con otros coronavirus, otros la confirman 37, de manera que no se puede descartar la presencia de falsos positivos 6.
Todo ello ha llevado a la OMS a no recomendar, hasta ahora, el uso de pruebas rápidas antigénicas para la atención del paciente, aunque invita encarecidamente a investigar su rendimiento y posible utilidad diagnóstica 21.
Rapidez, sencillez y facilidad de estandarización
Adecuada sensibilidad si se utiliza fluorimetría
Buena correlación con la qRT-PCR
Buen valor predictivo positivo
No presentan problemas de contaminación
Moleculares
Estas muestras se fundamentan en la amplificación isotérmica mediada por bucle de transcripción inversa RT-LAMP, método que se ha venido utilizado para la detección rápida de virus ARN como SARS-CoV, MERS-CoV e influenza 39,40,41,42. No obstante, aún están en desarrollo para la identificación de SARS-CoV-2 45,46,47,48,49,50 y no se ha avalado su utilidad en entornos clínicos 2.
Los ensayos LAMP incluyen la amplificación exponencial de secuencias específicas de ácido nucleico a una temperatura constante en la que se emplean un total de seis cebadores: dos internos, dos externos y dos de bucle, que facilitan y aceleran la amplificación y detección. Incorporan también una ADN polimerasa de desplazamiento de cadena, que actúa a una temperatura constante de 60 a 65 °C39,46,51. La formación en estructura de bucle elimina el paso de desnaturalización a 90 ºC que es esencial en la qRT-PCR para obtener ADN monocatenario 52,53.
Ventajas
Mayor velocidad de amplificación 45,51,54
Especificidad y sensibilidad comparables a la qRT-PCR 46
Se obtiene más ADN que en la qRT-PCR 51,55,56,57
Permiten muestras clínicas variables (hisopados orales, de garganta, nasales o nasofaríngeos y lavado broncoalveloar) 2
Evitan la inclusión de un control interno de inhibición de la PCR
Requieren de un sistema de diseño de cebador complejo
El tamaño del producto final limita aplicaciones posteriores como la clonación
Durante las manipulaciones del ensayo son sensibles a los contaminantes en aerosol
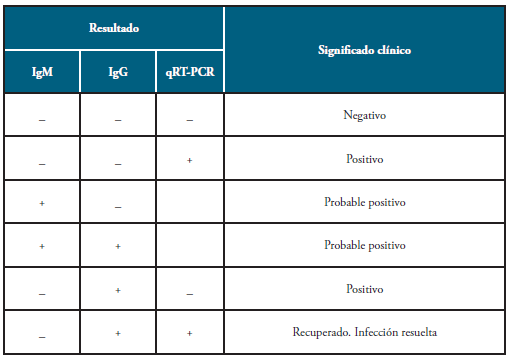
Otras POCT moleculares en ensayo son la metodología Sherlock 4,59,60 y un sistema de detección basado en Cas13a 61. ¿Son útiles o no estas pruebas? Depende de su correcta aplicación (figura 1) e interpretación clínica (tabla 1)