Introducción
En diciembre de 2019, se identificaron los primeros casos de neumonía atípica en la provincia de Hubei en China 1. Su agente causal se identificó como un nuevo coronavirus denominado SARS-CoV-2, caracterizado por una alta tasa de propagación y ser capaz de producir manifestaciones graves a nivel respiratorio o muerte, por lo que la Organización Mundial de la Salud (OMS) declaró el brote de este coronavirus como una pandemia 2.
A la fecha, varios grupos de investigación trabajan en la búsqueda de un tratamiento efectivo contra este nuevo virus. Sus esfuerzos están encaminados al desarrollo de vacunas, evaluación de la eficiencia de fármacos ya conocidos y la búsqueda de estrategias basadas en péptidos.
Características moleculares de SARS-CoV-2
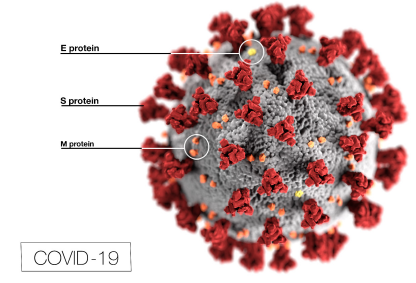
El SARS-CoV-2 es un virus de la familia Coronaviridae, que presenta una estructura esférica con una glicoproteína de espiga (S, spike protein), responsable de la unión inicial del virus a la célula pulmonar 3. Junto con la proteína de membrana (M), cooperan para el ingreso del virus a la célula; ambas constituyen la mayoría de las proteínas presentes en la envoltura viral. También, se encuentra la proteína de envoltura (E), responsable de la morfogénesis y patogénesis del virus 4 (figura 1).
Fuente: Librería de imágenes públicas (PHIL) 5.
Figura 1 Morfología estructural del coronavirus. Se observan las proteínas características del virus, Proteína S, color rojo; proteína E, color amarillo y proteína M, color naranja. Imagen tomada del Centro del Control de Enfermedades y Prevención CDC.
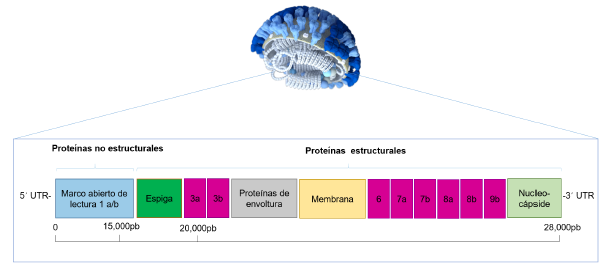
La envoltura viral encapsula el genoma ARN monocatenario (+ ssARN) de aproximadamente 30 Kb, el cual se encuentra rodeado de proteínas de la nucleocápside (N). Su genoma codifica cuatro proteínas estructurales (S, M, E, N) y 16 proteínas no estructurales o nsp por los ORF1a/b, donde se codifican las proteasas virales: nsp3 (proteasa similar a la papaína) y nsp5 (proteasa principal), nsp12 (ARN polimerasa dependiente de ARN) y nsp13 (helicasa); además de ocho proteínas accesorias ORF3a, 3b, 6, 7a, 7b ,8a, 8b, 9b 5. En la figura 2 se observa la organización de su genoma y el tipo de proteínas codificadas.
Fuente: Librería de imágenes públicas (PHIL) 5.
Figura 2 Organización del genoma de SAR- CoV-2. El genoma se compone de regiones que codifican para proteínas estructurales y no estructurales, en morado se identifican las proteínas accesorias. Imagen del virus tomada del Centro del Control de Enfermedades y Prevención CDC.
Mecanismo de ingreso de SARS-CoV-2 a la célula
Al ingresar al torrente respiratorio, el virus se une a los receptores de las células diana del huésped por la proteína S, constituida por dos subunidades S1 y S2, las cuales son escindidas por proteasas celulares. La subunidad S1 se enlaza al dominio peptidasa de la enzima convertidora de angiotensina 2 (ACE2) 6, mientras que la subunidad S2 se escinde y es activada por TMPRSS2 (serina proteasa transmembranal tipo II), lo cual permite la fusión de las membranas 7. Por su papel durante la infección las proteínas S, ACE2 y TMPRSS2 se han convertido en blancos terapéuticos.
Estrategias terapéuticas basadas en péptidos
Los péptidos son moléculas pequeñas de fácil diseño y síntesis que pueden actuar desde dos perspectivas principalmente, como péptidos miméticos que interactúan con los receptores de la célula o del virus para bloquear su interacción, o como vacunas, donde se sintetizan péptidos basados en epítopes como agentes inmunogénicos.
Péptidos como inhibidores de la interacción de proteínas
Las proteínas interactúan entre sí por un número limitado de aminoácidos que son críticos para la activación de una señal celular. Los péptidos se diseñan para imitar la estructura de una proteína y actuar como “interferentes”, de manera que enmascaran u ocultan un sitio de interacción proteica.
Para el caso del SARS-CoV-2 se están diseñando péptidos que impiden la unión, escisión y activación de proteínas fundamentales para la infección. Dutta 8 diseñó péptidos análogos a la glicoproteína S; en su trabajo se destacan las moléculas Seq12 y Seq12m por su baja toxicidad y capacidad de interferir con la proteína M de la membrana viral.
Asimismo, el grupo de Xia et al.9 demostró que el péptido EK1C4, inhibe la fusión de membranas mediada por SARS-CoV-2. En esta misma dirección, Zhang et al 2020 realizó el diseño de un inhibidor α-cetoamida, denominado 13b, similar a un péptido que bloquea la función de la proteasa principal 10. Todos estos estudios evidencian el potencial terapéutico de los péptidos sintéticos, pues son hasta el momento altamente efectivos en modelos murinos e infecciones in vitro.
Los péptidos como vacunas
Las vacunas administran componentes específicos que buscan generar inmunidad a través de la respuesta inmune terapéutica. Para ello se han empleado diferentes rutas como la administración de microorganismos vivos atenuados, inactivados o fracciones de sus proteínas; sin embargo, muchos microrganismos son de difícil cultivo y, por lo tanto, se reduce la posibilidad de tenerlos atenuados o inactivados. Además, al inyectar microorganismos atenuados o inactivados, la especificidad de la respuesta inmune puede desencadenar respuestas inmunológicas no deseadas. Por lo anterior, considerar una estrategia que se limite al uso de segmentos antigénicos es una ventaja considerable tanto en facilidad de producción como en seguridad 11,12.
Los péptidos basados en determinantes antigénicos o epítopes, que solo representan una porción inmunogénica mínima y que permiten una dirección precisa de la respuesta inmune, son candidatos para vacunas. El grupo de Pant et al. 13 evaluó más de 300 compuestos y reportó que moléculas pequeñas tipo péptido pueden actuar como inhibidores de la proteasa principal del virus y convertirse en candidatos promisorios 12. Abdelmageed et al 2020, emplearon un enfoque inmunoinformático con genómica comparativa y determinaron diez péptidos basados en epítopes de las proteínas de envoltura E, también potenciales candidatos para el diseño de vacunas 14. De acuerdo a la OMS, a la fecha se cuentan con 11 proyectos orientados al diseño, síntesis y evaluación de péptidos para el desarrollo de una vacuna 15.
Perspectivas
Aunque son muchos los desafíos para implementar el uso de péptidos, estos constituyen una herramienta que ofrece enormes posibilidades, especialmente su fácil producción y seguridad. Actualmente, más de 70 fármacos están aprobados en Estados Unidos y 200 se encuentran en ensayos clínicos, lo que evidencia el potencial de los péptidos como alternativa a los enfoques tradicionales de tratamiento para esta infección.














