Introducción
Diferentes campos de investigación a través del avance de la ciencia se han centrado en contribuir a la salud y al mejoramiento de la calidad de vida por medio de la producción de fármacos o sustancias que inhiban la progresión o desarrollo de una enfermedad, la clave para esto se basa en la comprensión de los organismos modelos para el estudio, donde se destacan animales primates, porcinos, roedores entre otros 1,2. A pesar de algunas limitaciones, los animales juegan un papel importante como anfitriones sustitutos para investigar los mecanismos fisiopatológicos de la enfermedad y para poner a prueba en ellos medicamentos contra dicha patología.
Es importante destacar que el uso de animales en la investigación se guía por la promoción de un marco ético como el descrito por William Russell y Rex Birch hace más de 50 años donde estipulan que los animales deben ser reemplazados cuando sea posible, o donde esto no sea posible, los estudios deben usar métodos que reduzcan el número de animales requeridos y los procedimientos deben minimizar al máximo el dolor, el sufrimiento y la angustia 1,3,4. Es por ello que se han popularizado huéspedes de reemplazo que incluyen invertebrados como la mosca de la fruta Drosophila melanogaster, las larvas de la polilla de cera mayor Galleria mellonella o el nematodo Caenorhabditis elegans1.
Dentro de las mayores problemáticas de salud pública a nivel mundial en el ámbito farmacológico se destaca la producción de antibióticos, pues actualmente las bacterias son un gran problema ya que a través del tiempo estos microorganismos han desarrollado la capacidad de infectar a una gran variedad de especies susceptibles y muchas de estas bacterias han generado "resistencia" a ciertos medicamentos libres en el mercado 1,3. Un informe de la Organización Mundial de la Salud describe cómo las bacterias resistentes a los antibióticos están presentes en todas las regiones del mundo, y que muchos países carecen incluso de los sistemas básicos para rastrear y controlar estos microbios peligrosos 1. A todo esto, se le suma la automedicación, el uso desmedido y el fácil acceso a los antibióticos existentes como predisposición para generar resistencia y como consecuencia el daño sustancial o la muerte de diferentes seres vivos en el mundo.
Es por ello, que en los últimos años los estudios se han centrado en desarrollar antimicrobianos efectivos capaces de inhibir bacterias patógenas, de hecho esto radica como prevención y urge la necesidad de incluir al sistema farmacológico medicamentos que den cobertura a aquellas cepas que actualmente ningún antibiótico existente logra controlar. Sin embargo, los antibióticos en desarrollo han tenido limitaciones para su validación, pues en su gran mayoría inicialmente se verifican en modelos In vitro, a partir de medios convencionales de crecimiento bacteriano o en líneas celulares, luego de aprobar este proceso, se usan animales complejos y finalmente la ratificación en humanos lo que prolonga el tiempo del estudio 5.
No obstante, cada vez resulta más difícil usar modelos animales por todas las limitaciones bioéticas existentes y aunque en la primera fase experimental a nivel In vitro es efectiva, después la mayoría de medicamentos fracasan porque no se tiene en cuenta que el huésped infectado está conformado por un sistema completo de células, proteínas y al mismo tiempo respuestas fisicoquímicas que vuelven contraproducente el tratamiento, lo que hace necesario usar un modelo desde los inicios de la propuesta para ahorrar tiempo 1,6. Para ello es necesario usar modelos simples en los estudios preliminares que permitan evaluar las interacciones huésped-patógeno-antimicrobiano.
Las características esenciales de un modelo infeccioso radican en la capacidad que tiene el animal de tener una homología significativa con el ser humano, así como montar respuestas inmunes completas y ser sensible a una gran variedad de infecciones con el fin de permitir entender diferentes condiciones que contribuyan a comprender dicha relación y agilizar los procesos investigativos de los fármacos propuestos. Por otro lado es importante determinar hospederos, que sean de fácil manejo y sin tantas restricciones éticas, de esta forma el modelo permitirá ofrecer soluciones a las problemáticas actuales que perjudican la calidad de vida de los seres vivos 1,2,4,5.
Hasta el momento, Caenorhabditis elegans ha demostrado ser susceptible a varias bacterias, con la capacidad de responder a estímulos ambientales con cambios observables en el comportamiento tras las infecciones. Por otro lado su estado de salud es estable al consumir su alimento normal de laboratorio (Escherichia coli OP50), es por ello que resulta muy útil usarlo en este tipo de investigaciones, adicionalmente C. elegans tiene una vida promedio corta y no cuenta con restricciones éticas para su uso 7. Por lo anterior, en este artículo se revisa la susceptibilidad que tiene C. elegans de infectarse con diferentes bacterias y al no estar validado completamente como modelo para poner a prueba antimicrobianos, se propone que este nematodo puede ser útil como modelo In vivo de infecciones y tratamientos bacterianos.
Generalidades del modelo Caenorhabditis elegans
Caenorhabditis elegans (C. elegans) fue inicialmente descrito y nombrado Rhabditis elegans, señalado por su valor potencial en la investigación genética 8, posteriormente fue colocado en el género Caenorhabditis9. Su nombre es una mezcla de griego y latín (Caeno; reciente, rabditis; vara, elegans; bonito) 10,11. Este nematodo está estrictamente relacionado con eucariotas y dado a los diferentes hallazgos hasta el momento existe una estrecha relación evolutiva con algunos mamíferos, haciendo correlación directa del 36% con el ser humano 12. C. elegans es de vía libre, tiene un tamaño inicial de 0.25 milímetros de largo y al llegar a la adultez mide 1.5 milímetro aproximadamente, se encuentra distribuido en todo el mundo, su principal alimento son las bacterias, su temperatura ideal oscila entre los 12-25°C durante todo su desarrollo y no son considerados patógenos para mamíferos, puesto que temperaturas superiores a los 30° inhabilitan su desarrollo 11,12.
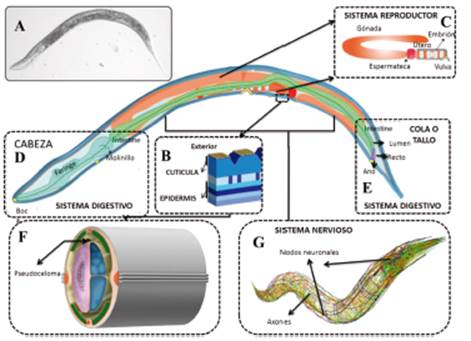
Este modelo tiene tejidos muy bien definidos y como todos los nematodos tienen una simetría corporal básica compuesto por dos tubos concéntricos separados por el "pseudoceloma" que aloja los sistemas de órganos principales y da la forma al animal, también, consta de una epidermis que protege los sistemas y a su vez cumple funciones neuromusculares junto con una cutícula colágena, además, la cavidad pseudocelómica, albergan los celomocitos, que actúan como carroñeros en la cavidad corporal. 11,12. En este nematodo se han descrito tres sistemas muy bien definidos; el sistema reproductor, el nervioso y el digestivo, es por ello que algunas enfermedades e infecciones que involucran estos sistemas se pueden esclarecer en este modelo de laboratorio, Figura 1.
Fuente: Tomado y Modificado de: Gonzalez-Moragas G. y colaboradores 13, Wormatlas 14, Prevedel R. y colaboradores 15.
Figura 1 Anatomía de C. elegans adulto. (A): se observa una fotografía real del nematodo. (B): Conformación externa del nematodo. (C): Sistema reproductor. (D y E): Sistema Digestivo. (F): Líquido Pseudoceloma. (G): Sistema Nervioso.
El ciclo de vida de este invertebrado dura aproximadamente 3 a 4 días desde la embriogénesis hasta el adulto que pone huevos y su tamaño va aumentando proporcionalmente durante cada etapa larval L1, L2, L3 y L4 11. Después del período reproductivo, los nematodos pueden vivir varias semanas más, antes de morir de senescencia 12. C. elegans tiene dos sexos naturales; XO machos y XX hembras hermafroditas. Los hermafroditas son simplemente hembras autofértiles y su puesta de huevos está entre los 300-350 por un solo animal, sin embargo, cuando ambos sexos están presentes, la hembra es muy receptiva y se aparea con el macho, en este caso la progenie es mucho más alta y logra producir más de 1000 descendientes cuando se aparea 16,17.
Susceptibilidad bacteriana en el modelo Caenorhabditis elegans
Uno de los primeros agentes infecciosos utilizados en el modelo de C. elegans es la bacteria Pseudomonas aeruginosa y Pseudomonas fluorescens quien tiene la capacidad de causar muerte en los nematodos en la cepa silvestre N2 18,19. La muerte de los nematodos está mediada por las toxinas de esta bacteria segregadas durante la infección 20. Un estudio revela que el cianuro de hidrógeno es un factor tóxico producido por P. aeruginosa y es responsable de la muerte del nematodo 21. Por otro lado algunas cepas de Pseudomonas spp pueden causar un efecto letal que se asocia con una parálisis neuromuscular 22.
Así mismo la enterobacteria Salmonella typhimurium, es capaz de infectar y matar a C. elegans23. Las bacterias se acumulan en la luz intestinal y los nematodos mueren en el transcurso de varios días 24. En consecuencia Salmonella enteritidis causa una infección persistente en el intestino de C. elegans y desencadena la muerte celular programada (PCD) 25. Es importante mencionar que gracias al modelo C. elegans - Salmonella es posible identificar factores de virulencia nuevos y previamente conocidos 26. Más aun, diferentes especies de Burkholderia spp matan a este nematodo utilizando una parálisis mediada por endo-toxinas 27. Uno de los estudios sobre Burkholderia pseudomallei y C. elegans mostró que la fuerza de la virulencia de diferentes aislados clínicos es similar tanto en nemato-dos como en vertebrados superiores, lo que sugiere la presencia de un mecanismo evolutivamente conservado 28.
Garsin D. y colaboradores demostraron que las bacterias Gram positivas como En-terococcus faecalis y Streptococcus pneumoniae matan a Caenorhabditis29, además, Streptococcus pyogenes asesina al nematodo, tanto en medios líquidos como en sólidos, esta muerte está mediada por peróxido de hidrógeno y para que el nematodo muera se requieren bacterias vivas, pues en algunos casos él se ve afectado por microorganismos incluso cuando están muertos, sin embargo, en algunas ocasiones la producción de cata-lasa puede inhibir la muerte. A diferencia de las infecciones con S. typhimurium o P. aeruginosa no se observa acumulación bacteriana de S. pyogenes en el interior C. elegans, lo que excluye la participación de procesos similares de infección por cada bacteria 30. Por el contrario Staphylococcus aureus extermina al nematodo en el transcurso de varios días en un proceso que se correlaciona con la acumulación de bacterias dentro de su tracto digestivo 31.
Otro de los hallazgos importantes es que algunos agentes pueden inducir cambios morfológicos en el nematodo como Microbacterium nematophilum, pues estas bacterias se adhieren a la cutícula rectal y posanal de los nematodos susceptibles, e inducen una hinchazón local sustancial del tejido hi-podérmico subyacente. La hinchazón conduce al estreñimiento y al crecimiento lento del animal infectado, pero la infección no es letal 32. En este orden de ideas Serratia marcescens, también es capaz de infectar al nematodo e inducir una respuesta inmune dentro del organismo del nematodo 33. Yersinia pestis, la bacteria causante de la peste bubónica fue utilizada en este invertebrado para determinar la formación de Biofilm dentro del nematodo, los resultados arrojan que este microorganismo obstruye las vías de alimentación causando su muerte, de hecho, algunas cepas de C. elegans mutantes son resistentes a la formación de biopelícu-las, por lo tanto, ofrece un sistema experimental potencialmente útil para investigar interacciones mediadas por Biofilm entre bacterias e invertebrados 34,35.
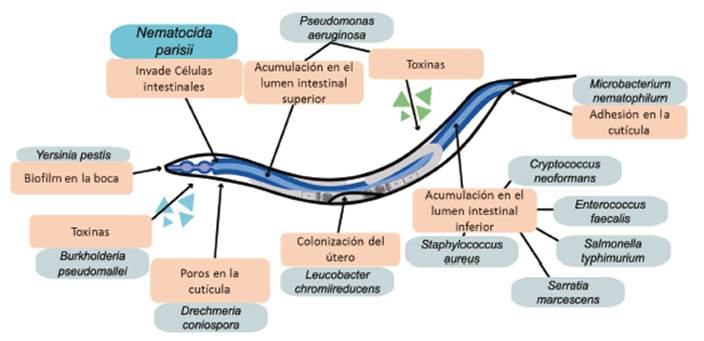
Otras investigaciones sugieren que cuando C. elegans es alimentado con; Streptoverticillium albireticuli, Bacillus megaterium, Shewanella frigidimarina, Shewanella massilia, Aeromonas hydrophila, Photorhabdus luminescens, Xenorhabdus nematophila, dichos microorganismos generan una reacción nematicida 19,36. Uno de los estudios que resumen algunas bacterias que afectan al nematodo por la acumulación bacteriana en cavidades corporales se expresa en la Figura 2, a través de un diagrama anatómico 37.
Fuente: Tomado y Modificado de: Hodgkin J. y colaboradores 37.
Figura 2 Microorganismos nematicidas en C. elegans.
Es importante mencionar que cuando los nematodos se alimentan con su dieta estándar de laboratorio (la cepa OP50 de Escherichia coli), las bacterias no se encuentran dentro de la luz intestinal hasta 48 horas después de la última etapa larval (L4), y no provocan daño tisular extenso hasta el día de su muerte, alguna inhibición en su ciclo de vida o morfología común; por el contrario funciona como una fuente de alimento prolongando a diferencia de las bacterias mencionadas anteriormente que causan la muerte o algún perjuicio en el nematodo 33. No obstante, aún no se conoce un perfil completo de la reacción de C. elegans ante todos los microorganismos o agentes infecciosos, sin embargo, se sabe que Bacillus subtilis, Erwinia chrysanthemi, Agrobacterium tumefaciens, Brucella sp cepa 96-566 y Mycobacterium fortuitum ATCC 6841 no causan enfermedades evidentes y por consiguiente no asesinan al nematodo 19,29. Por lo anterior se puede afirmar que estos agentes y muchos otros más sin estudiar pueden ser útiles para alimentar el nematodo. En la Figura 3, se puede observar la acumulación e infección bacteriana con respecto a aquellas bacterias que no afectan el nematodo.
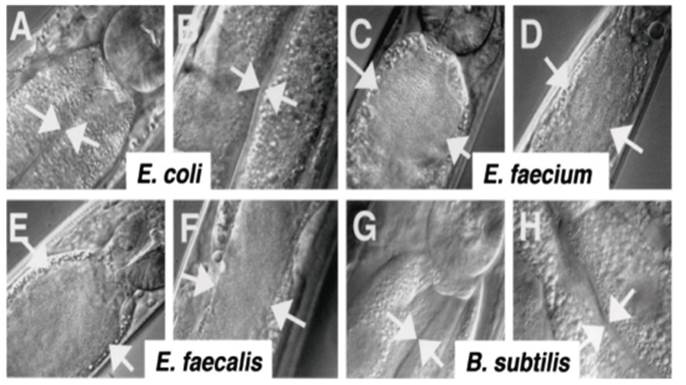
Fuente: Tomado de: Garsin DA. y colaboradores 29.
Figura 3 C. elegans alimentado con E. coli y B. subtilis no se ve afectado ni hay acumulación bacteriana en comparación del consumo con E. faecalis y E. faecium.
En cuanto a los agentes micóticos se sabe que C. elegans puede usar varias levaduras, incluidas Cryptococcus laurentii y Cryptococcus kuetzingii, como única fuente de alimento, produciendo tamaños de cría similares tras el consumo E.coli OP50. Sin embargo, la levadura patógena humana Cryptococcus neoformans mata al nematodo 38. De modo similar Candida albicans y Candida auris son ingeridas por C. elegans y establecen una infección letal persistente en la vía intestinal del nematodo 39. Es importante destacar que los componentes clave de la patogénesis de Candida sp en mamíferos también están involucrados en la muerte de C. elegans40. En los medios líquidos, las células de levadura experimentan un cambio morfológico para formar hifas, lo que resulta en una destrucción agresiva del tejido y la muerte del nematodo 41.
Mecanismos de defensa y patogenia de C. elegans ante infecciones mediadas por microorganismos
El contacto constante de C. elegans con microbios transmitidos por el suelo sugiere que este nematodo debe haber desarrollado respuestas protectoras contra los patógenos. Inicialmente no se tenía claro cómo funcionaba la inmunidad de este animal, es por ello que Mallo G. y colaboradores buscan comprender el sistema inmune de este invertebrado y realizan una infección con Serratia marcescens, obteniendo como resultado la expresión de gran cantidad de genes tras el contagio 33. Actualmente se sabe que C. elegans puede protegerse de los patógenos a través del uso de barreras físicas, proteínas efectoras y vías de señalización, la Tabla 1, resume estos mecanismos.
Tabla 1 Mecanismos de defensa de C. elegans. PNL: neuropéptidos, CNC: caenacinas, MAPK: proteína quinasas activadas por mitógenos, TGF- β: Factor de Crecimiento Transformante β, Daf-2: Gen que codifica un receptor insulínico en C. elegans. IGF-I: factor de crecimiento insulínico tipo I, TLR: Toll Like Receptor, ABF: Proteínas factor antibacteriano.
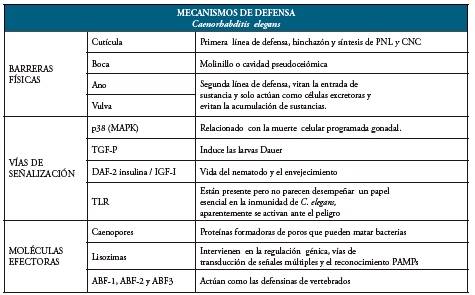
Fuente: Elaboración propia.
Las defensas físicas están mediadas inicialmente con la cutícula quien es la primera defensa del nematodo contra cualquier agente infeccioso que encuentre 42. La epidermis de C. elegans está en contacto con numerosos patógenos, existen innumerables microbios bacterianos y fúngicos que atacan a los nematodos después de una unión inicial a la cutícula, causando por ejemplo hinchazón o en otros casos la expresión de genes antimicrobianos que codifican neuropéptidos, (PNL) y una familia de péptidos estrechamente relacionados llamados caenacinas (CNC), 43. Cuando el ingreso patogénico va más allá de la cutícula se pueden ver involucrados la boca, el ano, la vulva o las aberturas sensoriales. Los microbios que ingresan por la boca se encuentran inmediatamente con el molinillo, formado por tres pares de células musculares que se contraen simultáneamente, con el fin de romper los agentes infecciosos a medida que pasan de regreso al intestino. Las cepas mutantes que carecen de molinillo son más susceptibles a ciertas bacterias 42.
Se sabe que los nematodos no acumulan normalmente bacterias en el intestino, sino que expulsan las mismas a través del ano, es por ello que al observar acumulación de gérmenes dentro de C. elegans es un signo claro de infección. Sin embargo una pequeña fracción de agentes infecciosos puede pasar intactos a través de la cutícula y entrar al intestino 42. Una vez en el intestino, algunas bacterias patógenas son capaces de proliferar como se mencionó anteriormente y matar a C. elegans mediante un proceso infeccioso o con el uso de toxinas. Por otro lado, este modelo produce una amplia variedad de proteínas y péptidos antimicrobianos potenciales, muchos de estos no tienen equivalentes estructurales en otras especies, y su espectro real de actividades aún no se ha probado 43.
Las primeras proteínas descrita fueron las de tipo saposina (SPP; también conocidas como caenopores, una familia numerosa, con 28 genes diferentes que codifican al menos 33 proteínas distintas), 44. Los caenopores comparten características estructurales y funcionales con los amebapores y con las proteínas citotóxicas de los vertebrados como la granulisina y NK-lisina, en común estas proteínas son formadoras de poros que pueden matar bacterias, se expresan predominantemente en el intestino de C. elegans y su activación es modulada por bacterias patógenas específicas 43,44.
Una segunda clase de proteínas antimicrobianas bien caracterizadas son las lisozimas, de las cuales hay 15 en C. elegans, se cree que se secretan en la luz intestinal, donde actúan directamente sobre los microbios, de hecho, ciertas lisozimas han demostrado ser importantes para la resistencia del nemato-do a patógenos bacterianos Gram-positivos y Gram-negativos; estas, muestran patrones complejos de regulación génica y parecen ser objetivos de vías de transducción de señales múltiples y el reconocimiento de patógenos 43.
Tras diversas investigaciones se han descrito seis péptidos del factor antibacteriano homólogo (ABF) en C. elegans. Estos péptidos son algo similares a las defensinas de vertebrados. Basado en su secuencia, se cree que las proteínas ABF mata microbios por disrupción de membrana, las cuales se expresan conjuntamente (ABF-1, ABF-2 y ABF-3); ABF-2 principalmente en los tejidos faríngeos, mientras que ABF-3 se detectan en el intestino y ABF1 en ambas zonas 33. Cabe resaltar que el ABF-2 recombinante tiene una actividad antimicrobiana de amplio espectro demostrada In Vitro, con el mayor efecto observado en bacterias Grampositivas 45.
En cuanto a las vías de señalización algunos estudios demuestran que los genes inducidos en infecciones por patógenos de mamíferos son homologas y se expresan durante la infección en el nematodo, 38. Por ejemplo, en el ser humano una de las vías más relevantes son la ruta p38 MAP quinasa y la vía de la muerte celular programada, tras diversos estudios se identificaron dos genes homólogos en el nematodo de la ruta de la proteína quinasa activada por mitógeno p38 (MAPK): NSY-1 y SEK-1 quienes codifican una MAPK quinasa quinasa y una MAPK quinasa respectivamente 31,42,46. Asimismo, la disminución de PMK-1 un "p38 MAPK" en nematodos mutados, conduce a mayor susceptibilidad de infección lo anterior permite concluir que esos genes traducen una señal de defensa en el animal 42.
Tras lo anterior algunos investigadores afirman que esta vía de señalización parece ser conservada a través de la filogenia, se sabe que la ruta de señalización MAPK de p38 de mamífero media las respuestas al estrés y se activa por choque térmico, luz ultravioleta (UV), las citoquinas proinflamatorias como la IL-1 o TNF-a, o Lipopolisacáridos bacterianos, 46. La consecuencia de la activación de p38 en células de mamíferos es la muerte celular programada (PCD) 38, En C. elegans, existen homólogos que inducen la PCD activada por estrés y ocurre en las células gonadales del nematodo, en respuesta a la irradiación Y o a la alimentación de Salmonella. Probablemente dicha muerte celular puede inducirse en otros agentes infecciosos patógenos para el invertebrado. Más aún cuando el animal consume E. coli OP50, que es su alimento habitual en el laboratorio se produce un bajo nivel de muerte celular programada gonadal 25.
En un estudio dirigido a examinar varios mutantes definidos de C. elegans que poseen pérdida de la función en los genes esp-2 / sek-1 y esp-8 / nsy-1 , relacionados con p38 MAPK quinasa parecen estar directamente involucrados en defensa de nematodos contra múltiples patógenos, pues tras la pérdida de dichos genes se encuentra que hay mayor susceptibilidad de muerte en todas las etapas de desarrollo a comparación de la cepa N2, lo que indica que el animal posee una vía de señalización conservada de la "p38 MAP quinasa" importante en la inmunidad del nematodo 31.
Otra vía de señalización asociada a infecciones en C. elegans es la relacionada con el factor de crecimiento transformante de tipo TGF-3 quien induce la formación de larvas Dauer en estos estadios la larva no se alimenta y presenta una cutícula más gruesa como defensa a factores ambientales externos 46. Una de las ultimas vías analizadas es la DAF-2 insulina / IGF-I que se asocia directamente con la vida del nematodo y el envejecimiento 47. Por otro lado, la vía tipo Toll aunque está presente de manera ortóloga no parece desempeñar un papel esencial en la mediación de la inmunidad innata de C. elegans. Esto se concluye porque los nematodos con una mutación en tol-1, pik-1, ikb-1 o trf-1 no difieren significativamente de los animales de tipo salvaje en respuesta a diversos patógenos. Sin embargo se relaciona a comportamiento, pues la vía Toll suele asociarse como mecanismo de defensa para mantener a C. elegans alejado de sustancias potencialmente dañinas 42.
Uso de C. elegans como modelo para validar y demostrar la infección por bacterias
Los organismos invertebrados son cada vez más reconocidos para el estudio de la patogénesis que causan diferentes microorganismos durante una infección. El nematodo C. elegans es susceptible a una variedad amplia de infecciones por bacterias, hongos y virus, lo que lo convierte en un modelo útil para el estudio de patógenos y simbiontes 19. Esta relación creciente entre C. elegans y microorganismos hace que el nematodo sea considerado un modelo de estudio atractivo a nivel de la interacción huésped-patógeno 42, además, la secuenciación completa del genoma de C. elegans indica que al menos el 36% de las 19,000 proteínas predichas tienen homólogos en humanos 22, por lo tanto, a pesar de la enorme distancia evolutiva entre humanos y nematodos, C. elegans es un modelo interesante para estudios de numerosos procesos de enfermedades infecciosas.
Sincronización de C. elegans como método inicial antes de cualquier ensayo
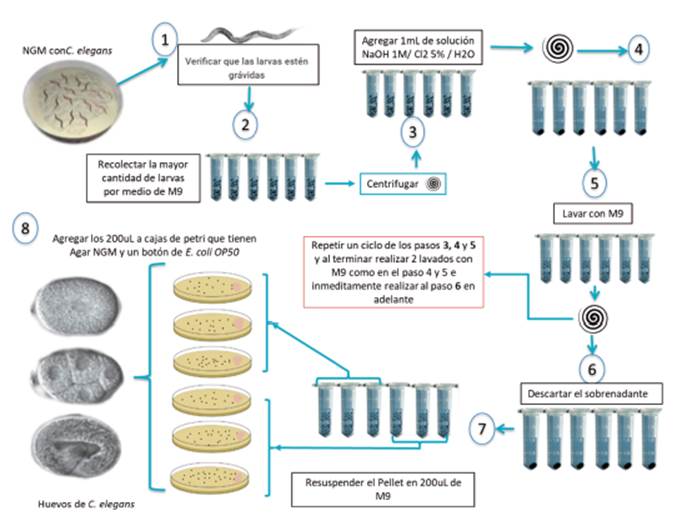
Una de las principales ventajas del modelo de C. elegans es su fácil manipulación en el laboratorio. Inicialmente, se pueden obtener nematodos en un mismo estadio del ciclo de vida de una manera rápida y sencilla, lo que permite obtener una población homogénea durante el estudio. Antes de realizar cualquier ensayo es necesario efectuar una sincronización de los nematodos, esto permite que los animales a estudiar se encuentren en su totalidad en el mismo estadio larvario, bajo las mismas condiciones de nutrición y ambiente. Además, con la sincronización, se obtienen una gran cantidad de huevos que permiten realizar estudios a nivel embriológico en el modelo. Por otro lado, estos huevos se pueden diferenciar y se obtienen poblaciones en un mismo estadio larvario, estas larvas se pueden exponer a una sustancia o una condición definida, para identificar cambios concluyentes en las características fisiológicas del nematodo 48,49.
La sincronización es clave para los estudios en el modelo infeccioso de C. elegans, pues permiten evaluar todo tipo de respuestas inmunes, ciclos de vida (antes, durante y después de la infección), observar cambios específicos a través del crecimiento y desarrollo pre y post contagio bacteriano, así como analizar la variabilidad morfológica o incluso la muerte. Todas estas características son esenciales para poner a prueba un tratamiento que promueva la mejoría del animal luego del contacto con el microorganismo problema. En la Figura 4, se evidencia el paso a paso de la sincronización según lo estandarizado por el grupo de investigación de Biotecnología y Genética UCMC de la Universidad Colegio Mayor de Cundinamarca luego de analizar el protocolo del Wormbook y diferentes protocolos propuestos por otros investigadores 8,45.
Ensayos fisiológicos en el modelo de infección de C. elegans
Los ensayos convencionales son muy útiles para determinar si los nematodos están infectados y si esta infección produjo algún cambio a nivel fenotípico o fisiológico, estos ensayos comienzan desde observar cambios por microscopia, hasta determinar las características de comportamiento que presenta el modelo como variaciones en longevidad, longitud, movilidad y reproducción. Los protocolos de estos ensayos son tomados del Wormbook, sin embargo, se deben realizar las estandarizaciones dependiendo de la cepa a trabajar así como las variaciones según el enfoque de la investigación 49.
Longevidad
C. elegans es un modelo popular para estudiar el envejecimiento o longevidad debido a su corta vida útil y capacidad de rastreo genético, la medición de longevidad es un método directo que determina las consecuencias del envejecimiento y la muerte. Este procedimiento se fundamenta en contar nematodos vivos y muertos en poblaciones sincronizadas a intervalos de tiempo regulares que se exponen a una condición como por ejemplo un agente patógeno, para luego generar curvas estadísticas de la vida útil e ilustrar los porcentajes de animales vivos en las poblaciones a lo largo del tiempo 50.
En un estudio realizado por Blanco A. y Kim S. luego de la sincronización de C. elegans, permitió evaluar la esperanza de vida tras la exposición del nematodo con E. coli y B. subtilis,Figura 5A y 5B. La evaluación se realizó durante el desarrollo del nematodo utilizando una cepa transgénica que contiene un marcador de envejecimiento (sod-3 unido con la proteína verde fluorescente (GFP)) cuya expresión disminuye con la edad. Este transgénico puede usarse para medir la edad fisiológica del nematodo, Figura 5C. Estos investigadores encontraron que los nematodos alimentados con B. subtilis viven más que los nematodos cultivados en E. coli y como se indicó anteriormente B. subtilis no afecta la vida del nematodo en comparación a otras bacterias que probablemente al usar esta cepa de C. elegans pueden disminuir significativamente su esperanza de vida 51.
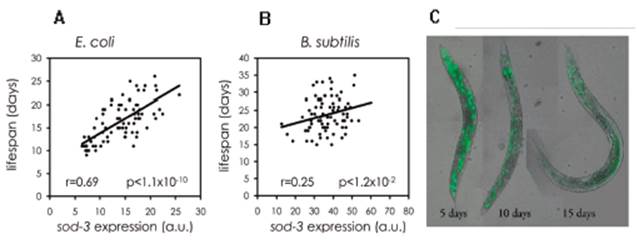
Fuente: Tomado y modificado de: Sanchez-Blanco A y colaboradores 51.
Figura 5 Esperanza de vida de C.elegans sod-3 marcado con GFP, (A): C.elegans alimentado con E.coli, (B) C.elegans marcado con B. subtilis (C) C.elegans sod-3 marcado con GFP.
Movilidad y Longitud
Para la movilidad se determinan las ondulaciones realizadas por el nematodo en estadio L4 durante 30 segundos y de la misma forma para el ensayo de longitud se miden los nematodos, usualmente tratados con levamisol con el fin de provocar la inmovilización de los nematodos y ser medidos en microscopio de retícula 52,53.
En un estudio realizado por Joshua P. y colaboradores; Figura 6A y 6B, se puede observar como el nematodo tiene ondulaciones aberrantes cuando consume Yersinia pseudotuberculosis en comparación de un movimiento sinusoidal normal de los nematodos cuando se exponen a E. coli OP50, lo cual indica que algunas bacterias pueden afectar significativamente su movilidad y si este modelo se usa con el fin de tratar con Rodriguez et al. Caenorhabditis elegans como modelo de infección para el estudio de antimicrobianos antibióticos a Y. pseudotuberculosis se esperaría que el animal recobrara su movimiento o mejorara el mismo a través del tiempo dependiendo de la dosis del fármaco 35. Por otro lado en un estudio realizado por Sugawara T. y colaboradores; Figura 6C., se observa como Bifidobacterium longum afecta la longitud del nematodo en comparación con aquellos C. elegans que consumieron E.coli OP50, lo que indica también que hay probabilidad de que otros microorganismos repercutan en el crecimiento normal del nematodo. 54.
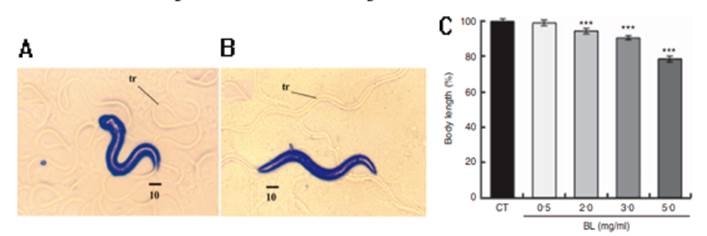
Fuente: Tomado y modificado de: Joshua GWP. y colaboradores 35 y Sugawara T. y colaboradores 54.
Figura 6 (A) C. elegans alimentado con Y. pseudotuberculosis genera un movimiento aberrante (curvas cerradas) (B) C. elegans cultivado en E. coli OP50 genera un movimiento sinusoidal normal. (C). Efectos de diferentes concentraciones de Bifidobacterium longum (BL) en el crecimiento de C.elegans, E.coli OP50 es el control (CT).
Reproducción
Para los ensayos de reproducción, una vez los nematodos son sincronizados y alcanzan el estadio larvario L4, se debe realizar una transferencia a medios NGM que contiene las bacterias en estudio, después del tratamiento, se deposita un nematodo por caja y diariamente cada nematodo es analizado durante su periodo reproductivo (4 días), las placas con los huevos puestos por los nematodos se observan un par de días después de la eclosión de los huevos, en donde se deben contar los nematodos en estado larvario L2 y L3; a la vez que se verifica la fertilidad de los huevos por medio del número de larvas eclosionadas. Es importante estandarizar el ensayo de reproducción como lo reportan Bustos AVG. y colaboradores en el 2017, quienes describen los ensayos de reproducción en una cepa mutante NB327 de C. elegans modelo para el estudio de cáncer de pulmón y próstata 52.
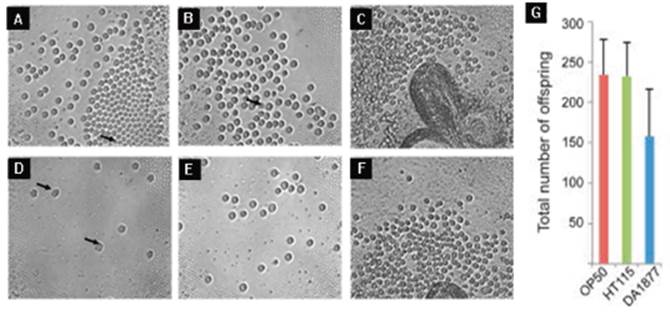
En el estudio realizado por MacNeil L y colaboradores. estudiaron el tamaño de la cría de C. elegans expuestos a tres bacterias diferentes, E. coli OP50, E. coli HT115 y Comomonas DA1877, encontraron que la descendencia disminuye o reduce la fecundidad significativamente entre E.coli y Comomonas como se observa en la Figura 7G, por otro lado Sharika L. y colaboradores; Figura 7A-F descubrieron que los exposición a Staphylococcus aureus y Vibrio alginolyticus tienen una diferencia en la morfología, el tamaño y la activación de los espermatozoides provenientes de machos C. elegans en comparación con la dieta convencional con E. coli OP50 55,56.
Evaluación de la infección en el Modelo de C. elegans
La utilidad de C. elegans como organismo modelo de infección es el resultado de su trazabilidad genética, tiempo de generación rápido, facilidad de propagación, un mapa de linaje celular bien definido y un genoma completamente secuenciado que contiene una gran cantidad de genes ortólogos de vertebrados 42. En comparación con otros animales este nematodo en particular tiene varias ventajas sobre otros modelos de mamíferos; no plantea tantas preocupaciones éticas como el uso de modelos vertebrados y los nematodos son fáciles de manipular en el laboratorio porque son lo suficientemente pequeños como para analizarlos en placas de microtitulación estándar 57.
Además la transparencia del cuerpo de C. elegans permite examinar el desarrollo y los cambios a nivel de una sola característica por medio de proteínas o sustancias fluorescentes en células vivas, de hecho la gran ventaja de este modelo como propuesta de infección radica en la capacidad que tienen las bacterias de ser teñidas fácilmente con sustancias fluorescentes o marcarse con GFP lo que facilita su observación al interior del nematodo. En la Figura 8, se observa a Pseudomonas putida marcada con GFP y a E. faecalis teñida con DAPI. Una de las propuestas del grupo de Biotecnología y Genética UCM de la Universidad Colegio Mayor de Cundinamarca radica en la posibilidad de usar tinciones más sencillas para observar bacterias al interior del nematodo diferente al uso de plásmidos y la proteína verde fluorescente.
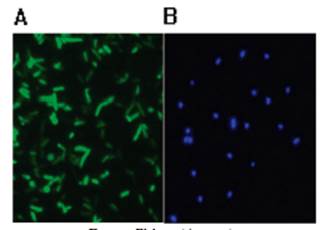
Fuente: Elaboración propia.
Figura 8 Bacterias fluorescentes. (A) Pseudomonasputida marcada con GFP. Tomado de: Duncan D. y colaboradores 58 y E. faecalis teñida con DAPI.
En un estudio elaborado por Aballay A, y colaboradores Figura 9 A-F, se puede observar la acumulación bacteriana y la presencia de las mismas por medio de la fluorescencia pues las bacterias E. coli OP50, S. typhimurium y P. aeruginosa fueron marcadas con plásmidos de expresión que llevan GFP, lo que permitió observarlas claramente en el intestino del nematodo por microscopio invertido y microscopia de fluorescencia, gracias a la transparencia del cuerpo de este modelo 24.
La fluorescencia de las bacterias marcadas con GFP también puede correlacionarse directamente con la inflamación del nemato-do luego de la infección, factor que es muy frecuente dependiendo del tipo de bacteria y de su colonización dentro del cuerpo del nematodo. Por ejemplo, en un estudio realizado por Sifri D. y colaboradores; Figura 9G y 9H, se observa la distensión del tracto digestivo de C. elegans alimentados con S. aureus, pero no con E. coli OP50. Gracias a la expresión del GFP los investigadores pueden correlacionar la morfología respectiva de los cocos o bacilos correspondientes a las bacterias en estudio y la respectiva respuesta frente a la inflamación en cada caso, concluyendo así que S. aureus afecta la morfología normal del modelo 31.
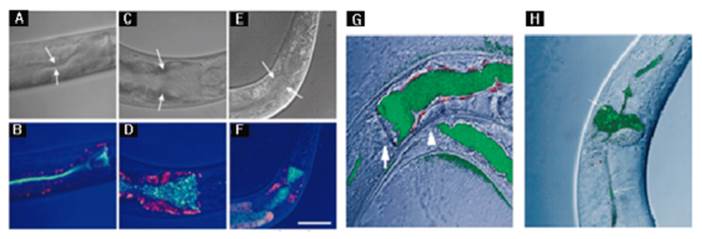
Fuente: Tomado de: Aballay A, y colaboradores 24 Y Sifri CD. y colaboradores 31.
Figura 9 Colonización bacteriana en el intestino de C. elegans (A, B) E. coli OP50 marcada con GFP por 72 h, (C, D) S. typhimurium marcada con GFP por 72 h, (E, F) P. aeruginosa marcada con GFP durante 24 h. (G) cocos abundantes compatibles a S. aureus dentro de C. elegans con distención del tracto digestivo. (H) Bacilos escasos compatibles con E.coli OP50 dentro de C. elegans sin distención del tracto digestivo.
Durante la evaluación de la infección los investigadores observaron que cuando los nematodos en etapa L4 se alimentan con una cepa bacteriana que es patógena, se encuentran bacterias intactas dentro de la luz intestinal aproximadamente a las 6 horas postcontagio, ellas proliferan rápidamente y en algunos casos se evidencia una distensión progresiva de la luz intestinal. Además, también reportan que los nematodos muestran pocos signos de infección durante las primeras 24 horas, su tasa de puesta de huevos es normal lo cual refleja que el estado general del nematodo es eficiente. Sin embargo, luego de este tiempo se observa que la locomoción del nematodo, el bombeo faríngeo y el forrajeo es anormal y posteriormente se evidencia una destrucción progresiva del epitelio intestinal y de la línea germinal, acompañado de una clara caída en la tasa de puesta de huevos después de 48 horas 31,33.
Después de todo lo anterior, los nematodos comienzan a morir, y quedan inmóviles alrededor de las 72 horas de contacto con el agente. Muchos nematodos muertos pierden toda la arquitectura celular aparente. Además, tras la infección en algunos casos se observa el llamado fenotipo de "bolsa de nematodos", en el cual los huevos de un hermafrodita grávido eclosionan internamente y las larvas resultantes consumen al progenitor 31. Es por ello que el tiempo de vida, así como el de infección pueden ser claves para poner a prueba antibióticos.
Mutantes para los estudios de infección en C. elegans
Actualmente, se han caracterizado mutantes que son susceptibles o resistentes a la muerte mediada por toxinas bacterianas o por infección. Por ejemplo los mutantes de C. elegans daf-2 de larga vida son resistentes a los patógenos bacterianos de mamíferos 59, y la cepa sek-1 (km4) provoca una mayor sensibilidad a diversos patógenos 57. Una de las ventajas que tiene el modelo y sus cepas mutantes es que la infección se hace evidente debido a que estas cepas poseen proteínas las cuales están marcadas usualmente con GFP y tras el consumo bacteriano en Rodriguez et al. Caenorhabditis elegans como modelo de infección para el estudio de antimicrobianos comparación con la cepa control de E. coli OP50 se puede observar fluorescencia ante aquellas bacterias que son perjudiciales para el nematodo.
Por ejemplo, Kniazeva M. y Ruvkun G. buscaron determinar si el desarrollo de los embriones de C. elegans adultos que tenía marcada la proteína sur-5 con GFP era normal o no al ser alimentado con E. coli OP50 y con Rhizobium huautlense, como resultado obtuvieron que los núcleos intestinales se encuentran anormalmente alargados y fragmentados tras el consumo de esta última bacteria, Figura 10. 60.
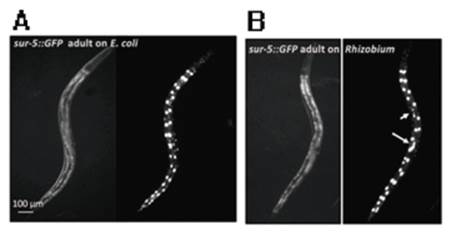
Fuente: Tomado de: Kniazeva M. y Ruvkun G.60.
Figura 10 (A) imágenes fluorescentes de C. elegans sur-5 GFP adulto alimentado E. coli OP50 desde el momento de la eclosión. (B) imágenes fluorescentes de C. elegans sur-5 GFP adulto alimentado con Rhizobium desde el momento de la eclosión. Las flechas largas y cortas apuntan a núcleos intestinales anormalmente alargados y fragmentados.
Por otro lado, también se ha demostrado que como mecanismo de defensa C. elegans deja de alimentarse en respuesta a los tóxicos ambientales o agentes que causan daño de forma dependiente de la dosis y reanuda la alimentación cuando se elimina el "tóxico" o la sustancia que causa daño en su organismo 27. Lo anterior sugiere que tanto la naturaleza del patógeno como la ruta de infección contribuyen a la inducción de genes de defensa específicos en C. elegans así como también inducir un tratamiento puede contribuir al mejoramiento de los signos presentados 61. Por esta razón, diversas investigaciones sugieren utilizar los medios Infusión Cerebro Corazón (BHI) o Luria Bertani (LB) para el cultivo de las bacterias que se van a exponer al nematodo, ya que se ha demostrado que otro tipo de medios de cultivo pueden aumentar la virulencia bacteriana y afectar notoriamente al nematodo 20,39.
Es importante mencionar que existen algunos requisitos a tener en cuenta para la funcionalidad de C. elegans como modelo huésped-patógeno, debido a que el nemato-do solo tiene defensas inmunes innatas, las preguntas dirigidas a comprender el sistema inmunitario adquirido deberán abordarse en un modelo de vertebrado. C. elegans también carece de un tipo de célula fagocítica móvil, como el macrófago de mamífero o el hemocito de Drosophila. Además, no todos los genes de inmunidad innata de vertebrados tienen ortólogos en este modelo. Estas limitaciones subrayan la necesidad de una variedad de sistemas de modelos complementarios de huésped-patógeno para comprender a fondo la complejidad total de las interacciones virulencia-defensa, sin embargo es importante considerar que este modelo puede ser muy útil para realizar tamizajes en el estudio de sustancias candidatas a fármacos 42.
Finamente, otro de los factores importantes para seleccionar este nematodo como modelo eucariota es la capacidad que tiene de crio preservarse a largo plazo, lo que permite realizar variados estudios con diferentes cepas transgénicas o mutantes 12. Por otro lado, dado a los estudios realizados en el nematodo infectado con patógenos, con el fin de comprobar la función y efectividad de compuestos o estandarizar alternativas de tratamiento eficientes, este animal puede ser propuesto como modelo In Vivo de infecciones. Se podrían evaluar sustancias, extractos o péptidos antimicrobianos entre otros, pues en él se pueden aplicar infinidad de ensayos arrojando resultados de manera rápida y concluyente 40,57. Además, es importante reconocer que durante muchos años C. elegans se ha utilizado para comprender enfermedades como Alzheimer, Diabetes Mellitus entre otros y ha proporcionado numerosos avances en estas áreas del conocimiento 12.
Conclusiones
Diversos son los estudios realizados hasta el momento enfatizados en demostrar que pese a que C. elegans se alimenta naturalmente de bacterias no todas pueden ser relativamente sanas o promover su ciclo de vida normal, de hecho a lo largo de este artículo se evidenció que el nematodo puede verse afectado al consumir ciertas bacterias en comparación a su alimento habitual en el laboratorio (la E. coli OP50), lo que indica que pese a la distancia evolutiva sigue existiendo una conexión entre una especie y otra, sobre todo en el caso de la sensibilidad a infecciones que atacan a diferentes mamíferos.
Además todo esto contribuye a la posibilidad de usar este nematodo como modelo In vivo de infecciones y no solamente con el fin de determinar aspectos netos de la inmunidad o las repercusiones que los microorganismos tienen en la vida del modelo, sino que es posible que a nivel de ciencia e investigación este pequeño invertebrado sea útil para descartar o no medicamentos en estudio en diferentes campos de investigación, sobre todo aquellos que involucran los sistemas que conforman a C. elegans.
Las infecciones son cada vez más persistentes y la necesidad de explorar nuevas sustancias para contrarrestarlos es evidente en la actualidad, a través de este modelo se puede contribuir sustancialmente para validar antibióticos candidatos de manera rápida y sencilla y al mismo tiempo evitar largas experimentaciones que en un final fracasan, porque comprometen la vida de otros modelos animales que tiene un periodo de vida más largo o que presenta restricciones éticas.
El semillero de Biotecnología y Genética UCMC de la Universidad Colegio Mayor de Cundinamarca propone utilizar a este modelo como fase inicial de experimentación a nivel de antibióticos, usando las técnicas In vitro convencionales y comparándolas con la infección y la resolución de la misma dentro del nematodo tras la administración del fármaco en estudio, para posteriormente hacer estudios más especializados con otros animales solo si el medicamento no compromete la vida del nematodo. De hecho, actualmente este grupo tiene en curso una investigación que tiene como objetivo validar y estandarizar a C. elegans como modelo In Vivo de infección para el estudio de antimicrobianos.