Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Lasallista de Investigación
Print version ISSN 1794-4449
Rev. Lasallista Investig. vol.8 no.1 Caldas Jan./June 2011
Artículo original / Original article / Artigo original
Identificación serológica y molecular de virus del tomate de árbol (Solanum betaceum) en cultivos de Córdoba (Nariño, Colombia)*
Serological and molecular identification of viruses in tree tomato (Solanum betaceum) in crops from Córdoba (Nariño, Colombia)
Identificação serológica e molecular de vírus do tomate de árvore (Solanum betaceum) em cultivos de Córdoba (Nariño, Colômbia)
Mora****, Pablo Andrés Gutiérrez***** y Mauricio Marín Montoya******
* Artículo producto de la investigación financiada por el proyecto COLCIENCIAS Código 1118-405-20317 y por la Universidad Nacional de Colombia sede Medellín a través del proyecto DIME BICENTENARIO 2008-2009: 20101007745.
** Bióloga, Universidad de Nariño, Aspirante a Magíster en Biotecnología.
*** Ingeniera Agrónoma, Magíster en Biotecnología, Docente Facultad de Ciencias Administrativas y Agropecuarias, Corporación Universitaria Lasallista. Es miembro del Grupo de Investigación en Producción, Desarrollo y Transformación Agropecuaria.
**** Bióloga, Magíster en Ciencias, Docente Facultad de Ciencias Exactas y Naturales, Universidad de Nariño.
***** Biólogo, PhD en Bioquímica, Docente Facultad de Ciencias, Universidad Nacional de Colombia sede Medellín.
****** Ingeniero Agronómo, PhD en Fitopatología, Docente Facultad de Ciencias, Universidad Nacional de Colombia sede Medellín.
Correspondencia: Mauricio Marín Montoya. e-mail: mamarinm@unal.edu.co
Artículo recibido: 03/12/2010; Artículo aprobado: 25/04/2011
Resumen
Introducción. Las enfermedades causadas por virus se han convertido en un factor limitante para los cultivos de tomate de árbol presentes en diferentes regiones de Colombia, incluyendo aquellos del departamento de Nariño. Estudios previos apuntan a un posible potyvirus y a diferentes virus isométricos como asociados a estas enfermedades, aunque su identidad taxonómica en el nivel de especie y su incidencia no se habían determinado. Dicha situación fue abordada en este estudio utilizando pruebas de ELISA, RT-PCR y secuenciación de la cápside viral a partir de muestras de cinco cultivos afectados por la virosis en el municipio de Córdoba (Nariño). Resultados. Los resultados indicaron la presencia de potyvirus en todas las muestras analizadas, mientras que los virus CMV (33%), ToMV (50%) y PLRV (75%) presentaban una alta incidencia en los cultivos de la región. El análisis molecular permitió identificar al potyvirus como miembro de la especie PVY, compartiendo niveles de identidad superiores al 92% con cepas obtenidas en cultivos de papa de diferentes países; mientras que las secuencias del polerovirus presentaron identidades superiores al 98% con cepas de PLRV de este mismo cultivo. Conclusión. Esta investigación confirma la ocurrencia de un complejo viral asociado a la virosis del tomate de árbol en el departamento de Nariño, y con base en los análisis de secuencias, plantea la posible infección cruzada de cepas de PVY y PLRV entre los cultivos de papa y tomate de árbol de la región, lo cual requiere confirmación experimental mediante evaluaciones de patogenicidad.
Palabras clave: CMV, ELISA, PLRV, potyvirus, PVY, secuenciación, ToMV.
Abstract
Introduction. The diseases caused by viruses have become an obstacle for tree tomato crops in several Colombian regions, including those from the Nariño province. Previous studies pointed at a possible potyvirus and at several isometric viruses as associated to these diseases, though their taxonomic identity in the species level and their incidence had not been determined yet. An approach to this situation was made by the use of ELISA, RT-PCR and viral sequence capsid tests from samples brought from five crops affected by viruses in Córdoba (Nariño). Results. The results indicated the presence of potyvirus in all of the samples analyzed, while CMV virus (33%), ToMV (50%) and PLRV (75%) had a great incidence in the crops from the region.The molecular analysis allowed the identification of potyvirus as a member of the PVY species, sharing identity levels above 92% with strains obtained in potato crops from several countries, while the polerovirus sequences obtained identities above 98% with PLRV strains from the same crop. Conclusion. This research work confirms that there is a viral complex associated to viruses in tree tomatoes from the Nariño province and, base on the sequence analysis, exposes the possible crossed infection of PVY and PLRV strains in potato and tomato plantations in the region, a fact which requires an experimental confirmation by pathogenicity evaluations.
Key words: CMV, ELISA, PLRV, potyvirus, PVY, sequence, ToMV.
Resumo
Introdução. As doenças causadas por vírus se converteram num fator limitante para os cultivos de tomate de árvore presentes em diferentes regiões da Colômbia, incluindo aqueles do departamento de Nariño. Estudos prévios apontam a um possível potyvirus e a diferentes vírus isométricos como sócios a estas doenças, ainda que sua identidade taxonômica no nível de espécie e sua incidência não se tinham determinado. Dita situação foi abordada neste estudo utilizando provas de ELISA, RT-PCR e na seqüencia da cápside viral a partir de mostras de cinco cultivos afetados pela viroses no município de Córdoba (Nariño). Resultados. Os resultados indicaram a presença de potyvirus em todas as mostras analisadas, enquanto os vírus CMV (33%), ToMV (50%) e PLRV (75%) apresentavam uma alta incidência nos cultivos da região. A análise molecular permitiu identificar ao potyvirus como membro da espécie PVY, compartilhando níveis de identidade superiores a 92% com cepas obtidas em cultivos de papa de diferentes países; enquanto as seqüências do polerovirus apresentaram identidades superiores ao 98% com cepas de PLRV deste mesmo cultivo. Conclusão. Esta investigação confirma a ocorrência de um complexo viral sócio à viroses do tomate de árvore no departamento de Nariño, e com base nas análises de seqüências, propõe a possível infecção cruzada de cepas de PVY e PLRV entre os cultivos de papa e tomate de árvore da região, o qual requer confirmação experimental mediante avaliações de patogenia.
Palavras Importantes: CMV, ELISA, PLRV, potyvirus, PVY, seqüencia, ToMV.
Introducción
Las enfermedades de plantas causadas por virus producen síntomas como disminución en el crecimiento, deformación de tejidos, mosaicos foliares y reducción en la producción, lo que lleva a grandes pérdidas en el rendimiento y/o la longevidad de una amplia variedad de cultivos de importancia agrícola. Las estrategias empleadas para su manejo se basan en prevenir que los virus lleguen o se establezcan en los cultivos mediante el manejo biológico o químico de los vectores que los transmiten, el uso de variedades resistentes, el empleo de material de siembra certificado y las prácticas de exclusión1.
En Colombia, los cultivos de solanáceas que se ven más afectados por estos patógenos son el de la papa (Solanum tuberosum), con enfermedades como el mosaico rugoso causado por PVY, el enrollamiento foliar (PLRV) y el amarillamiento de las venas (PYVV); y el de tomate de árbol (Solanum betaceum), con una enfermedad que en forma genérica se denomina como "Virosis" y que produce síntomas variados, pero cuyos agentes causales aún no han sido plenamente establecidos, aunque existen diferentes reportes que indican que al menos uno de los virus asociados a este cultivo pertenece a la familia Potyviridae2,3. Estos virus se caracterizan por tener forma de partícula flexuosa helicoidal de 730 x 11 nm, un genoma de ARN monocatenario positivo de 10 kb y diversos mecanismos de transmisión, entre los que se destacan los áfidos y los medios mecánicos. Recientemente, además de potyvirus, se han identificado en cultivos de varias regiones del país, diferentes virus isométricos de los géneros Polerovirus, Cucumovirus y Alfamovirus, entre otros4.
El tomate de árbol se ha convertido en una de las alternativas viables para reemplazar los cultivos tradicionales que dejan bajos niveles de rentabilidad y para sustituir los cultivos ilícitos en diversas regiones del país, dadas sus excelentes características organolépticas y su gran aporte nutricional, que lo hacen muy atractivo para la industria de bebidas y alimentos5. En Nariño, 18 municipios cultivan tomate de árbol lo que lo constituye en uno de los frutales con mayor importancia económica en el departamento. Los principales productores de tomate de árbol son Samaniego, Funes, Contadero y Santacruz, mientras que municipios como Córdoba, El Rosario y Taminango, a pesar de tener una baja producción, se consideran promisorios para la expansión de su siembra, por contar con condiciones climáticas y edáficas propicias para su cultivo6. Sin embargo, la fuerte incidencia y severidad de las enfermedades virales que se presenta en la actualidad en dichos municipios es una limitante significativa para el aumento de las áreas destinadas a la siembra de este frutal.
En Colombia la primera enfermedad viral del tomate de árbol se detectó en 1980 en Boyacá con la presencia del virus de la necrosis anular7, y en Antioquia con el mosaico del tomate de árbol8. En el departamento de Nariño, Betancourth; et al9, reportaron la presencia de un posible potyvirus que afecta los cultivos de tomate de árbol, caracterizado por la morfología de varilla flexuosa de 800 nm y su transmisión mediante áfidos de la especie Myzus persicae; sin embargo, su identificación en la especie era incierta.
La presencia de mezclas de virus que afectan el tomate de árbol se ha reportado en países como Nueva Zelanda y Ecuador, donde se han detectado miembros de las familias Potyviridae, Flexiviridae, Bromoviridae, Bunyaviridae, Luteoviridae y Comoviridae como agentes causales de las enfermedades virales de este cultivo10-12.
A pesar de la gran limitante que representan las "virosis" para el cultivo del tomate de árbol en Colombia, la identificación de las especies virales asociadas a los cultivos en las diferentes regiones del país es parcial. Esta situación es claramente inconveniente para el diseño de las estrategias de control de estas enfermedades y requiere ser abordada con celeridad. Apuntando a dicha necesidad, esta investigación tuvo como principal objetivo la determinación de los agentes causales de la "virosis" del cultivo de tomate de árbol en el municipio de Córdoba (Nariño), mediante la utilización de técnicas serológicas y moleculares.
Materiales y métodos
Localización. Las muestras de plantas de tomate de árbol con sintomatologías virales fueron colectadas en las veredas San Juan Chico, El Chair y Guacas del municipio de Córdoba (Nariño), que se encuentra ubicado a los 00° 51' 18" latitud Norte y 77° 31' 16" longitud Oeste, a una distancia de 90 Km de la ciudad de Pasto y a una altura de 2.800 m.s.n.m. Tiene una temperatura media de 12°C y precipitación media anual de 1.020 mm.
Descripción de sintomatologías virales. Se visitaron cuatro cultivos de tomate de árbol y en cada uno se tomaron 30 muestras aleatorias consistentes de cuatro hojas jóvenes. Se realizó una caracterización de las sintomatologías encontradas teniendo en cuenta el tejido foliar, frutos, flores y estado general de las plantas afectadas (presencia de enanismos, defoliaciones, etc.). Las muestras fueron transportadas al laboratorio de Biología celular y molecular de la Universidad Nacional de Colombia, Sede Medellín, para su análisis posterior.
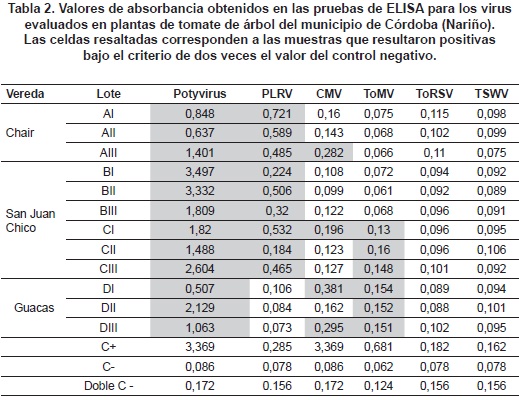
Detección serológica de los virus. Se realizaron pruebas de ELISA mediante el uso de anticuerpos específicos para los virus PLRV, Potyvirus, CMV, ToMV, ToRSV y TSWV, de la compañía AGDIA (Indiana, EE. UU.). Para esto se formaron 12 muestras compuestas (3 bulk/lote; 10 muestras/bulk) de 100 mg de tejido afectado, se maceraron en buffer de extracción 1X y se siguieron las instrucciones del fabricante. Los resultados colorimétricos fueron cuantificados en un equipo Multiscan (Labsystem, Finlandia). Cada prueba incluyó un control positivo (suministrado por el fabricante en forma liofilizada) y un control negativo (buffer de extracción). Los pozos considerados con reacción positiva fueron aquellos en los cuales la lectura de absorbancia a 405 nm tuvo un valor mínimo del doble de la lectura obtenida en el control negativo, siguiendo el criterio de Matthews13.
Identificacion molecular de los virus. Una vez detectada la presencia de los virus mediante ELISA, se llevo a cabo la extraccion de ARN total mediante el kit RNAeasy plant mini (Qiagen, California, EEUU), macerando 100 mg de tejido foliar con nitrogeno liquido y utilizando 450 μL de buffer RLT y 10 μL de B-mercaptoetanol, para evitar la oxidacion de la muestra. Posteriormente, se siguio el protocolo del fabricante hasta obtener ARN, el cual fue resuspendido en 30 μL de agua destilada esteril libre de ARNasas.
Las reacciones de RT-PCR se realizaron con el kit QIAGEN OneStep RT-PCR (Qiagen) e incluyeron 5 μL de ARN total, 0,6 μM de cada cebador especifico para la amplificacion de una region diagnostica de la capside de cada virus (Tabla 1), 1X de Buffer QIAGEN One-Step RT-PCR Buffer, 400 μM de dNTPs y 2 μL de mezcla de enzimas QIAGEN (transcriptasa reversa Omniscript, transcriptasa reversa Sensiscript y Polimerasa de ADN HotStarTaq). El programa de amplificacion utilizado consto de una fase inicial de sintesis de la primera cadena a 50°C por 30 min, seguido de la activacion de la Taq polimerasa HotStar a 95°C por 15 min y 35 ciclos a 94°C por 1 min, 52°C por 1 min, 72°C por 1,30 min y una extension final de 10 min a 72°C.
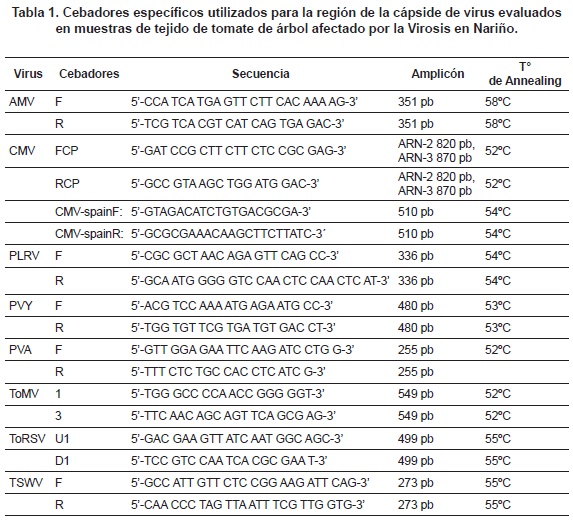
Los productos amplificados se separaron por electroforesis en gel de agarosa al 1,5%, suplementado con 4 μL de bromuro de etidio (10mg/mL), y fueron visualizados utilizando un transiluminador UV (Biometra, Göttingen, Alemania). Su tamaño se verificó comparando con el marcador de peso molecular Generuler 100 pb DNA ladder (Fermentas). La purificación de los amplicones de los virus que tuvieron el tamaño esperado se realizó mediante el kit PCR-purification (Qiagen) para su posterior secuenciación en la compañía Macrogen (Corea del Sur).
Las secuencias obtenidas con cada cebador fueron editadas mediante el software BioEdit 6.0.6, construyéndose secuencias consenso y confirmándose su identidad con genes virales por comparación con las bases de datos moleculares, mediante el programa BLASTn. Esta información permitió realizar comparaciones de los niveles de identidad entre las regiones secuenciadas de las cepas virales que afectan los cultivos de tomate de árbol en Córdoba (Nariño) y aquellas reportadas en el GenBank para otros cultivos y países, a partir del alineamiento de las secuencias mediante el software Clustal W.
Resultados
Descripción de sintomatologías virales
Se encontró una sintomatología general predominante, que corresponde a mosaicos foliares con ampollamientos, deformación de brotes y cambios en la apariencia (color, consistencia, forma) de flores y frutos. Estos síntomas estuvieron, además, acompañados por la presencia de manchas aceitosas en la superficie foliar y la formación de anillos necróticos en las hojas. Un síntoma generalizado en estados más avanzados de la infección fue la defoliación y reducción en el crecimiento de las plantas; aunque otros factores bióticos y abióticos también pudieran ser corresponsables de dichos cuadros sintomáticos
Detección serológica de los virus
Las pruebas de ELISA revelaron que la incidencia de potyvirus en los 12 bulk de muestras bajo análisis fue del 100%, al obtener valores de absorbancia mucho mayores que el doble del control negativo en todas las muestras. En cuanto al PLRV, se observó una incidencia del 75%, mientras que ToMV y CMV presentaron frecuencias de detección del 50% y 33%, respectivamente. Con la utilización de los anticuerpos utilizados no fue posible detectar los virus ToRSV, ni TSWV en ninguna de las muestras analizadas. En la tabla 2 se presentan los valores de absorbancia para los bulk de muestras realizados en cada lote.
Identificación molecular de los virus
Además de los virus PVA, PVY, PLRV, ToRSV y ToMV, se evaluaron cebadores para el virus PVX que afecta el cultivo de la papa y podría también presentarse en tomate de árbol y del virus AMV, reportado en este cultivo en otros países14, 15. Estas pruebas permitieron obtener los amplicones del tamaño esperado para PLRV y PVY, en muestras pertenecientes a cultivos de las veredas: San Juan Chico, El Chair y Guacas del municipio de Córdoba.
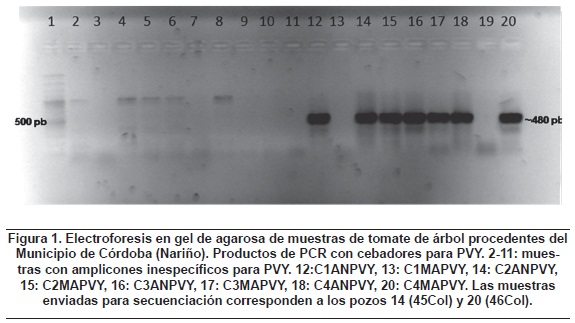
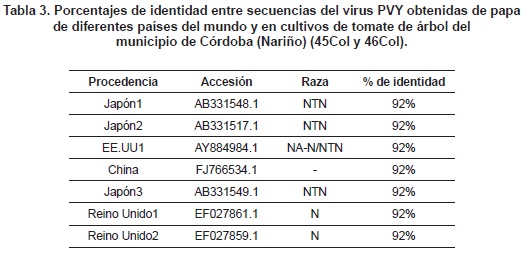
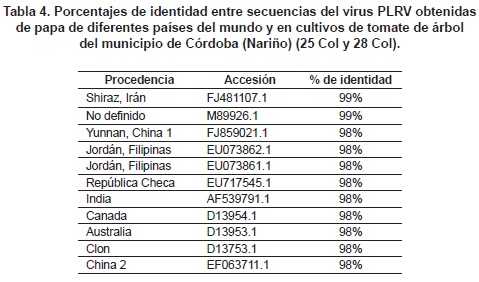
Para el caso del PVY, se obtuvieron bandas del tamaño esperado de 480 pb en siete muestras (figura 1), dos de las cuales fueron confirmadas como pertenecientes a una porción del gen de la cápside viral mediante su secuenciación posterior. Una vez establecidas las secuencias consenso, su comparación con cepas de referencia de este virus, obtenidas del GenBank y procedentes de diferentes lugares del mundo (Japón, Estados Unidos, China, Corea del Sur y Reino Unido), permitieron definir niveles de identidad superiores al 92% (Tabla 3). Para el virus PLRV se obtuvieron amplicones del tamaño esperado (336 pb) en dos muestras de la vereda El Chair del municipio de Córdoba (figura 2), las cuales fueron purifi cadas y enviadas para su secuenciación, y se confirmó su naturaleza viral, específi camente de una región que codifica para la cápside. Al alinearse las secuencias obtenidas con las depositadas en el Genbank, se obtuvieron porcentajes de identidad del 98% y 99% con cepas de PLRV procedentes de Irán, China, Filipinas, República Checa e India, entre otros, cuyos números de accesión son indicados en la tabla 4. Las secuencias obtenidas en esta investigación fueron depositadas en el GenBank bajo las accesiones: PVY45Col: HQ171921, PVY46Col HQ171922, PLRV25Col: HQ171923, PLRV28- Col: HQ171924.
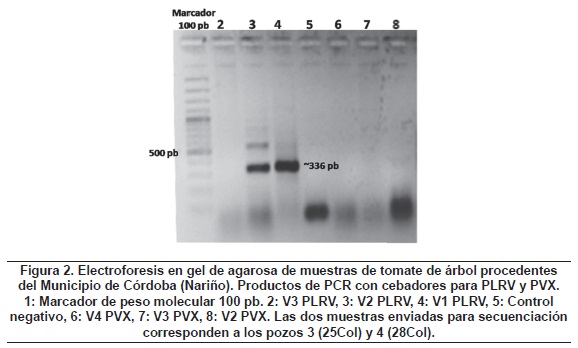
Discusión
Al analizarse las muestras foliares procedentes de cultivos de tomate de árbol en Córdoba (Nariño), se observaron diversas sintomatologías que incluyen la reducción en crecimiento de las plantas, distorsión en la forma de las hojas y de los brotes, mosaicos diversos, ampollamientos, manchas cloróticas y necróticas y amarillamiento de la superficie foliar, entre otros; todos estos, síntomas característicos de las infecciones virales de tomate de árbol16 y previamente reportados por varios autores del país17-19,20-23 y de otros lugares del mundo24-26.
Betancourth; et al27 reportaron la sintomatología de manchas aceitosas que afectan cultivos de tomate de árbol en Ipiales (Nariño) y a partir de observaciones de microscopía electrónica determinaron la presencia de partículas flexuosas de 800 nm probablemente asociadas a especies del género Potyvirus. En el presente estudio las muestras fueron evaluadas serológicamente con anticuerpos específicos para diferentes virus, y se logró confirmar la presencia de potyvirus en este cultivo, los que además resultaron con una incidencia del 100% en las muestras bajo análisis. De gran interés resultará en el futuro la realización de pruebas de ELISA específicas para las diferentes especies de potyvirus reportadas en tomate de árbol (PVY, TAMV, TaLMV y PVA), de manera que sea posible detallar su participación en la incidencia general de potyvirus, encontrada en este estudio. Adicionalmente, las pruebas de ELISA permitieron detectar la presencia de los virus PLRV, ToMV y en menor grado de CMV en dichas muestras, situación que confirma que la enfermedad denominada en términos genéricos como "Virosis" del tomate de árbol es causada por un complejo viral, que presenta al menos un potyvirus además de diferentes virus isométricos de los géneros Polerovirus Cucumovirus y de un tobamovirus (ToMV). Resultados similares fueron recientemente encontrados por Jaramillo28 para cultivos de Antioquia, Cundinamarca y Boyacá, y por Ochoa e Insuasti29 en Ecuador.
Con el fin de corroborar la identidad taxonómica de los virus detectados serológicamente, se realizaron pruebas de RT-PCR, y se logró la amplificación de fragmentos del gen que codifica para la proteína de la cápside de los virus PVY y PLRV, pero no para CMV. Dos de las secuencias obtenidas de cepas aisladas de hojas sintomáticas de tomate de árbol pudieron ser identificadas como pertenecientes a dicha región del genoma, dado que presentaron identidades superiores al 92% con cepas de diferentes razas de PVY. De acuerdo con lo anterior y teniendo en cuenta que la cápside es una proteína viral involucrada en la mayoría de procesos virales, como asistencia en la replicación del genoma, movimiento entre células y transmisión mediante vectores, la secuencia nucleotídica que codifica para dicha proteína presenta regiones altamente conservadas así como variables (responsables de la especificidad de la transmisión y de los hospederos de los virus), y su comparación con secuencias homólogas permite la identificación taxonómica de la mayoría de virus que afectan a plantas30.
Los criterios actuales para la identificación taxonómica de los virus se basan en el porcentaje de identidad en las secuencias de nucleótidos y de aminoácidos que forman diferentes proteínas. En el caso de los potyvirus, un criterio válido para la demarcación de especies dentro de la familia Potyviridae corresponde al porcentaje de identidad superior al 76% en la secuencia nucleotídica que codifica para la cápside; esto cuando no se cuenta con toda la secuencia del genoma viral31.
De acuerdo con lo mencionado anteriormente, se podría afirmar que las secuencias bajo análisis pertenecen al virus PVY, aunque no es posible definir su afinidad en razas, pues para esto se requeriría la secuenciación de regiones más variables como la proteína P1, que en estudios anteriores han permitido la identificación de cepas de PVYN y PVYO 32 ó del genoma viral completo, de tal forma que sea posible la ubicación de los sitios de recombinación reportados para cepas como PVYNTN y PVYN: Wilga 33.
Esto indica la necesidad de avanzar en la secuenciación del genoma completo de las cepas de PVY obtenidas en este estudio a partir de plantas de tomate de árbol, de tal manera que puedan abordarse importantes preguntas epidemiológicas como la posible patogenicidad cruzada de dicho virus entre solanáceas que comparten los agroecosistemas andinos de Colombia como papa, tomate de mesa, tomate de árbol, lulo y uchuva.
Por otra parte, los análisis moleculares también lograron confirmar la identidad del virus PLRV como un miembro del complejo viral que afecta el cultivo de tomate de árbol de Córdoba. Este virus es uno de los más frecuentemente encontrados en cultivos de papa en todo el mundo34. La raza que afecta papa está a su vez, dividida en tres linajes filogenéticos: el linaje II corresponde a cepas exclusivamente del continente australiano, mientras que los linajes I y III están conformados por cepas de diferentes regiones geográficas que no presentan diferencias en su eficiencia de transmisión por áfidos o grado de virulencia en papa35. No obstante, sus diferencias de origen geográfico no están reflejadas en el incremento de variabilidad genética, como ha ocurrido con otros virus; por el contrario PLRV es uno de los virus conocidos cuyos genomas son más conservados24. Dicha situación es evidente en el presente trabajo, puesto que las secuencias obtenidas de este virus compartieron altos niveles de identidad con las secuencias de PLRV provenientes de cultivos de papa de diferentes países del mundo. Un análisis adicional que se requiere realizar para evaluar la uniformidad en las secuencias de las cepas de Córdoba de PLRV bajo estudio, corresponde a la secuenciación de los ORF 0 y 1, que según Guyader & Ducray24, son más variables que aquellos que codifican para la proteína de la cápside (ORF 3) y para la proteína de movimiento (ORF4). La confirmación mediante secuenciación de las especies virales detectadas mediante ELISA solamente se obtuvo para PVY y PLRV, y no se logró obtener secuencias específicas para los demás virus bajo estudio.
Estos resultados pueden ser analizados desde dos perspectivas: a) los cebadores utilizados al ser diseñados con base en secuencias de cepas virales obtenidas en otros cultivos y regiones del mundo podrían no ser lo suficientemente genéricos para cubrir todo el rango de variación de cada especie viral, más cuando se considera que no existe ninguna secuencia depositada en las bases de datos moleculares para virus de estas especies procedentes de tomate de árbol; y b) que los cebadores presentan regiones complementarias en el genoma de S. betaceum, que conducen a amplificaciones inespecíficas, favorecidas por las relativamente bajas temperaturas de alineamiento (menores a 58°C) sugeridas por la literatura para el uso de dichos cebadores. La ausencia de amplificación en algunos de los virus evaluados hace necesario formular propuestas de investigación que permitan secuenciar completamente los genomas de las cepas virales presentes en cultivos de tomate de árbol, partiendo de metodologías clásicas de virología vegetal como la purificación de virus, separación de mezclas de virus a partir de inoculaciones en diferentes plantas indicadoras y la generación de ADN copia (ADNc) con cebadores genéricos como hexameros y oligo-dt (para aquellos virus que presenten colas de poli-A en sus extremos 3'). Este estudio representa un aporte científico para continuar aumentando la base de información disponible en el país sobre el complejo viral que afecta los cultivos de tomate de árbol y se espera contribuya al diseño de las estrategias de manejo de esta enfermedad en Colombia, de manera que este frutal se constituya nuevamente en una opción productiva para los agricultores de la región andina del país.
Conclusiones
La enfermedad denominada en campo como "Virosis" en el municipio de Córdoba (Nariño), es causada por un complejo viral constituido por miembros de los géneros Potyvirus (PVY), Cucumovirus (CMV), Tobamovirus (ToMV) y Polerovirus (PLRV) que generan diversidad de sintomatologías en este cultivo. Es necesario continuar con estudios que evalúen el efecto individual de estos virus sobre el cultivo, sus mecanismos de transmisión, niveles de varibilidad intraespecífica y posible patogenicidad cruzada con otros hospedantes.
Agradecimientos
Esta investigación fue financiada por el proyecto COLCIENCIAS Código 1118-405-20317 Contrato 408-2007 y por la Universidad Nacional de Colombia, sede Medellín, a través del proyecto DIME BICENTENARIO 2008-2009: 20101007745. Se agradece a los integrantes del Laboratorio de Biología Celular y Molecular y del Laboratorio de Microbiología de la Universidad de Nariño, por su colaboración durante el procesamiento de las muestras.
Referencias
1. ADAMS, Mike. Introduction to plant viruses. [en línea]. China: Rothamsted Research, 2007. [citado 20 Enero 2009]. Url disponible en: http://www.dpvweb.net/intro/index.php [ Links ]
2. BETANCOURTH, Carlos; GOYES, Rene y BRAVO, Darío Arturo. Caracterización biológica de un virus del tomate de árbol (Solanum betaceum Send) en el departamento de Nariño. En: Fitopatología Colombiana. 2003. Vol. 27, no 1, p. 7-10. [ Links ]
3. AYALA, Maryluz. Caracterización del Potyvirus asociado a la virosis del tomate de árbol en Antioquia. Tesis de Magíster en Ciencias Agrarias. Colombia: Universidad Nacional de Colombia. Facultad de Ciencias Agropecuarias. Medellín, Antioquia, 2009. 96 p. [ Links ]
4. JARAMILLO, Margarita. Análisis serológico y molecular de virus asociados al cultivo de tomate de árbol (Solanum betaceum) en Colombia. Tesis de Magíster en Ciencias - Biotecnología. Colombia: Universidad Nacional de Colombia. Facultad de Ciencias. Medellín, Antioquia, 2009. 127 p. [ Links ]
5. COLOMBIA. MINISTERIO DE AGRICULTURA Y DESARROLLO RURAL. Observatorio agrocadenas. [En línea]. Bogotá: El Ministerio, 2006 [citado 20 Octubre de 2008]. Url disponible en: http://www.agrocadenas.gov.co [ Links ]
6. COLOMBIA. SECRETARIA DE AGRICULTURA DE NARIÑO. Consolidado Agropecuario de Nariño. [en línea]. Pasto: La Secretaria, 2006. [citado 30 Noviembre de 2008]. Url disponible en: http://www.gobernacion-narino.gov.co/ [ Links ]
7. SÁNCHEZ DE LUQUE, M. y DE LA ROTTA, C., SUAREZ, A. Posible virus en tomate de árbol Cyphomandra betacea SENDT. En: ASCOLFI informa. 1981. Vol. 7, no. 3, p. 24. [ Links ]
8. TAMAYO, P.J. Mosaico del tomate de árbol. En: ASCOLFI Informa. 1990. Vol. 16, no. 6, p. 54-55. [ Links ]
9. BETANCOURTH, Carlos; GOYES, Rene y BRAVO, Darío Arturo. Op. Cit. p. 7-10. [ Links ]
10. VIZUETE B.; et al. Biological and Serological Characterization of Tree Tomato Virus Diseases in Ecuador. Quito, 1990. 3 p. [ Links ]
11. OCHOA, L. J. y INSUASTI, A. M. Etiología de las enfermedades virales del tomate de árbol en Ecuador. Quito: Instituto Nacional de Investigaciones Agropecuarias. Informe Técnico Anual - INIAP (Ecuador). Est. Exp. Santa Catalina. Departamento Nacional de Protección Vegetal estudios agronómicos, fitopatológicos y entomológicos de frutales nativos andinos, 2005. p. 1-4. [ Links ]
12. EAGLES, Robin M.; GARDNER, Richard C. y FORSTER, Richard. Incidence and distribution of six viruses infecting tamarillo (Cyphomandra betacea) in New Zealand. En: New Zealand Journal of Crop and Horticultural Science. 1994. Vol. 22, p. 453-458. [ Links ]
13. MATTHEWS, Ref. Plant Virology. 3 ed. New York: Academic press limited, 1991. 835 p. [ Links ]
14. VIZUETE B.; et al. Op Cit. 3 p. [ Links ]
15. EAGLES, Robin. Tamarillo mosaic potyvirus: characterization and resistance. Tesis PhD. Australia: University of Auckland. School of Biological Sciences, Auckland. 1994. [ Links ]
16. CANN, Alan. Principles of molecular virology. 3 ed. New York: Elsevier Academic Press, 2001. 323 p. [ Links ]
17. BETANCOURTH, Carlos; GOYES, Rene y BRAVO, Darío Arturo. Op. Cit.; p. 7-10. [ Links ]
18. AYALA, Maryluz. Op. Cit.; 96 p. [ Links ]
19. JARAMILLO, Margarita. Op. Cit.; 127 p. [ Links ]
20. SALDARRIAGA, A.; BERNAL, J.A. y TAMAYO, P. Virosis del tomate de árbol. En: CORPOICA. Enfermedades del cultivo de árbol en Antioquia: Guía de reconocimiento y control. Boletín técnico CORPOICA. 1997. 43 p. [ Links ]
21. CRUZ, Luisa Fernanda. Identificación de virus en Solanum betacea. Tesis de Ingeniero Agrónomo. Colombia: Universidad Nacional de Colombia. Facultad de Agronomía. Bogotá, Cundinamarca, 2005. 48 p. [ Links ]
22. CUSPOCA, J. Evaluación de virus en tomate de árbol (Solanum betacea) en plantas indicadoras y su detección por PCR. Tesis de Ingeniero Agrónomo. Colombia: Universidad Nacional de Colombia. Facultad de Agronomía. Bogotá, Cundinamarca, 2007. 43p. [ Links ]
23. GIL RAMÍREZ, José Fernando; et al. Identificación de potyvirus en cultivos de tomate de árbol (Solanum betaceum cav.) en Antioquia mediante detección serológica. En: Revista Politécnica. 2009. Vol. 5, p.112-120. [ Links ]
24. BETANCOURTH, Carlos; GOYES, Rene y BRAVO, Darío Arturo. Op. Cit. p. 7-10. [ Links ]
25. VIZUETE B.; et al. Op. Cit.; 3 p. [ Links ]
26. OCHOA, L. J. y INSUASTI, A. M. Op. Cit.; p. 1-4. [ Links ]
27. BETANCOURTH, Carlos; GOYES, Rene y BRAVO, Darío Arturo. Op. Cit.; p. 7-10. [ Links ]
28. JARAMILLO, Margarita. Op. Cit.; 127 p. [ Links ]
29. VIZUETE B.; et al. Op Cit. 3 p. [ Links ]
30. CALLAWAY, A.; et al. The multifunctional capsid proteins of plant RNA viruses. En: Annual Review of Phytopathology. 2001. Vol. 39, p. 419-460. [ Links ]
31. ADAMS, M.J.; ANTONIW, J.F. y FAUQUET, C.M. Molecular criteria for genus and species discrimination within the family Potyviridae. En: Archives of Virology. 2005. Vol. 150, p. 459-479. [ Links ]
32. NIE, Xianzhou y SINGH, Singh. Probable geographical grouping of PVYN and PVYNTN based on sequence variation in P1 and 5-UTR of PVY genome and methods for differentiating North American PVYNTN. En: Journal of Virological Methods. 2002. Vol. 103, p. 145-156. [ Links ]
33. KOGOVSEK, P.; et al. Single-step RT real-time PCR for sensitive detection and discrimination of Potato virus Y isolates. En: Journal of Virological Methods. 2008. Vol. 149, p. 1-11. [ Links ]
34. NIE, Xianzhou y SINGH, Singh. A novel usage of random primers for multiplex RT-PCR detection of virus and viroid in aphids, leaves, and tubers. En: Journal of Virological Methods. 2001. Vol. 91, p. 37-49. [ Links ]
35. GUYADER, Sébastien y DUCRAY, Danièle. Sequence analysis of Potato leafroll virus isolates reveals genetic stability, major evolutionary events and differential selection pressure between overlapping reading frame products. En: Journal of General Virology. 2002. Vol. 83, p. 1799-1807. [ Links ]














