Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Lasallista de Investigación
Print version ISSN 1794-4449
Rev. Lasallista Investig. vol.10 no.2 Caldas July/Dec. 2013
Artículo original / Original article / Artigo original
Asociación del polimorfismo del intrón 6 en el gen lactoferrina bovino con características de importancia en producción lechera*
Association of the intron 6 polymorphism of the bovine lactoferrin gene with characteristics important for the dairy industry
Associação do polimorfismo do intrón 6 no gene lactoferrina bovino com características de importância em produção leiteira
Nancy Rodríguez Colorado**, Albeiro López Herrera***, José Julián Echeverri Zuluaga****
*Artículo derivado de la investigación "Determinación de las frecuencias alélicas y genotípicas del gen CD18 en vacas Holstein lecheras y su asociación con características de importancia económica", realizada entre 2009 y 2010 con financiación de la Dirección de Investigaciones de la Universidad Nacional de Colombia, Sede Medellín (DIME).
**Estudiante de Maestría en Ciencias Agrarias. Universidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias Agropecuarias.
***Profesor Asociado. Universidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias Agropecuarias.
****Profesor Asistente. Universidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias Agropecuarias.
Correspondencia: Nancy Rodríguez Colorado, email: nrodrig@unal.edu.co.
Artículo recibido: 05/08/2012; Artículo aprobado: 01/11/2013
Resumen
Introducción. La lactoferrina estimula o inhibe diversos componentes humorales y celulares de la inmunidad; por sus cualidades puede desempeñar un papel en la prevención de la infección de la glándula mamaria. Objetivo. Explorar la asociación del polimorfismo (C/T) del intrón 6 en el gen de la lactoferrina bovino, con la producción de leche, porcentaje de grasa y proteína, y recuento de células somáticas en una población de vacas Holstein en Antioquia. Materiales y métodos. Esta investigación fue realizada con base en información correspondiente a 382 vacas Holstein del departamento de Antioquia. Fueron analizadas 1345 lactancias para el análisis de asociación con producción de leche y 293 lactancias para porcentaje de proteína, grasa y recuento de células somáticas (RCS). La producción y composición de la leche fue monitoreada durante al menos una lactancia completa. La genotipificación se realizó usando la técnica de PCR-RFLP. Resultados. Las frecuencias alélicas fueron 0.78 y 0.22 para A y B, respectivamente; las frecuencias genotípicas 0.60, 0.36 y 0.04 para AA, AB y BB, respectivamente. Se presentaron diferencias significativas (P<0.05) entre las medias de porcentaje de proteína y SCS entre los tres genotipos; para el porcentaje proteína el genotipo BB presentó una superioridad de 0.18 %, y para SCS el genotipo AB indicó un disminución en dicho puntaje de 0.77. Para las demás características no se encontraron diferencias significativas. Conclusiones. Estos resultados sugieren que el polimorfismo del gen LTF bovino es un buen indicador de la susceptibilidad o resistencia a mastitis subclínica y está asociado con el porcentaje de proteína en leche.
Palabras clave: marcadores moleculares, mejoramiento genético, producción de leche.
Abstract
Introduction. Lactoferrin stimulates or inhibits several humoral and cellular components of the immunity. Given its qualities, it can play a role in the prevention of mammary gland´s infections. Objective. To explore the association of the intron 6 polymorphism of the bovine lactoferrin gene with milk production, fat and protein percentages and somatic cells count in a population of Holstein cows, in Antioquia. Materials and methods. This research work was performed with base on information about 382 Holstein cows from the Antioquia province. 1345 lactations were taken for the association analysis with milk production and 293 lactations were taken for fat and protein percentages and for a somatic cells count (SCC). The production and the composition of the milk were monitored during, at least, a whole lactation. The genotyping was performed by the use of the PCR-TFLP technique. Results. The allelic frequencies were 0.78 and 0.22 for A and B, respectively. The genotypic frequencies were 0.60, 0.36 and 0.04 for AA, AB y BB, respectively. There were significant differences (P<0.05) between the measurements of protein percentages and SCC between the three genotypes. For the protein percentage, the BB genotype had 0.18% of superiority and for SCC the AB genotype showed a reduction of 0.77 in such rate. For the other characteristics, no significant differences were found. Conclusions. These results suggest that the LTF bovine gene is a good indicator of the susceptibility or the resistance to subclinical mastitis and is associated to the protein percentage in the milk.
Key words: molecular markers, genetic improvement, milk production.
Resumo
Introdução. A lactoferrina estimula ou inibe diversos componentes humorais e celulares da imunidade; por suas qualidades pode desempenhar um papel na prevenção da infecção da glândula mamária. Objetivo. Explorar a associação do polimorfismo (C/T) do intrón 6 no gene da lactoferrina bovino, com a produção de leite, porcentagem de gordura e proteína, e recontagem de células somáticas numa população de vacas Holstein em Antioquia. Materiais e métodos. Esta investigação foi realizada com base em informação correspondente a 382 vacas Holstein do departamento de Antioquia. Foram analisadas 1345 lactâncias para a análise de associação com produção de leite e 293 lactâncias para porcentagem de proteína, gordurosa e recontagem de células somáticas (RCS). A produção e composição do leite foi monitorada durante ao menos uma lactância completa. A genotipificação se realizou usando a técnica de PCR-RFLP. Resultados. As frequências alélicas foram 0.78 e 0.22 para A e B, respectivamente; as frequências genotípicas 0.60, 0.36 e 0.04 para AA, AB e BB, respectivamente. Apresentaram-se diferenças significativas (P<0.05) entre as médias de porcentagem de proteína e SCS entre os três genótipos; para a porcentagem proteína o genótipo BB apresentou uma superioridade de 0.18 %, e para SCS o genótipo AB indicou um diminuição em dita pontuação de 0.77. Para as demais características não se encontraram diferenças significativas. Conclusões. Estes resultados sugerem que o polimorfismo do gene LTF bovino é um bom indicador da susceptibilidade ou resistência a mastite subclínica e está associada com a porcentagem de proteína no leite.
Palavras importantes: marcadores moleculares, melhoramento genético, produção de leite.
Introducción
La lactoferrina (LTF) es una glicoproteína monomérica, no hémica de unión a hierro, considerada componente esencial de la inmunidad innata del hospedero, que estimula o inhibe diversos componentes humorales y celulares de la inmunidad implicados en la prevención o resolución de infecciones. Estructural y funcionalmente, la LTF humana y bovina han sido las más ampliamente estudiadas y, por ende, las mejor caracterizadas con hallazgos que incluyen un despliegue frente a un amplio espectro de microorganismos, actuando contra bacterias, algunos virus y hongos con actividad antitumoral (Orsi, 2004).
El gen bLTF bovino fue mapeado en el cromosoma 22q24 y presenta un tamaño de 34.5 kb. (Chaneton et al., 2008); presenta polimorfismos en varios exones sin generar cambios en la amino secuencia (Wojdak, Kmiec & Ziemak, 2006) y polimorfismos en el exón 4 y en el exón 11 que causan cambios importantes en la misma; en el exón 4, en la base 1155 A/G causa un cambio de Isoleucina a valina, y el exón 11 en la base 1415 C/T causa un cambio de histidina a tirosina. Un polimorfismo en el intrón 6 (C/T) está ligado a los cambios en los exones 4 y 11, separados por 14.5 kb. Cuando el intrón 6 se encuentra en su forma no mutada (citocina), el exón 4 contiene una adenina y el exón 11 una citosina; cuando el intrón 6 se encuentra con su forma mutada (Timina) el exón 4 se encuentra con una guanina y el exón 11 con una timina (Seyfert, Tuckoricz & Interthal, 1994).
En bovinos destinados a la producción lechera, la mastitis es una enfermedad con elevada incidencia, cuyo tratamiento y consecuencias generan altos costos al sistema productivo. Es una de las enfermedades infecciosas más frecuentes en los sistemas de produccion lechera en todo el mundo y, al mismo tiempo, causa efectos negativos sobre el bienestar animal. Las condiciones en la glándula mamaria bovina inmediatamente antes del parto y durante la involución uterina favorecen la actividad antimicrobiana de la LTF, ya que la concentración de bicarbonato es más alta, y menor la del citrato (Pawlik, Sender & Korwin, 2009). De este modo LTF puede desempeñar un papel en la prevención de la infección de la glándula mamaria, en especial en el parto y la involución uterina.
Entre las características de mayor importancia en ganado lechero, están la producción de leche y la calidad sanitaria y composicional de la misma. El contenido de proteína y grasa marca una diferencia importante en la economía del productor, ya que le permite obtener bonificaciones al mejorar los mencionados parámetros, además de obtener una ventaja competitiva fundamental para alcanzar y mantener las necesidades y exigencias de los consumidores.
Una correlación genética de -0.25 ha sido reportada entre la producción de leche y el contenido de LTF en leche (Arnould, Soyeurt & Colinet, 2009), lo cual indica que la selección para producción de leche produce un efecto adverso sobre los niveles de LTF en la misma; esto se traduce en un sistema inmune menos eficiente. Esta misma investigación reporta correlaciones de 0.37 y 0.52 entre LTF en leche, y porcentaje de grasa y proteína, respectivamente.
La industria lechera en Colombia ha basado su producción en la raza Holstein, lo que sugiere la necesidad de realizar investigaciones que conduzcan a buscar estrategias para mantener un equilibrio entre producción, reproducción y sanidad. El objetivo de esta investigacion es explorar la asociación del polimorfismo en el intron 6 del gen LTF con el SCS como indicativo de mastitis subclínica, producción de leche y porcentaje de proteína y grasa en leche.
Materiales y métodos
Localización y población de estudio
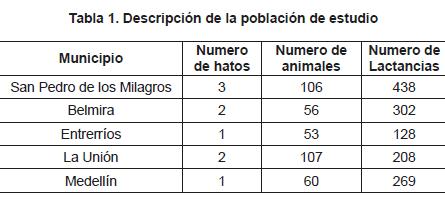
Esta investigación fue llevada a cabo en nueve hatos lecheros, ubicados en cinco municipios del departamento de Antioquia. Fueron utilizadas 1345 lactancias, correspondientes a 382 vacas de la raza Holstein. La descripción general de la población estudiada es presentada en la tabla 1.
Control de producción lechera
La información referente a producción, composición de la leche y calidad sanitaria, fue obtenida del programa de control de producción lechera, llevado a cabo por la cooperativa Colanta Ltda. y la Universidad Nacional de Colombia, entre los años 2009 y 2011. El análisis fisicoquímico de la leche se llevó a cabo en el laboratorio de la Cooperativa Colanta Ltda., mediante el uso del Milkoscan® FT120 y el RCS determinado con el uso del Fossomatic®.
Extracción de ADN y genotipificación
Para la obtención del ADN de sangre periférica se utilizó el método de "salting out" modificado, descrito por Miller y colaboradores(1988); el DNA fue almacenado a 4°C hasta el momento del análisis. La cantidad y calidad del ADN extraído se evaluó en gel de agarosa al 0.8 % teñido con bromuro de etidio, y se cuantificó en el espectrofotómetro NanoDrop ND 2000. Solo el ADN genómico con una pureza ideal entre 1.8-2.0 se consideró para los análisis moleculares posteriores.
El ADN aislado fue usado para la amplificación por reacción en cadena de la polimerasa (PCR) de un fragmento del gen LTF de 301 pb (pares de bases) con los siguientes: directo 5'-GCC TCA TGA CAA CTC CCA CAC-3' Reverso: 5'-CAG GTT GAC ACA TCG GTT GAC-3', según Seyfert y colaboradores (1994). El mix para PCR contenía 0.5 U de Taq polimerasa (Bioline®), 2.5 µL de Buffer para PCR 10x, 30ng de DNA, 0.5 µL de MgCl2 a 50nM, 0.5 µL de dNTPs a 20 nM y 0.5 µL de cada primer, se aforó con agua desionizada y estéril para un volumen total de 25 µL.
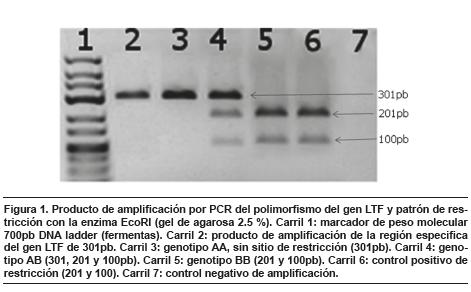
Las condiciones de la PCR estandarizada fueron: una desnaturalización inicial a 95 °C por 3 min, seguido de 28 ciclos a 95 °C por 45 s, 60°C por 30 s, 72 ºC por 20 s, con una extensión final a 72 °C por 3 min. El producto de la PCR se analizó por electroforesis en gel de agarosa al 2 % y bromuro de etidio. Luego se realizó un analisis de los polimorfismos de longitud de los fragmentos de restricción (RFLP) usando la endonucleasa EcoRI. El mix se realizó de la siguiente manera: 5 µL de reacción de PCR, 2 µL de Buffer EcoRI (Fermentas®), 0.5 µL EcoRI (Fermentas®) y 12.5 µL de agua desionizada y estéril, para un volumen total de 20 µL, se incubó por 3 horas a 37 °C. El producto de digestión se resolvió por electroforesis en gel de agarosa al 2.5 %, teñido con bromuro de etidio. El patrón de restricción esperado para el genotipo AA es un fragmento de 301pb, para el genotipo AB tres fragmentos (301pb, 201pb y 100pb) y para el genotipo BB dos fragmentos (201pb y 100pb).
Análisis estadístico
Las frecuencias alélicas y genotípicas del gen LTF se evaluaron mediante el programa GENEPOP versión 4.0 (Raymond & Rousset, 2008).
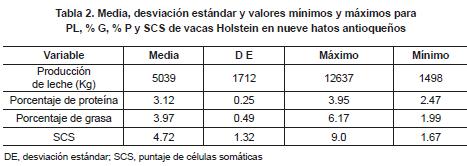
El RCS determinado en cada control fue transformado a una escala lineal para obtener el puntaje de células somáticas (SCS) de 1 a 9, mediante una transformación logarítmica, según la expresión Log2 (RCS/100,000) + 3, de acuerdo con lo descrito por Dabdoub y Shook (1984). Las lactancias fueron ajustadas a una duración de 305 días. Los porcentajes de proteína y grasa se calcularon como un promedio de las medidas obtenidas a lo largo de la lactancia completa.
Para determinar la asociación de cada una de las características con el genotipo para LTF, se ajustó un modelo lineal generalizado que incluyó diferentes fuentes de variación para cada una de las variables dependientes, producción de leche (PL), porcentaje de proteína (% P), porcentaje de grasa (% G) y puntaje de células somáticas (SCS). Un análisis de medias de Duncan fue utilizado para determinar las diferencias entre las medias para cada uno de los niveles de los efectos fijos incluidos en los modelos. Se utilizó el paquete estadístico SAS 9.0 (2004) para todos los análisis.
El modelo llevado a cabo fue el siguiente:
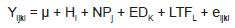
Dónde:

Posteriormente se llevó a cabo un ajuste de los efectos medioambientales que tuvieron significancia en los primeros análisis de varianza, con el objetivo de minimizar el efecto ambiental y determinar el efecto específico del genotipo para LTF. Para este análisis se utilizó la metodología descrita por Quijano (2004), como sigue:
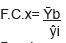
Donde:
F.C.x = Factor de corrección multiplicativo.
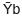 = Promedio base (PL, %P, %G o SCS).
= Promedio base (PL, %P, %G o SCS).
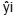 = promedio del grupo i. (i: Hato, número de partos)
= promedio del grupo i. (i: Hato, número de partos)
Se realizó un análisis de regresión lineal simple en SAS 9.0. (2004) para determinar el efecto de sustitución alélica de LTF sobre las características en estudio; para este fin de convirtió a una escala cuantitativa 0, 1 y 2 para los genotipos AA, AB y BB, respectivamente; de este modo el alelo base para sustitución fue el B. El modelo de regresión utilizado fue el siguiente:
Y1= β0 + β1 X1 + e1
Donde:
Yi: Valor de la variable dependiente (producción de leche, porcentaje de proteína, porcentaje de grasa y SCS) en función del número de alelos B.
β0 = Intercepto
β1 = Coefiente de regresión lineal estimado del alelo de sustitución (B)
Xi: Número de alelos B en el individuo i. (0, 1, 2).
ei: error residual
Resultados
Determinación de las frecuencias alélicas y genotípicas del gen LTF
Se obtuvo un fragmento amplificado de 301pb para cada una de las 382 muestras. El análisis de los fragmentos de restricción con la técnica RFLP usando la enzima EcoRI mostró fragmentos de 301pb (alelo A, sin sitio de restricción), 201 pb y 100 pb (alelo B) (figura 1). Las frecuencias de los alelos A y B fueron 0.78 y 0.22, respectivamente. Las combinaciones alélicas dieron lugar a tres genotipos AA, AB y BB, con frecuencias 0.60, 0.36 y 0.04, respectivamente.
Análisis descriptivo
Las 1345 lactancias utilizadas correspondían a 382 vacas que presentaron de 1 a 13 partos, con edades promedio de 7.9 ± 2.6 años y una producción de leche promedio de 5039 ± 1712 Kg. Para porcentaje de proteína, grasa y puntaje de células somáticas (SCS) se contó con la información de 293 lactancias correspondientes a 64 vacas; el promedio de porcentaje de proteína fue de 3.1 ± 0.25, el porcentaje grasa de 3.97 ± 0.49 y el SCS de 4.72 ± 1.32 (tabla 2).
Efecto del genotipo LTF, sobre las características productivas y sanitarias
De acuerdo con el analisis de varianza, producto del ajuste de un modelo lineal generalizado, el genotipo no presentó efecto significativo sobre la PL, SCS y porcentaje de grasa y proteína de la leche.
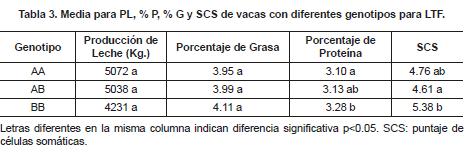
Un análisis de medias de Duncan llevado a cabo posteriormente, mostró que las vacas con genotipo AA produjeron 840.7 Kg de leche más por lactancia que las vacas con genotipo BB. De la misma forma los individuos AB fueron superiores en 807.3 Kg a los BB que presentaron el menor rendimiento. El porcentaje de grasa de las vacas con el genotipo BB fue mayor que el de las vacas con genotipos AA y AB, con diferencias de 0.16 % y 0.12 %, respectivamente; sin embargo, estadísticamente, para las características producción de grasa y porcentaje de grasa, no se detectaron diferencias significativas entre las medias (P>0.05).
Para el porcentaje de proteína el análisis de medias de Duncan mostró diferencias significativas (p<0.05) entre las medias de los tres genotipos, denotando el genotipo BB como el más favorable para porcentaje de proteína; estas diferencias son presentadas en la tabla 3.
El análisis para SCS mostró diferencias significativas (p<0.05) entre las medias de los tres genotipos, denotando el genotipo AB como el más favorable para el SCS con un puntaje menor (tabla 2).
Los modelos con las variables medioambientales que tuvieron significancia en los primeros análisis de varianza, hato y número de parto fueron ajustadas usando los factores de corrección multiplicativos; sin embargo, los análisis posteriores no mostraron diferencias significativas (P>0.05).
Efecto de sustitución alélica sobre características productivas y sanitarias
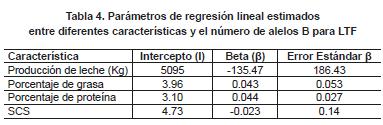
El coeficiente de regresión (β) estimado entre la producción de leche y el número de alelos B para LTF fue de -135.4 Kg, indicando que por cada alelo B para LTF que porte el individuo, la producción de leche se disminuye en 135.4 kg. de leche por lactancia. De acuerdo con los valores mostrados en la tabla 4, el coeficiente de regresión para SCS indica que por cada alelo B disminuye en 0.023 puntos este puntaje; en el caso del porcentaje de grasa, por cada alelo B, este parámetro aumenta en 0.043 %, situación similar para el porcentaje de proteína donde se observó que por cada alelo B la proteína aumenta en 0.044 %.
Discusión
Las frecuencias alélicas y genotípicas en la población de estudio difieren de las encontradas por Wojdak-maksymiec y colaboradores (2006) en una población de 124 vacas de la raza holstein en Polonia, donde se encontraron frecuencias de 0.68, y 0.33 para los alelos A y B del gen LTF y encontraron para los genotipos AA, AB, y BB frecuencias de 0.38, 0.60 y 0.02, respectivamente. En un trabajo realizado por Šrubařová y Dvořák (2009) en una población de 94 vacas Holstein en República Checa, se encontró una frecuencia de 0.57 para el genotipo AA 0.43 para el genotipo AB y no se encontró el genotipo BB; las frecuencias para el alelo A fueron de 0.79, y para el alelo B de 0.21, frecuencias muy similares a las encontradas en el presente estudio.
Kaminiski y colaboradores (2006) encontraron asociación entre el polimorfismo en la región promotora en la posición +32 (G/C) del gen lactoferrina con la producción de leche, el contenido de proteína y recuento de células somáticas. El autor menciona cómo el alelo G reduce la expresión de lactoferrina y reduce el recuento de células somáticas; por el contrario, el alelo C aumenta la expresión de lactoferrina lo cual conduce a una respuesta inmune más fuerte que trae como consecuencia un incremento en el recuento de células somáticas; para el caso de la proteína, el genotipo CC disminuye su contenido en leche, contrario de lo encontrado para el genotipo GG. Kaminiski (2006) reporta otro polimorfismo en la región promotora en la posición +216 que en ocurrencia simultánea con otro polimorfismo, en el gen receptor de la hormona del crecimiento, consigue diferencias significativas en el contenido de proteína y producción de leche.
Para el polimorfismo del presente estudio no se encontraron reportes de asociaciones con las características producción de leche, proteína y grasa, pero sí asociaciones con el recuento de células somáticas importantes. Wojdak-maksymiec y colaboradores (2006) encontraron asociación entre el genotipo AA y un recuento menor de células somáticas que el genotipo AB. En el mismo polimorfismo Sender (citado por Pawlik, 2009) encontró asociación entre el genotipo BB y un recuento menor de células somáticas en leche, acorde con los resultados obtenidos en este estudio.
La baja frecuencia del genotipo BB en las poblaciones es una limitación determinante a la hora de realizar las asociaciones en las poblaciones estudiadas; sin embargo, en la población estudiada de Antioquia se encontraron diferencias significativas (P <0.05) para el porcentaje de proteína y SCS, siendo el genotipo BB el mejor para proteína y el genotipo AB el mejor para disminuir el SCS.
En las poblaciones Holstein se han trabajado apareamientos dirigidos (selección artificial) con miras a lograr mayores volúmenes de producción de leche; esta selección podría explicar las frecuencias genotípicas del gen LTF bovino, ya que, aunque para producción de leche no se encontró diferencia significativa, existe una mayor proporción del genotipo AA.
Existen divergencias en algunos resultados obtenidos en este trabajo y los demás autores, que pueden ser explicadas por la influencia que tiene el medio ambiente sobre la expresión genotípica de las características. La baja frecuencia del genotipo BB en esta investigación genera una gran dificultad para determinar el grado de asociación que tiene cada uno de estos con características como producción de leche, grasa, proteína y SCS. Se requieren aún más esfuerzos por parte de los investigadores para aumentar el tamaño muestral y conseguir resultados que generen mayor confiabilidad en cuanto a los marcadores genéticos utilizados en los programas de mejoramiento animal, aunque son claras las tendencias en las asociaciones del gen LTF con características como proteína y SCS en la población estudiada.
Conclusión
Estos resultados son el primer reporte que caracteriza el gen de bLTF en ganado Holstein de trópico alto colombiano. Los mismos servirán de base para futuros estudios con este y otros marcadores moleculares, con el fin de contribuir con la conservación y permanencia de alelos de importancia, su asociación con enfermedades y características de importancia económica y como posterior marcador de susceptibilidad o resistencia a las mismas, lo cual es de gran utilidad como criterio de selección.
Referencias bibliográficas
• Arnould, V.; Soyeurt, H. & Colinet, G. (2009). Genetic analysis of lactoferrin content in bovine milk. Journal of Dairy Science, 92, 2151-2158. [ Links ]
• Chaneton, L.; Tirante, L.; Maito, J.; Chaves, J. & Bussmann, L. (2008). Relationship between milk lactoferrin and etiological agent in the mastitis bovine mammary gland. Journal of Dairy Science, 91, 1865-1873. [ Links ]
• Dabdoub, S. & Shook, G. (1984). Phenotypic relations among milk yield, somatic cell count and clinical mastitis. Journal of Dairy Science, 67, 163-164. [ Links ]
• Kaminiski, S., Olenski, K.; Brym, P.; Malewski, T. & Sazanov A. (2006). Single nucleotide polymorphism in the promoter region of the lactoferrin gene and its associations with milk performance traits in Polish Holstein -Friesian cows. Russian journal of genetics, 42, 924-927. [ Links ]
• Miller, S.; Dykes, D. & Polesky, H. (1988). A simple salting-out procedure for extracting DNA from human nucleated cells. Nucleic Acid Res, 16, 1215. [ Links ]
• Orsi, N. (2004). The antimicrobial activity of lactoferrin: current status and perspectives. Biometals, 17, 189-196. [ Links ]
• Pawlik, A.; Sender, G. & Korwin-Kossakowska, A. (2009). Bovine lactoferrin gene polymorphism and expression in relation to mastitis resistance - a review. Animal Science Papers and Reports, 27, 263-271. [ Links ]
• Quijano, B. (2004). Notas del curso de introducción a la genética de poblaciones. Universidad Nacional de Colombia Sede Medellín, p. 148. [ Links ]
• Raymond, M. & Rousset, F. (2008). Genepop'007: a complete re-implementation of the genepop software for Windows and Linux. Molecular Ecology Resources, 8, 103-106. [ Links ]
• SAS Institute Inc. (2004) SAS/STAT 9.0. Use's Guide. SAS Institute Inc. Cary, NC. [ Links ]
• Seyfert, H.; Tuckoricz, A.; Interthal, H., et al. (1994). Structure of the bovine lactoferrin encoding gene and its promoter. Gene, 143, 265-2691. [ Links ]
• Šrubařová, P. & Dvořák, J. (2010). Association between Lactoferrin gene polymorphism and bovine mammary gland inflammation. MendelNet´09 Agro Page. Recuperado de http://web2.mendelu.cz/af_291_mendelnet/mendelnet09agro/files/articles/bz_srubarova.pdf. [ Links ]
• Wojdak-Maksymiec, K.; Kmiec, M. & Ziemak J. (2006). Associations between bovine lactoferrin genepolymorphism and somatic cell count in milk. Veterinary Medicine, 51, 14-20. [ Links ]