Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista Lasallista de Investigación
versión impresa ISSN 1794-4449
Rev. Lasallista Investig. vol.13 no.1 Caldas ene./jun. 2016
Caracterización del polimorfismo genético del gen bola DRB3.2 en vacas holstein y bonxholstein de un hato lechero de Antioquia*
Characterization of the genetic polymorphism of the BoLA DRB3.2 gene in Holstein and Bonholstein cows from a dairy herd in Antioquia
Caracterização do polimorfismo genético do gene bola DRB3.2 em vacas holstein e bonx holstein de um rebanho leiteiro de Antioquia
Juan Carlos Zambrano Arteaga**, Julián Echeverri Zuluaga***, Albeiro López Herrera****
* Artículo original derivado de la investigación "Correlación entre mastitis clínica y subclínica con las variantes genotípicas del gen BoLA DRB3.2, presentes en las vacas y novillas de primer parto del hato lechero de la hacienda Paysandú de La Universidad Nacional de Colombia", código QUIPU 20101006713, financiado por la Dirección de Investigación de la Universidad Nacional de Colombia sede Medellín.
** Docente Ocasional. Facultad de Ciencias de la Salud. Institución Universitaria Colegio Mayor de Antioquia, Medellín, Colombia
*** Profesor Asociado. Facultad de Ciencias Agropecuarias. Universidad Nacional de Colombia, Sede Medellín, Medellín, Colombia
**** Profesor Asociado. Facultad de Ciencias Agropecuarias. Universidad Nacional de Colombia Sede Medellín, Medellín, Colombia
Correspondencia: Julián Echeverri Zuluaga, e-mail: jjecheve@unal.edu.co.
Artículo recibido:30/06/2011; Artículo aprobado: 29/02/2016
Resumen
Introducción. El gen DRB3 del antígeno leucocitario bovino (BoLA) ha sido ampliamente estudiado, dado que sus variantes alélicas han sido asociadas con enfermedades infecciosas y características productivas. Objetivo. Determinar las frecuencias de los alelos del exón 2 del gen BoLA DRB3 obtenidos mediante la técnica PCR-RFLP en vacas Holstein y BONxHolstein (BxH) de un hato lechero de Antioquia. Materiales y métodos. Fueron genotipificadas 101 vacas, 76 de raza Holstein y 25 del cruce BON x Holstein (BxH) para el exón 2 del gen DRB3 del antígeno leucocitario bovino (BoLA). Los alelos BoLA DRB3.2 fueron identificados por PCR-RFLP, empleando las enzimas de restricción RsaI, HaeIII y BstYI y confirmados por secueciación. Resultados. En este estudio fueron identificados 27 alelos BoLA DRB3.2 con un rango de frecuencias de 0.007 a 0.151 en la raza Holstein y de 0.02 a 0.20 en BxH. Los cuatro alelos más frecuentes para la raza Holstein fueron: BoLA DRB3.2* 23, 22, 24 y 16, con una frecuencia acumulada de 0.506 y los cuatro alelos más frecuentes en BxH fueron: BoLA DRB3.2*23, 24, 20 y 37, con una frecuencia acumulada de 0.54. Conclusiones. El gen BoLA DRB3.2 fue altamente polimórfico en los dos grupos genéticos (Holstein y BxH) y en los dos grupos se presentó un perfil de alelos similar.
Palabras clave: PCR-RFLP, Bovino, polimorfismo, Complejo Mayor de Histocompatibilidad.
Abstract
Introduction. DRB3 gene of the bovine leukocyte antigen (BoLA) has been widely studied, given the fact that its allelic variations have been associated to infectious diseases and production characteristics. Objective. To determine the frequencies of the alleles of the exon 2of the BoLA DRB3.2 gene obtained by means of the PCR-RFLP technique in Holstein y BONxHolstein (BxH) cows from a dairy herd in Antioquia, Colombia. Materials and methods. 101 cows were genotyped, 76 were Holstein and 25 of the BON X Holstein genetic cross (BxH) for the exon 2 of the DRB3 gene of the bovine leukocyte antigen (BoLA). The BoLA DRB3.2 alleles were identified by means of the PCR-RFLP technique using the Rsal, Haelll and BstYI restriction enzymes and were confirmed with a sequencing process. Results. 27 BoLA DRB3.2 alleles were identified with a frequency ratio from 0.007 to 0.151 in the Holstein cows and from 0.02 to 0.20 in BxH. The four most frequent alleles for Hostein cows were: BoLA DRB3.2 * 23, 22, 24 and 16, with an accumulated frequency of 0.506, and the four most frequent alleles in BxH were: BoLA DRB3.2*23, 24, 20 and 37, with an accumulated frequency of 0.54. Conclusions. The BoLA DRB3.2 gene was highly polymorphic in both genetic groups (Holstein and BxH) and also in both groups there was a similar allelic profile.
Key words: PCR-RFLP, Bovine, polymorphism, Major Histocompatibility Complex.
Resumo
Introdução. O gene DRB3 do antígeno leucocitário bovino (BoLA) ha sido amplamente estudado, dado que sus variantes alélicas hão sido associadas com doenças infecciosas e características produtivas. Objetivo. Determinar as frequências dos alelos do Exão 2 do gene BoLA DRB3 obtidos mediante a técnica PCR-RFLP em vacas Holstein e BONxHolstein (BxH) de um rebanho leiteiro de Antioquia.Materiales e métodos. Foram genotipificadas 101 vacas, 76 de raça Holstein e 25 do cruze BON x Holstein (BxH) para o Exão 2 do gene DRB3 do antígeno leucocitário bovino (BoLA). Os alelos BoLADRB3.2 foram identificados por PCRRFLP, empregando as enzimas de restrição RsaI, HaeIII e BstYI e confirmados por sequenciamento. Resultados. Neste estudo foram identificados 27 alelos BoLA DRB3.2 com um gama de frequências de 0.007 a 0.151 na raça Holstein e de 0.02 a 0.20 em BxH. Os quatro alelos mais frequentes para a raça Holstein foram: BoLA DRB3.2* 23, 22, 24 y 16, com uma frequência acumulada de 0.506 e os quatro alelos mais frequentes en BxH foram: BoLA DRB3.2*23, 24, 20 e 37, com uma frequência acumulada de 0.54. Conclusões. O gene BoLA DRB3.2 foi altamente polimórfico nos dos grupos genéticos (Holstein e BxH) e nos dois grupos se apresentou um perfil de alelos similar.
Palavras chave: PCR-RFLP, Bovino, polimorfismo, Complexo Maior de Histocompatibilidade.
Introducción
Las enfermedades infecciosas son parte de los problemas que afectan frecuentemente la salud de los bovinos, y son un factor limitante en la producción de carne y leche. Esto es reflejado en un aumento de animales descartados prematuramente, descarte de leche contaminada, bajos índice de fertilidad, incremento de gastos veterinarios (Rupp y Boichard, 2003). Teniendo en cuenta esta problemática, en los últimos años, el mejoramiento genético de bovinos para elevar la resistencia a enfermedades ha sido ampliamente usado como herramienta para seleccionar los animales más sanos y más productivos. Sin embargo, en Colombia, la selección para mejorar la salud animal ha sido dirigida mediante selección fenotípica, lo que ha conducido a realizar procesos de selección poco eficientes.
Con el desarrollo de técnicas moleculares específicas para análisis de ADN, se han identificado nuevos marcadores moleculares en genes responsables de la respuesta inmune que pueden ser usados como marcadores para mejorar la salud del animal, como algunos genes del complejo mayor de histocompatibilidad (CMH). El CMH en bovinos es conocido como antígeno leucocitario bovino (BoLA, del inglés bovine leukocyte antigen) y está localizado en el brazo corto del cromosoma 23 de los bovinos (Andersson y Davis, 1994). El BoLA ha sido divido en tres clases: clase I, clase II y clase III. Los genes clase I y los genes clase II (IIa y IIb) codifican glicoproteínas de membrana que se unen a péptidos exógenos. Los genes clase II son normalmente expresados por células del sistema inmune como macrófagos, celulas dendríticas y linfocitos B, las cuales procesan el antígeno y luego lo presentan a los linfocitos T ayudadores para desencadenar la respuesta inmune contra patógenos infecciosos (Banchereau & Steinman, 1998).
Dentro de los genes clase IIa se encuentra ubicado el locus DRB el cual presenta tres genes: el DRBP1, que es un pseudogen sin expresión debido a los múltiples codones de parada; el gen DRB2, que es pobremente expresado y su polimorfismo es muy bajo, y el gen DRB3, que es el de mayor expresión y tiene un alto polimorfismo, principalmente en el exón 2 que codifica el sitio de unión a antígeno (Russell, Marello, Gallagher, Mckeever y Spooner, 1994). Este exón ha sido utilizado como marcador molecular, y se han identificado más de 100 variantes alélicas (Davies, Andersson, Ellis, Hensen, Lewin, Mikko, Muggli-Cocket, Van Der Poel & Russell, 1997; Takeshima & Aida, 2006). Algunos alelos BoLA DRB3.2 han sido asociados a parásitos y enfermedades como la mastitis bovina, entre otros (Sharif, Mallard, Wilkie, Sargeant, Scott, Dekkers & Leslie, 1998; Rupp & Boichard, 2003; Martinez, Machado, Nascimento, Silva, Teodoro, Furlong, Prata, Campos, Guimarães, Azevedo, Pires & Verneque, 2006).
El objetivo de esta investigación fue determinar las frecuencias de los alelos del exón 2 del gen BoLA DRB3 obtenidos mediante la técnica PCR-RFLP en vacas Holstein y BONxHolstein (BxH) de un hato lechero de Antioquia.
Materiales y métodos
Población. Esta investigación se realizó con 101 vacas, 76 de raza Holstein y 25 del cruce genético BONxHolstein del hato Paysandú de la Universidad Nacional de Colombia, sede Medellín, ubicado en el departamento de Antioquia, corregimiento de Santa Elena, al oriente del municipio de Medellín en zona de bosque muy húmedo montañoso bajo (bmh-MB) con una temperatura media de 14 °C y a una altura de 2500 msnm.
Amplificación del exón 2 del gen DRB3. Para cada uno de los 101 individuos se colectaron 5 mL de sangre periférica en tubos al vacío con EDTA como anticoagulante y almacenadas a 4 °C hasta su procesamiento. El ADN fue extraído empleando el método de salting out y luego fue usado para las pruebas de genotipificación. Para la genotipificación de animales, se usó una PCR semianidada mediante la cual se amplificó el exón 2 del gen BoLA DRB3 como lo describen Van Eijk, Stewart-Haynes & Lewin, (1992), con algunas modificaciones. Los cebadores utilizados fueron: HL030 (5'-ATCCTCTCTCTGCAGCACATTTCC-3'), HL031 (5'- TTTAAATTCGCGCTCACCTCGCCGCT-3') y HL032 (5'TCGCCGCTGCACAGTGAAACTCTC-3). La primera ronda de amplificación se realizó en un volumen de reacción de 25 µL que contenía: 0.4 mM de cada dNTP, 0.5 µL de cebador HLO30 y HLO31, 2.5 µL de 10X de buffer termofílico libre de magnesio [500 mM KCl, 100 mM Tris-HCl (pH 9.0), 1 % Tritón X-100], 2.5 µL of 25 mM MgCl2, 12.8 µL de agua ultrapura, 1U de Taq ADN polimerasa (Fermentas, California USA) y 4 µL de ADN genómico. La mezcla de reacción se llevó a cabo en un termociclador (Biometra, Göttingen Germany) con el siguiente programa de ciclos térmicos: desnaturalización inicial a 94 °C por 4 minutos, seguido de 10 ciclos de desnaturalización a 94 °C por 1 minuto cada uno; alineación a 60 °C por 2 minutos; extensión a 72 °C por 1 minuto, y una extensión final a 72 °C por 5 minutos. La segunda ronda de PCR se llevó a cabo en un volumen final de 60 µL que contenía: 0.4mM de cada dNTP; 0.6 µM de cada cebador HLO30 y HLO32; 6 µL de buffer termofílico 10X libre de magnesio; 6 µL de 25 mM MgCl2, 36.9 µL de agua ultra pura; 1U de Taq ADN polimerasa (Fermentas, California U.S.A) y 2.5 µL de ADN amplificado en la primer ronda de PCR. Los ciclos de amplificación fueron los siguientes: desnaturalización inicial a 94 °C por 4 minutos, seguida de 35 ciclos: desnaturalización a 94 °C por 1 minuto, alineación a 67 °C por 2 minutos, extensión a 72 ºC por 1 minuto, seguido por una extensión final de 72 °C por 5 minutos. Como control negativo se hicieron reacciones en ausencia de ADN. El ADN amplificado fue verificado en geles de agarosa al 2 % y visualizado bajo luz ultravioleta en un equipo de fotodocumentación de geles (Biometra, Göttingen Germany).
Digestión con enzimas de restricción. El amplificado obtenido en la segunda ronda de PCR fue digerido con tres enzimas de restricción RsaI, BstYI (New England BioLabs, Ontario Canadá) y HaeIII (Fermentas, California U.S.A). La digestión del fragmento amplificado se llevó a cabo en un volumen final de 20 µL para cada enzima, el cual contenía 10 µL del producto de PCR, 5U de enzima y 2 µL de buffer de la enzima. La incubación para las enzimas RsaI y HaeIII se realizó a 37 °C, y para la enzima BstYI, a 60 °C por un tiempo de 90 minutos. Las muestras digeridas fueron resueltas en geles de agarosa como lo describen Gilliespie, Jayarao, Dowlen & Oliver (1999); la concentración de agarosa utilizada fue de 3 %, teñida con bromuro de etidio 10 µg/mL. Las muestras fueron corridas durante 3 horas a 100 voltios y luego visualizadas en un fotodocumentador de geles (Biometra, Göttingen Germany). La identificación de los alelos del gen BoLA DRB3.2 se realizó por combinación de los diferentes patrones de restricción obtenidos con las enzimas RsaI, BstYI, HaeIII, de acuerdo con la nomenclatura desarrollada por Van Eijk et al. (1992).
De las 101 muestras, 91 fueron amplificadas por PCR semianida, como se describió anteriormente, y fueron purificadas con el kit QIAquick (QIAGEN) y enviadas a la empresa Macrogene Inc. (Seúl Corea) para el análisis de secuenciación. Los productos de PCR fueron secuenciados directamente sin clonación o separación física de los alelos, como lo describen Miltiadou, Law y Russell (2003). Una vez obtenidos los resultados de secuenciación, se procedió a realizar el análisis de secuencias con el programa BioEdit: Sequence Alignment Editor Versión 6 (Hall, 2013), empleando la secuencia referencia, BoLA DRB3.2*1A que corresponde al alelo 24 la cual fue obtenida por Sigurdardottir, Borsch, Gustafsson y Andersson (1991). Con el mismo software fueron identificados los sitios de restricción de cada secuencia para las enzimas RsaI, HaeIII y BstYI, y confirmados usando los patrones de restricción obtenidos por PCR-RFLP.
Análisis de frecuencias alélicas y genotípicas y estructura genética poblacional. La frecuencia de los diferentes alelos se realizó determinando la proporción de cada forma del gen entre el número de copias totales de la población en estudio. Se identificaron los homocigóticos (dos copias del mismo alelo) y los heterocigóticos (una copia de cada alelo) y se determinó la frecuencia F de cada alelo contando los homocigóticos y añadiendo la mitad de los heterocigóticos. También se estimó la deficiencia o exceso de heterocigotos para cada locus en cada población y para la población total con la prueba exacta de Hardy Weinberg (HW) como lo describe Guo y Thompson (1992). Se estimó la diversidad genética comparando la proporción de los genotipos dentro de las subpoblaciones (Holstein y BxH), mediante la heterocigosidad observada (Ho) y heterocigosidad esperada (He). La estructura y diferenciación genética entre las poblaciones se determinaron mediante los estadísticos F de Wright (1969), tanto para la población global como para las subpoblaciones (Holstein y BxH). Todas las pruebas genéticas se estimaron usando el programa ARLEQUIN 3.5.1.2. (Excoffier & Lischer, 2010).
Resultados
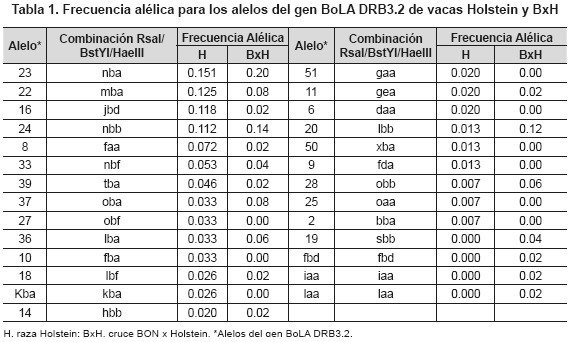
En este estudio fueron identificados 27 alelos BoLA DRB3.2 con un rango de frecuencias de 0.007-0.151 en la raza Holstein y 0.02-0.20 en BxH. Los cuatro alelos más frecuentes para la raza Holstein fueron: BoLA DRB3.2* 23, 22, 24 y 16, con una frecuencia acumulada de 0.506; los cuatro alelos más frecuentes en BxH fueron: BoLA DRB3.2* 23, 24, 20 y 37, con una frecuencia acumulada de 0.54 (Ver tabla 1).
Mediante secuenciación fueron confirmados
21 alelos BoLA DRB3.2, para los cuales se presentan las secuencias predichas de aminoácidos (figura 1). Los números de acceso para la base de datos GenBank de las secuencias nucleotídicas para los 21 alelos BoLA DRB3.2*6, 8, 9, 11, 16, 18, 19, 20, 22, 23, 24, 25, 27, 28, 33, 36, 37, 39, fbd, iaa, laa son en su orden: KF578527, KF578528, KF578525, KF578526, KF578522, KF578529, KF578537, KF578530, KF578519, KF578520, KF578521, KF578535, KF578523, KF578524, KF578531, KF578539, KF578538, KF578533, KF578532, KF578534, KF578536.
A partir de los alelos del gen BoLA DRB3.2 identificados se determinó que el 80.3 % de las vacas Holstein fueron heterocigóticas, y el 19.7 %, homocigóticas. Para BxH se obtuvo un resultado muy similar: 80 % de heterocigóticos y 20 % homocigóticos. Además, se encontraron desviaciones del equilibrio de Hardy-Weinberg (P<0.01) en las dos muestras evaluadas (Holstein y BxH), lo que indica que posiblemeten en este gen se han realizado procesos fuertes de selección.
La heterocigosidad observada (Ho = 0.803) fue menor que la heterocisosidad esperada (He = 0.917) en la raza Holstein; en BxH los resultados fueron similares, la Ho (0.800) y el valor de He fue 0.899. El valor del coeficiente de endogamia (Fis) en toda la población, que incluyó las vacas Holstein y BxH, fue 0.118, el cual no presentó diferencias significativas (P>0.05) y en cuanto a la estructura genética, se determinó un valor de diferenciación genética (Fst) de 0.011 (P>0.05), que igualmente no presentó diferencias significativas.
Discusión
En el hato Paysandú fueron identificados 27 alelos BoLA DRB3.2 en los dos grupos genéticos; Holstein y BxH (tabla 1), lo que constituye un indicativo de un alto polimorfismo para el gen. Para la raza Holstein los 10 alelos identificados con mayor frecuencia sumaron 0.776 de frecuencia acumulada. Siete de estos alelos BoLA DRB3.2*8, 16, 18, 22, 23, 24, y 27, fueron reportados por varios autores dentro de los 10 mas frecuentes en sus respectivos estudios (tabla 2). Esto indica que la raza Holstein tiende a presentar un perfil alélico característico para la raza y que difiere de otras, por ejemplo, en la raza Shorthorn Japonés en la cual los seis alelos más frecuentes fueron: 8, 9, 21, 27, 7 y 24 con 0.70 de frecuencia acumulada (Takeshima, Nakai, Ohta & Aida, 2002). En la raza criollo-argentino, los seis alelos más frecuentes reportados fueron: 15, 18, 24, 20, 27 y 5 con 0.73 de frecuencia acumulada (Giovambattista, Golijow, Dulout & Lojo, 1996).
En la raza Jersey, los siete alelos más frecuentes fueron: 10, 15, 8, 21, 36, 17 y el alelo "ibe" con una frecuencia acumulada de 0.794 (Gilliespie et al. 1999). En la raza Angus los siete alelos más frecuentes fueron: 36, 8, 4, 15, 22, 20 y 10 con 0.766 de frecuencia acumulada (Golijow, 1996) y en la raza Ayshire, los 5 alelos más frecuentes identificados fueron: 8, 7, 28, 10 y 24 (Udina, Haramyshera, Sulimova, Pavlenko, Turkova, Orlova & Ernst, 1998). Los alelos 33, 39, 50 y kba que fueron identificados en este estudio para la raza Holstein con frecuencias alélicas de 0.053, 0.046, 0.013 y 0.026, respectivamente, no han sido reportados en otros estudios para esta raza. El alelo 33 fue reportado solo en la raza Gyr lechero (Da Mota, Gabriel, Martinez & Coutinho, 2002) y el alelo kba fue reportado solo en la raza Jersey Gilliespie et al. 1999).
Para BxH, dentro de los 18 alelos identificados en este estudio, 10 fueron reportados por Martínez, Toro, Montoya, Burbano, Tobón, Gallego & Ariza (2005), para la raza BON dentro de los cuales están: BoLA DRB3.2*8, 11, 16, 18, 19, 20, 22, 23, 24, 37, con una frecuencia de 0.337 comparada con la de este estudio que fue de 0.74. Las discrepancias entre las dos investigaciones se deben a que en la actual investigación se estudió el cruce de razas (BONxHolstein), dentro de las cuales hay vacas F1 (50B:50H), F2 (25B:75H) y F3 (12.5B:87.5H) y por lo tanto el perfil alélico tiende a parecerse a la raza Holstein más que a la raza BON (tabla 2).
Hasta el momento han sido reportados 31 alelos BoLA DRB3.2 para la raza BON, de acuerdo con los resultados obtenidos por Martínez et al. (2005), y en este trabajo se identificaron 3 alelos en BxH (fbd, iaa y laa) que no fueron encontrados en la raza Holstein, lo que probablemente indica que estos alelos provengan de la raza BON o simplemente sea resultado del cruce de las dos razas. Los alelos "iaa" y "laa" han sido solo reportados en la raza Jersey (Gilliespie et al. 1999) y el alelo fbd es un alelo nuevo en bovinos encontrado en el cruce BxH.
Los alelos 22, 23, 24, 16, 33, y 37 fueron identificados en la raza Holstein y en BxH, y están dentro del grupo de los diez más frecuentes en los dos grupos genéticos (tabla 2). Según estos resultados, el perfil de frecuencias de BxH tiende a ser similar con el obtenido en la raza Holstein. Por otra parte, los alelos 19, 20 están dentro de los diez más frecuentes en el grupo genético BxH y han sido reportados en la raza BON (Martínez et al. 2005), con frecuencias de 0.023 y 0.031, respectivamente (dentro de los nueve más frecuentes en esta raza), aunque en la raza Holstein igualmente han sido identificados pero con frecuencias bajas; 0.002 y 0.004, respectivamente (Sharif et al. 1998). Starkenburg, Hansen, Kehrli & Chester-Jones (1997) y Nassiry, Sadeghi, Tohidi, Afshari & Khosravi, (2008) identificaron el alelo 20 en vacas Holstein con una frecuencia de 0.001 y también fue reportado por Dietz, Cohen, Timms & Kehrli (1997a) con una frecuencia de 0.003.
Es importante resaltar que en las dos muestras evaluadas, se encontró una alta diversidad genética para el gen BoLA DRB3.2 con un valor de He de 0.917 en Holstein y 0.899 en BxH y esto posiblemente se daba al número efectivo de alelos (Ne) que para Holstein fue 12.03 y en BxH de 9.92, siendo un indicativo del alto polimorfismo del gen. Resultados similares fueron obtenidos en ganado criollo colombiano (Blanco Orejinegro, Casanareño, Costeño con Cuernos, Chino Santandereano, Caqueteño, Hartón del Valle, Romosinuano, San Martinero y también en ganado especializado de raza Holstein (Hernández, Posso, Muñoz, Giovambattista & Álvarez, 2013).
Conclusiones
El gen BoLA DRB3.2 fue altamente polimórfico en el Hato Paysandú, tanto en la raza Holstein como en vacas BxH, lo que sugiere que en la población evaluada pueden encontrarse alelos tanto de susceptibilidad como de resistencia a enfermedades infecciosas. Además, los valores de heterocigosidad indican que hay una alta variabilidad genética en los dos grupos genéticos estudiados, incluso, fueron identificados algunos alelos en la raza Holstein (33, 39, 50 y kba) y en BxH (fbd, iaa y laa) que no han sido reportados en las respectivas razas. Finalmente, se considera que el gen BoLA DRB3.2 puede ser postulado como marcador molecular, candidato para evaluar características de importancia económica, especialmente relacionadas con salud animal, dado su alto polmorfismo y, por ser un gen que expresa proteínas presentadoras de antígeno.
Referencias bibliográficas
Andersson, L. y Davis, C. J. (1994). The major histocompatibility complex. In: Cell-mediated Immunity in ruminants (ed. by B. M. L. Goddeeris y W.I. Morrison) CRC Press, Boca Raton. [ Links ]
Banchereau, R. y Steinman, R. M. (1998). Dendritic cell and de control of immunity. Nature, 392, 245-252. [ Links ]
Da Mota, A. F., Gabriel, J. E., Martinez, M. L. y Coutinho, L. L. (2002). Distribution of bovine lymphocyte antigen (BoLA-DRB3) alleles in Brazilian dairy Gir cattle (Bos indicus). European Journal of Immunogenetics, 29(3), 223-227. [ Links ]
Davies, C. J., Andersson, L., Ellis, S. A., Hensen, E. J., Lewin, H.A., Mikko, S., Muggli-Cocket, E. N., Van Der Poel, J. J. y Russell, G. C. (1997). Nomenclature for factor of the BoLA system, 1996: report of the ISAG BoLA Nomenclature Commite. Animal Genetics, 28, 159-168. [ Links ]
Dietz, A, B., Detilleux, J. C., Freeman, A. E., Kelley, D. H., Stabel, J. R., Kehrli M. E. (1997b). Genetic association of bovine lymphocyte antigen DRB3 alleles with immunological traits of Holstein cattle. Journal of Dairy Science, 80(2), 400-405. [ Links ]
Dietz, A. B., Cohen, N. D., Timms, L. A. y Kehrli, M. E. (1997a). Bovine lymphocyte antigen class II alleles as risk factors for high somatic cell counts in milk of lactating dairy cows. Journal of Dairy Science, 1997a, 80(2), 406-412. [ Links ]
Excoffier, L. y Lischer, H. (2010). Software Arlequin 3.5.1.2. An integrated software package for population genetics data analysis. Institute of ecology and evolution & swiss institute of bioinformatics. University of Berne. Switzerland. [ Links ]
Gilliespie, B. E., Jayarao, B. M., Dowlen, H. H. y Oliver, S. P. (1999). Analysis and frequency of bovine lymphocyte antigen DRB3.2 alleles in Jersey cow. Journal of Dairy Science, 82, 2049-2053. [ Links ]
Giovambattista, G., Golijow, C. D., Dulout, F. N. y Lojo, M. M. (1996). Gene frequencies of DRB3.2 locus of Argentine Creole cattle. Animal Genetics. 27(1), 55-56. [ Links ]
Golijow, C. D. (1996). Estudio de la reducción de la variabilidad genética por acción de la selección artificial en poblaciones de Bos taurus, PhD Thesis, Universidad Nacional de La Plata, Argentina. [ Links ]
Guo, S. W. y Thompson, E. A. (1992). Performing the exact test of Hardy-Weinberg proportions for multiple alleles. Biometrics, 48, 361-372. [ Links ]
Hall, T. (2013). Bioedit - Biological sequence alignment editor for Win95/98/NT/2K/XP/7. Secuenciación y alineamiento de secuencias. Recuperado de http://www.mbio.ncsu.edu/bioedit/bioedit.html. [ Links ]
Hernández, D., Posso, A., Muñoz, J., Giovambattista., G. y Álvarez, L. (2013). Polimorfismo del gen BoLA DRB3.2 en ganado criollo colombiano. Revista MVZ Córdoba, 18, 3665-3671. [ Links ]
Ledwige, S. A., Mallard, B. A., Gibson, J. P., Jansen, G. B. y Jialing, Z. H. (2001). Multiprimer target PCR for rapid identification of bovine DRB3 alleles. Animal Genetics, 32(4), 219-221. [ Links ]
Martínez, M. L., Machado, M. A., Nascimento, C. S., Silva, M. V., Teodoro, R. L., Furlong, J., Prata, M.C., Campos, A. L., Guimarães, M. F., Azevedo, A. L., Pires, M. F. y Verneque, R. S. (2006). Association of BoLA-DRB3.2 alleles with tick (Boophilus microplus) resistance in cattle. Genetics Molecular Research, 5(3), 513-524. [ Links ]
Martínez, R., Toro, R., Montoya, F., Burbano, M., Tobón, J., Gallego, J. Y. y Ariza, F. (2005). Caracterización del locus BoLA-DRB3 en ganado criollo colombiano y asociación con resistencia a enfermedades. Archivos de Zootecnia, 54:349-356. [ Links ]
Miltiadou, D., Law, A. S. y Russell, G. C. (2003). Establishment of a sequence-based typing system for BoLA DRB3.2 exon 2. Tissue Antigen, 62(1), 55-65. [ Links ]
Nassiry, M. R., Eftekhar, S. F., Mosafer, J., Mohammadi, A., Manshad, E., Ghazanfari, S., Mohammad, M. R. & Sulimova, G. E. (2005). Analysis and Frequency of bovine lymphocyte Antigen (BoLA DRB3) Alleles in Iranian Holstein Cattle. Russian Journal of Genetics, 41(6), 664-668. [ Links ]
Nassiry, M. R., Sadeghi, B., Tohidi, R., Afshari, J. T. y Khosravi, M. (2008). Comparison of bovine lymphocyte antigen DRB3.2 allele frequencies between two subpopulations of Iranian Holstein cattle. African Journal Biotechnology; 7 (15), 2671-2675. [ Links ]
Rupp, R. y Boichard, D. (2003). Genetics of resistance to mastitis in dairy cattle. Veterinary Research, 34(5), 671-688. [ Links ]
Rupp, R., Hernandez, A. y Mallard, B, A. (2007). Association of bovine leukocyte antigen (BoLA) DRB3.2 with immune response, mastitis and production and type traits in Canadian Holsteins. Journal of Dairy Science, 90(2),1029-1038. [ Links ]
Russell, G. C., Marello, K. L., Gallagher, A. Mckeever, D. J. y Spooner, R. L. (1994). Amplification and sequencing of expressed DRB second exons from Bos indicus. Journal of Immunogenetics, 39(6), 432-436. [ Links ]
Sharif, S., Mallard, B. A., Wilkie, B. N., Sargeant, J. M., Scott, H. M., Dekkers, J. C. y Leslie, K. E. (1998). Associations of the bovine major histocompatibility complex DRB3 (BoLA-DRB3) alleles with occurrence of disease and milk somatic cell score in Canadian dairy cattle. Animal Genetics, 29(3), 185-193. [ Links ]
Sigurdardottir, S., Borsch, C., Gustafsson, K. y Andersson, L. (1991). Cloning and sequenceanalysis of 14 DRB alleles of the bovine major histocompatibility complex by using the polymerase chain reaction. Animal Genetics, 22(3), 199-209. [ Links ]
Starkenburg, R. J., Hansen, L. B., Kehrli, J. R., Chester-Jones, H. (1997). Frequencies and Effects of Alternative DRB3.2 Alleles of bovine lymphocyte antigen for Holstein in milk selection and control lines. Journal of Dairy Science, 80(12), 3411-3419. [ Links ]
Takeshima, S. N. y Aida, Y. (2006). Structure, Function and disease susceptibility of the bovine major histocompatibility complex. Animal Science Journal, 77(2), 138-150. [ Links ]
Takeshima, S. N., Nakai, Y., Ohta, M. y Aida, Y. (2002). Characterization of DRB3 alleles in the MHC of Japanese Shorthon Cattle by Polymerase Chain reaction-sequence based typing. Journal of Dairy Science, 85(6), 1630-1632. [ Links ]
Udina, I. G., Haramyshera, E. E., Sulimova, G. E., Pavlenko, S. P., Turkova, S. O., Orlova, A. R. y Ernst, L. K. (1998). Comparative analysis of Ayrshire and Black Pied cattle breeds by histocompatibility markers. Genetika, 34(12):1668-1674. [ Links ]
Van Eijk, M. J. T., Stewart-Haynes, J. A. y Lewin, H. A. (1992). Extensive polymorphism of the BoLA-DRB3 gene distinguished by PCR-RFLP. Animal Genetics, 23(6), 483-496. [ Links ]
Wright, S. (1969). Evolution and Genetics of Populations, The theory of gene frequencies. Vol. II Chicago: University of Chicago Press. [ Links ]