Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Ingeniería y Ciencia
versión impresa ISSN 1794-9165
ing.cienc. v.6 n.11 Medellín ene./jun. 2010
Dinámica del virus de la hepatitis C con carga viral y células T citotóxicas y monitoreo del daño hepático sin biopsias1
Dinâmica do vírus da hepatite C com ónus viral e células T citotóxicas e monitoramento do dano hepático sem biopsias
Hepatitis C virus dynamics with viral load and T killer cells and hepatic damage monitoring without liver biopsies
Justino Alavez–Ramírez2 y Jorge López–López3
1 Trabajo realizado en el marco del proyecto ”Estimación numérica de parámetros de un modelo matemático de la hepatitis C con observaciones parciales”. Registro PROMEP/103.5/08/3189. División Académica de Ciencias Básicas, Universidad Juárez Autónoma de Tabasco (UJAT), México.
2 Doctor en ciencias, justino.alavez@basicas.ujat.mx, profesor de la División Académica de Ciencias Básicas, UJAT, Villahermosa–México.
3 Maestro en ciencias, jorge.lopez@dacb.ujat.mx, profesor de la División Académica de Ciencias Básicas, UJAT, Tabasco–México.
(Recepción: 18-ago-2009. Modificación: 07-ene-2010. Aceptación: 20-ene-2010)
Resumen
En este trabajo se muestra la posibilidad de predecir el estado de un individuo infectado por el virus de la hepatitis C y el monitoreo de la evolución de su carga viral y daño hepático, sin necesidad de biopsias, mediante la estimación numérica de los parámetros de un modelo matemático de cuatro poblaciones (hepatocitos sanos y enfermos, carga viral y células T citotóxicas), usando mediciones de la carga viral y células T citotóxicas. También se muestra que la población de células T citotóxicas no influye significativamente en la estimación de los parámetros, por lo que es suficiente la medición de la carga viral y el modelo reducido a tres poblaciones (hepatocitos sanos, enfermos y carga viral) para diagnosticar la evolución de la enfermedad de un individuo infectado.
Palabras claves: hepatitis C, carga viral, células T citotóxicas, parámetro umbral, estimación de parámetros.
Resumo
Neste trabalho mostra-se a possibilidade de pré-dizer o estado de um indivíduo infectado pelo vírus da hepatite C e o monitoramento da evolução do seu ónus viral e dano hepático, sem necessidade de biopsias, mediante a estimação numérica dos parâmetros de um modelo matemático de quatro populações (hepatócitos sãos e doentes, ónus viral e células T citotóxicas), usando medições do ónus viral e células T citotóxicas. Também mostra que a população de células T citotóxicas não influi significativamente na estimação dos parâmetros, pelo qual é suficiente a medição do ónus viral e o modelo reduzido a três populações (hepatócitos sãos, doentes e ónus viral) para diagnosticar a evolução da doença de um indivíduo infectado.
Palavras chaves: hepatite C, ónus viral, células T citotóxicas, parâmetro umbral, estimação de parâmetros.
Abstract
In this work is shown the possibility to predict the stage of a person infected by hepatitis C virus, and the monitoring of the viral load and hepatic damage, without liver biopsies. This is done by numerically estimating the parameters of a mathematical model involving four populations (uninfected liver cells, infected liver cells, viral load and T killer cells) and using a few measurements of viral load and T killer cells. Also, is shown that the influence of the T killer cells population is insignificant in this process, concluding that measurements of viral load and the reduced model of three populations (uninfected liver cells, infected liver cells and viral load) is enough to predict the evolution of an infected person.
Key words: hepatitis C, viral load, cytotoxic T cells, threshold parameter, parameter estimation.
1 Introducción
La Organización Mundial de la Salud (OMS) ha reconocido que la infección crónica por el virus de la hepatitis C (VHC) representa un problema fuerte de la salud pública. Afecta a alrededor de 200 millones (aproximadamente el 3 %) de la población mundial [1], y entre el 0,7 y 1,6 % de la población mexicana [2, 3, 4]. No existe vacuna para la hepatitis C y tampoco una cura muy exitosa. Una vez que un niño contrae una infección crónica por el VHC, en la mayoría de los casos, la infección durará toda la vida [5]. Los pacientes con hepatitis C suelen estar totalmente asintomáticos durante largos periodos. Se ha estimado que el tiempo promedio entre la infección inicial y el diagnóstico de hepatitis C crónica, cirrosis, y carcinoma hepatocelular, es de 10, 20 y 30 años, respectivamente [6, 7]. Hay pacientes que pueden desarrollar carcinoma hepatocelular directamente de una hepatitis C crónica, sin desarrollar antes una cirrosis, especialmente aquéllos con genotipo 1b [7]. Entre el 5 y 6 % de los bebés nacidos de mujeres infectadas contraen la infección de sus madres y la mayoría de dichos infantes desarrollará una infección crónica. Entre el 15 y 25 % de los adultos expuestos al virus podrán combatir la infección y recuperarse, pero entre el 75 y 85 % restante padecerá una infección crónica de por vida [5]. Actualmente el tratamiento más eficaz del que se dispone para combatir la infección aguda y crónica de hepatitis C es el interferón pegilado α − 2a ó α − 2b combinado con ribavirina [8, 9, 10]. Aproximadamente el 50 % de los pacientes tratados en los ensayos clínicos, dependiendo del genotipo de la hepatitis C, son capaces de eliminar el VHC y curarse [10]. Este tratamiento es altamente beneficioso tanto para evitar la aparición de cirrosis, como para reducir el número de pacientes que hubiesen requerido un trasplante hepático por la evolución terminal de la cirrosis, o por haber desarrollado un carcinoma hepatocelular.
Una biopsia de hígado es una técnica invasiva que se usa para evaluar el grado del daño hepático (inflamación, fibrosis, cirrosis o carcinoma hepatocelular). ésta es la mejor herramienta de la que se dispone hasta ahora para una evaluación inicial del daño hepático, y para la evaluación de la eficacia del tratamiento con medicamentos [11, 12, 13]. La biopsia de hígado se utiliza comúnmente cuando el enfermo se encuentra en la fase inicial a media de la enfermedad, pues en otro caso, aumentan los riesgos de complicaciones relacionados [14, 15].
La evaluación de la carga viral del VHC es una técnica reciente que permite conocer la intensidad de la infección en función del número de partículas virales que circulan por la sangre. Así que un propósito central del tratamiento es conseguir una disminución significativa de la carga viral. Aunque no se ha logrado establecer una correlación entre la carga viral y el estado de salud del hígado, se acepta que mientras más baja sea la carga viral, hay mayores posibilidades de responder bien al tratamiento contra el VHC [16]. Con el uso del conteo de la carga viral para evaluar el grado de infección en el paciente, se podría evitar la biopsia.
Los modelos matemáticos pueden ser útiles para evaluar el daño hepático causado por el virus de la hepatitis C, evaluar la evolución de la carga viral y la dinámica de la respuesta inmune del individuo infectado, así como evaluar el grado de eficacia de la medicación frente al virus [1, 17, 18, 19, 20, 21,22, 23, 24, 25, 26, 27]. En [20] se presentan tres resultados sobre la dinámica de la hepatitis C, usando el modelo estándar reducido a tres poblaciones (hepatocitos sanos y enfermos, y carga viral), y el modelo de la dinámica viral con ALT. El primer resultado muestra que el parámetro umbral R0 es el mismo para ambos modelos; el segundo resultado muestra que la ecuación adicional que describe la dinámica del nivel de concentración de las enzimas ALT en el segundo modelo, no influye significativamente en la estimación de los parámetros que determinan R0 ; y el tercer resultado muestra que es posible el monitoreo de la evolución de la carga viral y el daño hepático, mediante la estimación numérica de los parámetros considerando solamente un número aceptable de las mediciones de la carga viral y del nivel de concentración de las enzimas ALT, y la propuesta de razonables condiciones iniciales.
En este trabajo se reafirma el tercer resultado de [20], pero empleando el modelo (1) propuesto por [26] de cuatro poblaciones (hepatocitos sanos y enfermos, carga viral y células T citotóxicas) para la dinámica de la hepatitis C, cuyo parámetro umbral R0 está dado en (2) y es el mismo parámetro umbral de los modelos que se dan en [20]. El primer objetivo consiste en mostrar que es posible predecir el estado de un individuo infectado por el virus de la hepatitis C, y el monitoreo de la evolución de su carga viral y daño hepático (sección 3), sin necesidad de biopsias, mediante la estimación numérica del parámetro umbral R0 y demás parámetros del modelo (1), pero ahora considerando solamente un número aceptable de mediciones de la carga viral y de células T citotóxicas, y la propuesta de razonables condiciones iniciales que bien la clínica puede aportar. El segundo objetivo consiste en mostrar que las células T citotóxicas, no influyen significativamente en la estimación de los parámetros del modelo (1), mediante un estudio comparativo (sección 4) de la estimación numérica de los parámetros de dicho modelo con los parámetros de los modelos que se reportan en [20].
2 Modelo matemático de la hepatitis C
Un modelo matemático que estudia la dinámica de la hepatitis C fue propuesto en [26], y está dado por el siguiente sistema de ecuaciones diferenciales ordinarias de primer orden:
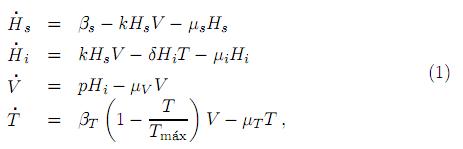
donde Hs(t) es la población (células/mm3) de hepatocitos sanos en el hígado al tiempo t. Se supone que se generan a una tasa constante βs y mueren a una tasa per cápita µs. Hi(t) es la población (células/mm3) de hepatocitos infectados (enfermos) en el hígado al tiempo t. Se supone que los hepatocitos sanos Hs son infectados y se convierten en Hi, con una tasa proporcional al producto del número de hepatocitos sanos Hs por la carga viral V, con constante de proporcionalidad k. V(t) es la carga viral (cantidad de ácido ribonucleico (ARN) o material genético del VHC) que circula en la sangre al tiempo t. Se mide en unidades internacionales por mililitro de sangre (UI/mL) o en copias/mL. Se supone que los VHC se producen dentro de un hepatocito infectado Hi a una razón de p viriones por hepatocito infectado por día, y mueren a una tasa de mortalidad per cápita µV. Dado que los hepatocitos infectados Hi mueren a causa de la replicación del VHC en su interior, se supondrá que mueren a una tasa per cápita µi, µi ≥ µs. T(t) es la población (células/mm3) de células T citotóxicas al tiempo t, también llamadas células T killer (células de tipo CD8+ que destruyen las células infectadas). Se supone que las células T citotóxicas destruyen a los hepatocitos infectados Hi a una tasa proporcional al producto del número de hepatocitos infectados Hi por el número de células T killer, con constante de proporcionalidad δ. Bajo la presencia de los VHC, las células T citotóxicas se producen de manera proporcional a la carga viral V con una tasa de saturación βT(1 − T/Tmáx), donde βT es la tasa de reproducción de las células T citotóxicas al ignorar el supuesto de saturación, y Tmáx es el nivel máximo de células T citotóxicas en el organismo. Por otro lado, se supone que dichas células mueren a una tasa per cápita µT.
Modelos similares han sido estudiados por [17, 19, 20, 21, 23, 24, 27, 28], entre otros. Un primer aspecto por resaltar es que el dominio de definición del sistema (1) con sentido biológico es el conjunto positivo
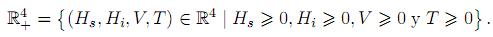
Aunque en realidad si µi ≥ µs, se demuestra en [26] que la dinámica de este sistema está restringida al subconjunto positivamente invariante: Ω ⊂ 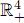
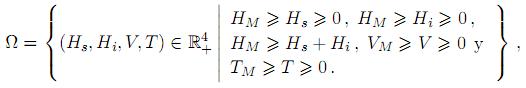
donde HM = βs/µs, VM= (p/µV) HM, TM = (βT/µ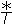 ) VM < Tmáx y µ
) VM < Tmáx y µ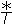 = µT + (βT/Tmáx) VM. El conjunto Ω es positivamente invariante con respecto a (1), en el sentido de que toda trayectoria solución de (1) con condiciones iniciales en Ω permanece en Ω para todo t ≥ 0. HM representa el tamaño máximo de la población de hepatocitos sanos en un milímetro cúbico de hígado de un individuo sano, VM la cantidad máxima de viriones que todos los hepatocitos pueden producir durante toda su vida media, y TM el número máximo de células T citotóxicas generadas en un individuo con hepatitis C. El parámetro umbral del modelo está dado por
= µT + (βT/Tmáx) VM. El conjunto Ω es positivamente invariante con respecto a (1), en el sentido de que toda trayectoria solución de (1) con condiciones iniciales en Ω permanece en Ω para todo t ≥ 0. HM representa el tamaño máximo de la población de hepatocitos sanos en un milímetro cúbico de hígado de un individuo sano, VM la cantidad máxima de viriones que todos los hepatocitos pueden producir durante toda su vida media, y TM el número máximo de células T citotóxicas generadas en un individuo con hepatitis C. El parámetro umbral del modelo está dado por

A R0 también se le llama número de reproductividad básico del virus, pues su valor juega un papel central en el estudio cualitativo del comportamiento global de las soluciones de (1) (es decir, del comportamiento de la evolución de la enfermedad hacia la cura o bien hacia la enfermedad crónica), con implicaciones relevantes para el tratamiento de la hepatitis C.
Los resultados centrales del análisis de (1) son:
A). Si R0 ≤ 1, entonces (1) tiene un solo estado de equilibrio admisible en Ω, el trivial I0 = (βs/µs, 0, 0, 0), el cual es globalmente asintóticamente estable (lo que dice que el individuo infectado, eventualmente siempre se cura).
B). Si R0 > 1, entonces (1) tiene dos estados de equilibrio admisibles en Ω:
i). El trivial I0 = (βs/µs, 0, 0, 0), que es ahora inestable.
ii). El no trivial
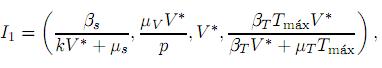
que está en el interior de Ω cuando µi > µs, es localmente asintóticamente estable y corresponde al enfermo endémico de hepatitis C. Aquí V∗ es la solución positiva de la ecuación cuadrática
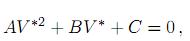
la cual existe, es única y satisface que 0 < V∗ < VM cuando R0 > 1. Los coeficientes de la ecuación cuadrática están dados por
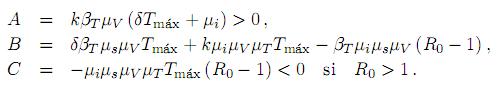
Una posible estrategia para la curación de un enfermo crónico de hepatitis C de acuerdo con los resultados de (1) fue propuesto por [18], y consiste en reducir el valor de su parámetro umbral R0 dado por (2) por debajo de la unidad. Dado que R0 = HMkp/µiµV, donde HM se puede considerar constante para un individuo, lo que se propone es modificar bajo tratamiento sólo los valores de k, p, µi y µV. Esto sugiere, para la cura de un enfermo crónico de hepatitis C, el uso de al menos cuatro medicamentos: uno que reduzca la tasa k de infección a una tasa crítica kc, otro que reduzca la tasa p de replicación viral a una tasa crítica pc, un tercero que aumente la tasa per cápita µede eliminación de células infectadas a una tasa per cápita crítica µ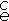 , y uno más que aumente la tasa per cápita µV de eliminación viral a una tasa per cápita crítica µ
, y uno más que aumente la tasa per cápita µV de eliminación viral a una tasa per cápita crítica µ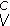 . Así que dada 0 < ε < 1 de tolerancia, si se reduce el parámetro umbral R0 del enfermo crónico a un parámetro umbral crítico: R
. Así que dada 0 < ε < 1 de tolerancia, si se reduce el parámetro umbral R0 del enfermo crónico a un parámetro umbral crítico: R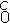 ≡ HMkcpc/µ
≡ HMkcpc/µ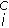 µ
µ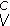 ≤ 1 − ε, entonces el enfermo se cura.
≤ 1 − ε, entonces el enfermo se cura.
3 Estimación numérica de parámetros
La importancia de estimar con exactitud el parámetro umbral R0 del modelo matemático de la hepatitis C, para un paciente en particular, es que permite diagnosticar la evolución de la enfermedad hacia la curación del paciente o bien hacia la enfermedad crónica. Pero para estimar R0 se necesita estimar primero los parámetros βs, k, p, µs, µi y µV del modelo (1). De hecho, estimando todos los parámetros involucrados en (1), así como las condiciones iniciales Hs0, Hi0, V0 y T0, será posible evaluar la evolución de la carga viral V a un paciente con hepatitis C, así como el comportamiento de la población de sus células hepáticas sanas Hs, células hepáticas infectadas Hi y células T citotóxicas. En esta sección, se investiga hasta qué punto es posible estimar numéricamente los parámetros k, p, µi, µV, µT, δ, βT y Tmáx, la carga viral inicial V0 y la población inicial de células T citotóxicas T0, contando con sólo un número razonable de observaciones de la carga viral V y de células T citotóxicas. Es importante observar que todos los parámetros y condiciones iniciales en el modelo son no negativos, por lo que el espacio admisible de parámetros Θ lo definen los vectores positivos de la forma
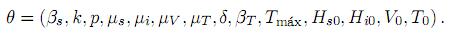
La forma de estimar los parámetros y condiciones iniciales, se hará en el sentido clásico de mínimos de cuadrados, es decir, bajo el supuesto de que sólo se cuenta con un número suficiente de observaciones de la carga viral V y de células T citotóxicas, dados por la tabla
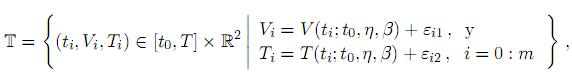
donde εij ∈ 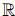 son errores de observaciones (variables aleatorias independientes e idénticamente distribuidos con media cero y varianza σ2), con 2(m + 1) > 14; la función objetivo g(θ) a minimizar en este caso [18, 29] es
son errores de observaciones (variables aleatorias independientes e idénticamente distribuidos con media cero y varianza σ2), con 2(m + 1) > 14; la función objetivo g(θ) a minimizar en este caso [18, 29] es
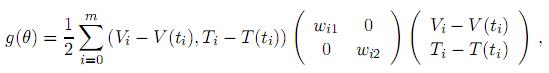
donde V(ti) = V(ti; T0, η, β), T(ti) = T (ti; T0, η, β), η = (Hs0, Hi0, V0, T0), β = (βs, k, p, µs, µi, µV, µT, δ, βT, Tmáx) y θ = (β, η). Los pesos wij se calcularon de acuerdo con [20, 29]:
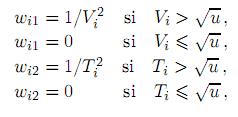
donde u es la unidad de redondeo de la aritmética de punto flotante usada.
El software que se está empleando para resolver el problema es el paquete DIFFPAR de [29], escrito en MATLAB. En DIFFPAR se usa un algoritmo que se basa en el método de Runge–Kutta de tercer orden para problemas no stiff, y un método BDF de tercer orden para problemas stiff. Es importante mencionar que existen otros métodos para estimar parámetros de modelos de la dinámica de la hepatitis C, como el que se usa en [17, 19, 27], entre otros. Otros métodos para identificar parámetros en ecuaciones diferenciales ordinarias, se pueden encontrar en [30, 31, 32].
3.1 Caso endémico
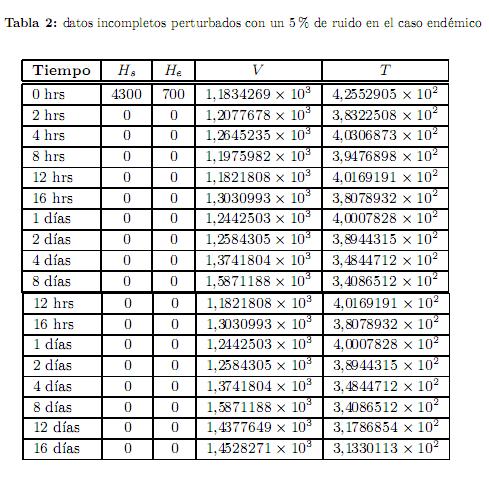
Para generar los datos que se necesitan para la estimación numérica de los parámetros, se resuelve (1) empleando los valores de los parámetros y condiciones iniciales que se muestran en la tabla 1, en un lapso de tiempo de 0 a 16 días. A los valores obtenidos de la carga viral V y del número de células T citotóxicas se les cargó un porcentaje de ruido, de acuerdo con la fórmula [18, 29]
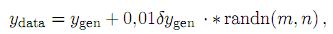
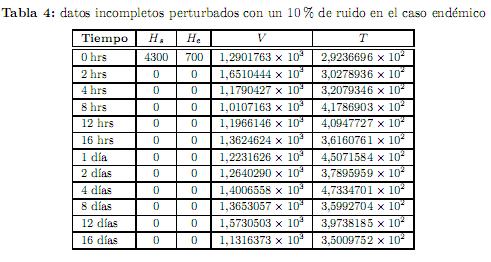
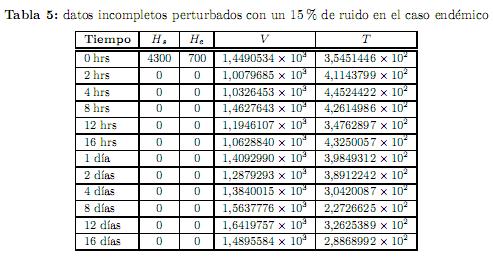
donde ygen es la matriz de datos exactos, δ es la perturbación dada, y randn es la función de MATLAB que genera números aleatorios con distribución normal con media 0 y varianza 1. Así que la matriz de datos perturbados y data, es una variable aleatoria con distribución normal con media ygen y varianza σ2= (0,01δygen)2. En todos los experimentos se suponen conocidos los valores numéricos de βs, µs, de la población inicial de células hepáticas sanas Hs0 y de la población inicial de células hepáticas enfermas Hi0, pero desconocidos los demás valores de Hsy Hi. Se reportan los resultados de cuatro experimentos de este caso.
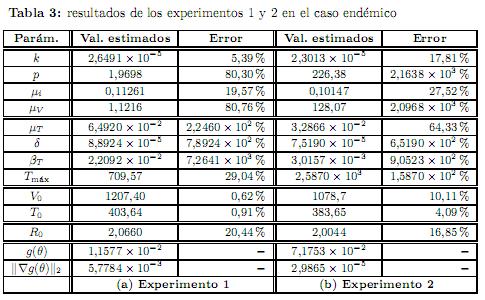
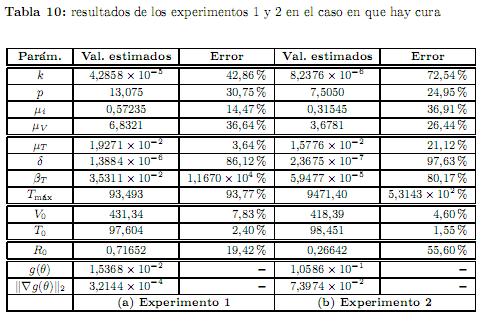
Experimento 1. Los datos utilizados para este primer experimento se obtuvieron cargando un ruido del orden del 5 % a los datos exactos, y son los que se muestran en la tabla 2. Iniciando el proceso de optimización desde β0= (15.4, 10−5,13, 3.08 y× 10−3,0.3, 5, 10−2,10−4,5.0×10−4,1200), junto con las condiciones iniciales y0= (4300, 700, 1000, 500), se obtuvieron los resultados que se dan en la tabla 3(a).
Experimento 2. Los datos que se usaron para este experimento se obtuvieron cargando un ruido del orden del 10 % a los datos exactos, mismos que se dan en la tabla 4. El punto de inicio que se usó en el proceso de optimización fue β0= (15.4, 6.0×10−5,15, 3.08×10−3,0.3, 9, 4.0×10−2,8.0×10−5,8.0×10−4,1100), junto con las condiciones iniciales y0= (4300, 700, 1500, 600). Los resultados obtenidos se muestran en la tabla 3(b).
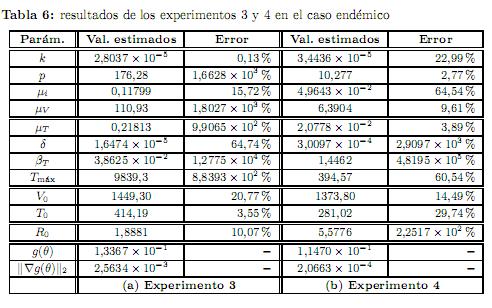
Experimento 3. Los datos empleados en este experimento se obtuvieron cargando un ruido del orden del 15 % a los datos exactos, mismos que se muestran en la tabla 5. El punto de inicio que se usó para el proceso de optimización fue β0= (15.4, 6.0×10−5,15, 3.08×10−3,0.2, 9, 7.0×10−2,2.0×10−5,6.0×10−5, 800), junto con las condiciones iniciales y0= (4300, 700, 1000, 600). Los resultados obtenidos se dan en la tabla 6(a).
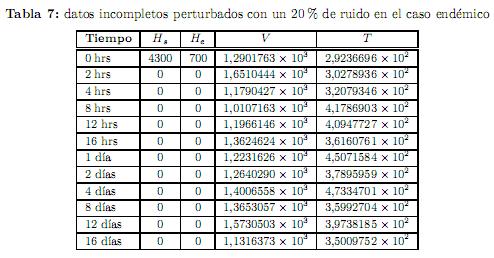
Experimento 4. Los datos empleados en este último experimento se obtuvieron cargando un ruido del orden del 20 % a los datos exactos, mismos que se muestran en la tabla 7. El punto de inicio que se usó para la optimización
fue β0= (15.4, 6.0×10−5,12, 3.08×10−3,0.3, 8, 4.0×10−2,1.5×10−4,2.0×10−2, 1500), junto con las condiciones iniciales y0= (4300, 700, 1000, 500). Los resultados obtenidos se muestran en la tabla 6(b).
3.2 Caso en que hay cura
Los valores de los parámetros y condiciones iniciales que se usaron para generar los datos exactos en un lapso de tiempo de 0 a 16 días para este caso, se tomaron de [26] y son los que se muestran en la tabla 8. También se reportan los resultados de cuatro experimentos de este caso.
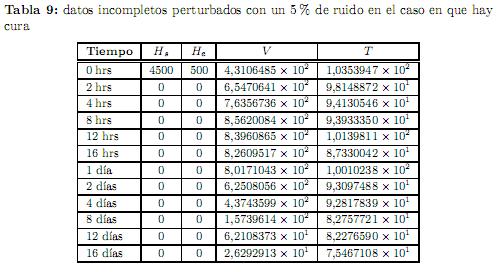
Experimento 1. Los datos que se usaron para este experimento se obtuvieron cargando un ruido del orden del 5 % a los datos exactos, y son los que se muestran en la tabla 9. El punto de inicio para la optimización fue β0= (100, 2.0×10−5,12, 2.0×10−2,0.1, 1, 5.0×10−2,4500.0×10−5,7.0×10−4,1100), junto con las condiciones iniciales y0= (4500, , 500, 30). Los resultados obtenidos se muestran en la tabla 10(a).
Experimento 2. Los datos que se usaron para este experimento se obtuvieron cargando un ruido del orden del 10 % a los datos exactos, mismos que se muestran en la tabla 11. El punto de inicio usado para la optimización fue β0= (100, 7.0×10−5,6, 2.0×10−2,0.2,3, 4.0×10−2,6.0×10−5,7.0×10−4,900), junto con las condiciones iniciales y0= (4500, 500, 500, 120). Los resultados obtenidos se muestran en la tabla 10(b).
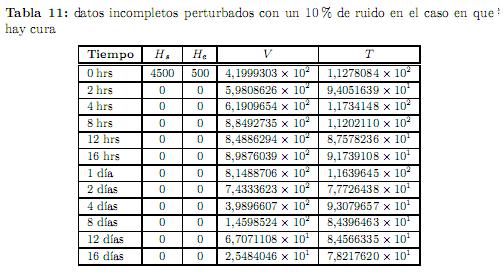
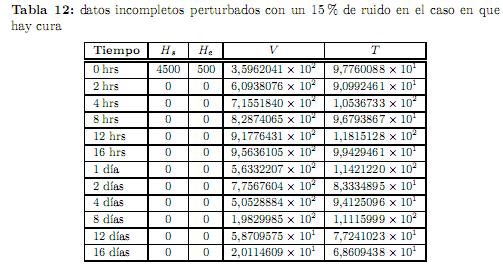
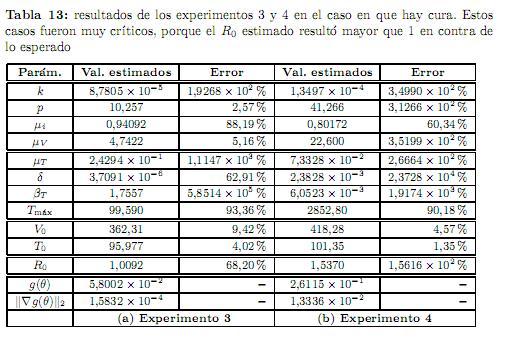
Experimento 3. Los datos que se utilizaron para realizar este experimento se obtuvieron cargando un ruido del orden del 15 % a los datos exactos, mismos que se muestran en la tabla 12. En este caso fue difícil de estimar los parámetros, se tuvo que eliminar el dato que corresponde a t = 2 hrs de la tabla 12 que resultó ser un dato aberrante. A pesar de ello, el mejor resultado se obtuvo iniciando el proceso de optimización desde el punto β0= (100, 2.0×10−5,14, 2.0×10−2,1, 10, 4.0×10−2,1.5×10−5,2.0×10−4,500),
junto con las condiciones iniciales y0= (4500, 500, 700, 300). Los resultados que se obtuvieron se muestran en la tabla 13(a).
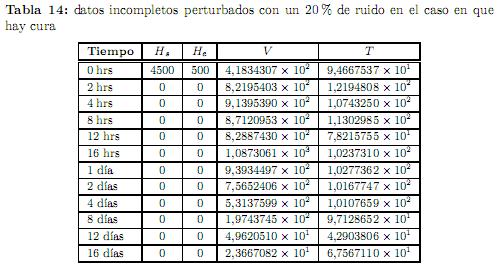
Experimento 4. Los datos que se usaron en este último experimento se obtuvieron cargando un ruido del orden del 20 % a los datos exactos, mismos que se muestran en la tabla 14. El punto de inicio que se usó para el proceso de optimización fue β0= (100, 1.5 x 10−5,15,2.0×10−2,1,9,3.0×−2, 1,5 x 10−5, 5.0 x 10−5,5.0 x 10−5, 5.0 x 10−4, 800), junto con las condiciones iniciales y0= (4500, , 700, 200). Los resultados obtenidos se muestran en la tabla 13(b).
4 Comparación numérica de los modelos de la hepatitis C
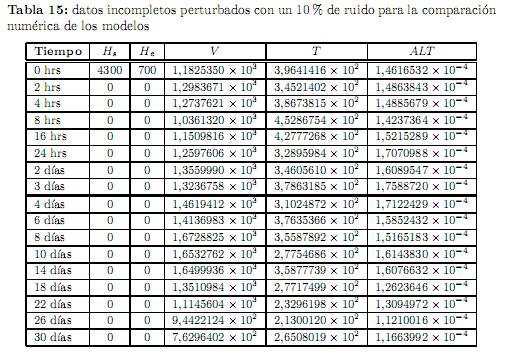
En esta sección se realiza un estudio comparativo del modelo completo de cuatro poblaciones dado en (1), con los modelos reducido de tres poblaciones y de la dinámica viral con ALT que se estudian en [23] y [20], mediante la estimación numérica de los parámetros correspondientes. Para ello, se generaron datos usando los valores de los parámetros y condiciones iniciales que se dan en la tabla 1 con el modelo completo (1), perturbados con un ruido del orden del 10 % en los valores numéricos para la carga viral V y del número de células T citotóxicas, en un lapso de tiempo de 0 a 30 días. Los datos generados se muestran en la cuarta y quinta columnas de la tabla 15. Los datos
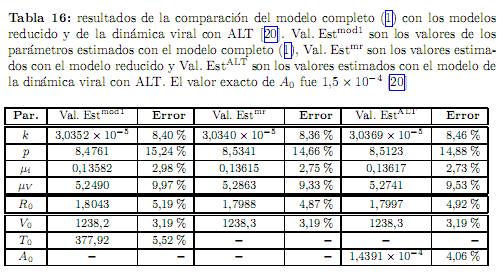
que aparecen en la sexta columna de dicha tabla se tomaron de [20], y corresponden al nivel de concentración de ALT perturbados con un ruido también del orden del 10 %. Los resultados de la comparación se muestran en la tabla 16. El punto de inicio para la optimización con el modelo completo (1) fue β0= (15.4, 1.5 x 10−5, 20, 3.08 x 10−3 0.10, 8, 10−2, 8.0 × 10−7, 2.0 × 10−4, 1100) junto con la condición inicial y0= (4300, 700, 800, 500). El punto de inicio con el modelo reducido a tres poblaciones fue β0= (15.4, 1.5, × 10−5, 3.08 × 10−3, β0=, 8.0 x 10−2 20, 10) junto con la condición inicial y0= (4300, 700, 850); y el punto de inicio con el modelo de la dinámica viral con ALT fue β0= (15.4, 4.0 x 10−5, 3.08 × 10−3 0.3, 20, 8,10−5,4.0×10−8, 4.5 ×10−8,1) junto con la condición inicial y0= (4300, 700, 800, 3,0 ×10−8).
Los intervalos de confianza al 95 % de los parámetros estimados con el con el modelo completo son: k = 3,0352 x 10−5 ± 3,0 × 10−9, p = 8,4761 ± 5,28 × 10−2 µi = 0,13582 ± 1,1 ×10−4 µV= 5,2490 ± 3,27 ×10−2, µT= 1,8008 ×10−2 ± 6,33 ×10−4, δ = 1,,6079 ×10−6 ± 3,949 ×10−7, βT = 1,3840 ×10−5 ± 1,02860 ×10−4, Tmáx= 1,1000 × 103 ± 3,1740 × 104, V0= 1,2382 × 103 ± 0,3 y T0= 3,7792 × 102 ± 0,028. Con el modelo reducido a tres poblaciones, dichos intervalos de confianza al 95 % resultaron ser: k= 3,0340 ×10−5 ±1,0 ×10−9, p = 8,5341 ± 1,47 ×10−2, µi = 0,13615 ± 1,0 ×10−5, µV = 5, ± 9,×10−3,2863 ± 9,1 ×10−3
y V0= 1,2383 × 103 ± 0,1; y con el modelo de la dinámica viral con ALT, los intervalos de confianza también al 95 % resultaron ser: k = 3,0369 × 105 ± 2,0 × 10−9 p = 8,5123 ± 2,74 × 10−2, µi = 0,13617 ± 1,0 × 10−5, µV = 5,2741 ± 1,7 × 10−2, βA = 5,6366 × 10−5 ± 1,1712, αs = 1,6012 × 10−8 ± 2,5813 × 10−4, αe = 2,1633 × 10−8 ± 8,6720 × 10−4, µA = 8,0801 × 10−1 ± 1,6975 × 10−4, V0= 1,2383 × 103 ± 0,1 y A0 = 1,4391 × 10−4 ± 1,2417 × 10−1.
5 Discusión
Las condiciones iniciales V0 y T0, que representan la carga viral inicial y la población inicial de células T citotóxicas de un individuo infectado, se pueden estimar muy bien como se muestra en todos los experimentos realizados en las secciones 3.1 y 3.2. La condición inicial V0 se estimó muy bien pues se obtuvieron estimaciones con menos del 21 % de error en el 100 % de los casos
analizados, y T0 se estimó muy bien con estimaciones menores del 5 % de error en el 87,5 % de los casos analizados, y solamente en un 12,5 % de los casos el error fue del orden del 30 %.
La tasa de infección k por el virus de la hepatitis C, se estimó razonablemente bien en el caso endémico, pues se obtuvieron estimaciones con menos del 23 %, mientras que para el caso en que hay cura los errores de las estimaciones fueron muy grandes. La tasa de replicación viral p se estimó con menos del 31 % de error en un 50 % de los casos analizados. La tasa de eliminación de células hepáticas infectadas µe, se estimó con menos del 28 % de error en un 50 % de los casos analizados. La tasa de mortalidad del virus µV, se estimó con menos del 27 % de error en solamente un 37,5 de los casos analizados.
El parámetro umbral R0 se estimó razonablemente bien con un error menor del 21 % en un 50 % de los casos analizados. En un 75 % de los casos analizados se mantuvo la tendencia de que R0 fuera mayor o menor que 1 según el caso; mientras que en un 25 % que corresponden en el caso en que hay cura, R0 resultó mayor que 1 en contra de lo esperado.
La tasa de mortalidad µT de las células T citotóxicas, se estimó con un error menor del 22 % en un 37,5 % de los casos analizados. Los parámetros δ, βT y Tmáx no se estimaron bien en casi todos los casos analizados. Estos últimos parámetros resultaron ser muy sensibles a perturbaciones en los datos y a parámetros iniciales.
Los resultados de la comparación numérica de los modelos son excelentes como se muestra en la tabla 16. El error en los parámetros estimados k, p, µi, µV, R0 y V0, son casi iguales y razonablemente pequeños en los tres modelos, y los intervalos de confianza al 95 % que corresponden a estos parámetros son también muy buenos. Esto muestra, por un lado, que el método de DIFFPAR es suficiente para la estimación numérica de los parámetros; y por otro, confirma que ni el nivel de concentración de las enzimas ALT ni la población de las células T killer, influyen significativamente en la estimación de los parámetros antes mencionados.
También es importante resaltar que todos los experimentos de la sección 3 se hicieron con 12 datos tomados de 0 a 16 días para cada variable de estado, mientras que el experimento de la sección 4 se hizo con 17 datos tomados de 0 a 30 días. Los resultados muestran que se obtienen mejores estimaciones de los parámetros al aumentar el número de datos (tablas 3(b) y 16).
Se resumen dos conclusiones importantes, la primera es que es posible predecir el estado de un individuo infectado por el virus de la hepatitis C mediante la estimación numérica del parámetro umbral R0 del modelo (1), y el monitoreo de dos de las variables (carga viral y daño hepático) más relevantes de la dinámica de la enfermedad, si se dispone de un número suficiente de mediciones de la carga viral y de células T citotóxicas y la propuesta de razonables condiciones iniciales, sin necesidad de efectuar biopsia alguna. La segunda conclusión, la más importante, es que la población de células T citotóxicas no influye significativamente en la estimación de los parámetros, por lo que es suficiente la medición de la carga viral y el modelo reducido para diagnosticar la evolución de la enfermedad de un individuo infectado.
Agradecimientos
Los autores agradecen a los árbitros anónimos por sus valiosos comentarios y sugerencias que contribuyeron para mejorar el manuscrito. En particular, a Angel C. Ruiz Ortiz por su ayuda en la traducción del resumen al portugués.
Referencias
1. H. Dahari, R. M. Ribeiro, Ch. M. Rice, and A. S. Perelson. Mathematical Modeling of Subgenomic Hepatitis C Virus Replication in Huh-7 Cells. Journal of Virology, ISSN 0022–538X, 81(2), 750–760 (2007). Referenciado en 11, 12 [ Links ]
2. E. F. White, R. S. Garfein, K. C. Brouwer, R. Lozada, R. Ramos, M. Firestone– Cruz, S. G. Pérez, C. Magis–Rodríguez, C. J. Conde–Glez, S. A. Strathdee. Prevalence of hepatitis C virus and HIV infection among injection drug users in two Mexican cities bordering the U. S. Salud Pública de México, ISSN 0036–3634, 49(3), 165–172 (2007). Referenciado en 11 [ Links ]
3. G. Benítez–Arvizu, R. Cortez–Gómez, B. A. Novelo–Garza, A. Malagón– Martínez, A. Guerra–Márquez, M. del C. Alvarado–Maldonado, M. Rodríguez– Bartolo, R. M. Argüelles–Pimentel, R. G. Sánchez–Barrera. Prevalencia del virus de hepatitis C en el Banco de Sangre del Centro Médico Nacional La Raza. Revista Médica del Instituto Mexicano del Seguro Social, ISSN 0484–7849, 44(3), 227–233 (2006). Referenciado en 11 [ Links ]
4. L. Vera de León, J. A. Juárez Navarro, M. Díaz Gómez, J. Méndez Navarro, R. A. Chirino Sprung, M. Dehesa Violante, L. Casillas Dávila, M. T. Rizo Robles, R. Torres Ivarra, C. Cano Domínguez, J. Nava Estrada, M. Ramos Gómez, M. E. Hernández, S. Wong González, M. Félix Tamayo, M. C. Corona Lechuga, A. R. Zárate Negrete, M. Rangel Jiménez, H. Rodríguez Hernández, V. González Ortiz, M. P. Tirado Estrella, M. A. Villanueva Carreto, R. Orta Flores, J. L. Manteca Argumedo, I. Vázquez ávila, J. González Macías, O. Razcón Hernández, P. Torres López, C. Bazán Pérez. Epidemiologic and situational Panorama of hepatitis C in Mexico. Revista de Gastroenterología de México, ISSN 0375–0906, 70(1), 25–32 (2005). Referenciado en 11 [ Links ]
5. T. Parnell, H. Margolis, L. Moyer, R. Conlon, Ch. Kukka, L. Brady, P. Rosenthal. Informe de PKIDs sobre Hepatitis Pediátrica, ISBN 1–929524–03–X. PKIDs, 2003. Referenciado en 11 [ Links ]
6. B. A. Cipra. Will Viruses Succumb To Mathematical Models?. SIAM News, ISSN 1557–9573, 32(2), 1–4 (1999). Referenciado en 11 [ Links ]
7. N. C. Tassopoulos. Patterns of progression: unpredictability and risk of decompen sated cirrhosis. Digestive Diseases and Sciences, ISSN 0163–2116, 41(12), 41S– 48S (1996). Referenciado en 11 [ Links ]
8. A. Mangia, R. Santoro, N. Minerva, G. L. Ricci, V. Carretta, M. Persico, F. Vinelli, G. Scotto, D. Bacca, M. Annese, M. Romano, F. Zechini, F. Sogari, F. Spirito, and A. Andriulli. Peginterferon alfa–2b and Ribavirin for 12 vs. 24 Weeks in HCV Genotype 2 or 3. The New England Journal of Medicine, ISSN 0028– 4793, 352(25), 2609–2617 (2005). Referenciado en 11 [ Links ]
9. S. D. Sullivan, D. M. Jensen, D. E. Bernstein, T. I. Hassanein, G. R. Foster, S. S. Lee, H. Cheinquer, A. Craxi, G. Cooksley, W. Klaskala, K. Pettit, K. K. Patel, J. Green. Cost-effectiveness of combination peginteferon alpha-2a and ribavirincompared with interferon alpha-2b and ribavirin in patients with chronic hepatitis C. The American Journal of Gastroenterology, ISSN 0002–9270, 99(8), 1490– 1496 (2004). Referenciado en 11 [ Links ]
10. C. Sarrazin, D. A. Hendricks, F. Sedarati, and S. Zeuzem. Assessment, by Transcription-Mediated Amplification, of Virologic Response in Patients with Chronic Hepatitis C Virus Treated with Peginterferon α − 2a. Journal of Clinical Microbiology, ISSN 0095–1137, 39(8), 2850–2855 (2001). Referenciado en 11 [ Links ]
11. L. S. Friedman. Controversies in liver biopsy: who, where, when, how, why?. Current Gastroenterology Reports, ISSN 1522–8037, 6(1), 30–36 (2004). Referenciado en 11 [ Links ]
12. R. Olsson, I. Hägerstrand, U. Broomé, Å. Danielsson, G. Järnerot, L. Lööf, H. Prytz, B-O Rydén, S. Wallerstedt. Sampling variability of percutaneous liver biopsy in primary sclerosing cholangitis. Journal of Clinical Pathology, ISSN 0021–9746, 48(10), 933–935 (1995). Referenciado en 11 [ Links ]
13. S. Sherlock. Needle biopsy of the liver: A review. Journal of Clinical Pathology, ISSN 0021–9746, 15(4), 291–304 (1962). Referenciado en 11 [ Links ]
14. M. Friedrich-Rust and S. Zeuzem. Transient Elastography (FibroScan) for the Non-Invasive Assessment of Liver Fibrosis: Current Status and Perspectives. Zeitschrift für Gastroenterologie, ISSN 0044–2771, 45(5), 387–394 (2007). Referenciado en 11 [ Links ]
15. A. S. Mahal, C. M. Knauer and P. B. Gregory. Bleeding After Liver Biopsy. Western Journal of Medicine, ISSN 0093–0415, 134(1), 11–14 (1981). Referenciado en 11 [ Links ]
16. M. E. Garassini. Tratamiento actual de la hepatitis C. Gastroenterología Latinoamericana, ISSN 0716–8594, 17(2), 253–255 (2006). Referenciado en 12 [ Links ]
17. T. C. Reluga, H. Dahari, and A. S. Perelson. Analysis of Hepatitis C virus infection models with hepatocyte homeostasis. SIAM Journal on Applied Mathematics, ISSN 0036–1399, 69(4), 999–1023 (2009). Referenciado en 12, 14, 17 [ Links ]
18. J. Alavez–Ramírez. Estimación de parámetros en ecuaciones diferenciales ordinarias: identificabilidad y aplicaciones a medicina. Foro RED-Mat, ISSN 14051745, 21(1), 2007. Referenciado en 12, 15, 17 [ Links ]
19. R. Thièbaut, J. Guedj, H. Jacqmin-Gadda, G. Chêne, P. Trimoulet, D. Neau, and D. Commenges. Estimation of dynamical model parameters taking into accountundetectable marker values. BMC Medical Research Methodology, ISSN 1471– 2288, 6(38), 2006. Referenciado en 12, 14, 17 [ Links ]
20. J. Alavez-Ramírez, Je. López-Estrada y G. Reyes-Terán. Dinámica del virus de la hepatitis C con carga viral y ALT y monitoreo del daño hepático libre de biopsias. Revista Ingeniería y Ciencia, ISSN 1794–9165, 2(4), 125–144 (2006). Referenciado en 12, 13, 14, 17, 18, 25, 26, 28 [ Links ]
21. J. Alavez-Ramírez, J. López-Estrada y G. Gómez-Alcaraz. Monitoreo del daño hepático sin biopsias vía modelación matemática. Revista de Ciencias Básicas UJAT, ISSN en trámite, 5(1), 37–56 (2006). Referenciado en 12, 14 [ Links ]
22. T. J. Layden, J. E. Layden, R. M. Ribeiro and A. S. Perelson. Mathematical modeling of viral kinetics: a tool to understand and optimize therapy. Clinics in Liver Disease, ISSN 1089–3261, 7, 163–178 (2003). Referenciado en 12 [ Links ]
23. R. M. Ribeiro, J. Layden-Almer, K. A. Powers, T. J. Layden and A. S. Perelson. Dynamics of alanine aminotransferase during hepatitis C virus treatment. Hepatology, ISSN 0270–9139, 38(2), 509–517 (2003). Referenciado en 12, 14, 25 [ Links ]
24. A. S. Perelson. Modelling viral and immune system dynamics. Nature Reviews Immunology, ISSN 1474–1733, 2(1), 28–36 (2002). Referenciado en 12, 14 [ Links ]
25. S. Zeuzem and E. Herrmann. Dynamics of hepatitis C virus infection. Annals of Hepatology, ISSN 1665–2681, 1(2), 56–63 (2002). Referenciado en 12 [ Links ]
26. R. Avendaño, L. Esteva, J. A. Flores, J. L. Fuentes Allen, G. Gómez and Je. López-Estrada. A mathematical model for the dynamics of hepatitis C. Computational and Mathematical Methods in Medicine, ISSN 1748-670X, 4(2), 109–118 (2002). Referenciado en 12, 13, 14, 18, 22 [ Links ]
27. A. U. Neumann, N. P. Lam, H. Dahari, D. R. Gretch, Th. E. Wiley, Th. J. Layden, A. S. Perelson. Hepatitis C Viral Dynamics in Vivo and the Antiviral Eficacy of Interferon-α Therapy. Science, ISSN 0036–8075, 282(5386), 103–107 (1998). Referenciado en 12, 14, 17 [ Links ]
28. D. Wodarz. Hepatitis C virus dynamics and pathology: the role of CTL and antibody responses. Journal of General Virology, ISSN 0022–1317, 84, 1743–1750 (2003). Referenciado en 14 [ Links ]
29. L. Edsberg and P.–Å. Wedin. Numerical tools for parameter estimation in ODE-systems. Optimization Methods and Software, ISSN 1055–6788, 6(3), 193–217 (1995). Referenciado en 17 [ Links ]
30. Z. Li, M. R. Osborne and T. Prvan. Parameter estimation of ordinary differential equations. IMA Journal of Numerical Analysis, ISSN 0272–4979, 25(2), 264–285 (2005). Referenciado en 17 [ Links ]
31. N. J-B. Brunel. Parameter estimation of ODE's via nonparametric estimators. Electronic Journal of Statistics, ISSN 1935–7524, 2, 1242–1267 (2008). Referenciado en 17 [ Links ]
32. J. O. Ramsay, G. Hooker, D. Campbell, and J. Cao. Parameter estimation for differential equations: a generalized smoothing approach. Journal of the Royal Statistical Society: Series B, ISSN 1467–9868, 69(5), 741–796 (2007). Referenciado en 17 [ Links ]