Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
CES Medicina Veterinaria y Zootecnia
versão On-line ISSN 1900-9607
Ces. Med. Vet. Zootec. vol.6 no.1 Medellín jan./jun. 2011
* Para citar este artículo: Castañeda RD, Peñuela LM . 2011. Técnicas de quantificação da síntese microbiana no rúmen: urna revisão. Rev CES Med Vet Zootec. Vol 6 (1): 46-53.
** Autor para correspondência: Román David Castañeda. Universidade Estadual de Maringá, Av. Colombo 5790, Maringá - Paraná - Brasil. Correo electrónico: romancaser@gmail.com
(Recibido: 7 de julio, 2010; aceptado: 5 de mayo, 2011)
Resumo
Quantificar a síntese de proteína microbiana em animais ruminantes é de grande importância para avaliar a qualidade nutritiva das dietas. A determinação dessa síntese se obtém por meio da utilização de marcadores microbianos, os quais são classificados em internos (subprodutos dos microrganismos ruminais) e externos (produtos administrados aos animais que são incorporados pelos microrganismos) e técnicas indiretas. Embora tenham ocorrido avanços ainda não se tem o marcador ideal. O objetivo desta revisão é detalhar a cada uma das vantagens e desvantagens das técnicas para estimar a síntese de proteína microbiana no rúmen e discutir os resultados obtidos utilizando cada técnica.
Palavras-chave: Ácidos nucléicos, marcadores externos, marcadores internos, purinas.
Abstract
The quantification of the microbial protein synthesis in the ruminants is of great importance to assess the nutritional quality of the diets. The determination of microbial synthesis is achieved through the use of microbial markers, which are classified: internal (by-products of rumen microorganisms) and external (substances administered to animals that are incorporated by rumen microorganisms). Although some advances has occurred lately an ideal marker still need to be developed. So the objective of this review is to detail each of the techniques for estimating microbial protein synthesis in the rumen, and discuss the results obtained using each technique.
Key words: External markers, internal markers, nucleic acids, purines.
Resumen
La cuantificación de la síntesis de proteína microbiana en los rumiantes es de gran importancia para evaluar la calidad nutricional de las dietas. La determinación de la síntesis microbiana se logra a través del uso de marcadores microbianos que se clasifican como: Internos (subproductos de los organismos de la panza) y externos (sustancias administradas a los animales que son incorporadas por los organismos de la panza). Aunque últimamente ha habido algunos avances, aún es necesario desarrollar un marcador ideal. Así que el objetivo de esta revisión es detallar cada una de las técnicas usadas para estimar la síntesis de proteína microbiana en la panza y discutir los resultados obtenidos al usar cada técnica.
Palabras Clave: Marcadores externos, marcadores internos, ácidos nucleicos, purinas.
Introdução
O aumento na produção de ruminantes pode ser obtido através da maximização da eficiência microbiana ruminal. Trabalhos de pesquisa indica que a proteína microbiana ruminal (PMR) responde, em média, por 59% da proteína que chega ao intestino delgado8. Essa proteína pode ser estimada por meio do conhecimento da síntese microbiana29.
A importância da proteína microbiana na nutrição de ruminantes está relacionada ao fato desta ser uma fonte de alta qualidade de aminoácidos (AAs) disponíveis para a absorção, possuir uma digestibilidade aparente intestinal de aproximadamente 85% e um perfil de AAs essenciais semelhantes àqueles do leite e dos tecidos.A determinação ou a estimativa do suprimento de proteína microbiana é uma importante área de estudo na nutrição protéica dos ruminantes. A quantificação da proteína microbiana no rúmen como resultado da fermentação microbiana é de interesse porque há evidencias que esta pode ser influenciada pela dieta9.Os estudos para determinar a contribuição das proteínas microbianas como fonte de proteína para o hospedeiro utiliza marcadores microbianos, que podem ser internos ou externos. O objetivo desta revisão é detalhar e descrever cada técnica, analisar as vantagens e desvantagens de cada uma para uatificar a síntese de proteína microbianano rúmen e discutir os resultados obtidos utilizando cada técnica.
Microrganismos ruminais
Os ruminantes não podem sintetizar celulose, mas podem digerí-la da parede celular de plantas não lignificadas, por meio da fermentação ruminal. Além disso, têm desenvolvido uma endosimbiose com os microrganismos, o que lhes permite viver de urna dieta cuja principal fonte de carboidratos é a celulose25.
O ecossistema ruminal não pode ser definido em termos de energia metabolizável ou energia bruta, somente pode ser descrito pela quantidade de bactérias próprias e pelos produtos finais da digestáo21; assim, a taxa de degradação da proteína no rúmen está influenciada pela ação proteolítica dos microrganismos ruminais e pelo fluxo do conteúdo ruminal3.
As populações de microrganismos variam dentro do mesmo animal segundo o tempo percorrido depois da última ingestão do alimento, nos dias após, e entre animais com igual alimentação em diferentes países20. As bactérias criam urna associação juntando-se com microrganismos afins, onde um microrganismo usa os produtos finais do outro para seu crescimento.
Existem marcadas interações entre os protozoários e as bactérias, assim muitos protozoários ingerem e digerem as bactérias produzindo urna diminuição da biomassa bacteriana, podendo reduzir a taxa de colonização das partículas do alimento pelas bactérias. Por outro lado, os protozoários competem eficientemente pelos açucares solúveis e pelo amido com as bactérias, armazenando carboidratos dentro de suas células 34. Para calcular a contribuição da proteína verdadeira microbiana digestível (PVMD) no intestino delgado, há necessidade do conhecimento da produção de proteína microbiana.
Marcadores microbianos
Os marcadores microbianos podem-se classificar em dois grandes grupos: marcadores internos e externos. Os marcadores internos são constituintes das células microbianas e por isso não precisam ser administrados aos animais experimentais. Por outro lado, os marcadores externos devem ser administrados aos animais experimentais para que sejam incorporados pelos microorganismos ruminais.
Marcadores microbianos internos
Os principais marcadores internos são: Derivados de Purinas, ácidos nucléicos (ácido ribonucleico (ARN) e ácido desoxirribonucléico (ADN)), ácidodiaminopimélico (DAPA), ácido aminoetilfos-fónico (AEPA), perfis de aminoácidos e o ATP Nesta revisão faremos ênfase nas primeiras duas técnicas, por serem as mais utilizadas e aceitas na atualidade.
Derivados de purinas. A obtenção de dados da produção de proteína microbiana no rúmen tem sido difíceis, principalmente pelo fato que os métodos estabelecidos são trabalhosos, demorados e requererem animais implantados cirurgicamente com çãnulas28. As pesquisas, ao longo dos últimos anos, confirmaram a relação entre produção de proteína microbiana e excreção urinária de derivados de purinas, porem assumiu-se que a absorção de purinas estaria condicionada á quantidade de proteína microbiana, estimada a partir da excreção urinária dos derivados de purinas (alantoína, ácido úrico, xantina e hipoxantina)12.
Por outro lado foi observada correlação positiva entre o fluxo de N microbiano e a excreção urinária de derivados de purinas, comparando os métodos dos ácidos nucléicos microbianos e a excreção de derivados de purinas na urina, observando urna alta correlação22. Também foi relatado que a excreção urinária de derivados de purinas apresentou correlação positiva na estimativa do fluxo de N microbiano no duodeno14,28.
A excreção de derivados de purinas está diretamente relacionada com a absorção de purinas e, com o conhecimento da relação N purina: N total, na massa microbiana, a absorção de N microbiano pode ser calculada a partir da quantidade de purina absorvida, que é estimada por intermédio da excreção urinária de derivados de purinas6.
A excreção urinária de derivados de purinas, pelos ruminantes, pode ser usada para estimar o fluxo intestinal de proteína microbiana, urna vez que a excreção endógena de derivados de purinas e a relação quantitativa entre a excreção de derivados de purinas e a absorção de purina tenham sido previamente determinadas7. Segundo os mesmos autores, há diferenças no metabolismo das purinas, sendo já estabelecido que ovinos e Bos taurus diferem quanto ao nível de excreção endógena de purinas e á habilidade de utilização de purinas de origem exógena.
As purinas são prontamente absorvidas como nucleosídeos e bases livres no lúmen do intestino delgado e estão sujeitas à degradação extensiva por enzimas específicas, como guanina deaminase, adenosina deaminase e xantina oxidase, em sua passagem pela da mucosa intestinal. É importante salientar que a extensão desta degradação determina a disponibilidade metabólica das purinas a serem utilizadas pelos animáis24.
Tem-se, por intermédio da ação da enzima-chave na degradação das purinas, a xantina oxidase, a conversão de hipoxantina em xantina e ácido úrico; este, então, é degradado em alantoína, pela ação da urease15.
Na urina de bovinos, ambas as purinas endógenas e exógenas têm composição semelhante de, aproximadamente, 85% de alantoína e 15% de ácido úrico; xantina e hipoxantina não estão presentes em quantidades significativas na urina de bovinos7,32.
A excreção urinária de derivados de purinas pode ser considerada método simples e não-invasivo para estimar a produção microbiana, entretanto, de acordo com Pérez et al. (1996)19, seu uso necessita de validação pela comparação de estimativas com outras técnicas.
A técnica de determinação de derivados de purinas (DP) para estimar a síntese microbiana assume que todos os ácidos nucléicos de origem dietética são degradados no rúmen e que, portanto, todos os ácidos nucléicos que deixam o rúmen são essencialmente de origem microbiana. A Figura 1 abaixo mostra de modo esquemático o principio deste método. As purinas dos ácidos nucléicos microbianos então são absorvidas, degradadas e excretadas na urina como seus derivados (produtos de degradação), hipoxantina, xantina, acido úrico e alantoína, conforme pode ser observado na Figura 2. A excreção de DP é diretamente proporcional a absorção de purinas6.
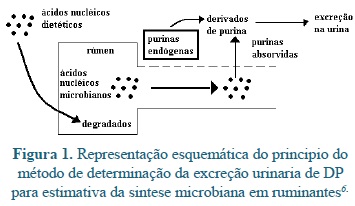
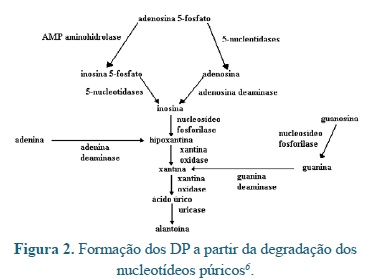
Os DP urinários compreendem, portante, hipoxantina, xantina, acido úrico e alantoina (Figura 2). Estes quatro estão presentes na urina de ovinos, caprinos, cervídeos e lhamas, mas apenas acido úrico e alantoina são excretados por bovinos e bubalinos. Isto porque bovinos e bubalinos tem urna alta atividade de xantina oxidase na mucosa intestinal, degradando, portanto, as bases púricas a seus derivados mais distantes: acido úrico e alantoina. Nos ovinos, a concentração de xantina oxidase no tecido intestinal é mínima6.
Diferenças especificas no metabolismo das purinas tem sido observadas (26). É importante notar que diferentes equações são necessárias para cada espécie animal. As equações desenvolvidas e validadas ate o momento são para ovinos e bovinos 32. Cabe salientar que foram desenvolvidas para animais europeus. Os resultados recentes demonstram que a excreção de DP por animais zebuínos é relativamente menor 26.
As principais limitações desta técnica residem no fato de estar baseada em duas suposições. A primeira é que todo acido nucléico que chega ao intestino delgado é de origem microbiana. Assumir tal fato não compromete a maioria dos estudos, pois realmente os ácidos nucléicos dietéticos são rapidamente e extensivamente degradados no rúmen. Porem há exceções que não podem ser desprezadas. Para certos alimentos de origem animal, principalmente farinha de peixe, tal fato não se aplica. O segundo ponto de limitação refere-se à relação entre purinas e proteína nos microrganismos ruminais ser constante. Ate o momento, não há informação suficiente para tal afirmação.
Para as coletas de urina, com duração de 24 horas, podem-se utilizar funis coletores, adaptados aos animais, conforme metodologia descrita por Valadares et al. 30. Da urina total coletada em recipiente contendo 200 mL de H2S04, a 10%, no período de 24 e 12 horas, após pesagem e homogeneização, se obtém alíquotas de 100 mL. Estas amostras devem ter o pH ajustado abaixo de 3 e foram diluídas 3 a 4 vezes, para evitar destruição bacteriana dos derivados de purinas urinários e precipitação do ácido úrico.
As análises de derivados de purinas (alantoína, ácido úrico, xantina e hipoxantina) podem ser feitas pelo método colorimétrico, conforme técnica 11, descrita por Chen y Gomes (1992)6. A determinação de alantoína foi descrita por Young e Conway (1942)35, e baseia-se na hidrólise alcalina a 100 °C da alantoína a ácido alantóico, que, posteriormente, é convertido em uréia e ácido glioxílico em solução ácida. O ácido glioxílico reage, entáo, com hidrocloreto de fenilhidrazina, para produzir fenilhidrazona do ácido. O produto forma um cromógeno instável, com ferricianeto de potássio, que pode ser dosado, colorimetricamente, a 522 nm.
O método descrito por Fujijara et al. (1987)11, para determinação de ácido úrico, baseia-se no tratamento da amostra de urina com, urease resultando em alantoína, que não absorve ultra-violeta (UV) a 293 nm. Então, a redução na leitura de absorbância a 293 nm foi correlacionada com a concentração de ácido úrico. Para determinação de xantina e hipoxantina, pode-se converter em ácido úrico pela xantina oxidase e medidas como ácido úrico a 293 nm6.
A técnica de determinação da produção de proteína microbiana pelas bases purinas, usando coletas de digestas de abomaso, pode ser adotada em experimentos simultâneos de digestibilidade. Aproximadamente, dois litros de digesta de rúmen de cada animal devem ser coletados via fístula ruminal, e as bactérias isoladas5. Ao final dos experimentos, devem ser feitas amostras compostas para cada ração e, para medir a produção de biomassa microbiana, deve-se utilizar as bases purinas, determinadas27.
O método da excreção urinária de derivados de purinas pode ser utilizado para estimar a produção de proteína microbiana com segurança. A produção de proteína microbiana pode ser estimada em função do consumo de matéria seca, consumo de NDT e da porcentagem de fbra em detergente neutro da ração23.
Ácidos nucléicos (ADN e ARN). A utilização do ADN e especialmente do ARN são baseadas nas altas concentrações destas substâncias que apresentam os microrganismos ruminais4. Sua utilização como marcadores microbianos remonta-se aos anos sessenta, quando diversos autores observaram que a síntese de proteína microbiana no rúmen estava relacionada com o fluxo total de polinucleótidos10. Posteriormente tem-se utilizado como marcadores microbianos o total de ácidos nucléicos27,36 o ADN1 e o ARN16,18.
Os dados sobre o conteúdo em ARN das bactérias mostram variação bastante acentuada, de 3,5 e 5,0 g/100 g MS para bactérias aderentes e livres, respectivamente18 até 12 g/100 g MS 13. Esta variação em parte é atribuída aos diferentes métodos de análise utilizados, mas, por outro lado, reflete também o nível de atividades metabólicas das bactérias2.
Para a estimativa da eficiência de síntese de compostos nitrogenados microbianos, pode-se utilizar as bases purinas como indicador microbiano, determinando-se a concentração do N-purínico ou ARN equivalente em isolado de células bacterianas ruminais de amostras de digesta de rúmen e abomaso27. Segundo estes últimos autores, o procedimento combina métodos por hidrólise de nucleotídeos com acido perclórico seguido por precipitação de purinas livres com nitrato de prata, para a separação das purinas de outros compostos. O procedimento é rápido, simples, preciso e económico.
A passagem duodenal de N microbiano é estimada por este procedimento seguindo alimentação com dietas purificadas e semipurificadas. A degradação rápida de ácidos nucléicos dietéticos no rumen17 conduziu ao uso amplo de ácidos nucléicos ou de suas bases constituintes, as purinas ou as pirimidinas como marcadores para a determinação da síntese microbial da proteína no rúmen. A quantidade de compostos nitrogenados (N) no abomaso pode ser calculada dividindo-se o fluxo abomasal de N-ARN pela relação N-ARN/N-total das bactérias isoladas no rúmen. Determinando o fluxo de MS bacteriana no abomaso pela relação entre o N microbiano no abomaso e a porcentagem de N na MS microbiana.
Marcadores microbianos externos
Os marcadores microbianos externos precisam ser administrados aos animais, para que estes sejam incorporados pelos microrganismos ruminais. O procedimento consiste em colocar urna infusão continua do marcador no rúmen dos animais, durante urna período experimental ate alcançar condições de equilíbrio. Os principais marcadores externos são 15N, 32P e 35S.
32P
A utilização de radiofósforo (32P) para a determinação da síntese microbiana tem mostrado ser bastante eficiente. Por meio da coleta de amostra do liquido do rúmen pode-se medir a taxa de incorporação do radiofósforo in vitro, avaliando dessa maneira a atividade microbiana.
O método baseia-se na relação entre a incorporação do fósforo na matéria microbiana e a síntese de proteína, utilização de amónia e produção de ácidos graxos láteis, em curtos períodos de incubações usando 32P como marcador31,33.
A técnica in vitro de incorporação de 32P para estimar síntese microbiana baseia-se na incubação de urna pequena quantidade de amostra com liquido ruminal em meio tamponado e urna diminuta quantidade de radiofósforo como marcador de proteína microbiana. Com base na atividade especifica do fósforo solúveis (AE) e na incorporação do radiomarcador pelos microrganismos, é possível calcular a quantidade total de massa microbiana gerada.
15N
A síntese microbiana é determinada in vivo, com a utilização do isótopo pesado do nitrogénio (15N). Urna fonte enriquecida em 15N (sulfato de amônio marcado, por exemplo) é fornecida aos animais, ou, ainda, colocada diretamente no rúmen. Esta fonte é metabolizada pelos microrganismos do rúmen e proteínas microbianas são sintetizadas com o nitrogênio marcado. Nas amostras da digesta coletadas no duodeno, determina-se então, a quantidade de proteína microbiana que passa para o restante do sistema digestorio77. O 15N inorgânico tem sido usado amplamente como um indicador para quantificar a produção de proteína bruta microbiana em vivo, o mesmo autor relata as diversas vantagens que resultam em utilizar o 15N como um marcador externo. 1) porque o 75N é um isótopo estável e não é um perigo ambiental. 2) geralmente, 15N não é encontrado na dieta acima do enriquecimento natural; de ai que, dosagem intraruminal com sais do amônio 15N em estudos a curto prazo marcaram somente N.3) 15N inorgânico é relativamente barato4.
35S
O isótopo 35S inorgánico pode infundido no rúmen e ser incorporado na proteína bruta microbiana. Os métodos típicos envolvem dosagem de Na235S04 no rúmen, onde é reduzido a sulfito 35S e logo é incorporado na proteína bacteriana através da síntese de novo de cistina e de metionina. O 35S será incorporado também em outros compostos de enxofre, tais como a co-enzima A. Indiretamente, o 35S marcara proteínas de protozoários. Urna amostra "típica" de micróbios ruminais deve ser isolada para fornecer a relação padrão do indicador. Todavia, a radioatividade acumulada nos tecidos e no leite impossibilita seu uso no alimento. Assim, o uso rotineiro de 35S como um indicador microbiano em vacas leiteiras seria inconveniente por causa dos problemas da eliminação4.
Conclusões
A utilização de metodologias para conhecer a síntese microbiana no rúmen, tem trazido informações importantes para o entendimento e analises de novos alimentos, subprodutos e aditivos na nutrição de ruminantes. Os marcadores microbianos mais utilizados são os derivados de purinas, os ácidos nucléicos (ARN) e o 15N, os três têm sido propostos como marcadores de eleição por vários autores. Todavia, são escassas as pesquisas comparando estes marcadores, para avaliar a correlação entre eles. Na atualidade a técnica das purinas é mais utilizada devido as vantagens que esta fornece.0 75N apresenta principalmente problemas econômicos, pelo alto preço do isótopo e da posterior analise que precisa o método. A recomendação na hora de determinar a síntese microbiana é utilizar mais de um marcador.
Referências
1. Arambel MJ, Bartley EE, Dufva GS, Nagaraja TG, Dayton AD. 1982. Effect of diet on amino and nucleic acids of rumen bacteria and protozoa. J Dairy Sci; 65:2095-210. [ Links ]
2. Bates DB, Gillett JA, Barao SA. 1985.The effect of specific growth rate and stage of growth on nucleic acid-protein values of pure cultures and mixed ruminal bacteria. J Anim Sci; 61 (3):713-724. [ Links ]
3. Boenker D. 1987. Importancia de la fermentación ruminal y su influencia sobre el comportamiento reproductivo del rumiante. Carta Ganadera; 24 (2):33. [ Links ]
4. Broderick GA, Merchen NR. 1992. Markers for quantifying microbial protein synthesis in the rumen. J Dairy Sci; 75 (9):2618-2632. [ Links ]
5. Cecava MJ, Merchen NR, Gay LC. et al. 1990. Composition of ruminal bacteria harvested from steers as influenced by dietary energy level, feeding frequency and isolation techniques. J Dairy Sci; 73 (9): 2480-2488. [ Links ]
6. Chen XB, Gomes MJ. 1992. Estimation of microbial protein supply to sheep and cattle based on urinary excretion of purine derivatives- an overview of technical details. INTERNATIONAL FEED RESEARCH UNIT. Aberdeen, UK: Rowett Research Institute, 21p. (Occasional publication). [ Links ]
7. Chen XB, Samaraweera L, Kyle, DL. et al. 1996. Urinary excretion of purine derivatives and tissue xantine oxidase (EC 1, 2, 3, 2) activity in buffaloes (Bubalis bubalis) with special reference to differences between buffaloes and Bos taurus cattle. Br. J Nut; 75:317-407. [ Links ]
8. Clark JH, Klusmeyer TH, Cameron MA. 1992. Microbial protein sinthesys and flows of nitrogen fractions to the duodenum of dairy cows. J Dairy Sci., 75 (8):2304-2323. [ Links ]
9. Dove H, Milne JA. 1994. Digesta flow and rumen microbial protein production in ewes grazing perennial ryegrass. Aus. J Agri Res; 45 (6):1229-1224. [ Links ]
10. Ellis WC, Pfander WH. 1965. Rumen microbial polynucleotide synthesis and its possible role in ruminant nitrogen synthesis. Nature (Lond.); 205: 974-975. [ Links ]
11. Fujihara T, Orskov ER, Reeds PJ. et al. 1987. The effect of protein infusión on urinary excretion of purine derivatives in ruminants nourished by intragastric nutrition. J Agrie Sci; 109:7-12. [ Links ]
12. Giesecke D, Ehrentreich L, Stangassinger M. 1994. Mammary and renal excretion of purine metabolites in relation to energy intake and milk yield in dairy cows. J Dairy Sci; 77 (8):2376-2381. [ Links ]
13. John, A. 1984. Effects of feeding frequency and level of feed intake on chemical composition of rumen bacteria. J Agrie Sci; 102 (1):45-57. [ Links ]
14. Johnson, L.M, Harrison, J.H., Riley, RE. 1998. Estimation of the flow of microbial nitrogen to the duodenum using urinary uric acid or allantoin. J Dairy Sci; 81 (9):2408-2420. [ Links ]
15. Leningher, A.L., Nelson, D.L., Cox, M.M. 1995. Princípios de bioquímica. 2.ed., São Paulo: Savier. 839p. [ Links ]
16. Ling J.R., Buttery P.J., 1978. The simultaneous use of ribonucleic acid, 35S, 2,6-diaminopimelic acid and 2-aminoethylphosphonic acid as markers of microbial nitrogen entering the duodenum of sheep. Br. J Nutr; 39:165-179. [ Links ]
17. McAllan AB, Sutton JP, Beever DE. 1994. Rumen fermentation characteristics and duodenal nutrient flow in lactantin cows receiving two types of grass silage with two levels of concentration. Ani Feed Sci and Tech; 46:277-291. [ Links ]
18. Merry RJ, McAllan AB. 1983. Comparison of the chemical composition of mixed bacteria harvested from the liquid and solid fractions of rumen digest. Br. J Nutr; 50 (3):701-709. [ Links ]
19. Pérez JF, Balcells J, Guada JA. et al. 1996. Determination of rumen microbial-nitrogen production in sheep: a comparison of urinary purine excretion with methods using 15N and purine bases as markers of microbial-nitrogen entering the duodenal. Br. J Nutr; 75:699-709. [ Links ]
20. Posgate JR, Ware DA. 1965. Manipulating rumen fermentation. J Anim Sci; 46 (3):585-599. [ Links ]
21. Preston TR. 1986. Propuestas de metodología de producción adecuadas a países tropicales. In: Seminario Universidad Nacional de Colombia, 1, Palmira, 1986. Resumos. Universidad Nacional de Colombia. Sede Palmira: Facultad de Ciencias Agropecuarias; 56-70. [ Links ]
22.Puchala R, Kulasek GW. 1992. Estimation of microbial protein flow firom the rumen of sheep using microbial nucleic acid and excretion of purine derivatives. Can. J Anim Sci; 72:821-830. [ Links ]
23. Rennó LN, Valadares RFD, Leáo MI. et al. 2000. Estimativa da produção de proteína microbiana em novilhos. Rev Bras Zoot; 29 (4):1223-1234. [ Links ]
24. Stangassinger M, Chen XB, Lindberg JE. et al. 1995. Metabolism of purines in relation to microbial prodution. In: Engelhardt, W.V., Leonhard-Marek S, Breves G, Giesecke D. (Eds.) Ruminant physiology: digestion, metabolism, growth and reprodution. Proceedings of the Eighth International Symposium on Ruminant Physilogy. Stuttgart: Ferdinand Enke Verlag: 387-406. [ Links ]
25. Stanier R. 1984. El mundo de la microbiología. Asociaciones simbióticas entre los miembros no fotosintéticos. 1 ed: 752. [ Links ]
26. Tamminga S, Chen XB. 2000. Animal-based techniques for the estimation of protein value. In: Givens DI, Owen E, Axford RFE, Omed HM (Ed). Forage evaluation in ruminant nutrition. Wallingford: CAB Publinshing. [ Links ]
27.Ushida K, Lassalas B, Jouank JP 1985. Determination of assay parameters for RNA analysis in bacterial and duodenal samples by spectrophotometry Influence of sample treatment and preservation. Reprod Nutr Dev; 25:1037-1044. [ Links ]
28. Vagnoni DB, Broderick MK, Clayton RD. et al. 1997. Excretion of purine derivatives by Holstein cows abomasally infused with incremental amounts of purines. J Dairy Sci; 80: 1695-1702. [ Links ]
29. Valadares Filho, S.C. 1995. Eficiência de síntese de proteína microbiana, degradação ruminal e digestibilidade intestinal de proteína bruta em bovinos. In: Simpósio Internacional Sobre Exigências Nutricionais em Ruminantes, Viçosa, MG. Anais... Viçosa: Jard, 355-388. [ Links ]
30. Valadares RFD, Gonçalves LC, Sampaio IB. et al. 1997. Níveis de proteína em dietas de bovinos. 3-pH, amônia e eficiência microbiana. Rev Bras Zootec; 26 (6):1264-1269. [ Links ]
31. Van Nevel C, Demeyer DI. 1977. Determination of rumen microbial growth in vitro form 32-P labeled phosphate incorporation. Br. J Nutr; 38:101-114. [ Links ]
32. Verbic J, Chen XB, Macleod NA. et al. 1990. Excretion of purine derivatives by ruminants. Effect of microbial nucleic acid infusion on purine derivative excretion by steers. J Agri Sci; 114 (3):243-246. [ Links ]
33. VItti DM, Silva Filho JC, Abdalla AL 1988. Phosphorus availability for rumen microorganisms: effect of different sources. J Nucí Agri Bio; 7:186-187. [ Links ]
34. Wolin MJ. 1989. The rumen fermentation: a modelform microbial interaction in anaerobic ecosystem. 1 ed: Condrit. [ Links ]
35. Young EG, Conway CF. 1942. On the estimation of allantoin by the Rimini-Schryver reaction. J Biol Chem; 142:839-53. [ Links ]
36. Zinn RA, Owens FN. 1986. A rapid procedure for purine measurement and its use for estimating net ruminal protein synthesis. Can. J Anim Sci; 66 (1):157-166. [ Links ]













