Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
CES Medicina Veterinaria y Zootecnia
On-line version ISSN 1900-9607
Ces. Med. Vet. Zootec. vol.10 no.1 Medellín Jan./June 2015
Artículos originales de investigación
Genetic diversity of BoLA-DRB3 gene in Colombian creole Hartón del Valle cattle¤
Diversidad genética del gen BoLA-DRB3 en el ganado criollo colombiano Hartón del Valle
Diversidade genética do gene BoLA-DRB3 em gado crioulo colombiano Hartón del Valle
Darwin Yovanny Hernández Herrera1*, Zoot, MSc, PhD;Jaime Eduardo Muñoz Flórez2, IA, Esp, PhD; Luz ángela álvarez Franco2, Zoot, MSc, PhD.
¤Para citar este artículo: Hernández Herrera DY, Muñoz Flórez JE, álvarrez Franco LA. Diversidad genética del gen BoLA-DRB3 en el ganado criollo colombiano Hartón del Valle. Rev CES Med Zootec. 2015; Vol 10 (1): 18-30.
*Autor para correspondencia: Darwin Yovanny Hernández Herrera. Facultad de Ciencias Agropecuarias, Universidad de Sucre
1 Grupo de Investigación en Reproducción y Mejoramiento Genético, Facultad de Ciencias Agropecuarias, Universidad de Sucre, Sede Puerta Verde. Granja Perico Km 7 Margen izquierdo vía Sincelejo - Sampués. darwin.hernandez@unisucre.edu.co;
2Grupo de Investigación en Recursos Zoogenéticos, Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia, Sede Palmira. Carrera 32 No 12-00 Palmira, Colombia
(Recibido: 6 de enero, 2015; aceptado: 4 de abril, 2015)
Abstract
The objective of this study was to evaluate the genetic diversity of ball-DRB3 gene in the Colombian creole Hartón del Valle cattle. A total of 93 Hartón del Valle (HV), 30 Lucerne (LUC), and 30 Holstein (HOL) animals were evaluated for DRB3 gene polymorphism using the PCR-SBT method. The number of alleles found were 27, 17 and 14 for HV, LUC, HOL, respectively. The most frequent alleles were DRB3*1101 (14.5%) in HV; LUC DRB3 * 0701 in LUC; and *1107, *1501 and *2301 in HOL (11.7%). Expected heterozygosity values were higher than observed, which resulted in a low fixing index, positive F IS (significant only for HV), and no significant deviations from Hardy- Weinberg equilibrium for LUC. The molecular analysis of variance reflected little variation between breeds (3%) and a moderate genetic differentiation (F ST = 0.056 p<0.01). Regarding sequence, LUC was the most diverse breed, no neutral selection is occurring, and none of the breeds is in selection equilibrium. Comparison with reports on other breeds showed high genetic diversity in Colombian breeds. We conclude that HV has high genetic diversity in BoLA-DRB3 locus. The cause of this polymorphism may be due in part to the origin of HV. This high variation can be maintained by selection equilibrium.
Key words: Genetic marker, major histocompatibility complex, PCR-SBT.
Resumen
El objetivo de esta investigación fue evaluar la diversidad genética del gen BoLA-DRB3 en el ganado criollo Colombiano Hartón del Valle. En 93 animales Hartón del Valle (HV), 30 Lucerna (LUC) y 30 Holstein (HOL) se evaluó el polimorfismo del gen DRB3 usando la metodología PCR-SBT. Se encontraron 27 alelos en la raza HV, 17 en LUC y 14 en HOL. El alelo más frecuente en HV fue el DRB3*1101 (14,5%), en la raza LUC los alelos DRB3*0701, *1107, *1501 y *2301 al igual que el alelo DRB3*0701 en la raza HOL tuvieron la frecuencia más alta (11,7%). Los valores de heterocigocidad esperada fueron más altos que la observada, lo que se tradujo en un valor bajo del índice de fijación, valores de F IS positivos solo significativo en el HV y desviaciones del equilibrio de Hardy-Weinberg no significativos en LUC. El análisis de varianza molecular reflejó poca variación entre razas (3%) y una diferenciación genética moderada (F ST= 0,056 p<0,01). A nivel de secuencia se resalta que, la raza más diversa es LUC, que no se está presentando selección neutral y que ninguna de las razas se encuentra en equilibrio de selección balanceadora. La comparación con otras razas reportadas, mostró alta diversidad genética en las razas colombianas. Se concluye que la raza HV tiene alta diversidad genética en el locus BoLA-DRB3, que el origen del polimorfismo puede deberse en parte al origen de la raza y que esta alta variación puede ser mantenida mediante equilibrio de selección.
Palabras clave: Complejo mayor de histocompatibilidad, marcador genético, PCR-SBT..
Resumo
O objetivo desta pesquisa foi avaliar a diversidade genética do gene BoLA-DRB3 no gado crioulo colombiano Hartón del Valle. O estudo se fez com 93 animais Hartón del Valle (HV), 30 Lucerna (LUC) e 30 Holandês (HOL). Avaliou-se o polimorfismo do gene DRB3 utilizando a metodologia PCR-SBT. Encontraram-se 27 alelos na raça HV, 17 em LUC e 14 em HOL. O alelo com maior frequência em HV foi o DRB3*1101 (14,5%), na raça LUC os alelos DRB3*0701, *1107, *1501 e *2301 ao igual que o alelo DRB3*0701 na raça HOL tiveram a maior frequência (11,7%). Os valores de heterocigocidade esperada foram maiores que a observada, o que se traduz em um valor baixo do índice de fixação, valores de F IS positivos só foram significativos no HV e desvio do equilíbrio de Hardy- Weinberg foram não significativos em LUC. O Analice de variância molecular reflexou pouca variação entre raças (3%) e uma diferenciação genética moderada (F ST= 0,056 p<0,01). No nível de sequência destacasse que, a raça mais diversa é LUC, que não está apresentando seleção neutral e que nenhuma das raças encontra-se em equilíbrio de seleção balanceadora. Se comparar com outras raças reportadas, as raças colombianas exibiram uma alta diversidade genética. Conclui-se que a raça HV tem alta diversidade genética no locus BoLA DRB3, que a origem do polimorfismo pode se dever em parte àorigem da raça e que essa alta variação pode ser mantida mediante o equilíbrio de seleção
Palavras-chave: Complexo maior de histocompatibilidade, marcador genético, PCR-SBT.
Introducción
El Complejo Mayor de Histocompatibilidad (CMH) de los bovinos es conocido como Antígenos de los Leucocitos Bovinos (BoLA) y se localiza en el cromosoma23. El sistema BoLa es un conjunto de glicoproteínas que se ubican en la superficie celular y su función principal es la presentación de péptidos 46.
En general la estructura del BoLA está conservada en los mamíferos y se divide en tres regiones, llamadas clase I, clase II y clase III, cada una con función diferente 13. El producto de los genes clase I son dos cadenas polipeptídicas, una de ellas atraviesa la membrana celular y tiene un peso molecular aproximado de 45.000 daltons; la cadena más corta llamada microglobulina β2 tiene un peso aproximado de 12.000 daltons. Estas proteínas están implicadas en el reconocimiento de las células hospederas que han sido infectadas, mediante la presentación de péptidos a las células citotóxicas T CD8+46. Los genes clase II están localizados en dos sitios diferentes del cromosoma23, poseen dos regiones llamadas clase IIa y clase IIb separadas por una distancia de 15 cM. Estos genes producen proteínas implicadas en la comunicación intercelular entre las células B y T, presentación de antígenos extracelulares con células T CD4+ y otras funciones inmunes. Los genes clase III codifican proteínas de complemento, implicadas en la destrucción de células extrañas 46.
Los genes y productos de la clase II han sido más estudiados, entre ellos, la clase IIa contiene los genes DR y DQ y la clase IIb los genes DYA, DYB, DMA, DMB, DOB, DOA, TAP1, TAP2, LAPM2 y LMP7. La clase IIa es la más polimórfica y contiene los genes: DRA, DRB (DRB1, DRB2, DRB3), DQA (DQA1, DQA2, DQA3, DQA4, DQA5) y DQB (DQB1, DQB2, DQB3, DQB4, DQB5) 2, mientras que, la clase IIb solo se encuentra en rumiantes 46.
El gen BoLA-DRB3 tiene una longitud de 11,4 Kbp desde el codón de inicio hasta la señal de poliadenilación, con cinco intrones y seis exones 39 y codifica elementos funcionales de restricción, proceso mediante el cual un linfocito puede reconocer un antígeno como propio o extraño 6. De estos exones el más polimórfico es el 2 (BoLA-DRB3.2) que tiene un tamaño aproximado de 270 bp; este codifica justo para el sitio de unión al peptido que se quiere presentar, y conociendo que la unión entre antigeno-anticuerpo es específica se requiere una alta diversidad en este gen. Al momento se han reportado 137 alelos BoLA-DRB3.2, que estan enlistados en la base de datos del EMBL-EBI IPD-MHC8.
El método de genotipación más utilizado para el estudio de este gen es la PCR-RFLP, con ella se han estudiado diferentes razas autóctonas, criollas y foráneas, en los cuales el número de alelos varió entre 11 y 37 alelos y la heterocigocidad esperada entre 0,88 y 0,91 10,11,38,40,53. Esta técnica es un método simple que permite un análisis rápido del polimorfismo de este gen, sin embargo, su utilidad es limitada en el caso de los alelos (tipos) que tienen alelos adicionales (subtipos), por lo que un mismo patrón de corte, codifica para varios alelos determinados por la secuencia del ADN. Por tanto, métodos de genotipaje basado en secuenciamiento del ADN como la PCR-SBT (sequence based typing) han sido desarrollados, pero menos utiizados, por la complejidad en la interpretación de las secuencias. Con PCR-SBT el número de alelos determinados es ligeramente mayor (11 a 39) así como el valor de heterocigocidad esperada (0,89 a 0,95) 32,43,44.
Los genes del BoLA son muy interesantes para los criadores de animales y genetistas. Algunos alelos del gen DRB3 han sido asociados con resistencia y/o susceptibilidad a una gran variedad de enfermedades como la mastitis y la leucosis viral bovina, a enfermedades transmitidas por garrapatas y con otras características como producción de leche, crecimiento y respuesta inmune 21,23,32,33,34,37.
El ganado criollo Colombiano Hartón del Valle ha sido caracterizado usando marcadores moleculares tipo STRs, cromosoma Y, D-Loop mitocondrial y para el gen DRB3 en menor medida por PCR-RFLP y PCR-SBT. Por tanto el objetivo de este trabajo fue evaluar la diversidad genética del gen DRB3 en el ganado criollo Colombiano Hartón del Valle mediante la técnica PCR-SBT y compararlo con otras razas previamente reportadas.
Materiales y métodos
Población y muestras
Se tomaron 93 muestras de animales puros Hartón del Valle (HV) de cuatro fincas localizadas en el departamento del Valle del Cauca, 30 muestras de animales de la raza Lucerna (LUC) y 30 animales de la raza Holstein (HOL); el menor grado de parentesco posible fue el criterio para la elección de los animales. Se recolectaron muestras de sangre en tubos de 5 ml, mediante punción de la vena coccígea con el sistema Vacutainer® con anticoagulante (K2EDTA 7,2 mg) y refrigerados hasta su transporte al laboratorio.
Extracción de ADN
El ADN se extrajo mediante el método de microextracción de Salting-Out 41. Rápidamente, 500 µl de sangre se mezclaron con 1 ml de solución de lisis I (0,32 M de Sacarosa, 10 mM de Tris HCl pH 7,5, 5 mM de MgCl2 y 1% de Triton X100) y se mezclaron por inversión a temperatura ambiente durante 1 minuto. Se centrifugó a 14000 rpm durante un 1 minuto y se descartó el sobrenadante, se realizaron sucesivos lavados con la solución de lisis I hasta obtener una pastilla de glóbulos blancos bien limpia. Se agregaron 400 μl de solución de lisis II (10 mM de Tris HCl pH 7,5, 10 mM de EDTA pH 8,0, 50 mM de NaCl, 0,2% de SDS) y 20 μl de proteinasa K (10 mg/μl) se incubó a 56 °C durante 1 hora. Se añadió 400 μl de NaCl 5 M, se hizo vórtex durante 1 minuto, y se centrifugó a 14000 rpm durante 14 minutos. 350 µl del sobrenadante fueron pasados a un tubo nuevo con 900 µl de etanol absoluto frio y se mezcló suavemente por inversión, se centrifugó a 14000 rpm durante 12 minutos y se descartó el sobrenadante. Se agregó 1 ml de etanol al 70%, se mezcló por inversión durante 2 minutos, y se centrifugó a 14000 rpm durante 5 minutos. Se dejó secar el pellet de ADN durante 20 minutos a 37 °C y luego se resuspendió en TE 1X (1 mM de Tris HCl pH 7,5, 0,1 mM de EDTA pH 8,0).
La calidad y concentración del ADN se evaluó usando geles de agarosa al 0,8% en una cámara SUB-CELL® GT, (BIO-RAD, USA) teñidos con bromuro de etidio y visualizados con luz ultravioleta, la concentración se determinó mediante la comparación de las muestras con concentraciones conocidas del bacteriófago lambda.
Amplificación y secuenciación (PCR-SBT) del gen DRB3
La genotipificación de los alelos BoLA-DRB3.2 se realizó utilizando la metodología PCR-SBT, en un protocolo de un paso con los cebadores:
D R B 3 F E W ( 5 ' - C G C T C C T G T G A C C A G AT C - TATCC-3') y DRB3REV (5'-GGTGAGCGCGGGGGTG- 3') 3,48 en una concentración de 10 mM, 25 ng de ADN, 0,2 mM de cada dNTP, 1X de tampón de PCR, 2,5 mM de MgCl2 y 1U de Taq DNA Polymerase.
El perfil térmico de la reacción incluyó una desnaturalización inicial a 95 °C durante 5 minutos, seguido por 35 ciclos de 95 °C por 30 segundos 62 °C por 30 segundos y 72 °C por 1 minuto, para terminar con una extensión final de 72 °C durante 5 minutos. Las amplificaciones fueron llevadas a cabo en un termociclador PTC-100® Teltier Thermal Cycler (BIO-RAD, USA).
Los amplificados se observaron en geles de agarosa al 1,2% teñidos con bromuro de etidio en una cámara SUBCELL ® GT, (BIO-RAD, USA) y visualizados con luz ultravioleta. El producto de la PCR fue un fragmento de aproximadamente 281 pb, y la concentración mínima de los amplificados fue 20 ng/μl. El secuenciamiento de las muestras se realizó en la empresa MACROGEN, USA.
Análisis estadístico
Las secuencias y sus electroferogramas fueron editados usando el programa GENEIOUS 6.1 (Biomatters development team, USA) y alineadas usando el programa MEGA 6 49. Los genotipos fueron determinados usando el programa ASSIGN 400ATF ver. 1.0.2.41 (Conexio Genomics, Fremantle, Australia).
El número de Alelos (Na), sus frecuencias, los alelos únicos, la heterocigocidad observada (Ho) y esperada (He), el índice de fijación (F) y las desviaciónes del equilibrio de Hardy-Weinberg (EHW) fueron estimadas con el programa ARLEQUIN versión 3.5 9. Se realizó un análisis de varianza molecular (AMOVA) con el fin de estimar la estructura y la diferenciación genética entre las poblaciones además de los estadísticos F de Wright's usando los programas ARLEQUIN versión 3.5 y GENALEX versión 6.5 35.
La distancia genética estándar de Nei entre las razas (HV, LUC y HOL y reportadas) fueron estimadas usando el programa GENALEX versión 6.5 a partir de las frecuencias alélicas e interpretadas mediante un análisis de clúster usando el algoritmo UPGMA (unweighted pair-group method with arithmetic mean) mediante el programa MEGA 6 49. La significancia de las ramas se determinó con un bootstrap de 1000 réplicas.
La diversidad genética a nivel de secuencia se estimó a partir de la diversidad nucleotídica (p), se realizó el test de neutralidad de Tajima (D-Tajima) y el test exacto de Ewens-Watterson-Slatkin (EWS) mediante el programa ARLEQUIN versión 3.5. La distancia genética entre pares de secuencias fue estimada con el modelo de Kimura 2-parámetros y el árbol construido usando el algoritmo neighbor-joining (NJ) con el programa MEGA 6 49. La significancia de las ramas se determinó con un bootstrap de 1000 réplicas, y se compararon con los 137 alelos del gen DRB3 reportados en la base de datos del EMBL-EBI IPD-MHC 8
ResultadosEl número de alelos y sus frecuencias para cada raza se pueden apreciar en la tabla 1. Los alelos con las frecuencias más altas están resaltados en negrilla. Se encontraron 27 alelos en la raza HV, 17 en LUC y 14 en HOL. En el HV la mayoría de los alelos (74%) presentaron frecuencias menores del 5% y solo un alelo (DRB3*1101) tuvo frecuencia mayor al 10%, los dos alelos que le siguen en frecuencia fueron el BoLADRB3* 0902 y *2703. El 23,5% de los alelos en la raza LUC mostraron frecuencias mayores al 10% entre ellos el DRB3*0701, *1107, *1501 y *2301, de otro lado, el 41% de los alelos presentaron frecuencias menores al 5%. En la raza HOL no se encontraron alelos con frecuencias menores al 5%, mientras que, el 85,8% de los mismos presentaron frecuencias entre el 5 y el 10%, los alelos más frecuentes fueron DRB3*0701 y *2701.
De los 39 alelos totales, 4 solo estuvieron en HOL todos con frecuencias entre el 5 y el 10%. La raza LUC presento 6 alelos únicos dos de los cuales mostraron altas frecuencias alélicas (DRB3*2301 y *2901). En la raza HV se encontraron 16 alelos únicos, 14 de ellos con frecuencias bajas y solo los alelos DRB3*0902 y *2703 con frecuencias mayores al 5% (Tabla 1).
La Ho en la raza HV fue ligeramente menor que en las demás razas (Tabla 2). Sin embargo, se observó que en todos los grupos genéticos la He fue mayor que la Ho, es esta diferencia se vio reflejada en los valores de positivos de FIS indicando un déficit de heterocigotos que solo fue significativo en el HV.
La diversidad genética (He) fue alta en todas las razas, mayor en HV y con valor similar en LUC y HOL, de aquí que el índice de fijación fue igualmente bajo en todas las razas. Solo en las razas HV y HOL se observaron desviaciones significativas de las proporciones teóricas del equilibrio de Hardy-Weinberg, mientras que, la raza LUC si se encontró en EHW (Tabla 2).
El AMOVA indicó que del total de la variación solo el 3% es debido a diferencias entre las razas y el 5% a las diferencias de los individuos dentro de las razas, como es común la mayor variación se presentó dentro de los individuos (Tabla 3). La diferencia genética (FST) entre las razas fue moderada y altamente significativa, en general se observó un déficit significativo de heterocigotos en cada raza (FIS) y toda la población de bovinos estudiada (F IT).
El análisis a nivel de secuencia de ADN mostró que la diversidad nucleotídica fue más alta en la raza LUC seguido por HV y HOL (Tabla 4). El test de neutralidad de Tajima indicó que no se está presentando selección neutral en las razas estudiadas. El test EWS mostró que ninguna de las razas se encuentra en equilibrio de selección balanceadora.
Con el fin de ver la asociación entre todos los alelos BoLA-DRB3, se generó un árbol usando la distancia genética del modelo de Kimura 2-parámetros y el método de reconstrucción de Neighbor-Joining con 137 secuencias de una longitud de 235 pares de bases (ver figura 1), se adjuntaron a este árbol los alelos reportados en las razas Yacumeño (Y), Hartón del Valle (HTV) 12,17; Holstein (H), Shorthorn Japonés (S), Negro Japonés (W), Jersey (J) 32,45 y Hanwoo (K) 25.
De forma general los tipos y subtipos de alelos se agruparon con valores medios y altos de bootstrap. De los 27 alelos encontrados en el HV siete estuvieron también presentes en otros ganados criollos, 19 alelos fueron compartidos con razas foráneas de América Latina y solo un alelo fue único para el HV (DRB3*2006). Por su parte la raza LUC mostró 6 alelos únicos (DRB3*20011, *2202, *2301, *2702, *2901 y *3801), los restantes 11 alelos del LUC se compartieron con otras razas. La raza HOL solo presentó un alelo único (DRB3*2704) (Figura 1).
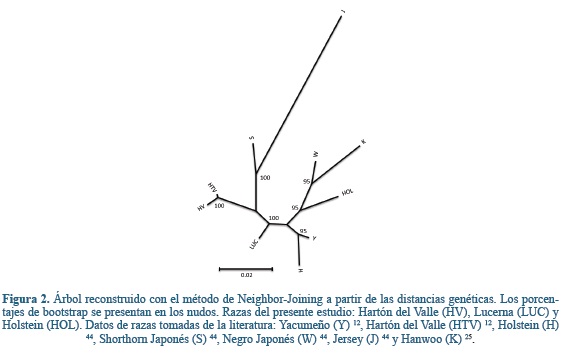
En la figura 2 se muestra un árbol de distancias genéticas estándar de Nei, agrupadas por el método UPGMA entre las razas estudiadas y otras reportadas en la literatura. La menor distancia genética se encontró entre el Hartón del Valle (HV) y un reporte anterior de esta misma raza (HTV) 12 (0,00546), la raza más divergente fue la Jersey (J) (0,10135) que se agrupo con la raza Shorthorn Japonés (S). En un tercer clúster, la raza Lucerna (LUC) mostró una distancia intermedia entre el criollo Hartón de Valle y el Holstein. La raza Yacumeño estuvo cercana al Holstein, mientras que, las razas Negro Japonés (W) y Hanwoo (K) se agruparon a una distancia de 0,03205.
Discusión
Para el ganado criollo Colombiano, solo existen reportes de la utilización de la técnica PCR-SBT para evaluar el polimorfismo del gen DRB3 en la raza Hartón del Valle, sin embargo, utilizando la técnica PCR-RFLP se han reportado en las razas Blanco Orejinegro entre 15 y 31 alelos 18,30, en la Casanareño 17 alelos, 12 alelos en la Costeño con Cuernos, 10 en la raza Chino Santandereano, en la raza Caqueteño 25, 12 alelos en la San Martinero, 16 en el Romosinuano y 14 en el Hartón del Valle. Por su parte, en las razas sintéticas Colombianas se reportan 13 y 12 alelos en Lucerna y Velásquez respectivamente 18. La PCR-SBT permite detectar más y con mejor precisión los alelos DRB3 que la PCR-RFLP, ya que un mismo patrón de corte con RFLP puede estar determinado por varias secuencias; esto explica porque el número de alelos aquí presentado es mayor para HV y LUC que lo reportado por Hernández et al., (2013) 18. El mismo autor también reporta valores de heterocigocidad esperada más bajos y valores de F IS más altos en todas las razas de ganado criollo Colombiano excepto en el HV (0,021ns) donde el valor fue similar.
En el HV si ha sido empleada la técnica PCR-SBT para estudiar el polimorfismo del gen DRB con reportes de 37 alelos 17, diez más de los aquí presentados, los más frecuentes fueron: BoLA-DRB3*1101 (0,204), *20012 (0,122), *2006 y *2801 (0,071); de estos solo el BoLADRB3* 1101 presentó también alta frecuencia. El mismo autor también presenta una He mucho más baja (0,734), un déficit alto de heterocigotos FIS=0,213, (p<0,01) y contrario a lo aquí indicado, reporta que la raza no mostraba desviaciones del EHW. Otro estudio 12 reporta 24 alelos, tres menos que el presente estudio, y coincide en que el alelo más frecuente es el BoLA-DRB3*1101 (0,121); por otro lado, muestra un exceso leve de heterocigotos, reportando un valor mayor de Ho que de He (0,94) y un FIS de -0,036, (p<0,01) lo que se tradujo en desviaciones significativas de EHW. Estas diferencias pueden atribuirse a la utilización de un menor número de animales y al origen de los mismos, pues las muestras empleadas para este estudio fueron recolectadas en cuatro hatos, mientras que, los reportes12, 24 solo son de una finca.
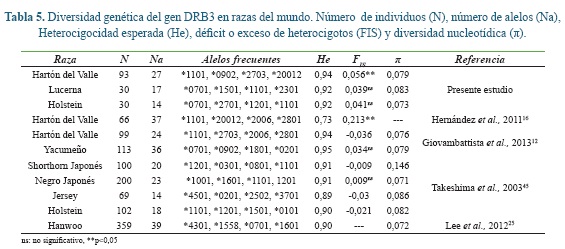
La comparación detallada de nuestros resultados con otras razas alrededor del mundo se presentan en la tabla 5. En general se observa que el número de alelos estuvomás alelos fueron la Yacumeño y el Hanwoo. El alelo DRB3*1101 es común en varias razas, aunque cada raza tiene un alelo en particular, diez alelos estuvieron solo presentes en ganados criollos (Hartón del Valle y Yacumeño). Estas diferencias en las frecuencias y distribución de los alelos en las razas puede deberse a varios factores, entre ellos: el origen de la raza, selección artificial o natural, endogamia o efecto fundador12, 45.
Pese al bajo número de animales utilizado en este estudio, los resultados revelan que el HV tiene una alta diversidad genética en el locus BoLA-DRB3. Estos valores obtenidos fueron similares a lo reportado en otras razas. Los valores He estuvieron por encima de 0,9 excepto en un reporte de Hartón del Valle 17 y en la Jersey 45 con leve déficit de heterocigotos. A nivel se secuencia la raza más diversa fue la Shorthorn Japonés (Tabla 5).
Estos valores de diversidad no son inesperados para genes clase II del complejo mayor de histocompatibilidad (CMH) conociendo su importante función biológica en el organismo.
Una posible explicación para la alta diversidad genética en este locus puede ser el origen múltiple de las poblaciones fundadoras de las razas26 como ocurre en el Hartón del Valle, donde según datos de ADN mitocondrial esta raza tiene un 91,7% de razas Europeas, 5,5% de razas africanas y 2,7% de razas del medio Oriente1 o en la raza Lucerna producto del cruce entre el Hartón del Valle, Holstein y Shorthorn. Luego la diversidad genética podría ser mantenida por algún mecanismo entre los que se cuentan la sobredominancia, la selección dependiente de las frecuencias y los sistemas reproductivos.
En los sistemas reproductivos se considera el apareamiento y aborto selectivo basado en el genotipo de CMH, los cuales han sido documentados en poblaciones de ratones y humanos y que puede ser lo suficientemente fuerte para mantener la diversidad genética el CMH36,51. Sin embargo, la alta diversidad de este locus en las razas estudiadas, así como en otros animales de granja27, 28, no puede ser explicada por preferencias sexuales ya que la reproducción en estas razas ha sido controlada por los criadores de cada raza.
En grandes poblaciones como las de humanos, se han reportado un déficit significativo de homocigotos para los genes del CMH5,19. El exceso de heterocigocidad se ha interpretado como una consecuencia de la sobredominancia, ya que los individuos heterocigotos son capaces de reconocer un amplio espectro antígenos, aumentando así la eficiencia de estos individuos en comparación con individuos homocigotos. Varios estudios han demostrado que la heterocigocidad confiere ventajas selectivas contra enfermedades infecciosas (ventaja del heterocigoto), por ejemplo, humanos heterocigotos en locus HLA clase II mostraron tener resistencia contra el virus de la hepatitis C y el virus de la inmunodeficiencia humana (VIH)4, 20. En vacas Holstein con mastitis causada por Escherichia o Streptococcus se encontraron desviaciones significativas de EHW en el locus clase II DQA1, mostrando una ventaja del heterocigoto a la progresión de la enfermedad47.
En el presente estudio las razas HV y HOL presentaron desviaciones significativas del EHW, con déficit de heterocigotos (FIS) significativo solo en HV; resultados similares para esta raza los presenta Hernández et al., (2011)16, mientras que, Giovambattista et al., (2013)12 presenta un leve exceso pero significativo de heterocigotos. La explicación más probable para el déficit de heterocigotos en las razas estudiadas y en otras reportadas es que el coeficiente de selección de sobredominancia para el locus en mención puede ser considerablemente bajo probablemente inferior a 0,02 31. Con este coeficiente de selección tan débil el aumento de heterocigotos solo aplicaría en poblaciones grandes y en ausencia de cuellos de botella, deriva genética y endogamia.
Una tercera hipótesis que podría explicar el alto nivel de polimorfismo del CMH propone que las frecuencias pueden ser explicadas por el equilibrio de selección, suponiendo que la presencia de un alelo en particular, en lugar del heterocigoto, es el factor crítico que termina la supervivencia y la aptitud diferencial. Si esto es correcto cada alelo DRB3 podría estar relacionado con la protección a alguna enfermedad infecciosa o asociados con distintos rasgos como la supervivencia o la fecundidad. El test apropiado para poner a prueba esta hipótesis es el de neutralidad (D), aunque las desviaciones de este test en las razas estudiadas no fueron significativas, los valores mayores de cero indican un posible efecto de selección balanceadora, mientras que el test de EWS mostró que ninguna de las razas se encontraba en equilibrio de selección balanceadora, lo que supone selección positiva o negativa contra algún alelo, donde los alelos más comunes en HV representan el 46,8% de las frecuencias alélicas acumuladas. Resultados similares son presentados en la raza Yacumeño 12 y Jersey 45. Igualmente se reportan varios microsatélites no neutrales en el locus BoLADRBP1 en razas bovinas Escandinavas22. Los efectos de la selección balanceadora también se reportan en otras especies como monos 29 y pecaríes 50. Adicionalmente, se ha reportado el efecto protector de varios alelos DRB3 a una gran variedad de enfermedades como la mastitis y la leucosis viral bovina, a enfermedades transmitidas por garrapatas, enfermedades parasitarias y con otras características como producción de leche, crecimiento y respuesta inmune2,7,14,15,42,52,53,54.
En conclusión los datos aquí mostrados son consistentes con la hipótesis que propone que el polimorfismo del gen en estudio puede ser explicado por el equilibrio de selección.
Conclusiones
La raza criolla Hartón del Valle tiene una alta diversidad genética en el locus BoLA-DRB3 representada por el alto número de alelos y su amplia distribución, en el valor de heterocigocidad, de diversidad nucleotídica y bajos de valores de endogamia y de FIS. Los datos sugieren que el origen del polimorfismo puede deberse en parte al origen de la raza, dado que el gen DRB3 codifica para el sitio de unión al péptido, estos valores altos de diversidad genética pueden ser entendidos como adaptaciones de cada raza, luego, esta alta variación puede ser mantenida mediante equilibrio de selección.
Agradecimientos
A la Dirección de Investigación de la Universidad Nacional de Colombia Sede Palmira por la financiación de la investigación.
Referencias
1. álvarez L, Vera V, Cárdenas H, Barreto G, Muñoz J. Assessing the genetic diversity and ancestry of Hartón del Valle cattle using mitochondrial DNA. Rev Colom. Cienc. Pecu. 2012; 25:14-26. [ Links ]
2. Baltian L, Ripoli M, Samfilippo M, Takeshima S, Aida Y, Giovambattista G. Association between BoLA-DRB3 and somatic cell count in Holstein cattle from Argentina. Mol Biol Rep 2012; 39(7): 7215-7220. [ Links ]
3. Baxter R, Hastings N, Law A, Glass E. A rapid and robust sequence-based genotyping method for BoLADRB3 alleles in large numbers of heterozygous cattle. Anim Genet 2008; 39(5): 561-563. [ Links ]
4. Carrington M, Nelson G, Martin M, Kissner T, Vlahov D, Goedert J. HLA and HIV-1: heterozygote advantage and B*35-Cw*04 disadvantage. Science 1999; 283 (5408): 1748-1752. [ Links ]
5. Cao K, Hollenbach J, Shi X, Shi W, Chopek M, Fernández-Viña MA. Analysis of the frequencies of HLA-A, B, and C alleles and haplotypes in the five major ethnic groups of the United States reveals high levels of diversity in these loci and contrasting distribution patterns in these populations. Hum Immunol 2001; 62(9): 1009-1030. [ Links ]
6. Davies C, Andersson L, Mikko S, Ellis S, Henen E, Lewin H et al., Nomenclature for factors of the BoLA system, 1996: report of the ISAG BoLA Nomenclature Committee. Anim Genet 1997; 28(3): 159-168. [ Links ]
7. Duangjindaa M, Jindatajak Y, Tipvong W, Sriwarothai J, Pattarajinda V, Katawtin, S et al., Association of BoLA-DRB3 alleles with tick-borne disease tolerance in dairy cattle in a tropical environment. Vet Parasitol 2013; 196 (3-4): 314-320. [ Links ]
8. EMBL-EBI IPD-MHC. International Society for Animal Science. [acceso: 27 de noviembre de 2014]. URL:http://www.ebi.ac.uk/ipd/mhc/bola/index.html. [ Links ]
9. Excoffier L, Lischer HEL. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Mol Ecol Resour 2010; 10(3): 564-567. [ Links ]
10. Fernández I, Rios J, Gayosso A, Ulloa R, Alonso R. Polymorphism of locus DRB3.2 in populations of Creole Cattle from Northern Mexico. Genet Mol Biol 2008; 31(4): 880-886. [ Links ]
11. Giovambattista G, Golijow CD, Dulout FN, Lojo MM. Gene frequencies of DRB3.2 locus of Argentine Creole cattle. Anim Genet 1996; 27(1): 55-56. [ Links ]
12. Giovambattista G, Takeshima SN, Ripoli V, Matsumoto Y, Alvarez L, Saito H et al., Characterization of bovine MHC DRB3 diversity in Latin American Creole cattle breeds. Gene 2013; 519(1): 150-158. [ Links ]
13. Glass EJ, Oliver RA, Russell GC. Duplicated DQ haplotypes increase the complexity of restriction element usage in cattle. J. Immunol 2000; 165(1): 134-138. [ Links ]
14. Gowane G, Sharma A, Vandre R, Sankar M. Association of DRB 3 EXON 2 Alleles with Productive and Reproductive. J Ad Vet Res 2013; 3(1): 31-35. [ Links ]
15. Gowane G, Sharma A, Sankar M, Narayanan K, Das B, Subramaniam S et al., Association of BoLA DRB3 alleles with variability in immune response among the crossbred cattle vaccinated for foot-and-mouth disease (FMD). Res Vete Sci 2013; 95(1): 156-163. [ Links ]
16. Hernández D, Posso A, Benavides J, Muñoz J, Giovambattista G, Alvarez L. Polimorfismos del gen BoLA-DRB3.2 en el ganado criollo Hartón del Valle por PCR-RFLP y PCR-SBT. Rev Colom Cienc Pecua 2011; 24(3): 390-390. [ Links ]
17. Hernández D, Posso A, Muñoz J, Giovambattista G, Alvarez L. Evaluación de la resistencia genética del ganado criollo Hartón del Valle al virus de la leucosis bovina. AICA 2011; 1:169-172. [ Links ]
18. Hernández D, Posso A, Muñoz J, Giovambattista G, Alvarez L. Polimorfismos del gen BoLA-DRB3.2* en ganado criollo Colombiano. Rev MVZ Córdoba 2013; 18(Suppl. 1): 3665-3671. [ Links ]
19. Hollenbach JA, Thompson G, Cao K, Fernandez- Vina M, Erlich H, Bugawan T et al., HLA diversity, differentiation, and haplotype evolution in Mesoamerican Natives. Hum Immunol 2001; 62(4): 378-390. [ Links ]
20. Hraber P, Kuiken C, Yusim K. Evidence for human leukocyte antigen heterozygote advantage against hepatitis C virus infection. Hepatology 2007; 46(6): 1713-1721. [ Links ]
21. Juliarena MA, Poli M, Ceriani C, Sala L, Rodriguez E, Gutierrez E, Dolcini G et al., Antibody response against three widespread bovine viruses is not impaired in Holstein cattle carrying bovine leukocyte antigen DRB3.2 alleles associated with bovine leukemia virus resistance. J Dairy Sci 2009; 92(1): 375-381. [ Links ]
22. Kantanen J, Olsaker I, Holm L, Lien S, Vilkki J, Brusgaard K et al., Genetic diversity and population structure of 20 north European cattle breeds. J Hered 2000; 91(6): 446-457. [ Links ]
23. Karimuriboa E, Morrison L, Black A, Turner C, Kambarage D, Ballingal K et al., Analysis of host genetic factors influencing African trypanosome species infection in a cohort of Tanzanian Bos indicus cattle. Vet Parasitol 2011; 179 (1-3): 35-42. [ Links ]
24. Kelly L, D'Angelo M, Nimo A, Piaggio J, Postiglioni A, Nicolini P. Polimorfismo del gen DRB3.2 en bovinos criollos del Uruguay. Arch Zootec 2003; 52(197): 77-80. [ Links ]
25. Lee B-Y, Hur T-Y, Jung Y-H, Kim H. Identification of BoLA-DRB3.2 alleles in Korean native cattle (Hanwoo) and Holstein populations using a next generation sequencer. Anim Genet 2012; 43(4): 438- 441. [ Links ]
26. Liron J, Bravi C, Mirol P, Peral-Garcia P, Giovambattista G. African matrilineages in American Creole cattle: evidence of two independent continental sources. Anim Genet 2006; 37(4): 379-382. [ Links ]
27. Liu Y, Yan GQ, Wang XF. Polymorphism analysis of MHC-DRB3 gene in Dolang sheep with PCR-RFLP. Yi Chuan 2004; 26(1): 59-62. [ Links ]
28. Lunney J, Ho C-S, Wysocki M, Smith D. Molecular genetics of the swine major histocompatibility complex, the SLA complex. Dev Comp Immunol 2009; 33(3): 362-374. [ Links ]
29. Luo M, Pan H, Liu Z, Li M. Balancing selection and genetic drift at major histocompatibility complex class II genes in isolated populations of golden snubnosed monkey (Rhinopithecus roxellana). BMC Evol Biol 2012; 12: 207-220. [ Links ]
30. Martínez R, Toro T, Montoya F, Burbano M, Tobón J, Ariza F. Caracterización del locus BoLA-DRB3 en ganado criollo Colombiano y asociación con resistencia a enfermedades. Arch Zootec 2005; 54(206-207): 349-356. [ Links ]
31. Mikko S, Roed K, Schmutz S, Andersson L. Monomorphism and polymorphism at MHC DRB loci in domestic and wild ruminants. Immunol Rev 1999; 167: 169-178. [ Links ]
32. Miyasaka T, Takeshima SN, Sentsui H, Aida Y. Identification and diversity of bovine major histocompatibility complex class II haplotypes in Japanese Black and Holstein cattle in Japan. J Dairy Sci 2012; 95(1): 420-431. [ Links ]
33. Miyasaka T, Takeshima S, Jimba N, Matsumoto Y, Kobayashi N, Matsuhashi T, et al. Identification of bovine leukocyte antigen class II haplotypes associated with variations in bovine leukemia virus proviral load in Japanese Black cattle. Tissue Antigens 2013; 81(2): 72-82. [ Links ]
34. Panei C, Suzuki K, Echeverria M, Serena M, Metz G, Gonzales E. Association of BoLA-DRB3.2 alleles with resistance and susceptibility to persistent lymphocytosis in BLV infected Cattle Argentina. Int J Dairy Sci 2009; 4(3): 123-128. [ Links ]
35. Peakall R, Smouse P. GENALEX 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics 2012; 28(19): 2537-2539. [ Links ]
36. Potts W, Manning C, Wakeland. Mating patterns in seminatural populations of mice influenced by MHC genotype. Nature 1991; 352(6336): 619–621. [ Links ]
37. Rastislav M, Mangesh B. BoLA-DRB3 exon 2 mutations associated with paratuberculosis in cattle. Vet J 2012; 192(3): 517-519. [ Links ]
38. Ripoli M, Lirón J, De Luca J, Rojas F, Dulout F, Giovambattista G. Gene Frequency Distribution of the BoLA-DRB3 Locus in Saavedreño Creole Dairy Cattle. Biochem Genet 2004; 42(7-8): 231-240. [ Links ]
39. Russell GC, Smith JA, Oliver RA. Structure of the BoLA-DRB3 gene and promoter. Eur J Immunogenet 2004; 31(3): 145-151. [ Links ]
40. Saama P, Jacob B, Kehrli M, Freeman A, Kelm S, Kuck A, et al. Genetic Variation in Bovine Mononuclear Leukocyte Responses to Dexamethasone. J Dairy Sci 2004; 87(11): 3928-3937 [ Links ]
41. Sambrook J, Russell D. Molecular Cloning: A laboratory Manual. 4th edition. USA: Cold Spring Harbor Laboratoy Press; 2012. [ Links ]
42. Schwaba E, Gearyb T, Baillargeonc P, Schwabd A, Fecteaua G. Association of BoLA DRB3 and DQA1 alleles with susceptibly to Neospora caninum and reproductive outcome in Quebec Holstein cattle. Vet Parasitol 2009; 165(1-2): 136-140. [ Links ]
43. Takeshima S, Ikegami M, Morita M, Nakai Y, Aida Y. Identification of new cattle BoLA-DRB3 alleles by sequence-based typing. Immunogenetics 2001; 53(1): 74-81. [ Links ]
44. Takeshima S, Nakai Y, Ohta M, Aida Y. Short Communication: Characterization of DRB3 Alleles in the MHC of Japanese Shorthorn Cattle by Polymerase Chain Reaction-Sequence-Based Typing. J Dairy Sci 2002; 85(6): 1630-1632. [ Links ]
45. Takeshima S, Saitou N, Morita M, Inoko H, Aida Y. The diversity of bovine MHC class II DRB3 genes in Japanese Black, Japanese Shorthorn, Jersey and Holstein cattle in Japan. Gene 2003; 316:111-118. [ Links ]
46. Takeshima S, Aida Y. Structure, function and disease susceptibility of the bovine major histocompatibility complex. Anim Sci J 2006; 77(2): 138-150. [ Links ]
47. Takeshima S, Matsumoto Y, Chen J, Yoshida T, Mukoyama H, Aida Y. Evidence for cattle major histocompatibility complex (BoLA) class II DQA1 gene heterozygote advantage against clinical mastitis caused by Streptococci and Escherichia species. Tissue Antigens 2008; 72(6): 525-531. [ Links ]
48. Takeshima S Matsumoto Y, Miyasaka T, Arainga- Ramirez M, Saito, Onuma M, et al. A new method for typing bovine major histocompatibility complex class II DRB3 alleles by combining two established PCR sequence-based techniques. Tissue Antigens 2011; 78(3): 208-213. [ Links ]
49. Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0. Mol Biol Evol 2013; 30(12): 2725-2729. [ Links ]
50. Taylor S, Jenkins D, Arcese P. Loss of MHC and neutral variation in Peary caribou: genetic drift is not mitigated by balancing selection or exacerbated by MHC allele distributions. PLoS One 2012; 7(5): e36748. [ Links ]
51. Wedekind C, Furi, S. Body odour preferences in men and women: do they aim for specific MHC combinations or simply heterozygosity? Proc Biol Sci 1997; 264(1387): 1471-1479. [ Links ]
52. Yoshida T, Furuta H, Kondo Y, Mukoyama H. Association of BoLA-DRB3 alleles with mastitis resistance and susceptibility in Japanese Holstein cows. Anim Sci J 2012; 83(5): 359-366. [ Links ]
53. Zambrano J, Echeverri J, Lopez A. Análisis y frecuencias de los alelos del antígeno leucocitario bovino BoLA DRB3.2 en vacas del hato Paysandú de la Universidad Nacional de Colombia. Rev Colom Cienc Pecua 2009; 22(3): 448-449. [ Links ]
54. Zambrano J, Echeverri J, López A. Alelos del gen BoLA DRB3.2 están asociados con mastitis en vacas lecheras. Rev Colom Cienc Pecua 2011; 24(2): 145- 156. [ Links ]