Introducción
La ganadería colombiana está constituida en un alto porcentaje por individuos de la subespecie Bos indicus (Novoa y Usaquén, 2010; Gómez et al., 2013) de la raza brahman, la cual llegó a Colombia a principios del siglo pasado (Rodríguez, 1993). Ésta es producto del cruce de las razas taurinas shorthorn y las cebuinas indubrasil, gyr y guzerá, sin embargo la mayoría de sus características fenotípicas (Rodríguez, 1993) y genéticas (Meirelles et al. 1999) corresponden a las cebuinas, dando lugar a los ganados brahman blanco y rojo (Sanders, 1980). Debido a sus características de rusticidad, los animales brahman se han adaptado a las condiciones ambientales del trópico bajo y a diferentes topografías en toda la geografía nacional, con un sinnúmero de ventajas comparativas frente a las razas de la subespecie Bos taurus (Vásquez et al., 2005), sin embargo, la carne de los animales brahman es menos tierna y jugosa (Wheeler et al.,1994).
La terneza es el parámetro más valorado por los consumidores de carne, y se sabe que es afectada por múltiples factores inherentes al ambiente o la genética del animal (Carragher et al., 2002, Warner et al., 2010). Desde el punto de vista genético se han descrito gran cantidad de parámetros que influyen en la terneza, los más importantes son los genes del complejo de la calpaína (CAPN) y su regulador la calpastatina (CAST), que están relacionados con el proceso de proteólisis de las fibras musculares durante la maduración de la carne, en los que se han descrito algunos alelos relacionadas con una mayor terneza (Carragher et al., 2002).
El gen CAPN1 (GenBank No. AF248054) codifica la enzima (-calpaína, está localizado en el cromosoma 29 y porta varios polimorfismos de un solo nucleótido (SNPs). El SNP CAPN316 (AF252504:g.5709C>G) es una transición C/G en el exón 9 que produce el cambio de alanina por glicina en la enzima (Page et al., 2002) y CAPN4751 (AF248054:g.6545C>T) es el producto de transición C/T en el intrón 17 (White et al., 2005). En ambos casos el alelo C ha sido asociado con una mayor terneza (Page et al., 2002; White et al., 2005). El gen CAST (GenBank No. AY008267) está localizado en el cromosoma 7, posee varios SNPs. UOGCAST ó CAST282 (AY008267:g282C>G) es una transversión C/G en el intrón 5 (Schenkel et al., 2006) y CAST2959 (AF159246:g2959G>A) es una transición A/G en el exón 3, ubicado en el extremo 3’ no traducido (UTR) (Morris et al., 2006). Los alelos C (Schenkel et al., 2006) y A (Morris et al., 2006) respectivamente, están asociados con una mayor terneza.
De igual manera el gen LEP u obeso (GenBank No. U50365) ubicado en el cromosoma 4, codifica para la leptina, una hormona que regula el apetito de los animales, el balance energético, el crecimiento, el depósito de grasa e influye en las características de la canal (Clarke y Henry, 1999, Houseknecht et al., 1998; Mácajová et al., 2004). El gen LEP porta algunos SNPs con variantes alélicas que favorecen el marmóreo, y este a su vez está íntimamente relacionado con la terneza (Schenkel et al., 2005). EL SNP E2FB (AY138588:g305C>T) es la transición C/T ubicada en el exón 2 (Buchanan et al., 2002) y E2JW (AY138588:g252A>T) es una transversión A/T ubicada en el exón 2 (Lagonigro et al., 2003). El alelo T de ambos polimorfismos se ha asociado a mayores porcentajes de grasa intramuscular (Buchanan et al., 2002; Lagonigro et al., 2003, Schenkel et al., 2005).
Con el fin de corroborar las bondades del ganado Brahman de la ganadería colombiana y establecer las bases para un programa de selección asistida por marcadores, el objetivo de este trabajo fue estimar las frecuencias alélicas y genotípicas, el equilibrio de Hardy-Weinberg, la estructura de la población y flujo genético para los SNPs CAPN316, CAPN4761, CAST282, CAST2959, E2FB E2JW y en ganado brahman comercial.
Metodología
Aval de ética y población de estudio
Con el aval del comité institucional para el uso y cuidado de los animales (CICUA) de la Universidad CES (acta número 5 de agosto 20 de 2013, proyecto 38), se evaluaron 270 machos brahman de los municipios de Puerto Berrío (n=29) en el Magdalena Medio Antioqueño (N:6.27.41 O:74.26.16, 107 msnm, 28,5ºC en promedio), Montería (n=86) en el Valle del río Sinú (N:08.33.45 O:76.00.87, 50 msnm, 28,1ºC en promedio) y Pueblo Nuevo (n=73) en la región del Alto San Jorge (N:8.26.45 O:75.19.59, 10 msnm, 27,8ºC en promedio) de Córdoba y en San Marcos (n=82) ubicado en la Depresión Momposina (N:8.37.22 O:75.16.57, 60 msnm, 27,8ºC en promedio) en el departamento de Sucre.
Genotipificación
De cada animal se extrajo el DNA genómico a partir de sangre periférica y se genotipificaron mediante la reacción en cadena de la polimerasa y el análisis de alta resolución en la temperatura de disociación (PCR-HRM), usando los cebadores específicos para los SNPs CAPN316 (F-5`TAGAGGCTGGGCAGG TCAGT3’ y R-5`CATCTTGACCCGGAGCTGCT3’) (López et al., 2016), CAPN4751 (F-5`CTCTGGGTGACCTGTCCTAAGA3’ y R-5`TCCCCCGTCACTTGACAC3’), CAST282 (F-5`TGGCATCAGCAGGTACTGC3’ y R-5`TTGTGTTTTATGTAGTCA ATTGTGAGA 3’), CAST2959 (F-5`GAAAACGATGCCTCAGGTGT3’ y R-5`TGTG CCCAATGCACAGTATT3’), E2FB (F-5`CGGGAAGGAAAATGCACTG3’ y R-5`CT ACCGTGTGTGAGATGTCATTG3’) y E2JW (F-5`GTGTTCTCGGAGATCGATGAT G3’ y R-5`CGTAAGACAGATAGGGCCAAAG3’) (López et al., 2017).
Análisis de la estructura y diferenciación poblacional
Utilizando el software GenAlEx (Peakal y Smouse, 2012) se calcularon las frecuencias alélicas para cada subpoblación y la población general usando las formulas fA=NA/2N y fa=Na/2N , donde N es el número de individuos en la población, NA y Na el número de alelos A y a. Para calcular las frecuencias genotípicas se utilizaron las formulas fAA=NAA/N, fAa=NAa/N y faa=Naa/N, donde NAA, NAa y Naa son el número de copias de los genotipos AA, Aa y aa. Además se determinó el estado de equilibrio de Hardy-Weinberg (HWE) comparando las frecuencias alélicas y genotípicas observadas y esperadas, se estableció la diversidad genética, definida como la heterocigosidad esperada (He) bajo condiciones HWE, y determinó la diferenciación genética comparando la heterocigocidad observada (Ho) y He.
La estructura poblacional se evaluó mediante el análisis de varianza molecular (amova) usando el coeficiente de diferenciación genética entre las subpoblaciones ( FST=(HT-He)/HT ), el coeficiente de endogamia en las subpoblaciones ( FIS=(He-Ho)/He ) y el coeficiente de endogamia total ( FIT=(HT-Ho)/HT ), donde Ho y He son la heterocigocidad observada y esperada entre las subpoblaciones y HT es la heterocigocidad total esperada. Se determinó el flujo genético expresado como el número de migrantes ( Nm=[(1/FST-1)/4] ) y se calculó el coeficiente de desequilibrio de ligamiento ( DAB=fAB-fAfB ), donde fAB es la frecuencia de la combinación de alelos AB, mientras que fA y fB son las frecuencias de los alelos de A y B (Peakal y Smouse, 2012).
Resultados
Frecuencias alélicas y genotípicas
Se obtuvieron los productos de PCR del tamaño esperado para cada marcador (López et al., 2017), lo anterior fue corroborado por electroforesis en gel de agarosa y/o la presencia de un solo perfil en la curva de disociación generada por la variación de la fluorescencia en función de la temperatura, que además es específico para cada genotipo, comparando con muestras de genotipo conocido usadas como control, lo cual permitió la genotipificación de cada uno de los SNPs estudiados, utilizando la técnica de PCR-HRM. A manera de ejemplo se muestran los perfiles de disociación (Figura 1A) y los perfiles normalizados (Figura 1B) de los genotipos AA, AG y GG del SNP CAST2959.
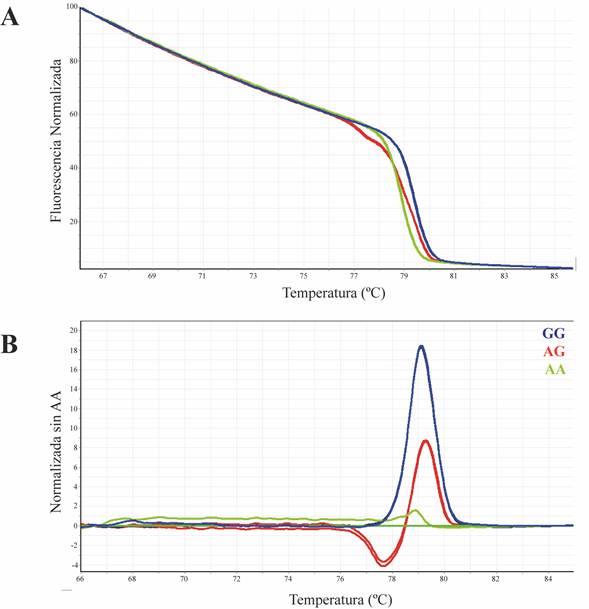
Figura 1 Genotipificación del SNP CAST2959: Perfil de HRM (A), (B), Perfil de HRM normalizada con el genotipo AA.
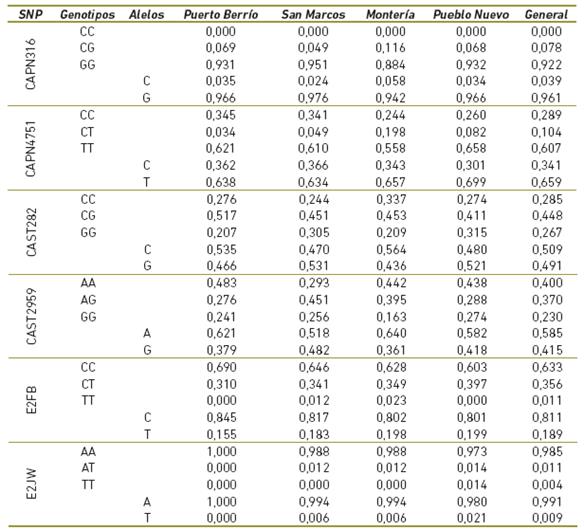
En la tabla 1 se resumen las frecuencias alélicas y genotípicas de los marcadores estudiados y al comparar sus frecuencias entre las subpoblaciones, no se encontraron diferencias (p>0,05). En la población general se evidencia baja frecuencia de los alelos C de CAPN316 (0,039), T de E2FB (0,189) y E2JW (0,009), los cuales han sido asociados con la producción carnes más tiernas y jugosas, mientras que las frecuencias genotípicas fueron: CAPN316 (CG = 0,078, GG = 0,922), CAPN4751 (CC = 0,289, CT = 0,104, TT = 0,606), CAST282 (CC = 0,285, CG = 0,448, GG = 0,267), CAST2959 (AA = 0,400, AG, = 0,370, GG = 0,230), E2FB (CC = 0,633, CT = 0,356, TT = 0,011) y E2JW (AA = 0,985, AT = 0,010, TT= 0,004).
Equilibrio de Hardy-Weinberg
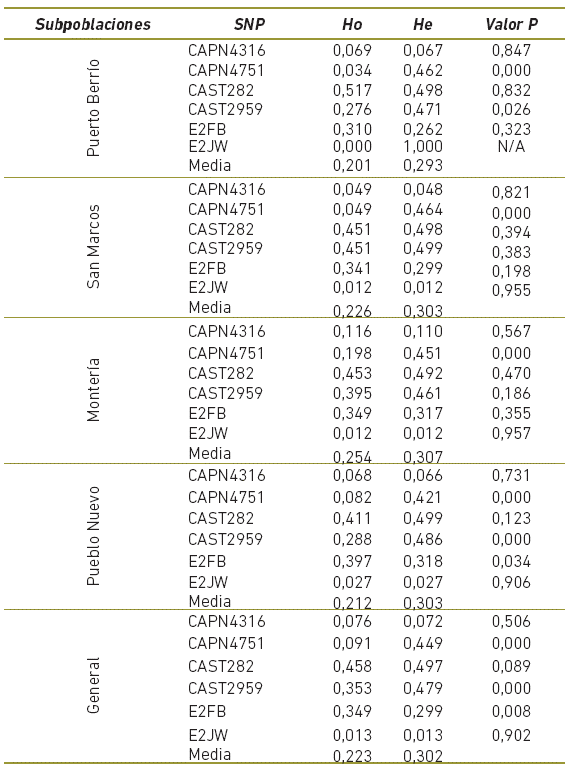
En la tabla 2 se muestran los valores de Ho, He y el valor de significancia de HWE de cada SNP en las subpoblaciones y en la población general. En la población general no se presentaron diferencias significativas (p>0,05) entre Ho y He para los SNPs CAPN316, CAST282 y E2JW, mientras que si se observó (p<0,05) para CAPN4751, CAST2959 y E2FB (Tabla 2). No se encontraron en HWE (p<0,05) los SNP CAPN4751 en ninguna subpoblación, de manera similar CAST2959 y E2JW en Puerto Berrío y CAST2959 y E2FB en Pueblo Nuevo.
Desequilibrio de ligamiento
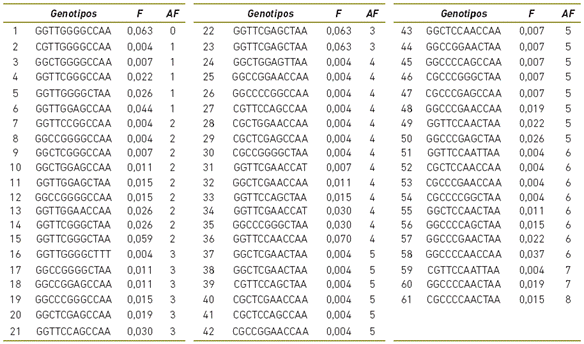
En la tabla 3 se muestran las combinaciones de genotipos de los SNPs encontrados en la población de ganado Brahman estudiada, la más frecuente fue GGTTCCAACCAA (0,070), portadora de cuatro alelos que favorecen la terneza y el marmóreo de la carne, seguida de GGTTGGGGCCAA (0,063), la cual no porta ningún alelo favorable. Dentro de los menos frecuentes se encuentran las portadoras de 6, 7 y 8 alelos favorables (Tabla 3). Los valores de D y r2 por población fueron diferentes de cero (p<0,05) para las combinaciones de SNPs CAPN316/CAPN4751 y CAST282/CAST2958. Las demás combinaciones no presentaron diferencias significativas (p>0,05) (Tabla 6), por lo que se espera que los alelos de cada locus segreguen de manera independiente.
Estructura genética
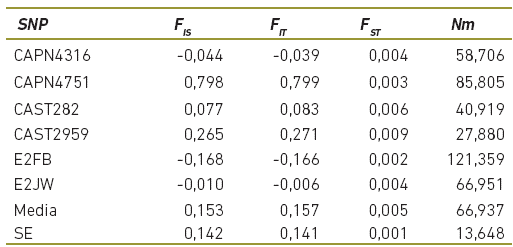
Se observó poca diferenciación genética entre las subpoblaciones dado que el valor del estadístico medio FST = 0,005 (p = 0,953), además el valor de FIT = 0,157 (p = 0,001) sugiere una tendencia baja a la endogamia entre las subpoblaciones, y dentro de las subpoblaciones, donde el valor de FIS = 0,153 (p=0,001) (Tabla 3).
Flujo genético y diferenciación genética
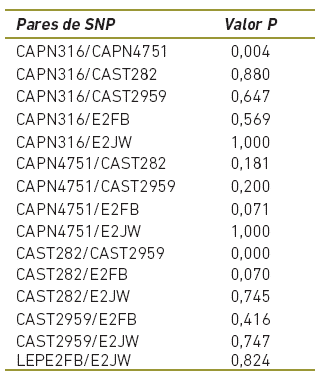
La media en el número de migrantes Nm = 66,937 (Tabla 3) sugiere un alto flujo de individuos o gametos entre las subpoblaciones por cada generación, promoviendo el intercambio genético entre éstas, lo cual explica la baja diferenciación genética de las subpoblaciones estudiadas, evidenciado en que los FST pareados presentaron poca variación, cuyos valores oscilan entre 0,001 y 0,007 (Tabla 4) y no presentaron diferencias significativas (p>0,005).
Tabla 4 Matrix pareada de poblaciones de los valores de FST, debajo de la diagonal se presentan los valores de FST y sobre la diagonal se muestra la probabilidad (P) (azar ( dato) basado en 999 permutaciones.
Tabla 5 Frecuencia de las combinaciones de genotipos de los SNPs CAPN316, CAPN4751, CAST282, CAST2959, E2FB y E2JW (F) y número de alelos favorables (AF) para la terneza y marmoreo de la carne de bovino.
Discusión
Los alelos C de CAPN316, CAPN4751 y CAST282 y el alelo A de CAST2959 han sido asociados con la producción de carnes más tiernas (Morris et al., 2006, Page et al., 2002, Schenkel et al., 2006, White et al., 2005), mientras que los alelos de T de E2FB y E2JW están relacionados con mayor marmóreo (Buchanan et al., 2002; Lagonigro et al., 2003, Schenkel et al., 2005). En la población estudiada, la frecuencia de éstos alelos es baja, excepto la de C de CAST282 y A de CAST2959 (Tabla 1). No obstante, los resultados muestran que los marcadores CAPN316, CAPN4751, CAST282, CAST2959 y E2FB en ganado Brahman son polimórficos, puesto que sus alelos no están fijados y sus frecuencias son superiores a 0,010 (Tabla 1).
Al comparar las frecuencias genotípicas obtenidas, no se encontró diferencia entre las subpoblaciones (p>0,05), además, los resultados son comparables con los obtenidos en los estudios realizados en ganado de la raza Brahman en Colombia (Cueitia, 2012, Castro, 2013), Australia (Cafe et al., 2010), Estados Unidos (Smith et al., 2009) y Puerto Rico (Bosques et al., 2015) y en ganado de la raza Nellore en Brasil (de Carvalho, 2012, da Silva et al., 2012, Oliveira et al., 2013, Pinto et al., 2011).
En la población estudiada los genotipos de los SNPs CAST282, CAST2959 y CAPN4751 y E2FB presentan buena segregación (Tabla 1). Las frecuencias del alelo C (0,509) y de los genotipos CC (0,285), CG (0,448) y GG (0,267) de CAST282 (Tabla 1) son similares a las reportadas por Castro (2013) y Pinto et al (2011), de igual manera las frecuencias del alelo A (0,585) y de los genotipos AA (0,40), AG (0,37) y GG (0,23) de CAST2959 (Tabla 1) son similares a las reportadas por Castro (2013), Cueitía (2012) y Pereira et al (2015).
Además, las frecuencias del alelo C (0,341) y del genotipo CC (0,289) de CAPN4751 (Tabla 1) son superiores a las reportadas por diferentes autores (Cafe et al., 2010, Castro, 2013, Bosques et al., 2015 y Smith et al., 2009, Pinto et al., 2011) quienes encontraron frecuencias de C y CC menores de 0,220 y 0,100, respectivamente, además, Cueitía (2012) no encontró individuos portadores del alelo C, debido quizá al tamaño de la muestra (n=22). La frecuencia del genotipo CT (0,104) es similar a la observada por Smith et al (2009), pero menor a la encontrada en otros estudios en los que se reportan valores entre 0,18 y 0,30 (Bosques et al., 2015, Pinto et al., 2011, Cafe et al., 2010, Castro, 2013).
Las frecuencias del alelo T (0,189) y de los genotipos CC (0,633), CT (0,356) y TT (0,011) de E2FB (Tabla 1) son similares a las encontradas por de Oliveira et al (2013) y da Silva et al (2012), quienes reportaron frecuencias bajas o ausencia de individuos con el genotipo TT, en contraste con lo observado por de Carvalho (2012) quien reportó una frecuencia mayor (0,159).
En contraste con lo anterior, los genotipos de CAPN316 y E2JW no presentan una adecuada segregación (Tabla 1). Las frecuencias del alelo C (0,039) y la de los genotipos CC (0,0), CG (0,078) y GG (0,922) de CAPN316 (Tabla 1) son similares a las reportadas en diferentes estudios (Bosques et al., 2015, Cafe et al., 2010 y Castro, 2013, Pinto et al., (2011) donde se encontraron valores infereiores a 0,05 en la frecuencia del alelo C, en contraste con lo reportado por Smith et al (2009) y Cueitía (2012), cuyos frecuencias son superiores a 0,30, además, no se encontraron animales portadores del genotipo CC, salvo en el trabajo realizado por Cafe et al., (2010), sin embargo, su frecuencia es muy baja (0,002).
Con respecto al SNP E2JW, las frecuencias del alelo T (0,009) y los genotipos AA (0,985), AT (0,010) y TT (0,004) (Tabla 1) son similares a las reportadas por da Silva et al (2012), pero diferentes a las observadas por de Oliveira et al (2013) quienes encontraron una frecuencia alta de animales portadores de AT (0,80).
En la población general y en todas las subpoblaciones el número de individuos heterocigotos observado (Ho) es menor al esperado (He) bajo condiciones de HWE (Tabla 2), esto sugiere una tendencia a la endogamia. Lo anterior puede ser explicado debido a que en las poblaciones existen fuerzas que alteran el HWE, entre otras, las practicas productivas en las ganaderías, el apareamiento selectivo, la endogamia, la estructura poblacional y la selección, quizás estas dos últimas son las principales razones, debido a la poca diferencia en las frecuencias alélicas entre las subpoblaciones (Nielsen y Slatkin, 2013). Se sugiere que las desviaciones del HWE (p<0,05) observadas en todos las subpoblaciones con respecto a CAPN4751 y de manera especial en Puerto Berrío para CAST4751, CAST2959 y E2JW y en Pueblo Nuevo para CAST4751, CAST2959 y E2FB (Tabla 2), podrían deberse a procesos de selección sobre características asociadas a estos marcadores, que afectarían sus frecuencias alélicas y genotípicas.
Excepto CAPN316/CAPN4751 y CAST282/CAST2959, las demás combinaciones de SNP analizados se encuentran en equilibrio de ligamiento, es decir que la frecuencia observada para la combinación de genotipos no difiere de manera significativa de su frecuencia esperada. De esta manera se puede afirmar que la presencia de cada alelo para cada uno de los genes analizados es independiente de la presencia de cualquier otro de ellos. Como en los bovinos los genes de CAPN1, CAST y LEP se encuentran en cromosomas diferentes, 29, 7 y 4 respectivamente, éstos poseen una tasa de recombinación máxima, es decir 0.5, confirmando su segregación independiente de acuerdo con la segunda ley de Mendel (Nielsen y Slatkin, 2013). El estado de equilibrio de ligamiento de la combinación E2FB/E2JW, puede explicarse por la baja frecuencia del alelo T, además es probable que se esté ejerciendo alguna fuerza selectiva sobre una característica relacionada con estos marcadores.
Como se mencionó antes, no se encontró el genotipo CC de CAPN316, y TT de E2JW se presenta en una frecuencia muy baja, mientras que los genotipos de los marcadores CAPN4751, CAST282, CAST2959 y E2FB presentan una adecuada segregación, sin embargo, en términos generales se observa una baja frecuencia de los alelos que han sido asociados con la producción de carnes más tiernas y jugosas. Lo anterior se puede constatar con las frecuencias de las combinaciones de genotipos, donde las frecuencias más altas las tienen aquellas que presentan un menor número de alelos favorables (Tabla 6).
El uso de los SNPs para evaluar la estructura genética de las poblaciones va en aumento debido a que algunos estudios demuestran que no hay diferencias significativas entre los resultados obtenidos con éstos, comparados con los realizados usando microsatélites. Aunque los SNPs son marcadores moleculares son poco polimórficos, son codominantes y presentan tasas de mutación bajas, lo que disminuye la posibilidad de cometer errores en la estimación de los parámetros poblacionales y aumenta la reproducibilidad de los resultados (Coates et al., 2009).
Teniendo en cuenta lo anterior se ha utilizado las frecuencias alélicas y genotípicas con el fin de caracterizar la estructura de las poblaciones estudiadas. Para ello, es importante saber que los estadísticos F permiten dividir el coeficiente de endogamia de una población subdividida (FIT) entre el componente debido a apareamientos no aleatorios dentro de las subpoblaciones (FIS) y la subdivisión entre poblaciones (FST) (Piñero et al., 2008). Para la población en estudio el FIT = 0,157 sugiere una baja tendencia la endogamia, de igual manera el FIS = 0,153 indica una leve tendencia a la homocigosidad debido al apareamiento no aleatorio entre los individuos de cada subpoblación (Tabla 3).
El valor de FST = 0,005 para la población general indica una baja diferenciación genética entre las poblaciones, esto representa una reducción en la heterocigosidad debida a la subdivisión poblacional. El mayor grado de diferenciación fue entre las subpoblaciones de Montería y San Marcos, no obstante estas no fueron significativas (p>0,05) (Tabla 3), mientras que en las poblaciones de Montería y Puerto Berrio la diferenciación fue menor.
Esto podría explicarse por el modelo de producción, las ganaderías de Puerto Berrío y San Marcos tienen un modelo de ciclo completo, en el que se seleccionan los animales que se van a aparear y se hace seguimiento desde el nacimiento de las crías hasta llevarlos al sacrificio, mientras que en las ganaderías estudiadas en Montería y Pueblo Nuevo, el modelo es de finalización, donde los animales son adquiridos en las subastas y cebados en las fincas, no obstante, los datos sugieren que las poblaciones son muy homogéneas con tendencias a la endogamia, y los valores de FST son menores de 0.007, lo que indica poca o nula diferenciación. Debido a esta condición las subpoblaciones evaluadas se comportan como una sola población (Tabla 4).
Gómez et al (2013) evaluaron la estructura y la diversidad genética de la ganadería Brahman en 23 regiones de Colombia (n = 3789) y encontró Ho = 0,6621, HWE (P < 0,001), FIT = 0,045, FST = 0,003 y FIS=0,042 y en otro estudio realizado por Novoa y Usaquén en 2010 en 20 regiones del país (n = 178), encontraron HWE (p <0,001 ), Ho = 0,637, FIS = 0,067. Estos estudios también sugieren que esta ganadería está constituida por una sola población, que no existe una subestructura genética en la población, al parecer debido a la alta tasa de migración de animales entre las subpoblaciones, lo cual homogeniza las frecuencias, además, esta población posee un modesto grado de heterocigocidad y diversidad alélica, comparado con otras razas, explicado por su origen de mezcla multiracial (Gómez et al., 2013, Novoa y Usaquén, 2010).
El Nm es el flujo de animales que entran a cada subpoblación en cada generación, esto influye en la disminución del índice de fijación, ejerciendo una fuerza importante contra la divergencia genética entre subpoblaciones (Hartl y Clark, 2007). Para la población analizada se encontró un Nm = 66,937 (Tabla 3), lo que confirma que no existe subdivisión poblacional (Nielsen y Slatkin, 2013). Lo anterior podría explicar la baja diferenciación genética entre las subpoblaciones estudiadas, puesto que el alto flujo genético entre ellas mantiene homogéneas las frecuencias alélicas. Lo anterior es coherente, con el origen de esta raza en Colombia, puesto que desde que el ganado Brahman llegó al país ha estado sometido a un proceso continuo de selección artificial dirigida, mediante reproducción endogámica, efectos de deriva genética causados por eventos fundadores, migración de ejemplares entre poblaciones y la importación de animales (Novoa y Usaquen, 2010).
Conclusiones
Las ganaderías Brahman estudiadas constituyen una sola población, en la que los marcadores CAPN316, CAPN4751, CAST282, CAST2959 y E2FB son polimórficos, aunque no ocurre lo mismo con E2JW, existe la posibilidad de producir carne de mejor calidad con estas ganaderías, debido a que aunque con frecuencias bajas, en esta raza se encuentran los alelos que han sido asociados con la producción de carnes más tiernas y jugosas. Por lo que el uso de los marcadores moleculares aquí presentados podría servir para realizar trabajos de selección asistida por marcadores, con el fin de aumentar la frecuencia de los alelos que favorecen la calidad.